2. 沈阳农业大学,辽宁沈阳 110866;
3. 中国农业科学院农业资源与农业区划研究所,北京 100081;
4. 中国农业科学院作物科学研究所,北京 100081
2. Shenyang Agricultural University, Shenyang, Liaoning 110866, China;
3. Institute of Agricultural Resources and Regional Planning, Chinese Academy of Agricultural Sciences, Beijing 100081, China;
4. Institute of Crop Sciences, Chinese Academy of Agricultural Sciences, Beijing 100081, China
CRISPR/Cas9是细菌和古细菌在防御外来核酸入侵时形成的一种适应性免疫系统,因其具有特异性、简单性和多功能性,故被视为一种新型的位点特异性基因组编辑工具,被用于生物和作物改良的研究[1–3]。此前我们所采用的基因组编辑方法主要有两种,锌指核酸酶 (ZFN) 和类转录激活效应因子核酸酶 (TALEN),它们主要依赖蛋白质和DNA相互作用来确定特异性[4]。在CRISPR/Cas9系统中,Cas9核酸内切酶由单个RNA (sgRNA) 引导,其引导原理包括两部分:其中一部分是通过DNA和RNA之间的WatsoneCrick碱基配对识别靶位点;另一部分是形成一种修复Cas9核酸内切酶的结构。对于靶位点的设计,仅要求必须具有PAM (NGG) 序列,这意味着基因组中几乎所有的基因都可以被CRISPR/Cas9系统进行靶向编辑[5]。CRISPR/Cas9系统可以准确地完全编辑目的基因,其获得的突变是可遗传的,再经过验证筛选出稳定的突变体株系[6]。目前,CRISPR/Cas9系统已经成功应用于植物中靶向基因组编辑,包括小麦[7]、烟草[8–9]、拟南芥[8, 10]、玉米[11–12]、大豆[13]等。虽然目前尚未有完全详细的CRISPR/Cas9系统的作用机制,但其作用机制的大体过程已经被研究出来,CRISPR/Cas9的作用机理可分为三个阶段:第一阶段,CRISPR/Cas9可变间隔区的获得,指噬菌体的一段DNA序列被整合到宿主基因组的CRISPR位点的5′端;第二阶段,CRISPR基因座的表达,插入的序列被转录形成crRNA;第三阶段,CRISPR/Cas9系统的激活以及其抵抗外源遗传物质的干扰能力[14–15]。
磷是植物生长和发育所需的必需元素,对植物的生理和生化过程具有积极的影响[16]。磷是核酸和磷脂的重要组分,其主要在光合作用、能量代谢、信号转导和基因表达等过程中起重要作用[17]。由于植物中的磷浓度远大于土壤中的浓度,因此植物吸收磷是通过逆浓度梯度能量耗散过程得以完成[18]。通常磷的缺乏已经成为植物生长的主要限制因素,磷转运蛋白基因对磷素缺乏有着积极的应答。磷转运蛋白系统有两种类型,高亲和力磷转运系统和低亲和力磷转运系统,两者都在磷吸收中起重要作用[19]。
实验室先前将冰叶日中花磷转运蛋白基因转入水稻中进行超表达,发现在低磷胁迫下,其对水稻的生长具有重要的作用。本研究利用CRISPR/Cas9基因编辑技术将Oryza sativa L. japonica. cv. Kitaake水稻中冰叶日中花磷转运蛋白基因McPht (GenBank:JQ343214.1) 的同源体,Pht3亚家族OsPht (GenBank:AK069397.1) 磷转运蛋白基因进行编辑,获得OsPht目标基因碱基缺失的突变体,并对突变体材料进行相关生理指标的测定,明确OsPht磷素转运蛋白基因碱基缺失下的突变体的生理功能变化,为深入探讨线粒体磷素转运蛋白生理功能提供科学的材料支持,同时也为冰叶日中花植物磷转运蛋白基因McPht导入OsPht基因碱基因突变或缺失的模式植物水稻株系,建立磷转运蛋白外源同源基因恢复磷转运功能提供可靠的材料支撑,依此系统深入研究外源同源体如McPht基因的生理生化功能。
1 材料与方法 1.1 敲除靶点引物序列设计根据编辑靶点引物序列设计的三点要求:1) 20 bp靶序列应紧接在5′-NGG PAM之前;2) 如果敲除目的是破坏基因功能,靶序列应该避免在编码区或内含子的3′末端;3) 为了减少脱靶效应,利用gRNA网站 (http://crispr.dbcls.jp/) 设计水稻磷转运蛋白基因敲除引物,上游引物的接头序列为5′-GGCA-3′,下游引物的接头序列为5′-AAAC-3′。用于基因编辑引导引物:F:5′-GGCACGCAGCCGTGGGTGTACGTG-3′;R:5′-AAACCACGTACACCCACGGCTGCG-3′。
1.2 载体构建稀释正向和反向目标特异性sgRNA寡核苷酸至10 μmol/L的终浓度。sgRNA寡核苷酸退火体系共20 μL,包括2 μL Annealing buffer (10 ×),F和R各9 μL。退火程序:95℃,5 min;95℃~25℃,1 min下降1℃;最后10℃保存。pCXUN-U3表达载体酶切体系共50 μL,包括5 μL AarI buffer (10 ×),1 μL Oligonucletide(50 ×),2 μL AarI,2 μL载体pCXUN-U3 (由作物科学研究所夏兰琴博士课题组友好捐赠),最后用灭菌水补充到50 μL。37℃反应3~6 h,跑胶,对酶切后的载体片段进行胶回收和纯化。将退火后的sgRNA寡核苷酸与用限制性内切酶AarI酶切后的载体连接,22℃反应3 h。
1.3 大肠杆菌转化及质粒的提取将5 μL连接后的载体与50 μL Trans1大肠杆菌感受态于2 mL EP管中轻轻混匀,冰浴30 min,42℃水浴30 s,再冰浴2 min,在超净台内向EP管加入500 μL不含抗生素的LB液体培养基,于37℃,200 rpm的摇床上培养1 h。8000 rpm离心1 min,弃上清液,保留100 μL培养液,用枪头吹打混匀,均匀涂在含卡那抗生素的LB固体培养基上,37℃黑暗条件下倒置培养基到有菌斑出现。挑取单克隆菌斑于2 mL含卡那抗生素的LB液体培养基的EP管中,于37℃,200 rpm的摇床上培养过夜。以菌液为模板,用表达载体通用引物进行PCR扩增,PCR扩增含有阴性对照,从而筛选出阳性菌斑。对于阳性菌斑用质粒小提试剂盒 (TIANprep Mini Plasmid Kit) 提取质粒DNA。
1.4 农杆菌转化将1 μg表达载体质粒DNA与200 μL EHA105农杆菌感受态于2 mL EP管中轻轻混匀,先冰浴 30 min,然后液氮冷冻5 min,迅速置于37℃水浴锅中水浴5 min,最后冰浴2 min。在超净台中向EP管中加入500 μL不含抗生素的LB液体培养基,于28℃,230 rpm摇床上过夜培养。8000 rpm离心1 min,弃上清液,保留100 μL培养液,用枪头吹打混匀,均匀涂在含卡那和利福平抗生素的LB固体培养基上,28℃黑暗条件下倒置培养基直到有单克隆菌斑长出。挑取单克隆菌斑于2 mL含卡那和利福平抗生素LB液体培养基的EP管中,于28℃,230 rpm的摇床上培养过夜,以扩大培养。以菌液为模板进行PCR扩增,筛选阳性菌落,在28℃、转速230 rpm培养24 h,向菌液内加入终浓度为50%的甘油,置于–80℃冰箱内。
1.5 突变体的筛选与鉴定取低温储存携带pCXUN表达载体的农杆菌EHA105阳性转化体按上述条件进行活化培养,待OD值达到期望值时,在无菌条件下利用活化的农杆菌液浸染Oryza sativa L. japonica cv. Kitaake水稻幼胚组织,并在含卡那和利福平抗生素MS固体培养基上培养2个月左右,将所获再生幼苗在光照生长箱进行适应性培养一个星期,然后移栽到草炭土中在温室培育3个半月,收获T0代转基因水稻种子。将所获的T0代转基因水稻种子在田间土壤进行栽培,对所有株系取相同位置叶片提取基因组DNA (EasyPure® Plant Genomic DNA Kit试剂盒) 。通过特异性引物:F:5′-ATGGCCGTCGTCTCCGAGAGC-3′和R:5′-CAGCATAGCCTTCTGCCTTGAC-3′,进行PCR检测分析,利用凝胶电泳回收纯化PCR扩增产物片段,用于测序。将测序得到的结果与原始序列通过DNAMAN进行序列比对,确定阳性转化材料,筛选出目标基因位点突变或缺失的水稻株系Cas2和Cas7用于后续实验。
1.6 试验材料准备将野生型和T1代突变体水稻种子Cas2和Cas7在盛水的玻璃培养皿 (直径9 cm) 中30℃黑暗条件下发芽3~4天,每天更换玻璃培养皿中的水,待种子发芽后将其转移到温室含有支撑网 (网孔面积为1 mm2) 的塑料盆中水培1周。选择长势均一的小苗,移栽到装有草炭土盆中,在网室自然光照下培养4个星期,对突变体和野生型水稻进行表型观测与取样用于相关的生理测试分析。
1.7 生物量测定与根系扫描用自来水将水稻冲洗干净,并用吸水纸吸干水分。用直尺测量野生型与突变体水稻的高度 (3个生物学重复),用天平称重。用根系扫描仪 (EPSONLA,UK) 对水稻根部进行扫描,用WINRHIZO Pro software of 2004c version (WINRHIZO,Regent,Co. Canada) 进行数据分析。
1.8 生理指标测定1.8.1 根系活力测定 标准曲线制作:取0.25 mL 0.4%的TTC溶液加入到10 mL试管中,加入少量次硫酸钠粉末,混合均匀后会有红色的甲腙产生。用乙酸乙酯定容至刻度并摇匀。取上述溶液0.25、0.5、1、1.5和2 mL分别加入10 mL容量瓶中并用乙酸乙酯定容,以空白作为参照,测定485 nm下的吸光度,制作标准曲线。
取野生型和突变体水稻根尖样品,剪碎,称取根尖样品约0.2 g,三次生物学重复,分别放入50 mL管中,加入5 mL 0.4%的TTC溶液和磷酸缓冲液,将根尖浸没于上述溶液中,37℃黑暗条件下反应2~3 h,加入2 mL的1 mol/L H2SO4终止反应,对照组先加1 mol/L H2SO4,再加根尖。用镊子把根尖取出,用吸水纸吸干水分,在研钵中与2 mL乙酸乙酯一起进行研磨,将红色研磨液放入10 mL试管中,用乙酸乙酯冲洗研钵,将冲洗液倒入试管中,用乙酸乙酯定容至10 mL,以空白作为参照,测定485 nm下的吸光度,制作标准曲线。根系活力计算公式为:
根系活力 [mg/(g·h)] = C/(W × t)
式中:C为标准曲线中查出的四氮唑还原量 (mg);W为样品质量;t为反应时间。
1.8.2 可溶性糖含量测定 标准曲线制作按照强晓晶方法[20]:取野生型和突变型水稻新鲜叶片,剪碎混匀,称取0.2 g新鲜叶片,三次生物学重复,分别放入25 mL试管中,加入5~10 mL蒸馏水,封口膜封口,于沸水中提取30 min (提取2次),提取液过滤到25 mL容量瓶中,用蒸馏水反复清洗并定容至刻度。取0.5 mL样品液于25 mL试管中,首先加入1.5 mL蒸馏水,然后加入0.5 mL蒽酮乙酸乙酯和5 mL浓 H2SO4充分混匀,放入沸水中保温1 min,自然冷却至室温,以空白作为对照,测定630 nm下的吸光度。可溶性糖含量按以下公式计算:
可溶性糖含量(%)=(C×VT×N×100)/(W×VS×106)
式中:C为从标准曲线中查得的糖量 (μg);VT为提取液总体积 (mL);N为稀释倍数;W为样品质量 (g);VS为测定时取用提取液的体积 (mL)。
1.8.3 叶绿素含量测定 标准曲线制作按照强晓晶方法[20]:取野生型和突变体水稻新鲜叶片,剪碎混匀,称取0.2 g新鲜叶片,三次生物学重复,分别放入研钵,加入少量石英砂和80%丙酮,研磨成匀浆,放入25 mL试管,向试管中加入10 mL 80%丙酮,将试管封口置于黑暗处直到叶片退为无色,期间不间断摇动试管。将滤纸放于漏斗中,先用80%丙酮湿润滤纸,再将提取液慢慢倒入漏斗并过滤到25 mL容量瓶中,用80%丙酮定容至25 mL,摇匀。以80%丙酮作为对照,测定470 nm、646 nm、663 nm下的吸光度。叶绿素含量的计算公式:
叶绿素含量 (mg/g) = C × V × N/(W × 1000) [21]
式中:C为色素含量 (mg/L);V为提取液体积 (mL);N为稀释倍数;W为样品鲜重 (g)。
2 结果与分析 2.1 载体构建与测序结果图1所示,冰叶日中花McPht磷转运蛋白与水稻OsPht磷转运蛋白氨基酸序列具有75%的高度同源性。
 |
| 图1 氨基酸同源性比较 Fig. 1 Comparison of amino acids |
OsPht基因靶位点敲除序列如图2所示,靶点序列长度为20 bp,位于编码区+ 95到+ 115处。经U3启动子驱动gRNA引导的pCXUN表达载体转化体浸染水稻幼胚组织后,获得具有抗生素抗性的水稻幼苗共计13株。对13株水稻苗期叶片DNA提取 (提取试剂盒EasyPure® Plant Genomic DNA Kit) 并进行PCR检测 (所用引物如方法1.5所述),检测结果表明都有与目标基因一致的DNA片段。为了证实水稻基因的敲除情况,将水稻幼苗DNA的PCR产物纯化并测序,测序结果显示13株水稻幼苗中7株编辑目标靶点展示出基因缺失或突变,这7株突变体碱基的编辑情况可概括为两类:一类为目标编辑片段20个碱基中后6个碱基缺失;另一类为目标编辑片段20个碱基中后8个碱基缺失或突变。
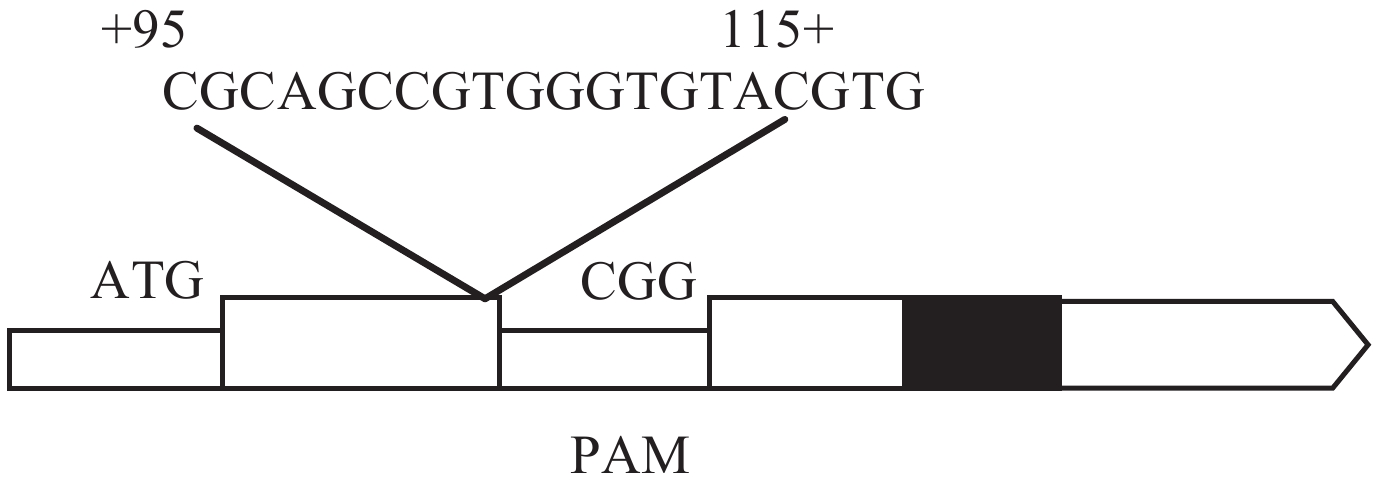 |
| 图2 OsPht基因靶位点 Fig. 2 Diagram of OsPht gene target site [注(Note):ATG 为起始密码子; CGG 为 PAM 序列; CGG 为 PAM 序列; 白色部分表示外显子,黑色为内含子 ATG is translation site; CGG is PAM motif;The white boxes indicate extron,and the black boxes indicate intron.] |
图3A为株系2和3的PCR检测电泳图,在株系2和3中,目标编辑片段20个碱基中后6个碱基被缺失 (图3B),图3C和3D显示的分别是野生型植株PCR产物测序峰图与突变体植株2和3的PCR产物测序峰图,与野生型相比,突变体株系后6个碱基TACGTG缺失。
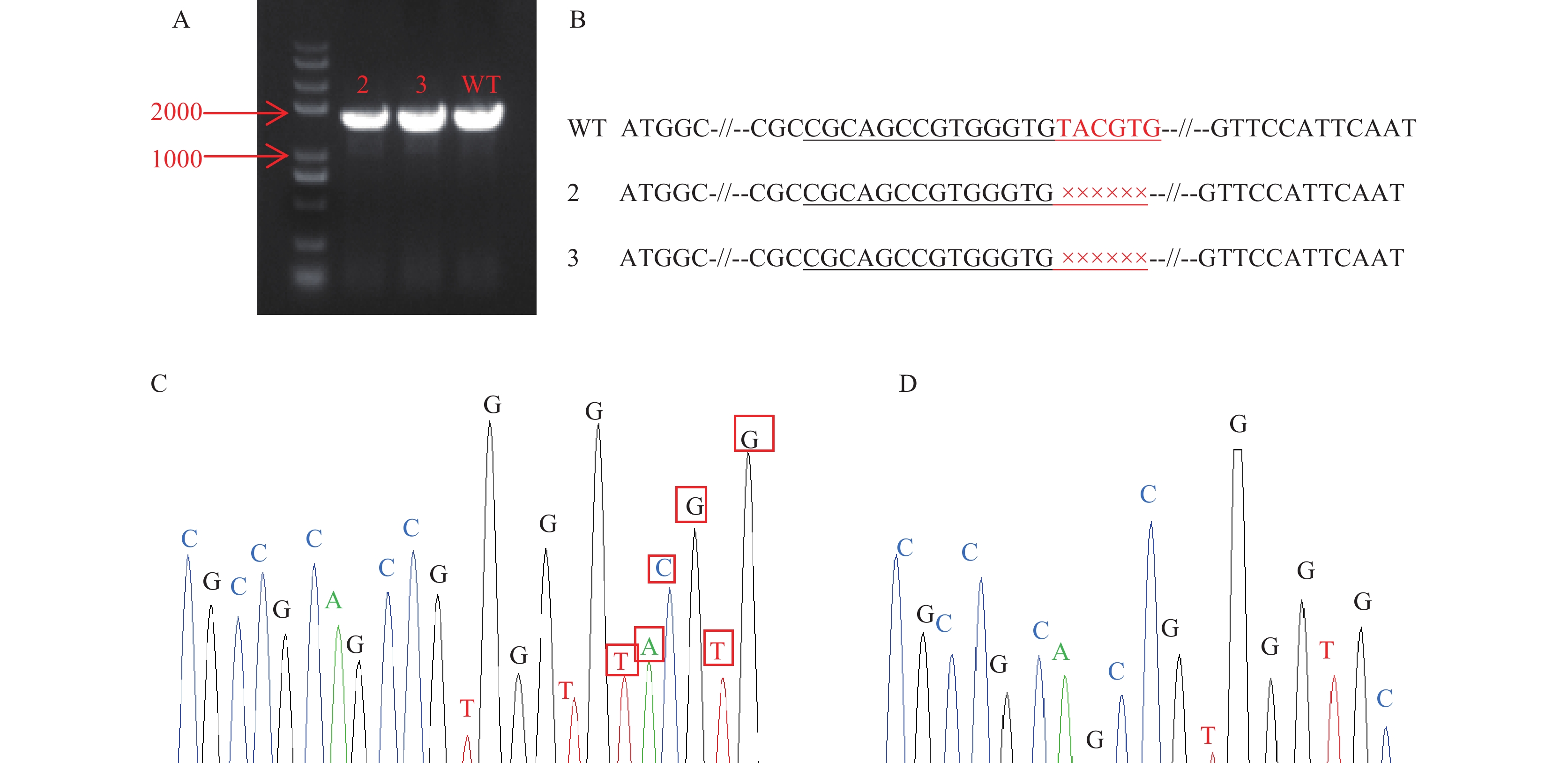 |
| 图3 PCR检测电泳图及测序结果 Fig. 3 PCR product analysis of electrophoresis and sequencing results [注 (Note):A— 突变体植株与野生型植株 PCR 电泳图; B— 野生型与突变体株系核苷酸序列对比图; C— 野生型株系目标基因靶点测序峰图; D— 突变体株系 2 和 3 目标基因靶点测序峰图; B 图中下划线为设计的基因编辑片段, 红色为实际测序敲除碱基; C 图中红框标记的是缺失或替换的碱基。A— PCR electrophoresis detection of wild type and mutants; B— Nucleotide sequence comparison of target gene site from wild type and mutants; C— Sequencing peak map of target gene site from wild type plants; D— Sequencing peak map of target gene site from mutant plants 2 and 3; Underlines in Fig. B represent the designed nucleotide sequences for gene editing fragment,red colors mean the actual bases deletion by sequencing; The red boxes in the Fig.C indicate the deleted or substituted bases.] |
图4A为株系7、10、11、12和9的PCR检测电泳图,株系10、11和12为目标编辑片段20个碱基中后8个碱基被完全敲除,株系7和9为后8个碱基发生缺失和突变 (图4B),其中碱基突变主要发生在G与T之间。与野生型相比,株系10、11和12的PCR测序峰图显示后8个碱基TGTACGTG完全缺失 (图4C和D);图4E所示,突变体7与野生型株系相比,倒数第7和8位碱基由TG变为GT;图4F所示,突变体9与野生型株系相比,倒数第7位碱基不变,倒数第8位碱基由T变为G。统计分析表明水稻株系总突变率为53.8%,后6个碱基成功敲除的株系占总突变株系的28.6%,后8个碱基成功敲除的株系占突变株系的42.9%,GT碱基突变株系占28.6%,证明CRISPR/Cas9基因编辑系统对水稻磷转运蛋白基因的敲除具有较高的编辑效率。通过DNAMAN对突变体氨基酸进行分析发现,后6个碱基缺失的突变体2和3其氨基酸序列与野生型株系相比,仅中间缺失2个氨基酸;而后8个碱基成功敲除的突变体10、11和12其氨基酸序列中共有14个终止密码子,第一个终止密码子位于第60个氨基酸;GT碱基突变体7中第37个氨基酸由V变为L;T碱基突变体9,氨基酸序列不变 (图5)。
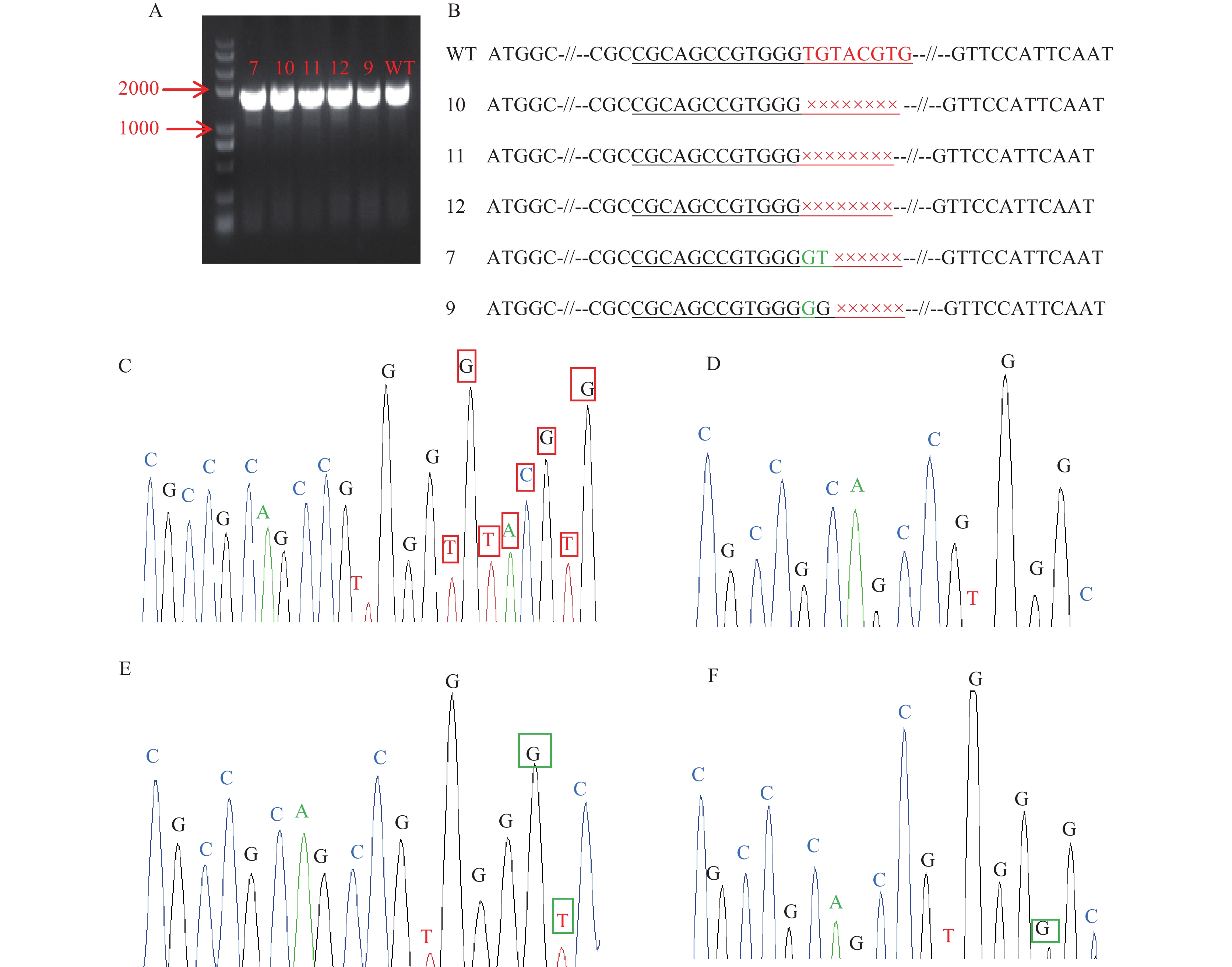 |
| 图4 株系7、10、11、12和9的PCR检测电泳图及测序结果 Fig. 4 PCR product analysis of electrophoresis and sequencing results for plants of 7, 10, 11, 12 and 9 [注 (Note):A— 突变体植株与野生型植株PCR电泳图; B— 突变体植株与野生型株系核苷酸序列对比; C— 野生型株系目标基因靶点测序峰图; D— 突变体株系 10、11 和 12 目标基因靶点测序峰图; E— 突变体株系 7 目标基因靶点测序峰图; F— 突变体株系 9 目标基因靶点测序峰图; B 图中下划线为设计的基因编辑片段,红色为实际测序缺失碱基,绿色为突变碱基; C 图红框指示突变体株系 10、11、12 与野生型相比敲除碱基; 图 E, F 中绿框代表突变株系 7 和 9 与野生型相比突变碱基。A— PCR electrophoresis map of wild tape and Mutants; B— Nucleotide sequence comparison of target gene site from wild type and mutants; C— Sequencing peak map of target gene site from wild type; D— Sequencing peak map of target gene site from mutant 10, 11 and 12; E— Sequencing peak map of target gene site from mutant 7; F— Sequencing peak map of target gene site from mutant 9; Underlines in Fig. B represent the designed nucleotide sequences for gene editing fragment, red colors mean the actual base deletion by sequencing, green color is a mutated base; Red boxes in Fig. C indicate the deleted bases in the mutants of 10, 11 and 12 relative to wild type; The green boxes in Fig. E and Fig. F indicate the mutated bases in mutant plants of 7 and 9.] |
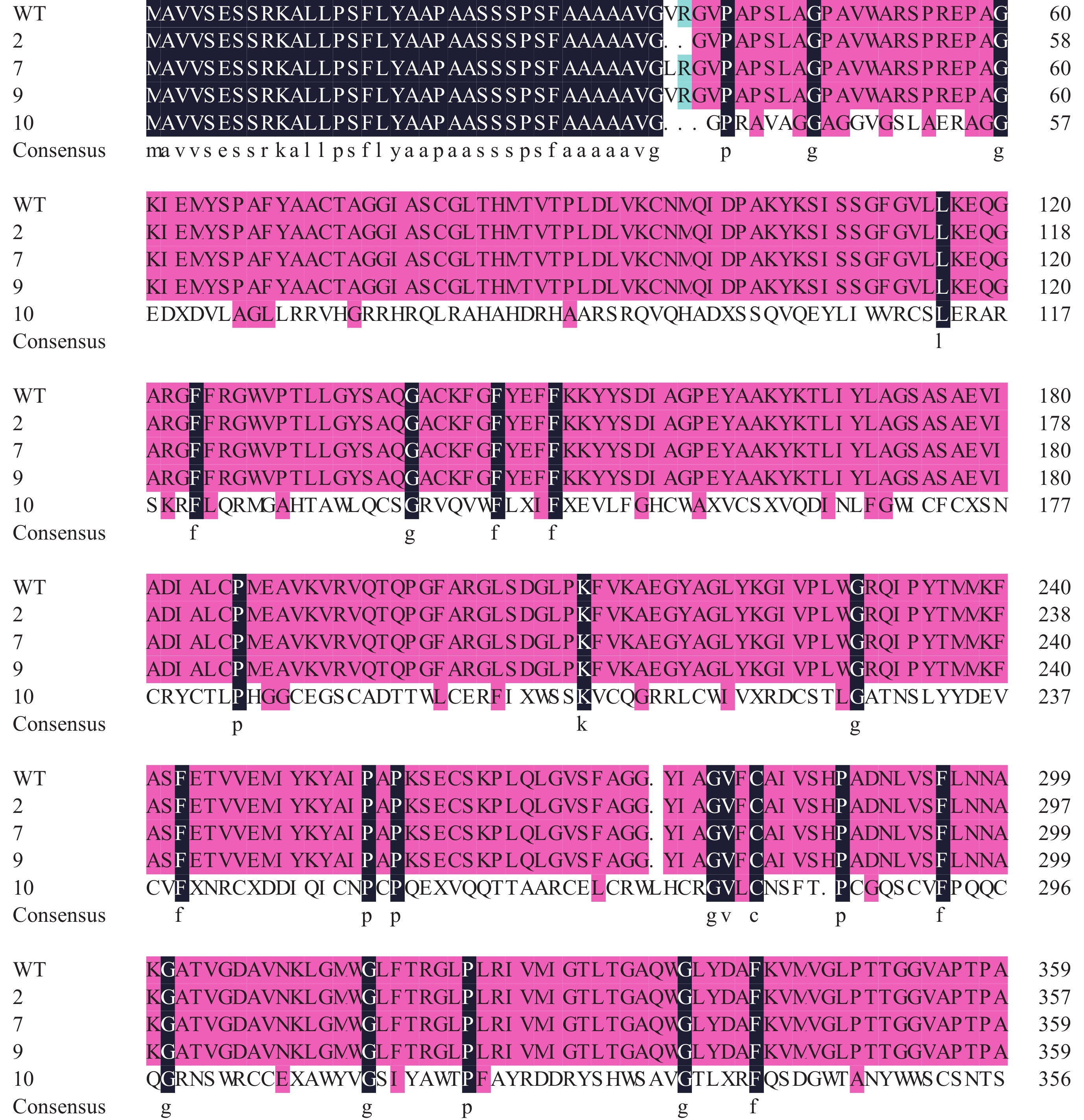 |
| 图5 CRISPR/Cas9对OsPP1基因编辑后蛋白序列比对结果 Fig. 5 Multiple alignments of OsPP1 protein between wild type and mutants [注 (Note):X—终止密码子Stop codon.] |
野生型和T1代突变体水稻表型如图6所示,水稻生物量和根系扫描如图7所示。突变体Cas-2和Cas-7与野生型水稻相比,鲜重分别减少0.01 g和0.02 g,经方差分析差异显著 (图7A);株高分别为野生型水稻的0.85和0.62倍 (图7B),差异极显著。水稻对营养元素的吸收主要通过根系进行,因此根系结构的变化可以间接反映植物对营养元素的吸收状况。突变体的总根长、总投影面积、总表面积和侧根数均高于野生型水稻。与野生型水稻相比,突变体Cas-2和Cas-7总根长分别增加了35.79 cm和20.69 cm,差异极显著 (图7C);总投影面积分别增加2.07 cm2和1.98 cm2 (图7D),Cas-2与野生型相比差异极显著;总表面积分别是野生型水稻的1.14和0.95倍 (图7E),Cas-2与野生型相比差异极显著;侧根数高于野生型水稻 (图7F),差异极显著。
 |
| 图6 野生型和突变体水稻表型 Fig. 6 Phenotypes of the wild type and mutant rice [注 (Note):WT—野生型Wild type; Cas-2 and Cas-7代表两株突变体Cas-2 and Cas-7 represent mutants.] |
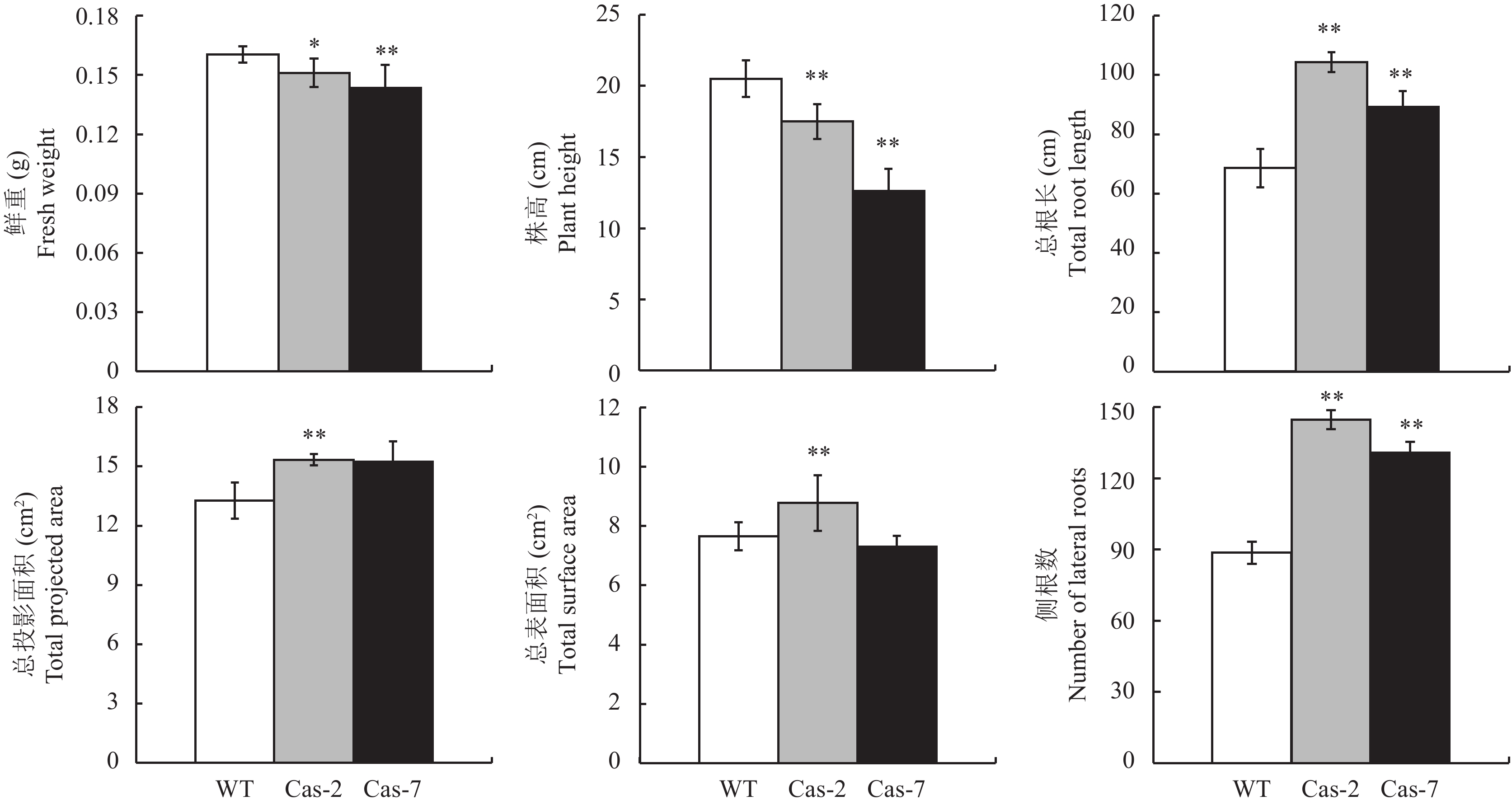 |
| 图7 生物量和根系扫描 Fig. 7 Biomass and roots scanning |
植物吸收土壤中的营养元素主要通过根系进行,植物生长情况、体内营养状况及其产量都可以通过根系生长情况及活力水平反映出来。突变体水稻Cas-2和Cas-7的根系活力分别是野生型的0.63和0.42倍 (图8A),差异显著;可溶性糖含量分别是野生型的1.29和0.83倍 (图8B),Cas-2与野生相比差异极显著;叶绿素含量是野生型水稻的1.01和0.95倍 (图8C),差异不显著。
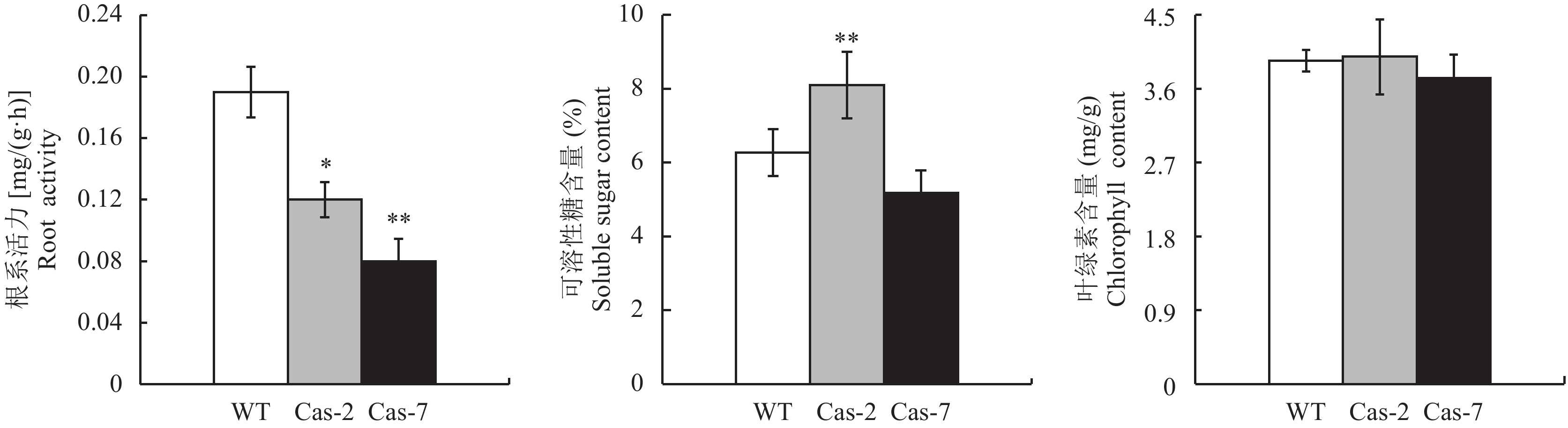 |
| 图8 水稻生理指标 Fig. 8 Physiological index of rice [注 (Note):WT—野生型Wild type; Cas-2 and Cas-7代表两株突变体Cas-2 and Cas-7 represent mutants.] |
早在1987年科学家就首次在E.coli碱性磷酸酶基因附近发现了短的回文重复序列,直到2002年将其命名为CRISPR/Cas9。最近几年,CRISPR/Cas9技术才逐渐趋成熟并被广泛应用[22]。在2013年8月8日,该技术在植物基因编辑中首次取得了巨大突破,Nature Biotechnology实验室成功对重要作物水稻、小麦以及模式植物拟南芥和本生烟的基因组进行定点编辑,主要包括多个基因定点敲除及插入,这说明了CRISPR/Cas9技术在植物基因组中可以完成定点编辑[23–25]。CRISPR/Cas9技术已经被成功应用于许多植物、动物和微生物中。在本研究中,我们利用CRISPR/Cas9技术对水稻磷转运蛋白基因组进行定点编辑,将其基因进行了定点敲除或突变,通过PCR及测序证实水稻基因OsPht被成功编辑;OsPht目标基因靶点编辑引发的突变的特异性与效率差异表明CRISPR/Cas9系统的编辑效率可能受多种因素影响,主要包括基因靶位点的特异性、sgRNA序列、用于Cas9和sgRNA表达的启动子及T-DNA插入位点[26]。有研究表明不同植物物种利用CRISPR/Cas9技术的敲除效率不相同,在拟南芥中的敲除率最低,在水稻中的编辑效率最高[27–28]。Li等[29]利用CRISPR/Cas9技术对水稻基因组进行编辑,结果表明水稻的基因组编辑效率很高,而且突变主要发生在G至A的改变,但也有G至C或G至T的改变。这与本研究结果相一致,水稻OsPht基因编辑效率较高,占水稻株系总突变率为53.8%,后6个碱基成功敲除的株系占总突变株系的28.6%,后8个碱基成功敲除的株系占突变株系的42.9%,GT碱基突变株系占突变株系的28.6%。高清松对水稻OsABC1K3突变体研究发现,OsABC1K3突变体的株高和叶绿素含量低于野生型[30];辛伟杰对OsPT2和OsPT6的突变体水稻进行研究发现,突变体水稻的生物量低于野生型,根长、总表面积和总体积等高于野生型[31],与我们的结果相一致,突变体水稻Cas-2和Cas-7的鲜重和株高均小于野生型,根长、表面积、投影面积和侧根数高于野生型,Cas-7的叶绿素含量小于野生型。有研究表明根是植物吸收营养的主要器官,根系的生长和活力直接影响植物地上部的生长情况[21],与我们的结果相符,野生型水稻地上部生长优于突变体,根系活力显著高于突变体。尽管我们没有测试植株体内磷素吸收量 (由于干重量不足无法满足测试需求),但突变体株系生物量的显著减少,无疑会影响水稻OsPht基因碱基缺失突变体对磷素的吸收。Anna等发现正常处理下,突变体522DK和527DK中葡萄糖及果糖含量高于野生型[32],我们的研究同样发现Cas-2中可溶性糖含量高于野生型。
CRISPR/Cas9基因编辑系统的问世给基因组定向编辑的研究带来突破性的技术创新,特别是对基因功能的研究有广阔的应用前景,使农作物水稻、小麦在性状改良与分子育种方面朝着对人类有益的方向发展[23]。水稻作为模式植物,具有基因组简单、结实率高和遗传操作简单等优点,被作为研究的首选植物。本研究对水稻磷转运蛋白OsPht基因的成功编辑获得的突变体测试结果表明,CRISPR/Cas9作为基因编辑的工具可以成功应用到植物养分转运蛋白基因的编辑修饰,以科学有效地解释植物养分蛋白基因对养分转运的分子调控机理。同时,利用所获得的养分转运蛋白基因碱基缺失或突变的水稻突变体,将外源磷转运蛋白基因如冰叶日中花McPht磷转运蛋白基因转入突变体中,获得磷转运蛋白回复株系材料,以进一步科学验证McPht基因的生理生化功能。本研究表明,基于CRISPR/Cas9所介导的基因编辑技术在植物养分分子调控机理的研究与应用中具有重要的科学意义。
| [1] | Komor A C, Badran A H, Liu D R. CRISPR-based technologies for the manipulation of eukaryotic genomes[J]. Cell, 2016, 168(1-2): 20–36. |
| [2] | Garneau J E, Dupuis M E, Villion M, et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA[J]. Nature, 2010, 468: 67–71. DOI:10.1038/nature09523 |
| [3] | Marraffini L A, Sontheimer E J. CRISPR interference: RNA-directed adaptive immunity in bacteria and archaea[J]. Nature Reviews Genetics, 2010, 11(11): 181–190. |
| [4] | Sander J D, Joung J K. CRISPR-Cas systems for editing, regulating and targeting genomes[J]. Nature, 2014, 32(4): 347–355. |
| [5] | Hsu P D, Lander E S, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering[J]. Cell, 2014, 157(6): 1262–1278. DOI:10.1016/j.cell.2014.05.010 |
| [6] | Feng Z Y, Mao Y F, Xu N F, et al. Multigeneration analysis reveals the inheritance, specificity, and patterns of CRISPR/Cas-induced gene modifications in Arabidopsis [J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(12): 4632–4637. DOI:10.1073/pnas.1400822111 |
| [7] | Shan Q, Wang Y, Li J, et al. Targeted genome modification of crop plants using a CRISPR-Cas system[J]. Nature Biotechnology, 2013, 31(8): 686–688. DOI:10.1038/nbt.2650 |
| [8] | Li J F, Norville J E, Aach J, et al. Multiplex and homologous recombination mediated genome editing in Arabidopsis and Nicotiana benthamiana using guide RNA and Cas9 [J]. Nature Biotechnology, 2013, 31(8): 688–691. DOI:10.1038/nbt.2654 |
| [9] | Nekrasov V, Staskawicz B, Weigel D, et al. Targeted mutagenesis in the model plant Nicotiana benthamiana using Cas9 RNA-guided endonuclease [J]. Nature Biotechnology, 2013, 31(8): 691–693. DOI:10.1038/nbt.2655 |
| [10] | Mao Y, Zhang H, Xu N, et al. Application of the CRISPR-Cas system for efficient genome engineering in plants[J]. Molecular Plant, 2013, 6(6): 2008–2011. DOI:10.1093/mp/sst121 |
| [11] | Liang Z, Zhang K, Chen K, et al. Targeted mutagenesis in Zea mays using TALENs and the CRISPR/Cas system [J]. Genetics and Genomics, 2014, 41(2): 63–68. DOI:10.1016/j.jgg.2013.12.001 |
| [12] | Xing H L, Li D, Wang Z P, et al. A CRISPR/Cas9 toolkit for multiplex genome editing in plants[J]. BMC Plant Biology, 2014, 14(1): 1–12. DOI:10.1186/1471-2229-14-1 |
| [13] | Jacobs T B, LaFayette P R, Schmitz R J, et al. Targeted genome modifications in soybean with CRISPR/Cas9[J]. BMC Biotechnology, 2015, 15(1): 1–10. DOI:10.1186/s12896-015-0115-2 |
| [14] |
方锐, 畅飞, 孙照霖, 等. CRISPR-Cas9介导的基因组定点编辑技术[J].
生物化学与生物物理进展, 2013, 40(8): 691–702.
Fang R, Chang F, Sun Z L, et al. The techniques of CRISPR-Cas9 mediated fixed-point editing of the genome[J]. Progress in Biochemistry and Biophysics, 2013, 40(8): 691–702. |
| [15] |
李铁民, 杜波. CRISPR-Cas系统与细菌和噬菌体的共进化[J].
遗传, 2011, 33(3): 213–218.
Li T M, Du B. CRISPR-Cas system and coevolution of bacteria and phages[J]. Hereditas, 2011, 33(3): 213–218. |
| [16] | Richardson A E. Regulating the phosphorus nutrition of plants: molecular biology meeting agronomic needs[J]. Plant and Soil, 2009, 322: 17–24. DOI:10.1007/s11104-009-0071-5 |
| [17] | Yang X J, Finnegan P M. Regulation of phosphate starvation responses in higher plants[J]. Annals of Botany, 2010, 105: 513–526. DOI:10.1093/aob/mcq015 |
| [18] |
常小箭. 水稻磷酸盐转运蛋白基因家族的功能分析[D]. 武汉: 华中农业大学硕士学位论文, 2012.
Chang X J. Functional analysis of phosphate transporter gene family in rice [D]. Wuhan: MS Thesis of Huazhong Agricultural University, 2012. |
| [19] | Christine R, Marcel B. Molecular mechanisms of phosphate transport in plants[J]. Planta, 2002, 216: 23–37. DOI:10.1007/s00425-002-0921-3 |
| [20] |
强晓晶. 小盐芥ThPIP1基因的水稻遗传转化及耐盐机理研究[D]. 北京: 中国农业科学院硕士学位论文, 2015.
Qiang X J. Thellungiella halophila ThPIPl gene transferring rice and mechanism of salt stress tolerance [D]. Beijing: MS Thesis of Chinese Academy of Agricultural Sciences, 2015. |
| [21] |
张殊慧. 番茄磷转运蛋白SlPht3基因最小表达框转化小麦对磷素营养的应答[D]. 沈阳: 沈阳农业大学硕士学位论文, 2016.
Zhang S H. Responsive regulation of transformed tomato SlPht3 gene minimal expression cassette in the transgenic wheat to phosphorus nutrient [D]. Shenyang: MS Thesis of Shenyang Agricultural University, 2016. |
| [22] |
王晓霞, 苏哲, 岳彩云, 等. CRISPR-Cas9植物基因编辑系统敲除拟南芥AG基因表达载体的构建
[J].
分子植物育种, 2016, 14(3): 593–597.
Wang X X, Su Z, Yue C Y, et al. The construction of expression vector of knocking out AG gene in Arabidopsis thaliana by CRISPR-Cas9 [J]. Molecular Plant Breeding, 2016, 14(3): 593–597. |
| [23] |
李君, 张毅, 陈坤玲, 等. CRISPR/Cas系统: RNA靶向的基因组定向编辑新技术[J].
遗传, 2013, 35(11): 1265–1273.
Li J, Zhang Y, Chen K L, et al. CRISPR/Cas: a novel way of RNA-guided genome editing[J]. Hereditas, 2013, 35(11): 1265–1273. |
| [24] | Feng Z Y, Zhang B T, Ding W N, et al. Efficient genome editing in plants using a CRISPR/Cas system[J]. Cell Research, 2013, 23(10): 1229–1232. DOI:10.1038/cr.2013.114 |
| [25] | Nekrasov V, Staskawicz B, Weigel D, et al. Targeted mutagenesis in the model plant Nicotiana benthamiana using Cas9 RNA-guided endonuclease[J]. Nature Biotechnology, 2013, 31(8): 691–693. DOI:10.1038/nbt.2655 |
| [26] | Johnson R A, Gurevich V, Filler S, et al. Comparative assessments of CRISPR-Cas nucleases’ cleavage efficiency in planta[J]. Plant Molecular Biology, 2015, 87(1): 143–156. |
| [27] | Fauser F, Schiml S, Puchta H. Both CRISPR/Cas-based nucleases and nickases can be used efficiently for genome engineering in Arabidopsis thaliana[J]. Plant, 2014, 79(2): 348–359. |
| [28] | Zhang H, Zhang J, Wei P, et al. The CRISPR/Cas9 system produces specific and homozygous targete gene editing in rice in one gerneration[J]. Plant Biotechnology Journal, 2014, 12(6): 797–807. DOI:10.1111/pbi.2014.12.issue-6 |
| [29] | Li J Y, Sun Y W, Du J L, et al. Generation of targeted point mutations in rice by a modified CRISPR/Cas9 system[J]. Molecular Plant, 2017, 10(3): 526–529. DOI:10.1016/j.molp.2016.12.001 |
| [30] |
高清松, 徐梦彬, 袁彩勇, 等. 水稻OsABC1K3突变体鉴定及其对强光胁迫的响应
[J].
中国农业科学, 2016, 49(4): 609–620.
Gao Q S, Xu M B, Yuan C Y, et al. Rice OsABC1K3 gene mutant and its response to high light stress [J]. Scientia Agricultura Sinica, 2016, 49(4): 609–620. DOI:10.3864/j.issn.0578-1752.2016.04.001 |
| [31] |
辛伟杰. 通过RNAi研究水稻磷转运蛋白基因功能[D]. 南京: 南京农业大学硕士学位论文, 2008.
Xin W J. Functional analysis of rice phosphate transporter genes through RNA interference [D]. Nanjing: MS Thesis of Nanjing Agricultural University, 2008. |
| [32] | Anna J, Damian G, Ewa P, et al. Physiological and biochemical characterisation of watered and drought-stressed barley mutants in the HvDWARF gene encoding C6-oxidase involved in brassinosteroid biosynthesis [J]. Plant Physiology and Biochemistry, 2016, 99: 126–141. DOI:10.1016/j.plaphy.2015.12.003 |
 2017, Vol. 23
2017, Vol. 23  doi:
doi: 

