文章信息
- 邵洋, 胡延平, 刘勇
- SHAO Yang, HU Yanping, LIU Yong
- 慢性淋巴细胞白血病患者长链非编码RNA RP5-180C18.1的表达及其预后评价作用
- Long Non-coding RNA RP5-180C18.1 Expression and Its Prognostic Significance in Patients with Chronic Lymphocytic Leukemia
- 中国医科大学学报, 2018, 47(4): 341-345
- Journal of China Medical University, 2018, 47(4): 341-345
-
文章历史
- 收稿日期:2017-04-05
- 网络出版时间:2018-04-09 11:15
2. 中国医科大学附属盛京医院 血液研究室, 沈阳 110004
2. Department of Hematology, Shengjing Hospital, China Medical University, Shenyang 110004, China
慢性淋巴细胞白血病(chronic lymphocytic leukemia,CLL)是成熟样B淋巴细胞在外周血、骨髓、淋巴结和脾脏大量蓄积为特征的低度恶性肿瘤。CLL在欧美较为常见,而在我国相对少见。目前对CLL的诊断主要依赖于WHO标准[1],依据白细胞计数、血涂片免疫表型等对CLL进行诊断,目前CLL治疗参考NCCN指南[2],需要根据患者年龄、细胞遗传学等分层治疗,预后判断参考Rai和Binet标准[3-4],这两个传统标准主要依赖于体检及血细胞计数来判断预后,并不能充分满足患者需要。近年来一些分子生物学指标(IGHV、ZAP70、17p-和11q-等)也参与CLL的诊断治疗及预后判断[5]。为弥补缺陷,CLL-IPI (CLL international prognostic index)积分系统纳入了TP53缺失/突变、IGHV突变、血清β2-微球蛋白、临床分期和年龄5个指标辅助CLL患者的预后判断[6]。
长链非编码RNA (long non-coding RNA,lncRNA)是一类本身不编码蛋白、转录本长度超过200 nt的长链非编码RNA分子,它可在多层面上(表观遗传调控、转录调控以及转录后调控等)调控基因的表达[7]。非编码RNA的异常表达可以辅助CLL的诊断及预后分层,已有研究证明,多种miRNA及LncRNA在CLL的发生发展中起到重要作用,miR-155提高BCR受体的敏感性,其高表达预示着较差的预后[8],而与之相反,miR-150降低BCR受体的敏感性[9],同时,也有多种lncRNA的下调被认为参与肿瘤的生成,包括HOTAIR、MALAT1和SChLAP1等[10]。本研究前期通过二代测序技术筛选CLL患者中相对于健康人B淋巴细胞中lncRNA的异常表达,结果发现超过20 000种lncRNA存在异常表达,其中RP5-180C18.1存在显著上调(P < 0.001),提示RP5-180C18.1可能在CLL的发生发展中起一定的作用。本研究分析RP5-180C18.1的表达,探讨其在CLL诊断及预后判断上的作用。
1 材料与方法 1.1 研究对象收集2006年1月至2017年1月本院血液科就诊的初诊CLL患者,采用样本库中留存的临床废弃骨髓样本60例,其中男性27例,女性33例,中位年龄65岁。并采自30例造血干细胞移植供者骨髓液为健康对照。所有患者经过形态学、免疫分型、细胞遗传学及分子生物学综合确诊,实验室特点见表 1,诊断标准参照WHO标准。本实验经过中国医科大学伦理委员会批准(No. 2016PS352K)。根据相关分子生物学指标,分别根据IGHV突变、CD38及ZAP70对CLL患者进行分组比较。并对60例CLL患者进行了随访。
| Variables | Control group (n = 30) | CLL group (n = 60) | P |
| Male/female | 14/16 | 27/33 | 0.529 |
| Age (year) | 59 (23-79) | 65 (28-87) | 0.103 |
| Leukocytes (1×109/L) | 8.63±0.92 | 18.5±2.66 | < 0.001 |
| Hemoglobin (g/L) | 141.54±15.27 | 127.79±17.83 | 0.005 |
| Platelets (1×109/L) | 194.01±18.34 | 121.9±14.12 | < 0.001 |
| Prolymphocyte (1×109/L) | NA | 6.1±0.73 | NA |
| NA,not applicable. | |||
1.2 方法 1.2.1 试剂及引物
淋巴细胞分离液Lymphoprep (中国达科公司),逆转录采用RrimeScript RT reagent Kit逆转录试剂盒(日本TaKaRa公司)。实时采用SYBR Green试剂盒(日本TaKaRa公司)。引物为RP5-180C18.1,F:GCAGCGTCCAGAGGCTGG,R:GGCAGCGAGGATGCTGAACC;TBP,F:CCACGGTGAATCTGTGCT,R:GGAGTCGTCCTCGCTCTT。
1.2.2 qPCR检测采用淋巴细胞分离液分离骨髓单个核细胞,然后Trizol-氯仿方法提取细胞总RNA,Oligo dT法进行反转录反应,以管家基因TBP为内参,同时进行RP5-180C18.1和TBP的实时反应。实时反应设置3个复孔,得出Ct均值,以正常人为对照,应用ΔΔCt相对定量法分析RP5-180C18.1表达水平的差异。ΔΔCt=ΔCt实验组-ΔCt对照组,ΔCt=Ct RP5-180C18.1-Ct TBP,2-ΔΔCt即为基因表达差异的倍数。
1.3 统计学分析应用SPSS 17.0进行统计学分析。计量资料采用x±s表示。计量资料比较采用独立样本t检验,计数资料比较采用χ2检验,预后分析采用log-rank检验。P < 0.05 (双向)为差异有统计学意义。
2 结果 2.1 RP5-180C18.1在不同CLL表型中的表达结果显示,与健康对照组(1.894±0.62)比较,RP5-180C18.1在CLL患者(12.93±2.98)表达较高(P= 0.013)。与突变型CLL患者(0.505±0.07)比较,IGHV野生型CLL患者(4.682±1.08) RP5-180C18.1表达高(P= 0.023);与ZAP70高表达CLL患者(0.262±0.04)比较,ZAP70低表达CLL患者(1.728±0.42) RP5-180C18.1表达较高(P= 0.002);CD38低表达(1.162±0.31)与高表达(2.183±0.67) CLL患者RP5-180C18.1表达差异无统计学意义(P= 0.206)。
2.2 RP5-180C18.1表达与CLL分型相关分析将60例CLL样本根据RP5-180C18.1相对表达量的中位数(2.863)进行分组[RP5-180C18.1低表达组(< 2.863)和RP5-180C18.1高表达组(> 2.863)],分析CLL患者临床特征、分型与RP5-180C18.1表达相关性。结果显示,RP5-180C18.1表达与CLL患者性别、年龄、血红蛋白含量及幼稚淋巴细胞比例无关(P分别为0.604、0.128、0.133、0.058);RP5-180C18.1高表达组CLL患者的白细胞计数较低(P < 0.001),血小板含量较高(P < 0.001)。根据分子生物学特征分组结果显示,RP5-180C18.1表达与CD38表达无关(P= 0.796);RP5-180C18.1高表达组CLL患者ZAP70表达率较高(P= 0.019),野生型IGHV比例较高(P= 0.030)。见表 2。
| Variables | RP5-180C18.1 low expression(n = 30) | RP5-180C18.1 high expression(n = 30) | P |
| Male/female | 18/12 | 15/15 | 0.604 |
| Age(year) | 64(28-81) | 67(30-87) | 0.128 |
| Leukocytes(×109/L) | 20.82±2.05 | 14.27±1.22 | < 0.001 |
| Hemoglobin(g/L) | 128.65±12.21 | 123.42±14.3 | 0.133 |
| Platelets(×109/L) | 109.13±10.31 | 124.7±9.15 | < 0.001 |
| Prolymphocyte(×109/L) | 6.3±0.48 | 5.7±0.44 | 0.058 |
| ZAP70 [n(%)] | 0.019 | ||
| Negative | 19(63.3) | 9(30.0) | |
| Positive | 11(36.7) | 21(70.0) | |
| CD38 [n(%)] | 0.796 | ||
| Negative | 15(50.0) | 13(43.3) | |
| Positive | 15(50.0) | 17(56.7) | |
| IGHV [n(%)] | 0.030 | ||
| Wild type | 19(63.3) | 27(90.0) | |
| Mutant type | 11(36.7) | 3(10.0) | |
2.3 RP5-180C18.1表达与CLL患者预后相关分析
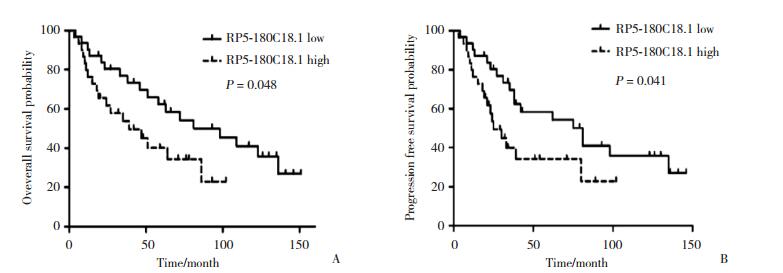
对60例CLL患者随访结果显示,中位总生存期(overall survival,OS) 93个月,中位无进展生存期(progression-free survival,PFS) 75个月。RP5-180C18.1低表达组中位OS 98个月,RP5-180C18.1高表达组中位OS 39个月,两者具有统计学差异(P= 0.048),见图 1A,RP5-180C18.1高表达CLL患者OS更短。如图 1B所示,RP5-180C18.1低表达组中位PFS 81个月,RP5-180C18.1高表达组中位PFS 25个月,两者具有统计学差异(P= 0.041),RP5-180C18.1高表达的CLL患者PFS更短。RP5-180C18.1高表达组预后相对于RP5-180C18.1低表达组更差。
|
| A, overall survival of patients with chronic lymphocytic leukemia (CLL) according to RP5-180C18.1 expression; B, progression-free survival of patients with CLL according to RP5-180C18.1 expression. 图 1 根据RP5-180C18.1表达量对CLL患者进行的Kaplan-Meier生存分析结果 Fig.1 Kaplan-Meier survival curves of patients with CLL according to RP5-180C18.1 expression |
3 讨论
RP5-180C18.1位于1号染色体,编码一段596 bp的转录本,基因ID为ENST00000413901,本研究发现lncRNA RP5-180C18.1在CLL不同表型中存在差异性表达,并且发现其在CLL诊断及预后判断具有辅助作用。结果显示RP5-180C18.1在CLL患者中相对于健康对照存在高表达,提示RP5-180C18.1有作为检测CLL诊断学标志物的潜力。已有研究证明,lncRNA在肿瘤中存在显著差异性表达,可以作为生物标志物辅助肿瘤检测。有研究[11]证明在患者尿液样本中检测lncRNA PCA3可以比前列腺特异性抗原更敏感更特异诊断前列腺癌。血清中检测rp11-445h22.4发现其在乳腺癌组织中高表达,并且具有92%敏感性和74%特异性,显著优于常规生物标志物(CEA、CA125、CA153、AFP) [12]。另外,在胃癌患者体液样本中检测lncRNA tincr,ccat2,aoc4p,bancr,linc00857,aa174084和H19发现,这些lncRNA可以有效鉴别胃癌患者与健康人,并且可以判断胃癌发展的阶段[13]。需要注意的是,本研究对照组设置相对于实验组在参数(幼稚细胞比例及血常规)上存在统计学差异,但这并不违背统计学原理,本研究目的是探索RP5-180C18.1作为CLL生物学标志物的潜力,需要把CLL从健康人中鉴别出来,因此必须采用健康人群作为对照,而幼稚细胞数及血常规指标正是CLL的常见异常。外周血标本来源便捷,临床上最理想的检测标志物采用外周血指标,但是因为标本收集受限,本研究未检测外周血RP5-180C18.1含量,本研究骨髓检测RP5-180C18.1表达可靠,同时期待应用于临床CLL诊断,后续将进一步完善外周血RP5-180C18.1表达检测。
对CLL患者随访分析结果显示,RP5-180C18.1高表达患者具有更差的预后。研究[14]证明lncRNA在肿瘤预后判断起重要作用,lncRNA CCAT2在多种肿瘤(结肠癌、前列腺癌和甲状腺癌等)中过表达,并且预示着较差预后。也有研究[15]认为treRNA降低CLL中DNA损伤,其高表达预示着较差预后。本研究发现RP5-180C18.1在野生型IGHV和ZAP70高表达CLL患者中表达更高,这是否与RP5-180C18.1高表达CLL患者预后较差有关尚未证实,需进一步研究。研究[16]证明IGHV未突变型或ZAP70高表达CLL患者具有较差预后,这也从另一方面支持本研究结果。
综上所述,lncRNA RP5-180C18.1在CLL中高表达,与CLL侵袭性表型相关,并且其高表达具有较差预后。今后应对RP5-180C18.1表达深入研究,为阐明CLL的发病机制提供科学依据。
| [1] |
HALLEK M. Chronic lymphocytic leukemia:2017 update on diagnosis, risk stratification, and treatment[J]. Am J Hematol, 2017, 92(9): 946-965. DOI:10.1002/ajh.24826 |
| [2] |
WIERDA WG, ZELENETZ AD, GORDON LI, et al. NCCN guidelines insights:chronic lymphocytic leukemia/small lymphocytic leukemia, version 1.2017[J]. J Nat Comprehensive Cancer Network, 2017, 15(3): 293-311. DOI:10.6004/jnccn.2017.0030 |
| [3] |
BINET JL, AUQUIER A, DIGHIERO G, et al. A new prognostic classification of chronic lymphocytic leukemia derived from a multivariate survival analysis[J]. Cancer, 1981, 48(1): 198-206. DOI:10.1002/(ISSN)1097-0142 |
| [4] |
RAI KR, HAN T. Prognostic factors and clinical staging in chronic lymphocytic leukemia[J]. Hematology/oncology Clini North Am, 1990, 4(2): 447-456. |
| [5] |
CLAUS R, LUCAS DM, STILGENBAUER S, et al. Quantitative DNA methylation analysis identifies a single CpG dinucleotide important for ZAP-70 expression and predictive of prognosis in chronic lymphocytic leukemia[J]. J Clin Oncol, 2012, 30(20): 2483-2491. DOI:10.1200/JCO.2011.39.3090 |
| [6] |
A Clinical Evaluation of the International Lymphoma Study Group Classification of Non-Hodgkin's Lymphoma. The non-Hodgkin's lymphoma classification project[J]. Blood, 1997, 89(11): 3909-3918. |
| [7] |
GUTTMAN M, DONAGHEY J, CAREY BW, et al. lncRNAs act in the circuitry controlling pluripotency and differentiation[J]. Nature, 2011, 477(7364): 295-300. DOI:10.1038/nature10398 |
| [8] |
GUINN D, RUPPERT AS, MADDOCKS K, et al. miR-155 expression is associated with chemoimmunotherapy outcome and is modulated by Bruton's tyrosine kinase inhibition with Ibrutinib[J]. Leukemia, 2015, 29(5): 1210-1213. DOI:10.1038/leu.2014.344 |
| [9] |
MRAZ M, CHEN L, RASSENTI LZ, et al. miR-150 influences B-cell receptor signaling in chronic lymphocytic leukemia by regulating expression of GAB1 and FOXP1[J]. Blood, 2014, 124(1): 84-95. DOI:10.1182/blood-2013-09-527234 |
| [10] |
PRENSNER JR, IYER MK, SAHU A, et al. The long noncoding RNA SChLAP1 promotes aggressive prostate cancer and antagonizes the SWI/SNF complex[J]. Nat Genet, 2013, 45(11): 1392-1398. DOI:10.1038/ng.2771 |
| [11] |
TINZL M, MARBERGER M, HORVATH S, et al. DD3PCA3 RNA analysis in urine——a new perspective for detecting prostate cancer[J]. Eur Urol, 2004, 46(2): 182-186. DOI:10.1016/j.eururo.2004.06.004 |
| [12] |
SHAPPELL SB. Clinical utility of prostate carcinoma molecular diagnostic tests[J]. Rev Urol, 2008, 10(1): 44-69. |
| [13] |
ZHANG K, SHI H, XI H, et al. Genome-wide lncRNA microarray profiling identifies novel circulating lncRNAs for detection of gastric cancer[J]. Theranostics, 2017, 7(1): 213-227. DOI:10.7150/thno.16044 |
| [14] |
WANG D, CHEN Z, XU H, et al. Long noncoding RNA CCAT2 as a novel biomaker of metastasis and prognosis in human cancer:a meta-analysis[J]. Oncotarget, 2017, 8(43): 75664-75674. DOI:10.18632/oncotarget.18161 |
| [15] |
MILLER CR, RUPPERT AS, FOBARE S, et al. The long noncoding RNA, treRNA, decreases DNA damage and is associated with poor response to chemotherapy in chronic lymphocytic leukemia[J]. Oncotarget, 2017, 8(16): 25942-25954. DOI:10.18632/oncotarget.15401 |
| [16] |
PARIKH SA, STRATI P, TSANG M, et al. Should IGHV status and FISH testing be performed in all CLL patients at diagnosis? A systematic review and meta-analysis[J]. Blood, 2016, 127(14): 1752-1760. DOI:10.1182/blood-2015-10-620864 |
 2018, Vol. 47
2018, Vol. 47