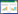文章信息
- 柳中洋, 卢颖, 韩丽英, 马艳萍, 齐莹, 黄郁晶, 阮强
- LIU Zhongyang, LU Ying, HAN Liying, MA Yanping, QI Ying, HUANG Yujing, RUAN Qiang
- 细菌人工染色体技术在人巨细胞病毒基因组小片段序列置换突变中的应用
- Application of Bacterial Artificial Chromosome in Small Fragment Mutation of the Human Cytomegalovirus Genome
- 中国医科大学学报, 2018, 47(2): 102-106
- Journal of China Medical University, 2018, 47(2): 102-106
-
文章历史
- 收稿日期:2017-07-14
- 网络出版时间:2018-01-08 10:54
2. 锦州医科大学基础医学院病原生物学教研室, 辽宁 锦州 121001;
3. 上海交通大学附属儿童医院新生儿科, 上海 200062
2. Department of Immunology and Pathogenic Biology, School of Basic Medicine, Jinzhou Medical University, Jinzhou 121001, China;
3. Neonatal Department, Children's Hospital of Shanghai Jiaotong University, Shanghai 200062, China
人巨细胞病毒(human cytomegalovirus,HCMV)是引起先天畸形的重要因素之一[1],分别研究每个基因的功能是揭示其致病机制的有效手段,单个基因的敲除与编辑是基因功能研究的重要方法。细菌人工染色体(bacterial artificial chromosome,BAC)具有拷贝数低、携带外源DNA片段大、易于操作等特点[2]。1999年,BORST等[3]构建了第1个HCMV-BAC,BAC成为研究HCMV基因功能的重要技术,但是BAC技术可置换HCMV DNA长度的下限尚无报道。LUNA位于复制必需基因UL80和UL82之间,其转录本与UL80和UL82的转录本都有重叠[4]。本研究通过置换突变HCMV-BAC中LUNA转录起始端短片段,探讨了HCMV-BAC技术在小片段序列置换突变中的应用,为HCMV基因小片段的编辑研究提供方法学支持。
1 材料与方法 1.1 材料HCMV Han株为中国医科大学附属盛京医院病毒研究室保存的临床分离株,Han株细菌人工染色体(Han-BAC)是病毒研究室与武汉病毒所合作构建[5];人胚肺成纤维细胞(human embryonic lung fibroblast,HELF)购自中国科学院上海细胞所;DH10B菌种购自美国Invitrogen公司;DY380菌种,pGEM-oriv/kana2质粒和pCDNA3-pp71质粒为武汉病毒所赠送。
1.2 方法 1.2.1 置换突变位置的选择分析LUNA转录起始位点附近结构[4],针对UL80与UL82转录方向相反的特点,选择UL80和UL82转录终止信号“AATAA”之间的31 bp作为置换突变位点,其侧翼各50 bp序列作为同源臂,见图 1。

|
| The underlined nucleotides represent 5'segment of LUNA transcript. The nucleotides in boldface represent the terminal signal of UL80 and UL82 or the promoter of LUNA. The nucleotides highlighted in grey represent the 31 bp target mutation sequence. 图 1 LUNA及其附近基因结构 Fig.1 Structure and sequence of LUNA and its neighboring genes |
1.2.2 PCR扩增卡那霉素抗性基因的序列
使用5’末端带有LUNA同源臂序列的卡那霉素抗性基因特异性引物(表 1),以pGEM-oriv/kana2质粒作为模板,按照La Taq(日本TaKaRa公司)推荐的反应体系,PCR扩增用于替换LUNA序列的卡那霉素抗性基因。卡那霉素抗性基因末端具有终止密码子“AATAA”。使用琼脂糖凝胶电泳检测PCR扩增产物。使用Gel and PCR Clean-Up System(美国Promega公司)纯化回收PCR产物,-20 ℃保存。
| Primer name | Sequence |
| Kanamycine resistant gene primer F | 5'-atttagggtttccctctctttttttttctacaccgtgataccctaataaaCTGTCTCTTATACACATCTCAACCATC-3' |
| Kanamycine resistant gene primer R | 5'-tcccccgcgtgtttttcccattccctgtatttatttctaaataataaaaaCTGTCTCTTATACACATCTCAACCCTG-3' |
| Identification primer F | 5'-TTTTCTACACCGTGATACCCT-3' |
| Identification primer R | 5'-CGCATTACGACCTACCTCTT-3' |
| LUNA outer primer | 5'-ccgttgttaccgcttcgacgtc-3' |
| LUNA inner primer | 5'-agtccggcttgggtgtccgag-3' |
| UL80 outer primer | 5'-caatagcgacggtggaagtggc-3' |
| UL80 inner primer | 5'-atctgtttgccgcgcggcagag-3' |
| UL82 outer primer | 5'-ccgagtacatcgtgcagatccag-3' |
| UL82 inner primer | 5'-accaatcagactcacgacaccatc-3' |
1.2.3 同源重组置换突变LUNA基因转录起始端序列
制备含有Han-BAC的DY380电转化感受态细菌[2],当细菌增殖至600 nm吸光度达到0.4时转移到水浴摇床中,42 ℃ 225 r/min,热诱导Red重组酶表达15 min。将1 μg纯化的卡那霉素抗性基因片段与100 μL的感受态细菌混合,转移到冰浴的1 mm电转杯中(美国Biorad公司),1.8 kV,25 μF,200 Ω电脉冲处理1次。迅速向电转化的细菌中加入800 μL LB(-)培养基,32 ℃,225 r/min培养1 h,涂于卡那霉素抗性LB平皿,32 ℃培养30 h。使用PCR方法,利用突变位点外侧的鉴定引物(表 1)对单克隆菌落进行鉴定,选择扩增鉴定阳性克隆进行保种和测序。
1.2.4 电转化拯救病毒按照试剂盒推荐程序,使用质粒DNA纯化试剂盒(德国MACHEREY-NAGEL公司)提取LUNAΔ株质粒,使用DNA Purification System(美国Promega公司)提取pCDNA3-pp71质粒,使用NanoDrop ND-1000(美国Thermo Fisher Scientific公司)检测质粒浓度。使用0.25%的胰酶(美国Gibco公司)消化培养成单层的HELF细胞,调整细胞密度至5×105/100 μL。取500 μL细胞悬液至预冷的1.5 mL EP管中,加入LUNAΔ质粒5 μg,pCDNA3-pp71质粒1 μg,吹打混匀后转入冰上预冷的4 mm电转杯(美国Biorad公司)中,260 V,975 μF,电阻∞,电脉冲处理1次。将电转化的细胞放回T25细胞培养瓶中,使用含有10% FBS(以色列BI公司)的MEM(美国Hyclone公司)于37 ℃,5% CO2条件下培养,每3 d换液1次。荧光显微镜(日本尼康公司)下观察绿色荧光和细胞病变效应(cytopathic effect,CPE)情况。待CPE达到100%时收获病毒,作为毒种保存。
1.2.5 实时PCR联合cDNA克隆测序检测缺失突变后LUNA、UL80和UL82的转录情况和转录本结构分别使用Han株和LUNAΔ株接种HELF,待病变率 > 80%时使用Trizol(美国Invitrogen公司)收获感染细胞,酚-氯仿法抽提总RNA。使用TURBO DNA-freeTM Kit(美国Ambio公司)对总RNA进行去DNA处理。使用3’-FULL RACE Core Set with PrimeScript Rtase(日本TaKaRa公司)试剂盒中的oligo dT将总RNA反转录成cDNA,以基因特异性引物作为上游引物(表 1),oligo dT引入的通用引物作为下游引物,采用巢式PCR分别扩增LUNA、UL80和UL82的mRNA 3’端序列。PCR产物经过切胶纯化,TA克隆到PCR2.1载体(美国Invitrogen公司)后进行测序。使用Bioedit软件比对Han与LUNAΔ株的测序结果。
2 结果 2.1 缺失突变结果缺失突变后挑取32个单克隆菌落进行PCR及琼脂糖凝胶电泳鉴定,其中1个菌落的鉴定结果显示,目的片段被卡那霉素抗性片段成功替换,见图 2A。测序结果显示,该菌落中的Han-BAC的LUNA目的片段完全按照预期缺失,缺失位点侧翼的UL80和UL82基因结构未受影响,见图 2B。
|
| A, PCR identification of partial monoclones. The white arrow indicates that the target sequence was replaced by kanamycin resistance sequence successfully. B, the result of sequencing around the mutation site of Han wild type strain and LUNAΔ strain. The upper sequence is of the target site and the flanking sequence of the wild type strain. The lower sequence is of the LUNAΔ strain. The nucleotides in capital represent the mutation site and the kanamycin resistance gene sequence. The nucleotides in lowercase represent the homologous arm region. The underlined nucleotides represent the transcription stop signal of UL80 and UL82. 图 2 缺失突变克隆鉴定 Fig.2 Identification of the mutated clones |
2.2 病毒拯救与病毒活力检测结果
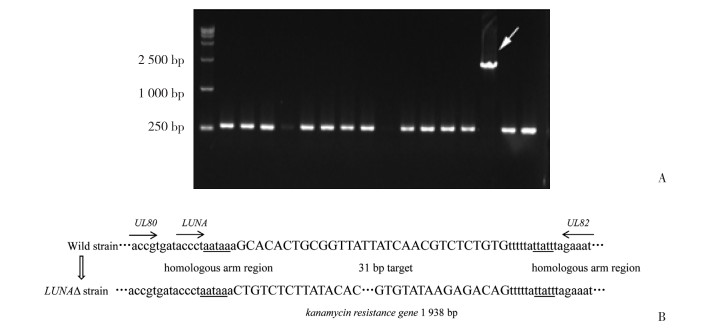
电转化拯救10 d后出现HCMV典型CPE,荧光显微镜下可见来自于病毒表达的绿色荧光蛋白信号,说明LUNAΔ株成功包装成病毒颗粒,并且具有感染细胞的能力,见图 3。
|
| A, the typical HCMV CPE formation of rescued LUNAΔ strain; B, the same field under ultraviolet excitation. The GFP signals are consistent with those of CPE. 图 3 LUNAΔ株拯救结果×200 Fig.3 The rescue result of LUNAΔ strain ×200 |
2.3 实时PCR与cDNA克隆测序结果
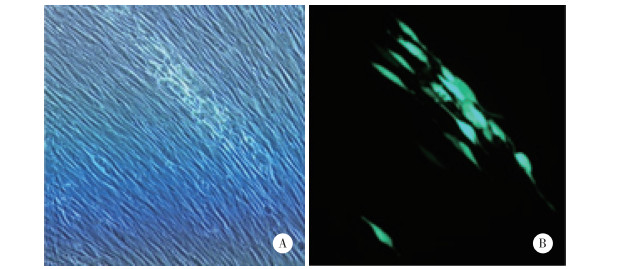
在LUNAΔ株感染的HLEF中,UL80与UL82转录正常(图 4A)。野生Han株与LUNAΔ株感染的HELF中均未检测到明确的LUNA mRNA转录。实时PCR产物测序分析结果显示,尽管突变株在置换突变后导致终止信号“AATAA”之后的序列发生了改变,但UL80与UL82仍然在既定位置切割并且添加了polyA尾(图 4B),其转录本3’末端结构完整。电泳中不符合预期产物长度的其他条带,经测序证实均为非特异扩增产物。
|
| A, agarose gel electrophoresis of the real-time PCR products. The white arrows indicate the prospective real-time PCR products of UL80 and UL82. B, sequencing of the 3'end of the UL80 and UL82 transcripts. The nucleotides in italics represent sequences after replacement mutation. The underlined nucleotides represent transcription stop signals. 图 4 病毒感染后基因转录本实时PCR鉴定结果 Fig.4 Identification of viral gene transcriptions after infection by real-time PCR |
3 讨论
在病毒基因功能研究方面,突变待研究的病毒基因是最具说服力的实验证据。采用BAC技术进行特定病毒基因突变具有诸多优势。在既往使用Han-BAC进行病毒基因突变时,均将目的基因完全替换,目的片段与进行替换的卡纳那霉素抗性基因片段长度相近,同源置换的空间阻力较小,成功率较高。由于HCMV的部分基因存在重叠,缺失突变掉过大的片段可能会造成临近基因结构的破坏,解决基因小片段突变势在必行。目的片段的缩短使2个同源重组位点彼此靠近,导致卡那霉素抗性基因两翼的同源臂捕获靶位点的阻力增大,同源重组的效率可能明显下降。能否以较大的卡那霉素抗性基因重组置换小片段的病毒基因序列尚无报道,Han-BAC系统可替换的病毒基因片段的下限也不清楚。
本研究以Han-BAC为基础,使用同源重组技术,成功以1 938 bp的卡那霉素抗性基因替换了31 bp的LUNA转录起始端序列。在插入的卡那霉素抗性基因末端存在转录终止信号AATAA,可以阻断LUNA启动子与其编码区的转录启动关系。这一结果表明,使用Han-BAC可以实现小至31 bp序列的有效置换。以小片段置换突变为基础,同时通过在卡那霉素抗性基因引物与同源臂上之间引入启动子、终止信号、终止密码子等序列[6]来调控基因的转录和表达,对于缺少足够空间进行大片段缺失的基因是一个可行的解决方法。
除了本研究所采用的BAC技术可以进行病毒基因突变以外,现有的galk系统也可以实现小片段缺失突变。该系统在体外构建好含有目的突变位点的打靶序列,经过两重同源重组与筛选过程,使用打靶序列替换目的序列,并且没有外源基因残留[7]。在某些情况下,如果单次缺失突变即可达到目的,可以有效节省时间与成本。本研究结果显示,在目的片段缩小到31 bp的条件下,缺失突变的效率仍然达到3%以上,可以满足实验需要。通过在制备感受态过程中提高重组酶的表达量,预期可以进一步提高重组效率。
本研究中,使用实时PCR技术在未突变的Han株感染的HLEF细胞中没有检测到LUNA转录本。分析其原因认为,LUNA的表达量低[8]导致PCR扩增难以得到单一产物;同时LUNA是潜伏感染相关基因,裂解感染状态下其转录可能受到抑制。即便如此,对突变病毒株进行DNA测序的结果依然表明,使用BAC技术可以成功置换小片段序列,而这一突变对临近基因的转录和转录本结构没有影响,提示相关技术可以用于病毒基因小片段的缺失突变和有关基因功能的研究。
| [1] |
MANICKLAL S, EMERY VC, LAZZAROTTO T, et al. The "silent" global burden of congenital cytomegalovirus[J]. Clin Microbiol Rev, 2013, 26(1): 86-102. DOI:10.1128/CMR.00062-12 |
| [2] |
J.萨姆布鲁克, D.W.拉塞尔, 黄培堂. 分子克隆实验指南[M]. 北京: 科学出版社, 2002, 101-102, 340-343.
|
| [3] |
BORST EM, HAHN G, KOSZINOWSKI UH, et al. Cloning of the human cytomegalovirus (HCMV) genome as an infectious bacterial artificial chromosome in Escherichia coli:a new approach for construction of HCMV mutants[J]. J Virol, 1999, 73(10): 8320-8329. |
| [4] |
BEGO M, MACIEJEWSKI J, KHAIBOULLINA S, et al. Characterization of an antisense transcript spanning the UL81-82 locus of human cytomegalovirus[J]. J Virol, 2005, 79(17): 11022-11034. DOI:10.1128/JVI.79.17.11022-11034.2005 |
| [5] |
ZHAO F, SHEN ZZ, LIU ZY, et al. Identification and BAC construction of Han, the first characterized HCMV clinical strain in China[J]. J Med Virol, 2016, 88(5): 859-870. DOI:10.1002/jmv.24396 |
| [6] |
XU Z, ZIKOS D, OSTERRIEDER N, et al. Generation of a complete single-gene knockout bacterial artificial chromosome library of cowpox virus and identification of its essential genes[J]. J Virol, 2014, 88(1): 490-502. DOI:10.1128/JVI.02385-13 |
| [7] |
WARDEN C, TANG Q, ZHU H. Herpesvirus BACs:past, present, and future[J]. J Biomed Biotechnol, 2011, 2011: 124595. DOI:10.1155/2011/124595 |
| [8] |
ROSSETTO CC, TARRANT-ELORZA M, PARI GS. Cis and trans acting factors involved in human cytomegalovirus experimental and natural latent infection of CD14(+) monocytes and CD34(+) cells[J]. PLoS Pathog, 2013, 9(5): e1003366. DOI:10.1371/journal.ppat.1003366 |
 2018, Vol. 47
2018, Vol. 47