2. 泰州市金鹏鹅业专业合作社, 泰州 225319
2. Taizhou City Jinpeng Geese Professional Cooperative, Taizhou 225319, China
肌肉的发育过程包括卫星细胞的激活,成肌细胞的生成、分化和融合,以及多核肌纤维的形成等。研究显示,生肌调节因子(myogenic regulatory factors, MRFs)家族是肌纤维生长发育的重要调控基因,该家族基因表达沉默将导致生肌节形成失败,最终无法形成肌肉[1]。肌细胞生成素(myogenin,MyoG)基因是MRFs家族的重要成员,在促进成肌细胞增殖和融合为多核肌细胞过程中起着重要的作用,是唯一不可替代的成肌调节因子[2],同时,MyoG还与家族内其他成员相互作用,共同调节畜禽产肉量和肉质性状[3]。鸡和鸭MyoG基因存在多处SNPs位点,并且与肉质性状和屠宰性状显著相关[4-5],说明MyoG基因是家禽肌肉发育的重要候选基因。
MyoG基因的转录和表达受到多种因素的影响,如营养水平、运动等外部环境因素[6-7]和组蛋白脱乙酰基酶(HDAC)、乙酰胆碱(Ach)等神经递质[8-9]以及激素[10-11]等多种方式。此外,表观遗传修饰研究发现,鸭MyoG基因启动子区CpG岛通过甲基化修饰影响基因在腿肌中的转录[12]。研究基因启动子结构可以更深入了解MyoG的表达调控机制,目前多个物种MyoG基因启动子已经被成功克隆,转录调控机制研究取得一定进展,发现多个转录因子如MZF1、CdxA、E-box等可能参与MyoG启动子调控[3, 13-14]。然而,鹅MyoG基因转录调控机制目前尚未见报道。
鹅肉具有高蛋白、低脂肪的优点,而且富含不饱和脂肪酸,近年来的消费量稳步上涨,鹅的肌肉发育调控值得深入研究。本研究拟克隆鹅MyoG基因的启动子片段,初步预测启动子区转录因子结合位点,扩增启动子系列缺失片段,对转录因子进行定点突变,应用双荧光素酶检测方法确定调控鹅MyoG基因转录调控的关键元件,为进一步探索MyoG基因对鹅肌肉发育的调控机理提供理论基础。
1 材料与方法 1.1 材料和试剂70日龄泰州鹅胸肌、腿肌、心、肝、脾、肺、肾和下丘脑8种组织样品由本实验室保存。
Tks GflexTM DNA Polymerase、T4 DNA连接酶、pMD19-T载体、DH5α感受态细胞和琼脂糖凝胶DNA回收试剂盒购自TaKaRa公司;限制性内切酶NheⅠ、Bgl Ⅱ购自美国NEB公司;DNA提取试剂盒、无内毒素质粒小量提取试剂盒购自鼎国生物公司;荧光素酶报告质粒pGL3-Enhancer、pRL-TK、pGL3-basic以及双荧光素酶报告基因检测试剂盒购自Promega公司;DNA Marker购自上海生工;Mut Express® Ⅱ Fast Mutagenesis Kit V2定点突变试剂盒购自Vazyme公司;胎牛血清、0.25%胰蛋白酶、Opti-MEM培养基和Lipofectamine LTX购自Thermo Fisher Science公司(美国);人胚肾细胞(C2C12)由本实验室保存。
1.2 鹅MyoG基因5′侧翼区片段扩增以泰州鹅腿肌DNA为模板,已知鹅MyoG基因部分序列(GenBank No: DQ294735.1),根据同源鸭(Anas platyrhynchos)MyoG基因编码区(GenBank No: NW_004676592.1)和启动子区(GenBank No: KX981598)序列信息,设计引物M0F/R扩增基因上游预测序列获得M0片段。生物信息学分析确定M0序列正确且包含启动子片段后,以M0序列为模板设计4条上游引物(M1F、M2F、M3F、M4F)递减扩增启动子区片段(引物序列见表 1)。对所有上游引物引入NheⅠ限制性酶切位点及保护碱基,下游引物引入Bgl Ⅱ酶切位点及对应的保护碱基,引物由上海生工合成。PCR扩增体系为50 μL: 1 μL Tks Gflex DNA Polymerase,22 μL ddH2O,25 μL 2×Gflx PCR Buffer,1 μL DNA模板,上下游引物各0.5 μL。PCR反应条件:98 ℃预变性5 min;98 ℃变性10 s,退火温度退火15 s,68 ℃延伸3 min,共30个循环;68 ℃最终延伸10 min,9 ℃保存。扩增产物通过1.5%凝胶电泳检测,产物回收纯化后连接到pMD19-T载体上,重组质粒经NheⅠ和Bgl Ⅱ酶切鉴定正确后,送上海生工测序。
|
|
表 1 扩增鹅MyoG基因启动子序列的引物 Table 1 Primers for amplifying promoter prediction sequence of goose MyoG gene |
利用在线数据库GPminer(http://gpminer.mbc.nctu.edu.tw/index.php)对扩增产物M0序列进行转录起始位点、TATA-box、CAAT-box和GC-box位点等预测。使用Signal Scan(http://http://www-bimas.cit.nih.gov/molbio/signal)和JASPAR数据库(http://jaspar.genereg.net/)对核心启动子区内潜在转录因子结合位点进行预测。利用DNAstar软件中Megalign程序进行序列同源性比对。
1.4 鹅MyoG报告基因载体构建及鉴定将PCR扩增得到的递减长度的鹅MyoG基因启动子片段进行NheⅠ和Bgl Ⅱ双酶切,经凝胶回收、T4 DNA连接酶连接后,定向克隆到同样双酶切的pGL3-Enhancer载体中,连接产物转化DH5α感受态细胞,挑选阳性单克隆菌落,进一步测序鉴定。利用NheⅠ和Bgl Ⅱ双酶切和测序验证,筛选构建正确的鹅MyoG基因pGL3-MyoG(-1 114~+37)、pGL3-MyoG(-774~+37)、pGL3-MyoG(-624~+37)和pGL3-MyoG(-154~+37)重组双荧光素酶报告基因质粒。
1.5 鹅MyoG基因突变位点报告基因载体的构建根据启动子活性检测结果,本试验运用在线软件对启动子核心区潜在的转录因子结合位点进行了预测,relative profile score阈值设定为0.95,总共筛选出3个转录因子结合位点:HNF4(-521~-503 bp)、USF(-379~-370 bp)和E2(-296~-281 bp)。利用点突变技术对3个转录因子结合位点进行敲除,设计并合成转录因子结合位点缺失的引物,引物序列见表 1。以HNF4位点突变试验为例,以pGL3-MyoG(-624~+37)质粒为模板,Mut1-HNF4-F/Mut1-HNF4-R为引物,PCR扩增整个质粒,扩增完成后,回收扩增片段,加入Dpn Ⅰ酶,去除模板质粒,再次回收,回收产物加T4 DNA连接酶连接,转化大肠杆菌,涂布平板,过夜培养,挑取单克隆,提取质粒进行双酶切验证后测序鉴定。构建突变位点报告基因载体,分别命名为pGL3-HNF4-MyoG、pGL3-USF-MyoG和pGL3-E2-MyoG。
1.6 鹅MyoG和USF基因组织表达分析以β-actin为内参基因,荧光定量PCR检测泰州鹅胸肌、腿肌、心、肝、脾、肺、肾、下丘脑8种组织中MyoG和USF的表达情况,定量引物见表 2。每个样品重复3次。
|
|
表 2 荧光定量引物 Table 2 Primers for fluorescence quantitative detection |
接种C2C12细胞于96孔板,添加DMEM/F12培养液(含有10%胎牛血清、0.10 μL·L-1卡那霉素、0.10 μL·L-1氨苄霉素),37 ℃、5% CO2条件下培养,当细胞生长至汇合率70%~80%时,将构建的重组双荧光素酶报告载体采用脂质体Lipo LTX转染到以上细胞内,作为试验组,同时转染pGL3-basic为阴性对照组,pRL-TK为内参质粒,试验重复3次,每组3个平行试验。质粒瞬时转染参照Lipofectamine LTX试剂盒说明书进行。质粒转染24 h后,收集细胞裂解液。根据双荧光素酶检测试剂盒(Promega)说明书测定Firefly和Renilla荧光素酶活性,通过计算其比值确定启动子片段相对活性。
1.8 数据分析qRT-PCR测定的基因表达量利用2-△△Ct方法计算,运用GraphPad prism软件进行单因素方差分析,使用SPSS 17.0对数据进行多重比较,P<0.05认为差异显著,以*表示,P<0.01认为差异极显著,以**表示。
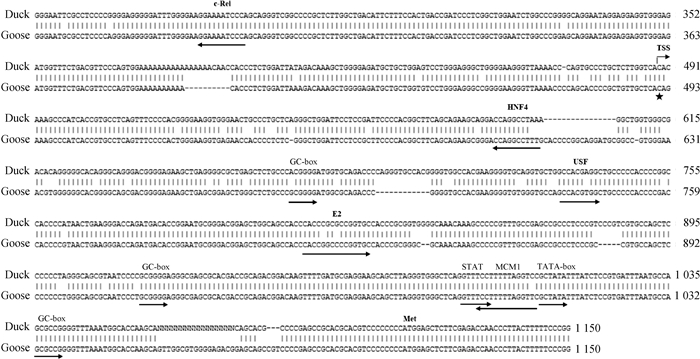
2 结果 2.1 鹅MyoG基因5′侧翼序列生物信息学分析对鹅MyoG基因5′端侧翼区序列进行在线生物信息学分析发现,鹅MyoG基因存在1个TATA - box,3个GC-box,无CAAT-box,BDGP在线预测鹅MyoG基因转录起始位点(TSS)位于-623 bp处。将鸭MyoG基因编码区(GenBank No: NW_004676592.1)和启动子区(GenBank No: KX981598)序列进行拼接,获得鸭MyoG基因5′侧翼区序列,将其与鸡MyoG基因(GenBank No: NW_020109686.1)和本研究获得的鹅MyoG基因起始密码子前1 114 bp进行多重比对,发现鹅和鸭同源性是88%,鹅和鸡的同源性仅为59.1%,说明MyoG基因启动子调控机制在鸭和鹅相对保守。
结合Signal Scan和JASPAR预测鹅MyoG基因启动子转录因子结合位点,比较发现,鸭和鹅MyoG基因启动子区存在多个保守的潜在转录因子结合位点,包括HNF4、E2、USF和c-Rel等,且主要集中在TSS和起始密码子ATG之间区域(图 1)。
|
星号表示转录起始位点;黑色箭头表示潜在转录因子结合位点和方向 The asterisk indicates the transcription start site; The black arrows indicate the binding site and direction of potential transcription factors 图 1 鸭和鹅MyoG基因启动子序列比较分析 Fig. 1 Aligment of MyoG gene promoter sequence between goose and duck |
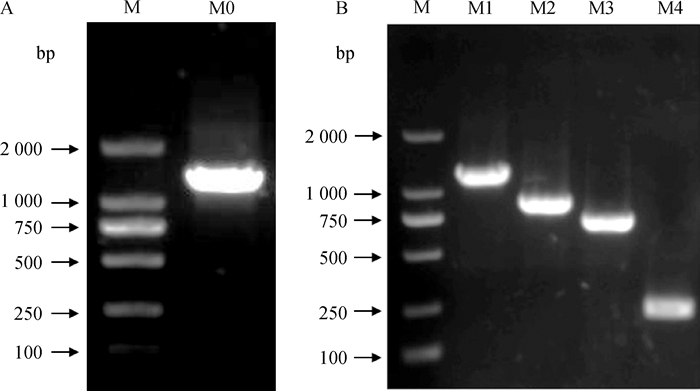
凝胶电泳结果显示,鹅MyoG基因5′侧翼区扩增序列M0(1 245 bp)符合预期长度(图 2A),系列缺失启动子片段M1(1 150 bp)、M2(810 bp)、M3(660 bp)和M4(190 bp)也符合预期长度(图 2B)。
|
A. 鹅MyoG基因5′侧翼区;B. 鹅MyoG基因系列缺失片段。M. DNA相对分子质量标准;M0. 1 245 bp;M1. 1 150 bp;M2. 810 bp;M3. 660 bp;M4. 190 bp A. PCR analysis of goose MyoG 5′-flanking region; B. Serial deletion fragments of goose MyoG promoter. M. DL2000 marker; M0. 1 245 bp; M1.1 150 bp; M2. 810 bp; M3. 660 bp; M4. 190 bp 图 2 鹅MyoG基因启动子克隆 Fig. 2 Cloning of goose MyoG gene promoter |
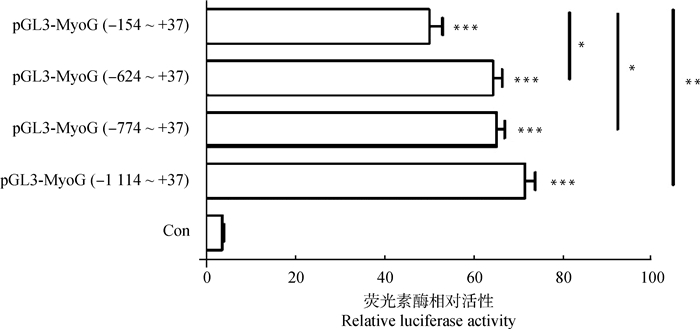
将构建的pGL3-MyoG(-1 114~+37)、pGL3-MyoG(-774~+37)、pGL3-MyoG(-624~+37)和pGL3-MyoG(-154~+37)启动子报告载体及对照质粒pGL3-Basic分别转染至C2C12细胞,双荧光素酶报告基因检测结果表明,不同长度MyoG启动子在C2C12细胞均具有活性,其中pGL3-MyoG(-1 114 ~+37)、pGL3-MyoG(-774~ +37)、pGL3-MyoG(-624~+37)相互之间差异不显著。pGL3-MyoG(-154~+37)的荧光素酶相对活性最低,显著低于pGL3-MyoG(-774~+37)和pGL3-MyoG(-624~+37)活性(P<0.05),同时极显著低于pGL3-MyoG(-1 114~+37)活性(P<0.01)。因此推测,-624~+37 bp是鹅MyoG基因核心启动子区,在-624~-154 bp区域存在关键顺式调控元件(图 3)。
|
图 3 鹅MyoG基因启动子不同片段在细胞系内的相对荧光素酶活性 Fig. 3 Relative luciferase activities of different segments of goose MyoG promoter in C2C12 cells |
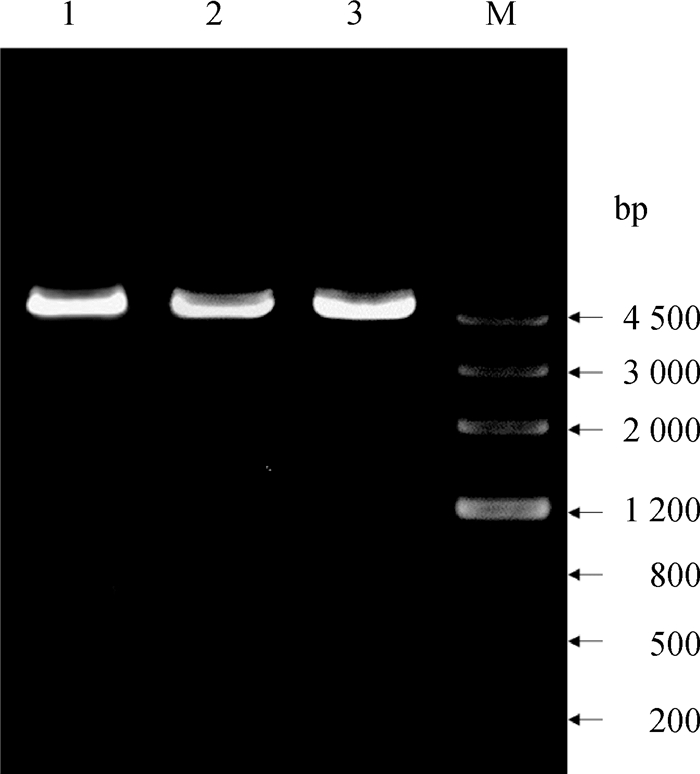
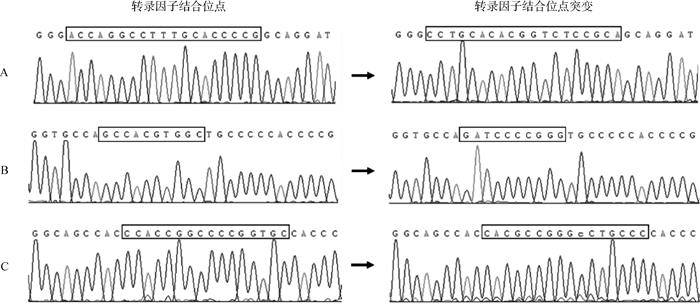
以pGL3-MyoG(-624~+37)为模板,以表 2所示引物对核心启动子区潜在转录因子结合位点(HNF4、USF和E2)进行点突变扩增,扩增产物加T4 DNA连接酶连接,转化大肠杆菌,挑取单克隆提质粒,进行双酶切(NheⅠ和Bgl Ⅱ)验证,同时对质粒进行测序验证(图 4),结果表明,成功构建了pGL3-HNF4-MyoG、pGL3-USF-MyoG和pGL3-E2-MyoG共3个突变位点报告基因载体(图 5)。
|
M. DNA相对分子质量标准;1. pGL3-HNF4-MyoG双酶切产物;2. pGL3-USF-MyoG双酶切产物;3. pGL3-E2-MyoG双酶切产物 M. DNA marker; 1. Double digestion products of pGL3-HNF4-MyoG vector; 2. Double digestion products of pGL3-USF-MyoG vector; 3. Double digestion products of pGL3-E2-MyoG vector 图 4 鹅MyoG突变载体双酶切产物凝胶电泳图 Fig. 4 Agarose gel electrophoresis of double digestion products of MyoG mutant vectors |
|
A. HNF4结合位点;B. USF结合位点;C. E2结合位点。方框内表示转录因子结合位点突变对比 A. HNF4 binding site; B. USF binding site; C. E2 binding site. Bases in box denote mutant transcription factor binding sites 图 5 转录因子结合位点突变载体测序 Fig. 5 Sequencing analysis of mutant transcription factor binding sites vectors |
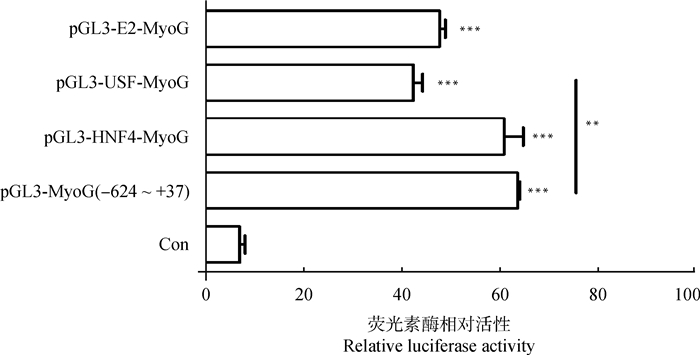
将构建好的pGL3-HNF4-MyoG、pGL3-USF-MyoG和pGL3-E2-MyoG突变位点报告基因载体和对照载体pGL3-MyoG(-624~+37 bp)分别转染至C2C12细胞,双荧光素酶报告基因检测结果表明,突变位点报告基因在C2C12细胞均有活性,3个突变位点报告基因的荧光素酶相对活性均比pGL3-MyoG(-624~+37 bp)降低,其中pGL3-USF-MyoG的活性极显著下降(P<0.01,图 6)。因此推测,USF是调控鹅MyoG基因转录的核心顺式调控元件。
|
图 6 定点突变转录因子在细胞系内的相对荧光素酶活性 Fig. 6 Relative luciferase activities of site-directed mutant transcription factors |
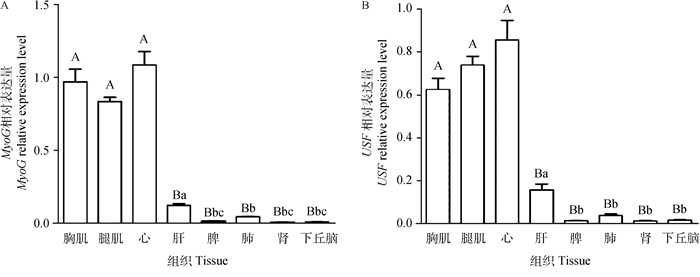
利用qRT-PCR对鹅胸肌、腿肌、心、肝、脾、肺、肾和下丘脑8种组织中MyoG、USF和内参基因β-actin片段进行扩增,检测MyoG和USF的组织相对表达量,结果见图 7。MyoG在胸肌、腿肌和心的表达量最高,极显著高于其它组织(P<0.01),USF的组织表达模式和MyoG相似,二者在肌肉组织内同时表达,且相对表达量较高。结合转录因子突变试验结果推测,USF和MyoG在鹅肌肉发育过程中共同发挥重要作用。
|
A. MyoG基因mRNA相对表达量;B. USF基因mRNA相对表达量。不同小写字母表示差异显著(P<0.05),不同大写字母差异极显著(P<0.01) A. MyoG mRNA relative expression level; B. USF mRNA relative expression level. Different superscript lowercase and captital letters indicate significant difference (P < 0.05) and extremely significant difference (P < 0.01), respectively 图 7 鹅MyoG和USF基因mRNA在不同组织的相对表达量 Fig. 7 The expression level of MyoG and USF mRNA in various tissues of goose |
MyoG在哺乳动物骨骼肌发育的整个过程中都起着十分重要的调控作用,国内外学者对其调控机理的研究获得了一系列重要的成果。畜禽个体肌肉的形成源自于胚胎时期成肌细胞的增殖与分化,肌纤维数量在胚胎期就基本固定下来,出生(壳)后不再增加,出生(壳)后肌肉产量的增加主要是通过肌纤维的增粗和变长实现[15]。由于家禽生理特点的差异,家禽胚胎期,尤其是胚胎后期肌肉发育规律与哺乳动物有显著不同,表现为肌纤维直径的缩小和胸肌产量的降低[16],与此同时,胸肌中卫星细胞的增殖活性和数量随着胚龄增加而减少[15],直到出壳当天,卫星细胞的增殖能力才显著恢复[17],这与MyoG基因在鹅胚孵化期的表达规律相似[18],因此推测,MyoG在家禽和哺乳动物的调控机制存在差异。本研究对鹅MyoG基因启动子区序列进行了克隆与分析,筛选出核心启动子区并初步明确了核心转录调控因子,为下一步分析其在胚胎期作用机制,阐明鹅肌肉发育的遗传机制奠定了基础。
本研究获得了鹅MyoG基因5′侧翼区序列,生物信息学分析发现,该区域包含MyoG基因启动子区,包含TATA-box和GC-box等典型的真核生物转录调控元件,但缺失CAAT-box,这与鸭MyoG启动子存在CAAT-box(位于-2 514~-2 508 bp)的研究结果不同[12],推测其可能存在于已知序列前端,还需要进一步试验验证。而且,CAAT-box也并不总是存在于一个基因的启动子区域,猪UCP2和SIM1基因启动子区存在相似的情况[19-20]。
为了研究MyoG基因启动子活性及其转录调控机制,本试验采用扩增启动子缺失片段的方法,通过构建双荧光素酶重组载体,转染至C2C12细胞,利用双荧光素酶检测系统对4个不同长度启动子片段进行活性检测。结果显示,-624~+37 bp区域是鹅MyoG基因核心启动子区,-624~-154 bp存在关键顺式调控元件。采用JASPAR在线程序MatInspector分析-624~-154 bp区域潜在转录因子结合位点,筛选到了HNF4(-521~-503 bp)、USF(-379~-370 bp)和E2(-296~-281 bp)的结合位点。通过采用点突变技术对3个转录因子结合位点进行敲除,构建双荧光素酶重组载体,转染至C2C12细胞,利用双荧光素酶检测系统对3个突变位点报告基因载体进行活性检测。结果显示,USF转录因子突变载体pGL3-USF-MyoG的活性极显著下降(P<0.01),其它两处元件(HNF4和E2)突变对启动子活性影响不显著,因此推测,USF是鹅MyoG基因核心转录调控因子。本课题组在C2C12细胞系内对USF基因进行敲降的研究中发现,USF表达量被显著抑制(P<0.05)后,MyoG基因表达量极显著降低(P<0.01),说明在C2C12细胞中USF可以调控MyoG的表达,与本研究得到的结果相似(数据未发表)。
上游刺激因子(upstream stimulatory factor, USF)属于碱性螺旋-环-螺旋亮氨酸锌指(bHLH-LZ)转录因子家族,USF蛋白结构域在进化上保守性较高[21],通常以同源或异源蛋白二聚体的形式特异结合靶基因DNA启动子区E-box元件,从而发挥转录调控作用[22-23],而E-box元件已被证明是一个潜在的顺式调控位点[24]。研究发现,大多数的骨骼肌相关基因控制区均含有一个或多个E-box元件[25],USF是在大鼠骨骼肌、心肌等肌肉组织中大量表达的调控因子[26],其可能广泛参与肌肉组织的生长调控。对猪USF基因的研究表明,其多态性与眼肌面积、瘦肉率、屠宰率等胴体性状存在显著相关[27]。
理论上,功能相关的基因通常有相似的表达模式,基因表达相关性分析已经成为发掘基因间相互作用的一种有效手段[28],转录因子对靶基因的调控具有组织特异性[29],某些转录因子与靶基因倾向于共表达[30],对牛肌肉发育相关基因与转录因子调控网络研究发现,MyoG基因与候选转录因子具有稳定的共表达模式[31]。本研究组织表达谱分析发现,USF和MyoG在鹅体内存在共表达趋势,尤其在胸肌、腿肌、心脏表达量较高,且极显著高于其它组织(P<0.01),结果进一步说明,USF在调控鹅肌肉发育中具有潜在的重要功能。
MyoG所在的MRFs家族同样具有bHLH结构域,结合本研究结果,推测USF首先与MyoG形成二聚体或多聚体,然后碱性区域与MyoG启动子区E-box序列结合,从而激活MyoG基因转录。USF是动物体内极其重要的转录因子,和细胞生长、糖脂代谢、排卵等生理过程紧密相关[23, 32],但已有的研究结果尚不能完全说明其所代表的生理意义,本研究首次揭示了USF通过调控MyoG基因转录参与调控肌肉发育过程。
来源于不同物种的同源基因分子结构不尽相同,其基因功能、转录调控机制等都因基因结构的差异而有所不同。鸡MyoG基因131 bp核心启动子区内包含E-box和MEF-2两个元件,二者协同激活MyoG基因启动子活性,并且介导E蛋白E2-5和蛋白激酶C(protein kinase C, PKC)调控MyoG转录活性[33]。民猪MyoG基因启动子核心调控元件是CdxA和MZF1转录因子[3],这与本研究结果不同。结合鸡、鸭MyoG基因启动子研究结果[12],进一步说明MyoG基因转录调控机制非常复杂,下一步可以通过染色质免疫共沉淀(EMSA)和凝胶阻滞分析试验等方法验证USF调控鹅MyoG转录活性的功能,进一步研究其作用机制。
4 结论本试验成功克隆得到鹅MyoG基因启动子区序列,序列比对表明,鹅和鸭MyoG基因启动子区同源性较高。启动子活性检测发现,-624~+37 bp区域是鹅MyoG基因核心启动子区,-624~-154 bp区域存在关键顺式调控元件。对核心区转录因子进行定点突变,构建转录因子突变表达载体后测定活性,结果表明,USF是启动子核心顺式调控因子。本研究结果为进一步开展鹅肌肉发育调控提供了理论基础。
| [1] | FONG A P, TAPSCOTT S J. Skeletal muscle programming and re-programming[J]. Curr Opin Genet Dev, 2013, 23(5): 568–573. DOI: 10.1016/j.gde.2013.05.002 |
| [2] | BERGSTROM D A, TAPSCOTT S J. Molecular distinction between specification and differentiation in the myogenic basic helix-loop-helix transcription factor family[J]. Mol Cell Biol, 2001, 21(7): 2404–2412. DOI: 10.1128/MCB.21.7.2404-2412.2001 |
| [3] |
张晓卉. 民猪MyoG基因的克隆、表达与启动子核心区的定位[D]. 哈尔滨: 东北农业大学, 2012.
ZHANG X H. The cloning, expression and the core area promoter positioning of Min pig MyoG gene[D]. Harbin: Northeast Agricultural University, 2012. (in Chinese) |
| [4] |
王琼, 刘益平, 蒋小松, 等. MyoG基因多态性与优质肉鸡屠宰性状和肉质性状的相关性分析[J]. 遗传, 2007, 29(9): 1089–1096.
WANG Q, LIU Y P, JIANG X S, et al. Correlation analysis of relationships between polymorphisms of high quality chicken myogenin gene and slaughter and meat quality traits[J]. Hereditas (Beijing), 2007, 29(9): 1089–1096. DOI: 10.3321/j.issn:0253-9772.2007.09.011 (in Chinese) |
| [5] |
赵忠海, 李辉, 易恒杰, 等. MyoG基因多态性对鸭屠宰性状的影响[J]. 基因组学与应用生物学, 2015, 34(6): 1189–1194.
ZHAO Z H, LI H, YI H J, et al. Effects of MyoG gene polymorphism on slaughter traits in ducks[J]. Genomics and Applied Biology, 2015, 34(6): 1189–1194. (in Chinese) |
| [6] | WILLOUGHBY D S, NELSON M J. Myosin heavy-chain mRNA expression after a single session of heavy-resistance exercise[J]. Med Sci Sport Exer, 2002, 34(8): 1262–1269. DOI: 10.1097/00005768-200208000-00006 |
| [7] | POWELL D J, MCFARLAND D C, COWIESON A J, et al. The effect of nutritional status on myogenic gene expression of satellite cells derived from different muscle types[J]. Poult Sci, 2014, 93(9): 2278–2288. DOI: 10.3382/ps.2013-03810 |
| [8] | COHEN T J, WADDELL D S, BARRIENTOS T, et al. The histone deacetylase HDAC4 connects neural activity to muscle transcriptional reprogramming[J]. Biol Chem, 2007, 282(46): 33752–33759. DOI: 10.1074/jbc.M706268200 |
| [9] |
刘晓伟. Myogenin在周期性牵张与Ach刺激的C2C12细胞中表达的实验研究[D]. 天津: 南开大学, 2010.
LIU X W. The effects of periodic tesion and Ach on the expression of myogenin in C2C12 myoblast[D]. Tianjin: Nankai University, 2010. (in Chinese) |
| [10] | SHIMA A, PHAM J, BLANCO E, et al. IGF-Ⅰ and vitamin C promote myogenic differentiation of mouse and human skeletal muscle cells at low temperatures[J]. Exp Cell Res, 2011, 317(3): 356–366. DOI: 10.1016/j.yexcr.2010.11.001 |
| [11] | MCFARLAND D C, PESALL J E, COY C S, et al. Effects of 17β-estradiol on turkey myogenic satellite cell proliferation, differentiation, and expression of glypican-1, MyoD and myogenin[J]. Comp Biochem Phys A Mol Integr Phys, 2013, 164(4): 565–571. DOI: 10.1016/j.cbpa.2013.01.001 |
| [12] |
吴乾锋, 刘贺贺, 张涛, 等. 鸭MyoG基因启动子的生物信息学分析及其甲基化频率对基因表达的影响[J]. 湖南农业大学学报: 自然科学版, 2017, 43(2): 187–194.
WU Q F, LIU H H, ZHANG T, et al. Bioinformatics analysis on cloned promoter MyoG in duck, and the influence of its methylation levels on gene transcription[J]. Journal of Hunan Agricultural University: Natural Sciences, 2017, 43(2): 187–194. (in Chinese) |
| [13] |
王秋华, 曹允考, 李树峰, 等. 牛MyoG基因启动子的克隆及功能的初步分析[J]. 畜牧兽医学报, 2012, 43(1): 37–43.
WANG Q H, CAO Y K, LI S F, et al. Cloning and preliminary functional analysis of bovine MyoG promoter[J]. Acta Veterinaria et Zootechnica Sinica, 2012, 43(1): 37–43. (in Chinese) |
| [14] |
刘铮铸, 巩元芳, 张文香, 等. 山羊MyoG基因启动子区序列分析与结构预测[J]. 畜牧兽医学报, 2012, 43(1): 29–36.
LIU Z Z, GONG Y F, ZHANG W X, et al. Sequence analysis and structure prediction of the promoter region of MyoG gene in goats[J]. Acta Veterinaria et Zootechnica Sinica, 2012, 43(1): 29–36. (in Chinese) |
| [15] |
刘贺贺. IGF-1对鸭胚肌肉发育影响及其分子机理研究[D]. 雅安: 四川农业大学, 2012.
LIU H H. Functional analysis of IGF-1 on duck embryonic muscle development and research about its molecular mechanism[D]. Ya'an: Sichuan Agricultural University, 2012. (in Chinese) |
| [16] |
陈伟. 孵化后期外源二糖和谷氨酰胺调控肉鸭骨骼肌蛋白质代谢机制研究[D]. 武汉: 华中农业大学, 2010.
CHEN W. Study on the regulatory role of administrating exogenous disaccharide and glutamine to late-term duck embryos in affecting the protein metabolism in skeletal muscle[D]. Wuhan: Huazhong Agricultural University, 2010. (in Chinese) |
| [17] | MOORE D T, FERKET P R, MOZDZIAK P E. Muscle development in the late embryonic and early post-hatch poult[J]. Int J Poult Sci, 2005, 4(3): 138–142. DOI: 10.3923/ijps.2005.138.142 |
| [18] |
腾战伟, 李德生, 徐晗, 等. 家禽胚期能量储备和肌肉发育特点及其调控[J]. 中国畜牧杂志, 2016, 52(21): 95–99.
TENG Z W, LI D S, XU H, et al. Energy reserves and muscle development of poultry embryos and its manipulation[J]. Chinese Journal of Animal Science, 2016, 52(21): 95–99. DOI: 10.3969/j.issn.0258-7033.2016.21.021 (in Chinese) |
| [19] | YAMADA M, HASHIDA T, SHIBUSAWA N, et al. Genomic organization and promoter function of the mouse uncoupling protein 2 (UCP2) gene[J]. FEBS Lett, 1998, 432(1-2): 65–69. DOI: 10.1016/S0014-5793(98)00835-7 |
| [20] |
陈哲, 雷明明, 施振旦. 猪SIM1基因启动子活性及表达模式分析[J]. 华北农学报, 2015, 30(2): 6–11.
CHEN Z, LEI M M, SHI Z D. Analysis of promoter activity of porcine SIM1 gene and its expression pattern[J]. Acta Agriculturae Boreali-Sinca, 2015, 30(2): 6–11. (in Chinese) |
| [21] | GUO Y X, LI T S, LI Q W, et al. A novel protein upstream stimulatory factor 2 identified in lamprey, Lethenteron reissneri[J]. Dev Genes Evol, 2020, 230(5): 347–357. DOI: 10.1007/s00427-020-00666-5 |
| [22] | GAYRAUD-MOREL B, CHRÉTIEN F, FLAMANT P, et al. A role for the myogenic determination gene Myf5 in adult regenerative myogenesis[J]. Dev Biol, 2007, 312(1): 13–28. DOI: 10.1016/j.ydbio.2007.08.059 |
| [23] |
谭雅文. 上游刺激因子2(USF2)对Smurf1和Smurf2的功能调控研究[D]. 济南: 山东大学, 2013.
TAN Y W. Transcriptional down-regulation of Smurf1/2 ubiquitin ligase by upstream stimulate factor 2 (USF2)[D]. Ji'nan: Shandong University, 2013. (in Chinese) |
| [24] |
李淑锋, 华子春. 人转录因子USF基因在大肠杆菌中的表达、纯化及在FAK启动子中的结合研究[J]. 东南大学学报: 医学版, 2007, 26(4): 267–270.
LI S F, HUA Z C. Expression, purification of human transcription factor USF and its DNA binding analysis in FAK promoter[J]. Journal of Southeast University: Medical Science Edition, 2007, 26(4): 267–270. DOI: 10.3969/j.issn.1671-6264.2007.04.007 (in Chinese) |
| [25] | YIN H D, LI D Y, WANG Y, et al. Myogenic regulatory factor (MRF) expression is affected by exercise in postnatal chicken skeletal muscles[J]. Gene, 2015, 561(2): 292–299. DOI: 10.1016/j.gene.2015.02.044 |
| [26] |
邹东霆, 李冰, 付欣, 等. 转录因子上游刺激因子在大鼠不同组织中的分布[J]. 广州医学院学报, 2010, 38(3): 28–30.
ZOU D T, LI B, FU X, et al. Distribution of transcription factor upstream stimulating factors in different tissues of rats[J]. Academic Journal of Guangzhou Medical College, 2010, 38(3): 28–30. DOI: 10.3969/j.issn.1008-1836.2010.03.008 (in Chinese) |
| [27] |
武华玉. 猪USF1和MBF1基因的克隆、鉴定及多态性分析[D]. 武汉: 华中农业大学, 2007.
WU H Y. Cloning, character and polymorphism of porcine USF1 and MBF1 gene[D]. Wuhan: Huazhong Agricultural University, 2007. (in Chinese) |
| [28] |
刘文苏, 田巍, 周静, 等. 利用RNA转录组数据库挖掘肿瘤细胞EZH2基因候选转录因子[J]. 中国生物化学与分子生物学报, 2020, 36(6): 665–673.
LIU W S, TIAN W, ZHOU J, et al. The RNA transcriptome database was used to mine the candidate transcription factors of EZH2 gene in tumor cells[J]. Chinese Journal of Biochemistry and Molecular Biology, 2020, 36(6): 665–673. (in Chinese) |
| [29] | CHEN K, RAJEWSKY N. The evolution of gene regulation by transcription factors and microRNAs[J]. Nat Rev Genet, 2007, 8(2): 93–103. |
| [30] |
陈靖祺, 柳靓婧, 田卫东. TF-coEx: 一种基于基因共表达网络的转录因子功能预测新方法[J]. 复旦学报: 自然科学版, 2012, 51(6): 803–812.
CHEN J Q, LIU J J, TIAN W D. TF-coEx: Transcription factor function prediction based on gene co-expression network[J]. Journal of Fudan University: Natural Science, 2012, 51(6): 803–812. (in Chinese) |
| [31] | GU Q, NAGARAJ S H, HUDSON N J, et al. Genome-wide patterns of promoter sharing and co-expression in bovine skeletal muscle[J]. BMC Genomics, 2011, 12(1): 23. DOI: 10.1186/1471-2164-12-23 |
| [32] | TAN Y W, CHEN Y J, DU M G, et al. USF2 inhibits the transcriptional activity of Smurf1 and Smurf2 to promote breast cancer tumorigenesis[J]. Cell Signal, 2019, 53: 49–58. DOI: 10.1016/j.cellsig.2018.09.013 |
| [33] | MALIK C, HUANG C F, SCHMIDT J. The role of the CANNTG promoter element (E-box) and the myocyter-enhancer-binding-factor-2 (MEF-2) site in the transcriptional regulation of the chick myogenin gene[J]. Eur J Biochem, 1995, 230(1): 88–96. DOI: 10.1111/j.1432-1033.1995.0088i.x |



