2. 广东省农业科学院农业质量标准与监测技术研究所, 广州 510640;
3. 贵州大学动物科学学院, 贵阳 550025
2. Institute of Quality Standard and Monitoring Technology for Agro-products of Guangdong Academy of Agricultural Sciences, Guangzhou 510640, China;
3. College of Animal Science, Guizhou University, Guiyang 550025, China
多杀性巴氏杆菌(Pasteurella multocida)是革兰阴性菌,能在世界范围内引起多种动物疾病,如禽霍乱、猪萎缩性鼻炎、兔败血症和牛肺炎等[1-3],在某些情况下,还可通过动物咬伤或抓伤引起人类感染[4-5]。目前,由于缺乏有效的多血清型疫苗,抗生素仍是治疗巴氏杆菌感染的首选方法[6],但随着抗生素的不合理使用,多国出现了对包括头孢噻呋、头孢噻肟、头孢曲松等第3代头孢菌素类抗生素在内的多种抗菌药物耐药现象,这给抗菌药物的使用带来了严峻的挑战[7-9]。
毒力因子是细菌入侵并在宿主体内增殖的关键因子,同时还具有较强的免疫原性[10]。毒力基因的丰度与细菌的致病性相关,目前已被证实与P. multocida致病性密切相关的毒力基因除荚膜(capsule)和脂多糖(lipopolysaccharide,LPS)相关基因外,还包括菌毛和黏附素(fimbriae and adhesins)、毒素(Pasteurella multocida toxin)、铁调节蛋白和铁获取蛋白(iron regulation and iron acquisition proteins)、唾液酸代谢相关蛋白(sialic acid metabolism)、透明质酸酶(hyaluronidases)及外膜蛋白(outer membrane proteins)的毒力基因[3, 11]。其中,荚膜可增强P. multocida的侵袭力和繁殖能力,由荚膜编码基因簇编码,该区域分为3个功能区(荚膜多糖输出区、荚膜合成区和荚膜多糖磷脂替换区),按照结构可分为5种不同的荚膜基因型(A、B、D、E、F)[12];按照每个区域的基因数目,可分成两大类:A、D、F型一类,10个编码基因紧密相邻;B和E型为另一类,15个编码基因,在荚膜多糖输出区分散排列[13]。LPS有黏附、侵入和抗血清的作用,其致病作用跟LPS的完整性相关[14-15]。LPS由类脂A和核心寡糖组成,根据核心寡糖外核基因在不同菌株间的差异可分为8个LPS基因型,即L1~L8[16]。此外,菌毛和黏附素在P. multocida感染致病过程中扮演着十分关键的作用,能黏附并定植于宿主细胞或组织,如IV型菌毛亚单位蛋白(由ptfA基因编码)、黏附相关蛋白(包括FimA、ComE1、FhaB、Flp1、Flp2、Hsf_1、Hsf_2、TadG、TadF、TadF、RcpB等)、菌毛低分子蛋白(fimbrial low-molecular-weight protein, Flp)(由Flp操纵子基因编码)等[11]。在P.multocida流行病学研究中,基于不同毒力基因谱检测的毒力基因分型已发展成为一种有用的基因分型方法,因此对于鸭源P. multocida毒力基因多样性的检测显得尤为重要[17]。
P. multocida分离株的感染表现出宿主偏好,如A:L1:ST129、D:L6:ST50、A:L3:ST9分别在禽类、猪、兔的感染中占优势[3]。应用荚膜基因型、LPS基因型和多位点序列分型(multilocus sequence typing, MLST)相结合的基因分型方法将有助于临床分离株的鉴定,为研究P. multocida的全球流行性和分子进化提供依据[17]。
本研究从贵州省出现的疑似禽霍乱的56日龄的病(死)花边鸭组织中分离得到5株P. multocida,对分离株进行分子生物学鉴定,同时采用细菌全基因组测序技术对分离得到的P. multocida强毒株PmCW1进行测序,并对其耐药基因及毒力基因进行分析,探究分离株的生物学、毒力及耐药特征,对鸭霍乱的临床用药及疫苗研发具有一定指导意义。
1 材料与方法 1.1 主要仪器与试剂胰蛋白胨大豆琼脂培养基(TSA)、胰蛋白胨大豆肉汤(TSB)等购自青岛海博生物生物技术有限公司;新生牛血清购自北京鼎国昌盛生物技术有限责任公司;无菌脱纤维绵羊血购自北京索莱宝科技有限公司;细菌基因组DNA提取试剂盒购自天根生化科技有限公司;DL2000 DNA Marker、Premix Taq (EX Taq version 2.0 plus dye)等购自宝生物(大连)工程有限公司;琼脂糖,购自Oxoid公司。普通梯度PCR仪、核酸电泳仪、凝胶成像系统,均购自Bio-Rad公司;超净工作台,购自苏州净化设备有限公司。
1.2 菌株的分离与鉴定2020年6月,贵州省三穗县某鸭场56日龄花边鸭群3 000余羽,3 d内发病死亡1 185羽,疑似感染禽霍乱,从典型的临床症状和剖检症状的病死鸭中,随机无菌采集5只病(死)鸭的脑和肝组织分别进行细菌的分离,观察菌株在含有5%新生牛血清的TSA平板及鲜血琼脂平板上的生长情况并进行菌株的纯化。采用kmt1基因特异性PCR方法进行分子生物学鉴定,测序并Blast分析,以确认其多杀性。引物序列:kmt1-F(5′-ACCGACAAGCCCACTCACAACA-3′);kmt1-R(5′-ATCATCCTAACCGCCTGAAAGC-3′)。
根据Townsend等[12]报道的荚膜基因型分型方法和Harper等[16]建立的脂多糖基因型分型方法,送生工生物工程(上海)股份有限公司合成PCR引物。PCR产物经1%琼脂糖进行凝胶电泳分析。
采用文献[12-13]报道的肠杆菌科细菌基因间重复序列聚合酶链式反应(enterobacterial repetitive intergenic consensus sequence,ERIC)-PCR方法鉴定5株分离株的同源性。
1.3 多位点序列分型法(MLST)本研究采用多宿主来源P. multocida MLST数据库(Multiple host MLST database)对5株分离株进行MLST分型。根据P. multocida MLST官网(http://pubmLst.org/pmultocida_multihost)上推荐的PCR方法分别扩增7个管家基因(adk、aroA、deoD、gdhA、g6pd、mdh、pgi)。PCR产物利用1%琼脂糖进行凝胶电泳分析,条带大小正确的扩增产物送往生工生物工程(上海)股份有限公司利用对应的测序引物进行测序,并将序列提交至Multiple host MLST database进行MLST基因分型。
1.4 动物回归试验健康未接种禽巴氏杆菌疫苗的15日龄三穗鸭24只,随机分成6组,每组4只,公母各半。5组试验组分别肌肉接种P. multocida分离菌悬液, 0.2 mL·只-1,浓度依次为1.1×102、1.1×103、1.1×104、1.1×105、1.1×106 cfu·mL-1,对照组注射等量生理盐水,逐日观察动物发病及死亡情况,及时对死亡鸭进行剖检、观察, 并进行病原菌的分离鉴定,同时采用ERIC-PCR方法鉴定攻毒前、后分离株的同源性。
1.5 药物敏感性试验参照美国临床实验室标准协会(CLSI)标准[18],采用微量肉汤二倍稀释法测定18种抗菌药物的MIC值,被测试的抗菌药物包括阿莫西林、氨苄西林、头孢噻肟、头孢曲松、头孢噻呋、头孢他啶、黏菌素、链霉素、庆大霉素、阿米卡星、环丙沙星、左氧氟沙星、恩诺沙星、林可霉素、红霉素、阿奇霉素、氟苯尼考和多西环素(北京索莱宝科技有限公司,中国)。以大肠埃希菌ATCCⓇ25922和金黄色葡萄球菌ATCCⓇ29213作为质控菌。
1.6 全基因组测序与耐药性、毒力性分析用细菌基因组DNA提取试剂盒提取PmCW1基因组DNA,提取的基因组DNA样品由北京诺禾致源基因科技有限公司进行全基因组测序。基因组测序在Nanopore ProMethation和Illumina NovaSeq PE150上进行;使用Unicycler软件[19](https://github.com/rrwick/Unicycler)用于基因组组装。使用GeneMarkS(Version 4.17)[20] (http://topaz.gatech.edu/GeneMark/)软件对新测序的基因组进行编码基因预测。使用Comprehensive Antibiotic Research Database(CARD)数据库(https://card.mcmaster.ca/)提供的Resistance Gene Identifier(RGI)软件将PmCW1的氨基酸序列与CARD数据库进行比对(RGI内置blastp,默认e value≤1×10-30),根据RGI的比对结果,统计注释到数据库的抗性基因信息。对注释得到的PmCW1耐药基因与其耐药表型进行比较分析,分析其耐药表型和耐药性的关系。使用Diamond软件[21],把目标物种的氨基酸序列与Virulence Factors of Pathogenic Bacteria(VFDB)数据库(http://www.mgc.ac.cn/VFs/)进行比对,把目标物种的基因和其相对应的毒力因子功能注释信息结合起来,得到注释结果。使用Easyfig[22](http://mjsull.github.io/Easyfig/)分析PmCW1毒力相关的脂多糖核心寡糖外核、荚膜编码基因簇及Flp操纵子基因簇与HN141014(LUCX00000000)、P1059(CM001581)、Pm70(AE004439)、ATCC1702(LUDD00000000)及HN06(CP003313)之间基因结构与同源性的差异。
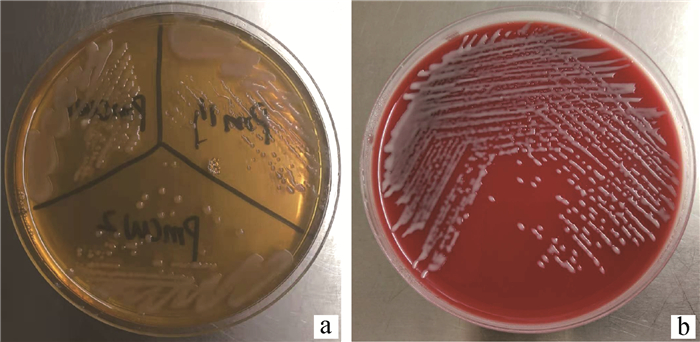
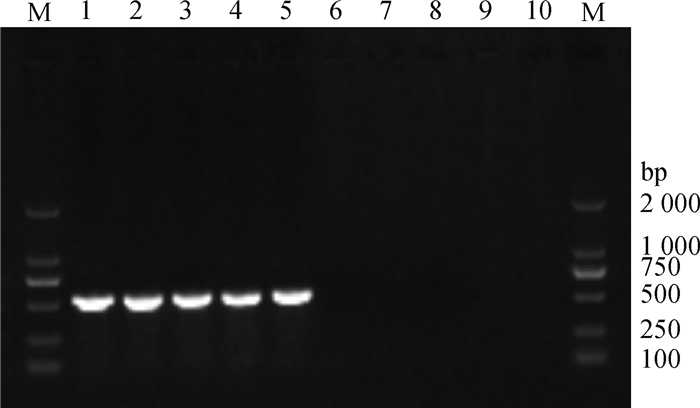
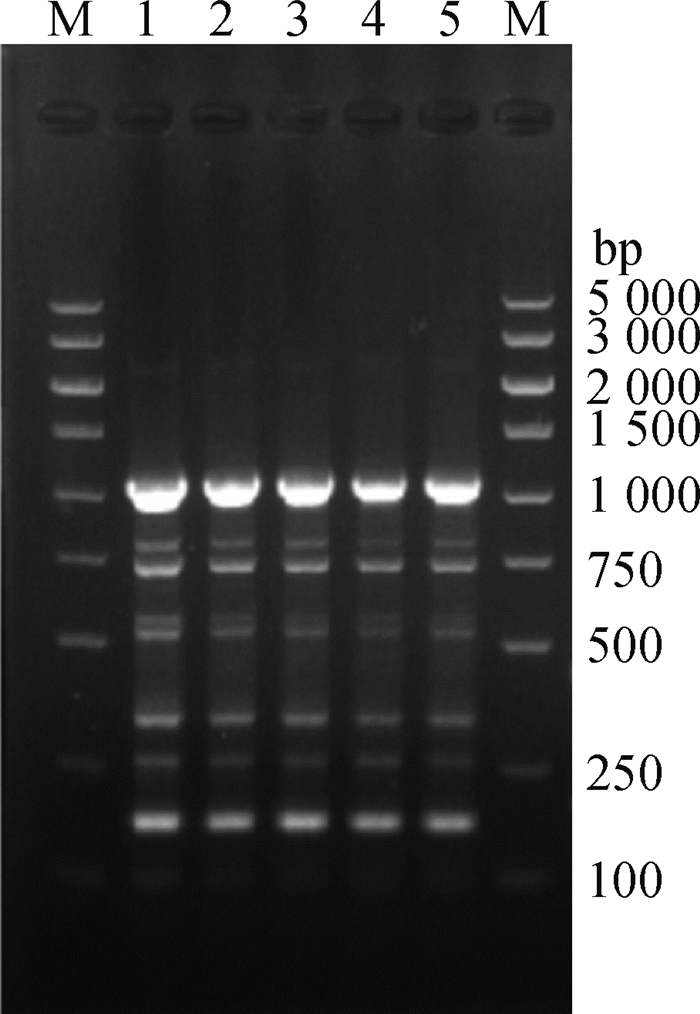
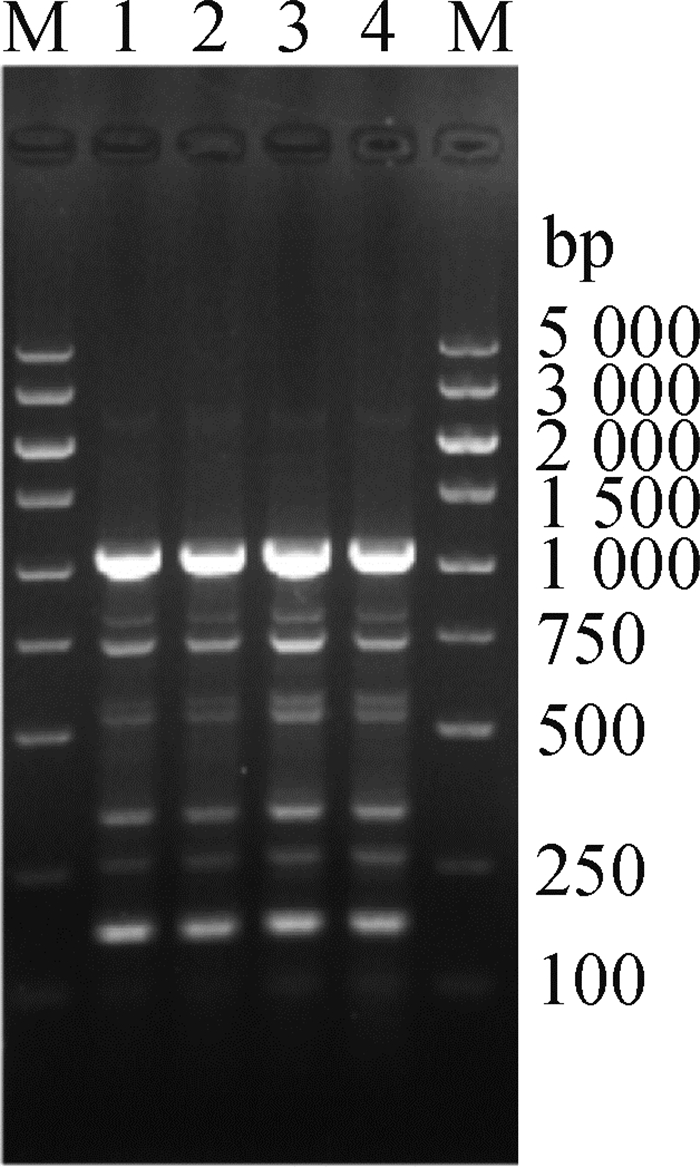
2 结果 2.1 菌株的分离鉴定2020年6月,从5只病(死)鸭的脑和肝中均分离出较纯的巴氏杆菌,同一个体不同部位分离出来的菌株算作1株,共分离得到5株巴氏杆菌。在含5%新生牛血清的TSA平板上生长良好,在鲜血平板上长成水滴状小菌落,不溶血(图 1)。kmt1-PCR扩增结果与预期相符(图 2),测序结果与NCBI公布的kmt1基因序列比对,相似性达99%以上,确定5株均为多杀性巴氏杆菌。从ERIC-PCR的电泳结果(图 3)可以看出,本次分离得到的5株菌具有相同的扩增片段,表明菌株间同源性高,克隆传播的可能性大。
|
a.含5%新生牛血清的TSA平板;b.鲜血平板 a.TSA plate containing 5% newborn bovine serum; b.Blood plate 图 1 鸭源巴氏杆菌在不同培养基上的菌落形态 Fig. 1 Colony morphology of Pasteurella from ducks on different media |
|
M. DL2000 DNA相对分子质量标准;1~5.PmCW1~5;6~10.阴性对照 M. DL2000 DNA marker; 1-5. PmCW1-5; 6-10. Negative control 图 2 kmt1-PCR鉴定结果 Fig. 2 Results of kmt1-PCR identification |
|
M.DL5000 DNA相对分子质量标准;1~5.PmCW1~5 M.DL5000 DNA marker; 1-5.PmCW1-5 respectively 图 3 5株巴氏杆菌分离株的ERIC-PCR结果 Fig. 3 ERIC-PCR results of five P. multocida isolates |
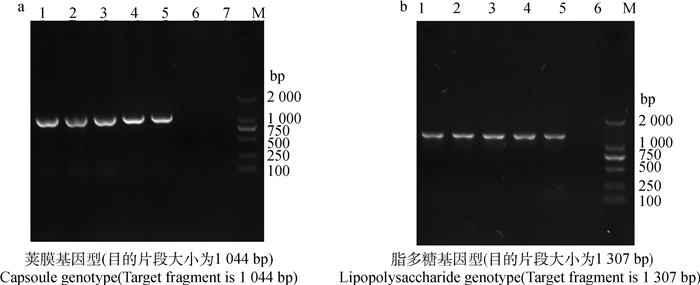
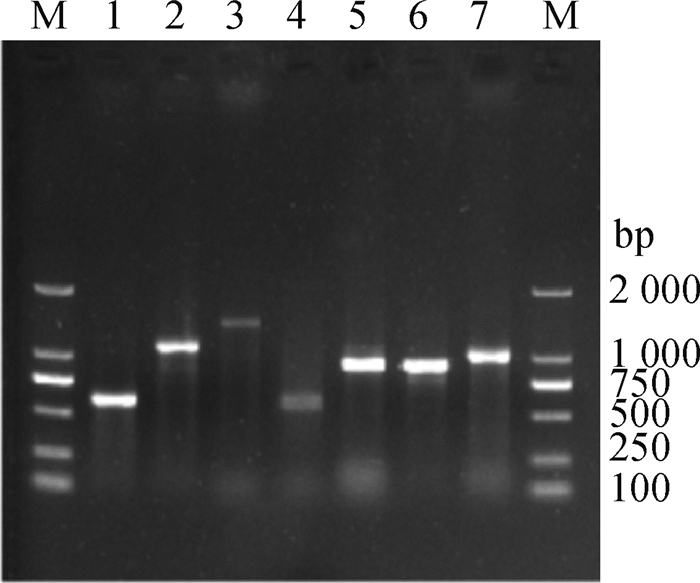
利用PCR方法对5株菌进行血清基因型鉴定,结果显示均为A:L1型(图 4)。对5株菌分别进行7个管家基因的扩增和测序,测序结果提交至http://pubmLst.org/pmultocida_multihost,结果显示, 5株均为ST128型,图 5为MLST所用7个管家基因PCR阳性图。
|
M.DL2000 DNA相对分子质量标准;1~5.PmCW1-5;6~7(图a)/6(图b).阴性对照 M.DL2000 DNA marker; 1-5.PmCW1-5 respectively; 6-7(Fig.a)/6(Fig.b).Negative control 图 4 荚膜基因型(a)和脂多糖基因型(b)PCR结果 Fig. 4 PCR for Capsules genotype (a) and lipopolysaccharide genotype(b) |
|
M.DL2000 DNA相对分子质量标准;1~7.deoD、gdhA、g6pd、adk、pgi、mdh、aroA基因阳性结果 M.DL2000 DNA marker; 1-7 are positive results of deoD, gdhA, g6pd, adk, pgi, mdh, aroA gene, respectively 图 5 MLST所用7个管家基因PCR阳性图 Fig. 5 Positive PCR results of seven housekeeping genes used in MLST |
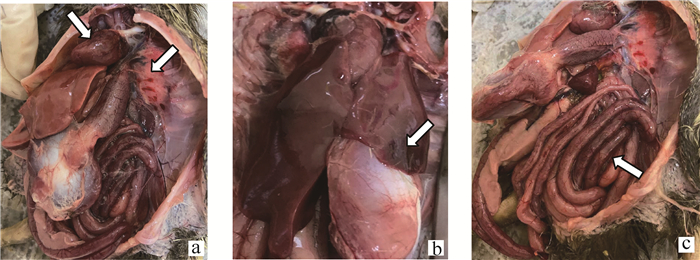
P. multocida PmCW1动物回归试验结果显示,1.1×104、1.1×105、1.1×106这三个试验组中鸭12 h内全部死亡,24 h内5组试验组鸭全部死亡,对照组正常。死亡鸭剖检观察到全身性出血(图 6),分离鉴定结果显示在5%新生牛血清的TSA平板均有较纯的菌落生长,经菌落PCR鉴定均为P. multocida。同时,ERIC-PCR鉴定结果显示与攻毒所用的PmCW1具有相同的扩增片段,可以判定为同一株P. multocida(图 7)。
|
a.心、肺出血;b.肝出血、坏死;c.肠道弥漫性出血 a. Heart and lung bleeding; b. Liver hemorrhage and necrosis; c. Diffuse intestinal bleeding 图 6 攻毒致死鸭的剖检图 Fig. 6 Necropsy diagram of ducks killed by challenge poison |
|
M.DL5000 DNA相对分子质量标准;1.PmCW1;2~4.攻毒后分离株 ERIC-PCR results of isolates before and after challenge 图 7 攻毒前后分离株的ERIC-PCR结果 Fig. 7 ERIC-PCR results of isolates before and after challenge |
由表 1可见,在对5株菌进行18种抗菌药物敏感性检测中,阿莫西林的MIC值最高,均>128 mg·L-1;其次为链霉素,MIC值为32~8 mg·L-1;后面依次为林可霉素(MIC值均为8 mg·L-1)、阿米卡星(MIC值为8~2 mg·L-1)、氨苄西林(MIC值为4~2 mg·L-1);对黏菌素的MIC值差异较大,MIC最大值为4 mg·L-1(PmCW1),而最小的则为 < 0.06 mg·L-1(PmCW5),存在至少64倍的差异;对庆大霉素的MIC值,除PmCW1为2 mg·L-1,其余均为1 mg·L-1;5株菌对其余11种抗菌药物的MIC值均≤0.5 mg·L-1。受试药物的耐药折点值判断标准主要参考CLSI VET01-S中描述的值,CLSI VET01-S未给出的则参考欧盟委员会药物敏感性测验(EUCAST)标准[23]。5株菌均对氨苄西林、阿莫西林、左氧氟沙星和林可霉素4种药物耐药,其中PmCW1还对环丙沙星低水平耐药。
|
|
表 1 抗菌药物对5株鸭源多杀性巴氏杆菌的 MIC值 Table 1 MIC value of five P. multocida isolates derived from duck |
通过对PmCW1基因组测序数据分析发现,该菌存在多个耐药基因,包括2个β-内酰胺类耐药基因(青霉素结合蛋白pbp2和pbp1a),5个四环素耐药基因(tetA、tetB、tetT、tet34、tet35),1个磷霉素耐药基因(murA),1个磺胺类耐药基因(sul3),1个维吉尼亚霉素乙酰转移酶vatB,1个甲氧苄啶耐药基因(dfrA3),1个广谱抗菌剂三氯生耐药基因fabI,3个多黏菌素抗性决定区基因(PmrE、PmrC和basS)。同时检测到9个抗生素耐药基因簇/操纵子(CpxR、vanRE、vanRN、vanTrL、vanHA、vanHB、vanL、mecC、arlR)和18个与外排泵复合体相关的基因(mdtk、bcr-1、cmeC、cpxA、CRP、farB、hmrM、H-NS、macA、macB、MexI、MexV、msbA、msrB、opmE、patA、sav1866、TaeA),它们介导对多种抗生素耐药。此外,还预测出4个喹诺酮耐药决定区基因(gyrA、gyrB、parC、parE)和1个与氟喹诺酮耐药相关基因mfd,经比对发现gyrA的80位出现突变(Ser80Gly)、parC的84位出现突变(Leu84Ser)。
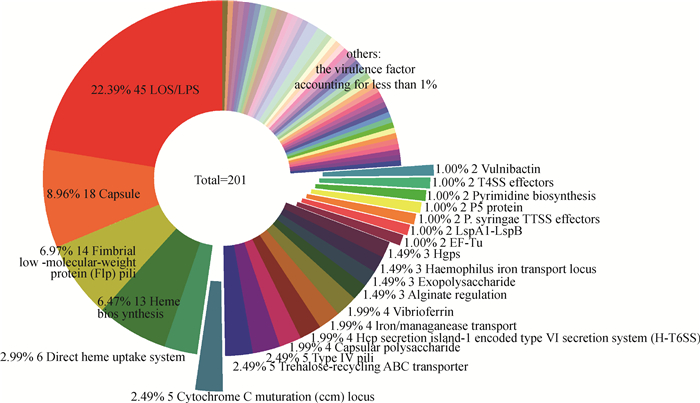
2.6 PmCW1毒力基因的分析经与VFDB数据库比对,预测PmCW1中存在的毒力基因总数为201;根据毒力基因所属类别名称(即VF_name)进行归类,毒力因子有71种;某类毒力基因的占比=(毒力基因所属类别名称即VF_name)÷(预测出来的毒力基因总数即201)×100%(图 8);如数量为1的即占比 < 1%,图 8中未将具体名称列出。PmCW1中存在的主要是与脂寡糖/脂多糖(LOS/LPS)(galE、galU、gmhA、htrB、kdkA、kdsA、kdsB、kdtA、kpsF、lex2B、lgtF、licA、licC、lpxA、lpxB、lpxC、lpxD、lpxH、lpxK、lsgA、lsgD、lsgE、lsgF、msbA、msbB、neuA、rfaC、orfM、rfaD、rfaE、rfaF、rffG、waq、wecA、yhxB/manB等)、荚膜(capsule)(phyBA、hyaEDCB、hexDCBA、bexA、bexC、bexD等)、黏附因子(fimbriae and adhesins)(flp1-flp2-tadV-rcpCAB-tadZABCDEFG、ptfA、comEA、hofBC等)等相关基因,此外,还有铁摄取相关蛋白基因(ccmABCEF、hgbBC、fur、hscB等)以及部分外膜蛋白基因(ompP5等),绝大多数在不同宿主来源的P. multocida中间高度保守。PmCW1中有些基因存在多个拷贝数,如脂寡糖/脂多糖相关基因rfaC存在2个拷贝、血红素生物合成相关基因hemN存在2个拷贝等。
|
占比 < 1%的毒力因子名称未列出 The names of virulence factors with a percentage < 1% are not listed 图 8 PmCW1毒力决定因子 Fig. 8 PmCW1 virulence determining factor |
脂多糖变异区(即外部核心多糖编码基因簇)比较基因组学分析显示(图 9a),PmCW1(A:L1,ST128)与HN141014(A:L1,ST129)相比,它们之间基因结构一致;而与P1059(A:L3,ST8)、Pm70(F:L3,ST9)、ATCC1702(A:L2,ST156)相比,它们之间的变异区主要集中在hptE与priA基因之间,这一区间内的基因主要为糖基转移酶编码基因。
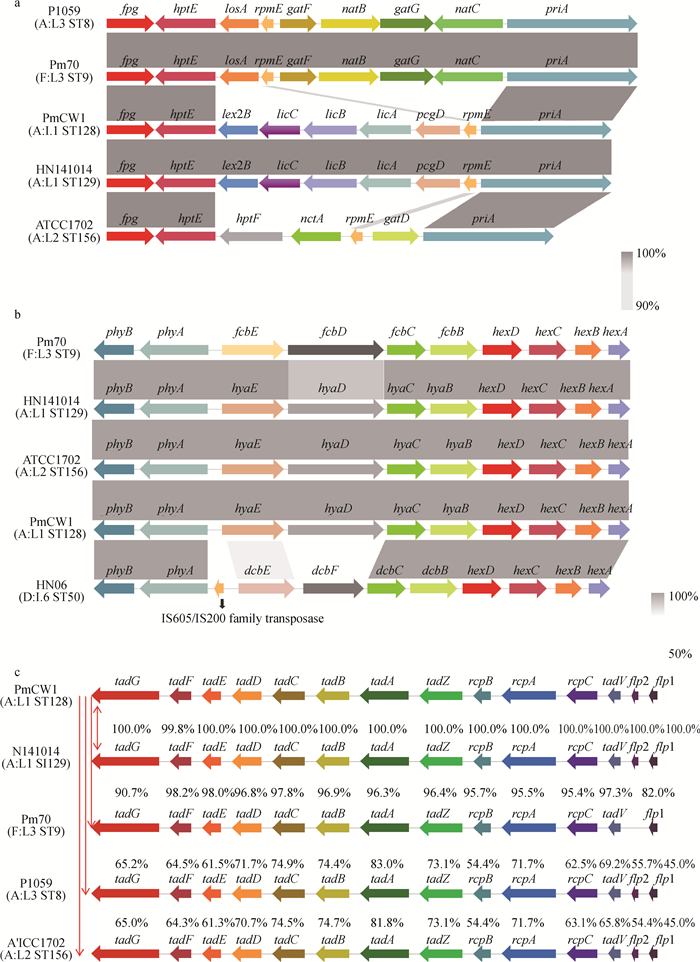
|
图 9 多杀性巴氏杆菌脂多糖核心寡糖外核(a)、荚膜合成区(b)及Flp操纵子(c)比较基因组学分析 Fig. 9 Comparative genomic analysis of LPS outer core biosynthesis gene clusters(a), capsular loci (b), and Flp-operons(c) among different P. multocida |
荚膜多糖磷脂替换区phyBA和荚膜多糖输出区hexDCBA,在不同血清型之间高度保守,而合成区基因簇存在差异,A型由hyaEDCB基因编码,D型由dcbEFCB基因编码,F型由fcbEDCB基因编码。PmCW1(A:L1,ST128)与HN141014(A:L1,ST129)、ATCC1702(A:L2,ST156)相比,各基因结构一致;与Pm70(F:L3,ST9)相比,除hyaD与fcbD的相似性为82.7%,其余各基因相似性均高于98.1%;与HN06(D:L6,ST50)相比,hyaED与dcbEF之间相似性在61.1%~50.4%,且HN06在phyBA和hyaED之间存在一个IS605/IS200家族转座酶基因(图 9b)。
Flp操纵子由14个基因(flp1-flp2-tadV-rcpCAB-tadZABCDEFG)组成,承担细菌的黏附功能。在本研究中分析5株巴氏杆菌Flp操纵子相似情况如图 9c所示,除Pm70(F:L3,ST9)缺失flp2基因,其余4株的Flp操纵子均是经典的14基因编码组成。PmCW1(A:L1,ST128)的Flp操纵子与HN141014(A:L1,ST129)的各基因结构一致;与Pm70(F:L3,ST9)除了flp1基因外各基因相似性均高于82.0%;而与P1059(A:L3,ST8)、ATCC1702(A:L2,ST156)虽然基因结构一致,但基因相似性较低。
3 讨论荚膜和LPS对P. multocida的毒力和宿主特异性起着重要作用。本研究从贵州规模化鸭场分离得到5株鸭源P.multocida强毒株,经鉴定血清型(荚膜基因型:脂多糖基因型)为A:L1型,这与王林柏等[24]、Li等[25]报道的国内部分地区禽源P.multocida血清型主要为A:L1型的结果一致,也与Peng等[3]报道全球不同地区的流行情况相同,说明在全球范围内流行的禽源P.multocida仍以A:L1型为主。需要注意的是,Ujvári等[26]、Ewers等[27]报道A:L1在猫和人分离株中最常见,Turni等[28]、Ujvári等[29]也报道过人类和各种动物物种之间存在P.multocida的共享基因型,同时还有报道[26]称人类不是P.multocida的自然宿主,但是人类可能通过与家庭宠物或养殖场动物接触而感染,造成不同血清型的P.multocida跨物种水平传播到人类。
已有的流行病学调查数据表明,导致某些特定疾病的P. multocida集中于某一特定的ST型,例如有报道与出血性败血症相关的P. multocida多为ST122,而与禽霍乱相关P.multocida多为ST129[30-31]。这些结果意味着P. multocida的ST型与疾病之间也可能存在一定的相关性。目前无论是我国还是全球范围内禽源P. multocida的多位点序列分型以ST129型为主,偶见ST122、ST107、ST6、ST44、ST8、ST58等[1, 24, 32-33]。本研究分离得到的鸭源P. multocida均为ST128型,这在禽源P. multocida中鲜有报道,提示在贵州地区鸭源P. multocida出现了不同于全国流行的ST型。
本研究中P. multocida分离株仅对氨苄西林、阿莫西林、左氧氟沙星、林可霉素和环丙沙星5种药物耐药,不同于陈国权等[34]、Holschbach等[7]报道的P. multocida分离株耐药性严重,说明P. multocida的耐药性受地理位置、用药史、管理水平及环境等多方面因素影响。对食品动物上禁用抗生素(如头孢曲松、头孢噻肟、头孢他啶、阿奇霉素等)的MIC值较低,说明此规模化鸭场对抗菌药物的使用相对合理。同时结合采样调研,说明此鸭场未乱用这些抗生素。本研究中的分离株ST型相同,但药敏试验结果存在差异,如不同菌株对黏菌素的MIC值存在至少64倍的差异,这可能与在动物体内不同抗生素压力下的选择作用有关。
PmCW1的耐药表型与耐药基因检测结果基本相符,如PmCW1中仅存在2个β-内酰胺类耐药基因(青霉素结合蛋白pbp2和pbp1a),这与PmCW1对青霉素类药物氨苄西林和半合成青霉素类药物阿莫西林均耐药,而对其他4种三代头孢菌素类药物MIC值均 < 0.06 mg·L-1的结果相一致。喹诺酮耐药决定区(QRDR)靶位基因突变通常定位于gyrA(第67-106位)或parC(第63-102位)的氨基末端结构域,gyrB和parE的突变也会导致喹诺酮类药物耐药,但突变频率相对少[35]。本研究中发现PmCW1的QRDR存在突变,gyrA的80位出现突变(Ser80Gly)和parC的84位出现突变(Leu84Ser),Kong等[36]也报道过P.multocida中parC的84位突变,这可能是PmCW1对喹诺酮类药物耐药的原因之一。此外,还预测到1个与氟喹诺酮耐药相关基因mfd,在空肠弯曲杆菌中mfd的过度表达提高了环丙沙星耐药性的自发突变率,mfd对氟喹诺酮类药物耐药性的产生中起重要作用[37],但mfd在P. multocida中对氟喹诺酮类药物耐药性的作用还需要进一步验证。
在流行病学研究中,毒力基因分型已发展成为一种有用的基因分型方法,所用基因一部分为P. multocida的广泛特征基因,它们在不同基因型和/或不同寄主种类的菌株中具有较高的检出率,如ptfA、fimA、hsf-2、exbB、exbD、tonB、fur、sodA、sodC、ompA、ompH、oma87和plpB等;另一部分毒力基因只在有限数量的分离株中检测到,这主要与特定类型的疾病和/或某些寄主物种有关,例如toxA只在与进行性萎缩性鼻炎相关的菌株中发现,而tbpA只在牛和羊源的分离株中发现[17]。本研究中预测到参与编码PmCW1的毒力基因有201个,除荚膜、LPS和Flp相关基因外,还有IV型菌毛基因(ptfA、comE、hofB、hofC、vfr)、铁摄取相关蛋白(ccmABCEF、hgbBC、fur、hscB等)以及部分外膜蛋白(ompP5等)等重要毒力基因。IV型菌毛基因ptfA已经在荚膜A型、B型和D型菌株中被发现[38],ptfA和comE编码的假定蛋白与菌毛帮助P.multocida黏附到宿主细胞上面,这与在其他细菌中发现的黏附素结构相似[39];ccmABCDEF操纵子,编码参与血红素翻译后附着的蛋白质,在大肠杆菌中也有发现[40];fur编码铁摄取调节因子Fur,与在胸膜肺炎放线杆菌中发现的相似[41];这些毒力基因的存在、缺失或丰度可能受到寄主、地理位置或环境因素的影响,对P. multocida毒力的增强、减弱具有一定的作用。
通过局部比较基因组学分析显示,强毒株PmCW1在脂多糖变异区和荚膜合成区的基因簇与A:L1型P. multocida各基因结构一致,说明PmCW1具有典型的A:L1型P. multocida的特征。脂多糖变异区(即外部核心多糖编码基因簇)常位于两个保守基因fpg和priA之间[42],而在本研究中,L1型与其他两种LSP型(L2、L3型)菌株间差异区缩小,主要集中在hptE和priA之间,这种差异可能是引起P. multocida菌体脂多糖血清型不同的原因[43]。值得注意的是rpmE基因在不同菌株间相似性高于90%,且在hptE和priA之间随机排列。对荚膜合成区进行比较,发现A型与D型的各基因结构及相似性差异大,而A型与F型的差异非常小,这可能与P. multocida分离株感染的宿主偏好性有关,如A型和F型常引起禽类的感染,D型在猪的感染中占优势[3]。Flp操纵子通常由14个基因编码,但也存在部分基因缺失的情况[44],如禽源弱毒株Pm70(F:L3,ST9)的Flp操纵子缺失flp2基因;PmCW1具有经典的14基因编码,与其余4株A型P.multocida之间基因结构相同,但相似性存在差异。本研究系统分析了鸭源A:L1,ST128型P.multocida强毒株PmCW1的耐药性及毒力特征,为贵州地区鸭霍乱的临床用药及疫苗研发提供理论依据。
4 结论通过对临床疑似鸭霍乱的病原菌进行系统研究,发现在鸭源P. multocida鲜有报道的ST128型PmCW1,且为强毒株。PmCW1携带多种耐药基因(pbp2、pbp1a、tetA、tetB、tetT、tet34、tet35、sul3以及dfrA3)介导对青霉素类、四环素类、磺胺类以及甲氧苄啶等药物耐药;此外,还存在耐药基因簇/操纵子和外排泵复合体相关的基因,可介导P. multocida对多种抗生素耐药。PmCW1具有典型的A:L1型P. multocida的特征,毒力基因主要是脂寡糖/脂多糖(LOS/LPS)、荚膜、黏附因子等相关编码基因;此外,还检测到还有IV型菌毛基因(ptfA、comE、hofB、hofC、vfr)、铁摄取相关蛋白(ccmABCEF、hgbBC、fur、hscB等)以及部分外膜蛋白(ompP5等)等重要毒力基因。
| [1] |
WILKIE I W, HARPER M, BOYCE J D, et al. Pasteurella multocida: diseases and pathogenesis[M]//AKTORIES K, ORTH J H C, ADLER B. Pasteurella Multocida. Berlin: Springer, 2012: 1-22.
|
| [2] |
WILSON B A, HO M. Pasteurella multocida: from zoonosis to cellular microbiology[J]. Clin Microbiol Rev, 2013, 26(3): 631-655. DOI:10.1128/CMR.00024-13 |
| [3] |
PENG Z, LIANG W, WANG F, et al. Genetic and phylogenetic characteristics of Pasteurella multocida isolates from different host species[J]. Front Microbiol, 2018, 9: 1408. DOI:10.3389/fmicb.2018.01408 |
| [4] |
HONNORAT E, SENG P, SAVINI H, et al. Prosthetic joint infection caused by Pasteurella multocida: a case series and review of literature[J]. BMC Infect Dis, 2016, 16(1): 435. DOI:10.1186/s12879-016-1763-0 |
| [5] |
RYAN J M, FEDER JR H M. Dog licks baby. Baby gets Pasteurella multocida meningitis[J]. Lancet, 2019, 393: E41. DOI:10.1016/S0140-6736(19)30953-5 |
| [6] |
LIU Q, HU Y L, LI P, et al. Identification of fur in Pasteurella multocida and the potential of its mutant as an attenuated live vaccine[J]. Front Vet Sci, 2019, 6: 5. DOI:10.3389/fvets.2019.00005 |
| [7] |
HOLSCHBACH C L, AULIK N, POULSEN K, et al. Prevalence and temporal trends in antimicrobial resistance of bovine respiratory disease pathogen isolates submitted to the Wisconsin Veterinary Diagnostic Laboratory: 2008-2017[J]. J Dairy Sci, 2020, 103(10): 9464-9472. DOI:10.3168/jds.2019-17570 |
| [8] |
刘海燕, 杨有文, 王远微, 等. 6株山羊源多杀性巴氏杆菌血清型、耐药性及致病性分析[J]. 黑龙江畜牧兽医, 2020(16): 65-68. LIU H Y, YANG Y W, WANG W Y, et al. Analysis of serotype, drug resistance and pathogenicity of 6 strains of Pasteurella multocida from goats[J]. Heilongjiang Animal Science and Veterinary Medicine, 2020(16): 65-68. (in Chinese) |
| [9] |
PRAJAPATI A, CHANDA M M, DHAYALAN A, et al. Variability in in vitro biofilm production and antimicrobial sensitivity pattern among Pasteurella multocida strains[J]. Biofouling, 2020, 36(8): 938-950. DOI:10.1080/08927014.2020.1833192 |
| [10] |
DABO S M, TAYLOR J D, CONFER A W. Pasteurella multocida and bovine respiratory disease[J]. Anim Health Res Rev, 2007, 8(2): 129-150. DOI:10.1017/S1466252307001399 |
| [11] |
HARPER M, BOYCE J D, ADLER B. Pasteurella multocida pathogenesis: 125 years after pasteur[J]. FEMS Microbiol Lett, 2006, 265(1): 1-10. DOI:10.1111/j.1574-6968.2006.00442.x |
| [12] |
TOWNSEND K M, BOYCE J D, CHUNG J Y, et al. Genetic organization of Pasteurella multocida cap loci and development of a multiplex capsular PCR typing system[J]. J Clin Microbiol, 2001, 39(3): 924-929. DOI:10.1128/JCM.39.3.924-929.2001 |
| [13] |
PENG Z, LIANG W, LIU W J, et al. Genomic characterization of Pasteurella multocida HB01, a serotype A bovine isolate from China[J]. Gene, 2016, 581(1): 85-93. DOI:10.1016/j.gene.2016.01.041 |
| [14] |
MARZA A D, JESSE ABDULLAH F F, AHMED I M, et al. The ability of lipopolysaccharide (LPS) of Pasteurella multocida B: 2 to induce clinical and pathological lesions in the nervous system of buffalo calves following experimental inoculation[J]. Microb Pathog, 2017, 104: 340-347. DOI:10.1016/j.micpath.2017.01.031 |
| [15] |
PERIASAMY S, PRAVEENA P E, SINGH N. Effects of Pasteurella multocida lipopolysaccharides on bovine leukocytes[J]. Microb Pathog, 2018, 119: 225-232. DOI:10.1016/j.micpath.2018.04.030 |
| [16] |
HARPER M, JOHN M, TURNI C, et al. Development of a rapid multiplex PCR assay to genotype Pasteurella multocida strains by use of the lipopolysaccharide outer core biosynthesis locus[J]. J Clin Microbiol, 2015, 53(2): 477-485. DOI:10.1128/JCM.02824-14 |
| [17] |
PENG Z, WANG X R, ZHOU R, et al. Pasteurella multocida: genotypes and genomics[J]. Microbiol Mol Biol Rev, 2019, 83(4): e00014-19. |
| [18] |
Clinical Laboratory Standards Institute (CLSI). Performance standards for antimicrobial disk and dilution susceptibility tests for bacteria isolated from animals; Approved standard-fourth edition. CLSI document VET01-A4[R]. Wayne, PA, USA: CLSI, 2013.
|
| [19] |
WICK R R, JUDD L M, GORRIE C L, et al. Unicycler: resolving bacterial genome assemblies from short and long sequencing reads[J]. PLoS Comput Biol, 2017, 13(6): e1005595. DOI:10.1371/journal.pcbi.1005595 |
| [20] |
BESEMER J, LOMSADZE A, BORODOVSKY M. GeneMarkS: a self-training method for prediction of gene starts in microbial genomes. Implications for finding sequence motifs in regulatory regions[J]. Nucleic Acids Res, 2001, 29(12): 2607-2618. DOI:10.1093/nar/29.12.2607 |
| [21] |
BUCHFINK B, XIE C, HUSON D H. Fast and sensitive protein alignment using DIAMOND[J]. Nat Methods, 2015, 12(1): 59-60. DOI:10.1038/nmeth.3176 |
| [22] |
SULLIVAN M J, PETTY N K, BEATSON S A. Easyfig: a genome comparison visualizer[J]. Bioinformatics, 2011, 27(7): 1009-1010. DOI:10.1093/bioinformatics/btr039 |
| [23] |
刘玉庆, 李璐璐, 骆延波, 等. EUCAST 2017欧盟药敏试验标准[M]. 北京: 中国标准出版社, 2016. LIU Y Q, LI L L, LUO Y B, et al. European committee on antimicrobial susceptibility testing[M]. Beijing: Standards Press of China, 2016. (in Chinese) |
| [24] |
王林柏, 孙久鹤, 郭东春, 等. 国内部分地区多杀性巴氏杆菌荚膜血清型和基因型的研究[J]. 中国预防兽医学报, 2016, 38(2): 116-119. WANG L B, SUN J H, GUO D C, et al. Identification of capsule serotype and genotype of Pasteurella multocida in some areas of China[J]. Chinese Journal of Preventive Veterinary Medicine, 2016, 38(2): 116-119. (in Chinese) |
| [25] |
LI Z C, CHENG F J, LAN S M, et al. Investigation of genetic diversity and epidemiological characteristics of Pasteurella multocida isolates from poultry in southwest China by population structure, multi-locus sequence typing and virulence-associated gene profile analysis[J]. J Vet Med Sci, 2018, 80(6): 921-929. DOI:10.1292/jvms.18-0049 |
| [26] |
UJVÁRI B, MAKRAI L, MAGYAR T. Virulence gene profiling and ompA sequence analysis of Pasteurella multocida and their correlation with host species[J]. Vet Microbiol, 2019, 233: 190-195. DOI:10.1016/j.vetmic.2019.05.005 |
| [27] |
EWERS C, LUBKE-BECKER A, BETHE A, et al. Virulence genotype of Pasteurella multocida strains isolated from different hosts with various disease status[J]. Vet Microbiol, 2006, 114(3-4): 304-317. DOI:10.1016/j.vetmic.2005.12.012 |
| [28] |
TURNI C, SINGH R, BLACKALL P J. Genotypic diversity of Pasteurella multocida isolates from pigs and poultry in Australia[J]. Aust Vet J, 2018, 96(10): 390-394. DOI:10.1111/avj.12748 |
| [29] |
UJVÁRI B, WEICZNER R, DEIM Z, et al. Characterization of Pasteurella multocida strains isolated from human infections[J]. Comp Immunol, Microbiol Infect Dis, 2019, 63: 37-43. DOI:10.1016/j.cimid.2018.12.008 |
| [30] |
PETERSEN A, BISGAARD M, TOWNSEND K, et al. MLST typing of Pasteurella multocida associated with haemorrhagic septicaemia and development of a real-time PCR specific for haemorrhagic septicaemia associated isolates[J]. Vet Microbiol, 2014, 170(3-4): 335-341. DOI:10.1016/j.vetmic.2014.02.022 |
| [31] |
WANG Y H, ZHU J, LU C P, et al. Evidence of circulation of an epidemic strain of Pasteurella multocida in Jiangsu, China by multi-locus sequence typing (MLST)[J]. Infect, Genet Evol, 2013, 20: 34-38. DOI:10.1016/j.meegid.2013.07.027 |
| [32] |
李伟杰, 田野, 岂晓鑫, 等. 禽源多杀性巴氏杆菌多位点序列分型研究[J]. 中国兽药杂志, 2017, 51(2): 1-6. LI W J, TIAN Y, QI X X, et al. Multi-locus sequence typing of Pasteurella multocida isolated from Avian[J]. Chinese Journal of Veterinary Drug, 2017, 51(2): 1-6. (in Chinese) |
| [33] |
陈红梅, 程龙飞, 邓辉, 等. 鸭源多杀性巴氏杆菌对磺胺类药物的耐药性检测及耐药基因分析[J]. 中国畜牧兽医, 2019, 46(1): 223-230. CHEN H M, CHENG L F, DENG H, et al. Detection of sulfonamides sensitivity and analysis of resistant genes of Pasteurella multocida isolated from ducks[J]. China Animal Husbandry & Veterinary Medicine, 2019, 46(1): 223-230. (in Chinese) |
| [34] |
陈国权, 张旭, 阎朝华, 等. 鸭源多杀性巴氏杆菌的分离鉴定及耐药性分析[J]. 中国畜牧兽医, 2020, 47(2): 620-628. CHEN G Q, ZHANG X, YAN C H, et al. Isolation, identification and drug resistance analysis of Pasteurella multocida from ducks[J]. China Animal Husbandry & Veterinary Medicine, 2020, 47(2): 620-628. (in Chinese) |
| [35] |
HOOPER D C, JACOBY G A. Mechanisms of drug resistance: quinolone resistance[J]. Ann N Y Acad Sci, 2015, 1354(1): 12-31. DOI:10.1111/nyas.12830 |
| [36] |
KONG L C, GAO D, GAO Y H, et al. Fluoroquinolone resistance mechanism of clinical isolates and selected mutants of Pasteurella multocida from bovine respiratory disease in China[J]. J Vet Med Sci, 2014, 76(12): 1655-1657. DOI:10.1292/jvms.14-0240 |
| [37] |
HAN J, SAHIN O, BARTON Y W, et al. Key role of Mfd in the development of Fluoroquinolone resistance in Campylobacter jejuni[J]. PLoS Pathog, 2008, 4(6): e1000083. DOI:10.1371/journal.ppat.1000083 |
| [38] |
RUFFOLO C G, TENNENT J M, MICHALSKI W P, et al. Identification, purification, and characterization of the type 4 fimbriae of Pasteurella multocida[J]. Infect Immun, 1997, 65(1): 339-343. DOI:10.1128/iai.65.1.339-343.1997 |
| [39] |
HATFALUDI T, AL-HASANI K, BOYCE J D, et al. Outer membrane proteins of Pasteurella multocida[J]. Vet Microbiol, 2010, 144(1-2): 1-17. DOI:10.1016/j.vetmic.2010.01.027 |
| [40] |
ALLEN J W A, LEACH N, FERGUSON S J. The histidine of the c-type cytochrome CXXCH haem-binding motif is essential for haem attachment by the Escherichia coli cytochrome c maturation (Ccm) apparatus[J]. Biochem J, 2005, 389: 587-592. DOI:10.1042/BJ20041894 |
| [41] |
JACOBSEN I, GERSTENBERGER J, GRUBER A D, et al. Deletion of the ferric uptake regulator Fur impairs the in vitro growth and virulence of Actinobacillus pleuropneumoniae[J]. Infect Immun, 2005, 73(6): 3740-3744. DOI:10.1128/IAI.73.6.3740-3744.2005 |
| [42] |
HARPER M, COX A D, ADLER B, et al. Pasteurella multocida lipopolysaccharide: the long and the short of it[J]. Vet Microbiol, 2011, 153(1-2): 109-115. DOI:10.1016/j.vetmic.2011.05.022 |
| [43] |
PETRUZZI B, BRIGGS R E, SWORDS W E, et al. Capsular polysaccharide interferes with biofilm formation by Pasteurella multocida Serogroup A[J]. mBio, 2017, 8(6): e01843-17. |
| [44] |
TOMICH M, PLANET P J, FIGURSKI D H. The tad locus: Postcards from the widespread colonization island[J]. Nat Rev Microbiol, 2007, 5(5): 363-375. DOI:10.1038/nrmicro1636 |
(编辑 白永平)



