2. 河南省农业科学院动物免疫学重点实验室, 农业部动物免疫学重点实验室, 河南省动物免疫学重点实验室, 郑州 450002;
3. 河南省农业科学院, 中英禽病国际研究中心, 郑州 450002;
4. 河南科技大学动物科技学院, 动物疫病与公共安全重点实验室, 洛阳 471003;
5. 扬州大学江苏高校动物重要疫病与人兽共患病防控协同创新中心, 扬州 225009
2. Key Laboratory of Animal Immunology of Ministry of Agriculture&Henan Provincial Key Laboratory of Animal Immunology, Henan Academy of Agricultural Sciences, Zhengzhou 450002, China;
3. UK-China Centre of Excellence for Research on Avian Diseases, Henan Academy of Agricultural Sciences, Zhengzhou 450002, China;
4. Key Laboratory of Animal Disease and Public Safety, College of Animal Science and Technology, Henan University of Science and Technology, Luoyang 471003, China;
5. Jiangsu Co-Innovation Center for the Prevention and Control of Important Animal Infectious Diseases and Zoonoses, Yangzhou University, Yangzhou 225009, China
马立克病病毒编码的miR-M4-5p调控宿主靶基因的筛选及鉴定马立克病病毒(Marek’s disease virus, MDV)是甲型疱疹病毒亚家族的重要成员,在其自然感染宿主鸡中可引起免疫抑制和快速发作的T细胞淋巴瘤,即马立克病(Marek’s disease, MD)[1]。MDV有3个血清型:MDV-1(GaHV2)、MDV-2(GaHV3)和MDV-3(MeHV1)[2],其中MDV-1包括对宿主具有毒力或致瘤性的强毒分离株以及它们的致弱变异株,MDV-2包括可产生小型病毒蚀斑但对宿主无致病性的毒株,MDV-3是从火鸡体内分离得到的非致病性家禽疱疹病毒,即火鸡疱疹病毒(herpesvirus of turkeys,HVT)。MD是世界上首例可通过病毒疫苗免疫预防进行有效控制的肿瘤性疾病,所以MDV感染和致瘤一直被认为是研究病毒性肿瘤病发生机制的一种良好的天然模型[3]。
微小RNA(microRNA,miRNA)是一类长20~24 nt的小分子非编码RNA,可以调控多种细胞生物学过程,主要通过降低mRNA稳定性、抑制基因转录后表达水平及蛋白质翻译[4-5]。1993年,生物学家在线虫中首次报道miRNA[6],2004年在病毒基因组也发现可以编码和表达miRNA[7]。2006年,首次报道了MDV-1编码的miRNA[8],在MDV-1基因组中,共编码有26种病毒miRNA,它们可能在病毒复制、潜伏感染和/或肿瘤发生中发挥关键作用[9-17]。这些miRNA形成3个明显的基因簇[18],分别被命名为Meq基因簇、Mid基因簇和LAT基因簇,其中Meq基因簇miRNA对MDV-1的致瘤表型具有重要调控作用[19]。miR-M4-5p是Meq基因簇的重要成员,已经被鉴定为病毒编码的宿主癌基因miR-155同源物,两者具有相同的种子序列[20-22]。miR-M4-5p在病毒感染诱导的肿瘤组织中表达量最高,可能在T淋巴细胞转化和肿瘤发生过程中发挥重要作用[23]。此前的研究也发现,miR-M4-5p参与调控MD肿瘤形成[19, 24-25],但其具体的调控分子机制有待进一步深入研究。
本研究中,首先利用hybrid-PCR技术[26]构建了1个cDNA文库,用于筛选miR-M4-5p潜在调控的宿主mRNA靶基因;然后构建报告基因载体质粒,通过双荧光素酶报告试验(dual luciferase reporter assay, DLRA)对miR-M4-5p与候选靶基因3′-UTR之间的相互作用进行体外验证;最后在CEF中过表达miR-M4-5p,利用qRT-PCR对候选靶基因mRNA的表达水平进行体内分析,以期最终鉴定宿主靶基因,为进一步阐明miR-M4-5p在MD肿瘤发生中的调控机制奠定重要基础。
1 材料与方法 1.1 细胞鸡胚成纤维细胞(chicken embryo fibroblast, CEF)分离自9~10日龄SPF鸡胚(北京勃林格殷格翰维通生物技术有限公司);HEK 293T细胞由河南省农业科学院动物免疫学重点实验室保存。
1.2 主要试剂与载体Lipofectamine 2000和TRIzol购自Invitrogen公司;3′-Full RACE Core Set with PrimeScript RTase、PrimeScriptTM RT Master Mix、pMD19-T(simple)vector、Primix TaqTM、LA Taq和E.coli JM109 Competent Cells,均购自宝生物工程(大连)有限公司;DNA限制性内切酶Xho Ⅰ和NotⅠ-HF购自NEB(北京)有限公司;Annealing Buffer for DNA Oligos(5×)购自碧云天生物技术公司;psiCHECK-2、pcDNA-6.2、pcDNA6.2-miR-neg、pcDNA6.2-miR-M4和pcDNA6.2-mut-miR-M4质粒和菌种由河南省农业科学院动物免疫学重点实验室制备并保存。
1.3 引物设计与合成本研究所使用的DNA引物及DNA片段均使用Beacon Designer 7软件设计,由生工生物工程(上海)股份有限公司合成。引物名称和序列等相关背景信息见表 1。
|
|
表 1 用于构建cDNA文库、制备3′-UTR及qRT-PCR分析的引物 Table 1 List of the primers used for the cDNA library construction, 3′-UTR preparation and qRT-PCR analysis |
TRIzol法提取CEF细胞总RNA,用NanoDrop 2000全波长紫外分光光度计(Invitrogen公司)测定RNA浓度和纯度,确保RNA的OD260 nm/OD280 nm的比值为1.8~2.1。按照3′-Full RACE Core Set with PrimeScript RTase试剂盒说明书,将提取的RNA样品反转录合成cDNA,然后进行套式PCR反应,包括两步:Outer PCR和Inner PCR。Outer PCR反应中以miR-M4-5p-RACE (表 1)为上游引物,以试剂盒自备的3′RACE Outer Primer为下游引物,以上述反转录得到的cDNA为模板进行PCR反应。Inner PCR反应中以miR-M4-5p-RACE为上游引物,以试剂盒自备的3′RACE Inner Primer为下游引物,以Outer PCR产物为模板进行反应。最后,将Inner PCR产物进行琼脂糖凝脂电泳分析,用胶回收试剂盒纯化回收100~ 1 000 bp的全部PCR产物,连接pMD19-T载体,转化至E. coli JM109感受态细胞并涂Amp抗性琼脂平板培养过夜,挑单菌落扩大培养,用miR-M4-5p-RACE和3′ RACE Inner Primer为上下游引物进行菌液PCR鉴定,将阳性菌液送样至生工生物工程(上海)股份有限公司测序。用BLASTn软件对测序结果进行序列比对分析,确定候选靶基因名称并下载全序列,构建miR-M4-5p候选靶基因文库。
1.5 载体质粒的构建取合成的含有miR-M4-5p结合靶点的候选基因3′ -UTR寡核苷酸链(表 1),分别用ddH2O溶解至终浓度为50 μmol·L-1,进行退火反应。反应体系为:Nuclease-Free Water 40 μL,Annealing Buffer for DNA Oligos(5×)20 μL,正、反核苷酸链各20 μL(50 μmol·L-1)。反应程序为95 ℃变性2 min,然后每90 s下降1 ℃,直至25 ℃。将退火形成的双链DNA冻存于-40 ℃备用。将psiCHECK-2质粒进行Xho Ⅰ和Not Ⅰ-HF双酶切,然后进行琼脂糖凝胶电泳分析,将psiCHECK-2载体大片段进行割胶回收。将退火得到的3′-UTR双链DNA片段与psiCHECK-2载体回收产物于4 ℃连接过夜,然后转化至E. coli JM109感受态细胞,涂板(Amp抗性)培养过夜,挑取单克隆菌落扩大培养,并送样测序,测序结果符合预期的菌种储存于-40 ℃备用。同上所述,将表 1列示合成的3′-UTR突变寡核苷酸链复性制备DNA双链后,插入psiCHECK-2载体,构建双荧光素酶报告突变载体质粒。
1.6 双荧光素酶报告试验将HEK 293T细胞铺于48孔细胞板中,37 ℃、5% CO2培养箱中培养至汇合度约80%~90%。取psiCHECK-2-3′-UTR和pcDNA6.2-miR-M4质粒各300 ng,均匀混于12.5 μL无血清培养基中;以1 μL·孔-1的量取Lipofectamine 2000稀释于12.5 μL无血清培养基中预混脂质体;两个体系各自孵育20 min,混合在一起再次孵育20 min。然后按照说明书,将孵育后的混合质粒/转染试剂(25 μL·孔-1)共转染HEK 293T细胞。以pcDNA6.2-miR-neg为阴性对照组,pcDNA6.2-mut-miR-M4为突变对照组,每组均设置3孔重复,在37 ℃、5% CO2培养箱中静置培养。转染48 h后,弃去细胞培养基,用PBS洗2次,然后用DLRA系统进行报告基因检测。首先用60 μL细胞裂解液裂解转染细胞,室温孵育15 min。然后将细胞裂解物转移至96孔白底不透明酶标板中,每孔加入100 μL LAR Ⅱ,轻柔混合均匀后, 用Ω酶标仪(OMG Labtech)测定萤火虫荧光素酶值。最后向每孔加入100 μL Stop and Glo Reagent,轻柔混合均匀后,同上测定海肾荧光素酶数值。最后统计海肾荧光素酶/萤火虫荧光素酶活性比值, 并进行差异显著性分析。
1.7 双突变DLRA验证试验同“1.6”所述,各取psiCHECK-2-mut-3′-UTR和pcDNA6.2-miR-M4质粒300 ng,与Lipofectamine 2000预混孵育后共转染HEK 293T细胞。以pcDNA6.2-miR-neg为阴性对照组,pcDNA6.2-mut-miR-M4为突变对照组,每组均设置3孔重复,转染后在37 ℃、5% CO2培养箱中静置培养。转染48 h后,同“1.6”所述处理转染细胞样品,分别测定萤火虫荧光酶素值和海肾荧光素酶数值,并统计分析海肾荧光素酶/萤火虫荧光素酶活性比值。
1.8 miRNA过表达和cDNA制备将CEF铺于6孔板中,37 ℃、5% CO2培养,待细胞汇合度至80%~90%时,用Lipofectamine 2000将pcDNA6.2-miR-neg、pcDNA6.2-miR-M4和pcDNA6.2-mut-miR-M4质粒分别转染至CEF中过表达miRNA。分别收集转染后24和48 h的细胞样品,按RNesay Mini Kit说明书提取细胞总RNA,并测定RNA样品的OD260nm/OD280nm,确保其比值介于1.8~2.1。使用PrimeScriptTM RT Master Mix一步式试剂盒将RNA反转录为cDNA的同时去除基因组DNA,-40 ℃保存备用。
1.9 qPCR以转染pcDNA6.2-miR-neg的CEF组cDNA为阴性对照,转染pcDNA6.2-mut-miR-M4的CEF组cDNA为突变对照,以鸡的管家基因GAPDH为内参基因进行qPCR反应,每个反应体系均设置3个重复。qPCR反应体系(20 μL)包括:cDNA模板1.5 μL、上下游引物各0.5 μL(10 μmol·L-1)、2×SYBR Premix Ex Taq 10 μL,加ddH2O补充体系至20 μL。反应程序:95 ℃ 2 min;95 ℃ 20 s,60 ℃ 34 s。熔解曲线生成条件为荧光定量PCR仪(7500 Fast,ABI)默认参数。所有qPCR试验数据均采用2-△△Ct法进行统计分析。
2 结果 2.1 miR-M4-5p候选靶基因cDNA文库的构建及鉴定将hybrid-PCR产物进行核酸凝胶电泳分析(图 1),然后割胶纯化长度100~1 000 nt的PCR产物,连接至pMD19-T载体后,转化E. coli JM109感受态细胞并涂板培养,挑取约2 000个单克隆菌落,活化后进行菌液PCR鉴定,共有1 446个样品可扩增出目的条带(图 2),随机挑选1 133个活化后的阳性菌样进行测序,获得436个有效的基因序列。经BLASTn比对分析后,获得128个单一序列基因。如表 2所示,miR-M4-5p结合靶点位于3′-UTR、CDS或5′-UTR的候选靶基因分别有73、46和9个。其中,结合靶点位于3′-UTR中的73个候选基因中,编号为1~23的候选宿主基因的结合靶点与miR-M4-5p种子序列形成完全互补(包括G:U配对)(表 2)。因此,优先选择这23个候选基因的3′-UTR进行下一步分析。
|
M. DNA相对分子质量标准 M. DNA marker 图 1 hybrid-PCR产物的琼脂糖凝胶电泳分析 Fig. 1 Agarose gel electrophoresis analysis of the hybrid-PCR products |
|
M. DNA相对分子质量标准; 1~20.菌样克隆号 M. DNA marker; Lanes 1-20. Bacterial clone number 图 2 cDNA基因文库部分克隆菌的PCR鉴定分析 Fig. 2 PCR analysis of partial clones of the cDNA library |
|
|
表 2 miR-M4-5p候选靶基因cDNA文库基因列表 Table 2 Candidate target genes for miR-M4-5p obtained from the cDNA library |
将人工合成的含有miR-M4-5p结合靶点的候选基因3′-UTR的DNA双链及相应3′-UTR突变体双链DNA,分别定向插入到psiCHECK-2载体中,构建psiCHECK-2-3′-UTR报告质粒,然后转化E. coli JM109感受态细胞中。经过PCR分析和测序鉴定,结果显示,表 1所列的23个psiCHECK-2-3′-UTR质粒和3个psiCHECK-2-mut-3′-UTR质粒均构建成功。
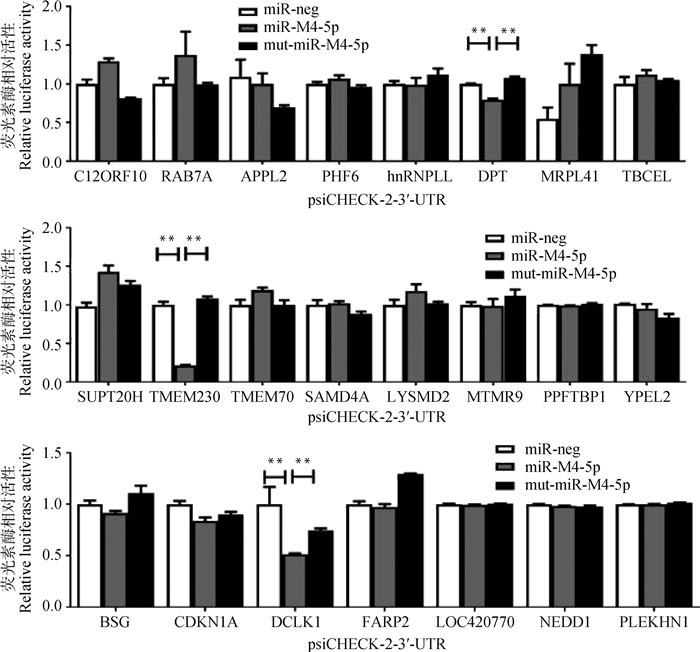
2.3 双荧光素酶报告试验(DLRA)分析将“2.2”构建的23个psiCHECK-3′-UTR质粒分别与pcDNA6.2-neg、pcDNA6.2-miR-M4或pcDNA6.2-mut-miR-M4载体共转染HEK 293T细胞,转染48 h后用双荧光素酶报告系统进行检测分析。如图 3所示,与阴性对照组和突变对照组相比,在23个被检测分析的候选基因3′-UTR质粒中,只有3个宿主基因的3′-UTR质粒(psiCHECK-2-DPT-3′-UTR、psiCHECK-2-TMEM230-3′-UTR和psiCHECK-2-DCLK1-3′-UTR)转染细胞的海肾荧光素酶/萤火虫荧光素酶活性比值出现了显著下调,表明miR-M4-5p可结合DPT、TMEM230和DCLK1的3′-UTR并抑制海肾荧光素酶的表达,这3个宿主基因很可能是miR-M4-5p调控的靶基因。
|
误差线表示3次独立重复试验的标准差(x),*、**.差异显著或极显著(P < 0.05或P < 0.01),下图同 Error bars indicate the standard deviation (x) for triplicate experiment repeats. *, **. Significant differences (P < 0.05 or P < 0.01), the same as below 图 3 DLRA分析miR-M4-5p与23个候选基因3′-UTR的体外相互作用 Fig. 3 In vitro interactions between miR-M4-5p and the 3′-UTR of 23 candidate host genes determined by dual luciferase reporter assay (DLRA) |
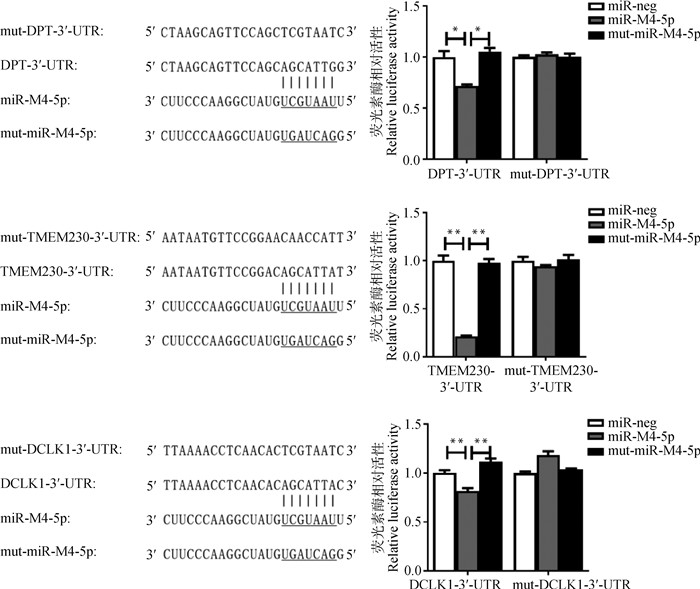
为了进一步验证miR-M4-5p与DPT、TMEM230和DCLK1的3′-UTR之间相互作用的序列特异性和依赖性,将psiCHECK-2-mut-DPT-3′-UTR、psiCHECK-2-mut-TMEM230-3′-UTR和psiCHECK-2-mut-DCLK1-3′-UTR分别与pcDNA6.2-miR-neg、pcDNA6.2-miR-M4、pcDNA6.2-mut-miR-M4质粒共转染至HEK 293T细胞中,于48 h后用双荧光素酶报告系统进行检测分析。结果如图 4所示,当这3个基因中的3′-UTR结合靶点序列发生突变之后,miR-M4-5p对海肾荧光素酶/萤火虫荧光素酶活性比值的下调均得到显著的解除,表明miR-M4-5p与DPT、TMEM230和DCLK1的3′-UTR之间的相互作用具有序列依赖性。
|
图 4 DLRA分析miR-M4-5p与候选靶基因3′-UTR的特异性相互作用 Fig. 4 Specific interactions between miR-M4-5p and candidate target genes determined by dual luciferase reporter assay |
为了进一步验证miR-M4-5p与候选宿主靶基因的相互作用,利用qRT-PCR分析了上述3个候选靶基因DPT、TMEM230和DCLK1在miR-M4-5p过表达的CEF细胞中的表达水平。分别用pcDNA6.2-miR-M4和pcDNA6.2-mut-miR-M4质粒转染CEF细胞,以pcDNA6.2-miR-neg转染的CEF组为阴性对照。如图 5所示,与表达miR-neg的阴性对照组比,在过表达miR-M4-5p的CEF细胞中,DPT、TMEM230和DCLK1的mRNA表达水平均发生极显著下调(P < 0.01);而当miR-M4-5p种子序列发生突变后,CEF细胞中DPT、TMEM230和DCLK1的表达水平则会恢复正常表达。上述数据表明,miR-M4-5p的表达与这3个宿主基因的表达水平呈显著的负相关关系,证实miR-M4-5p在体内可特异性抑制DPT、TMEM230和DCLK1的表达。
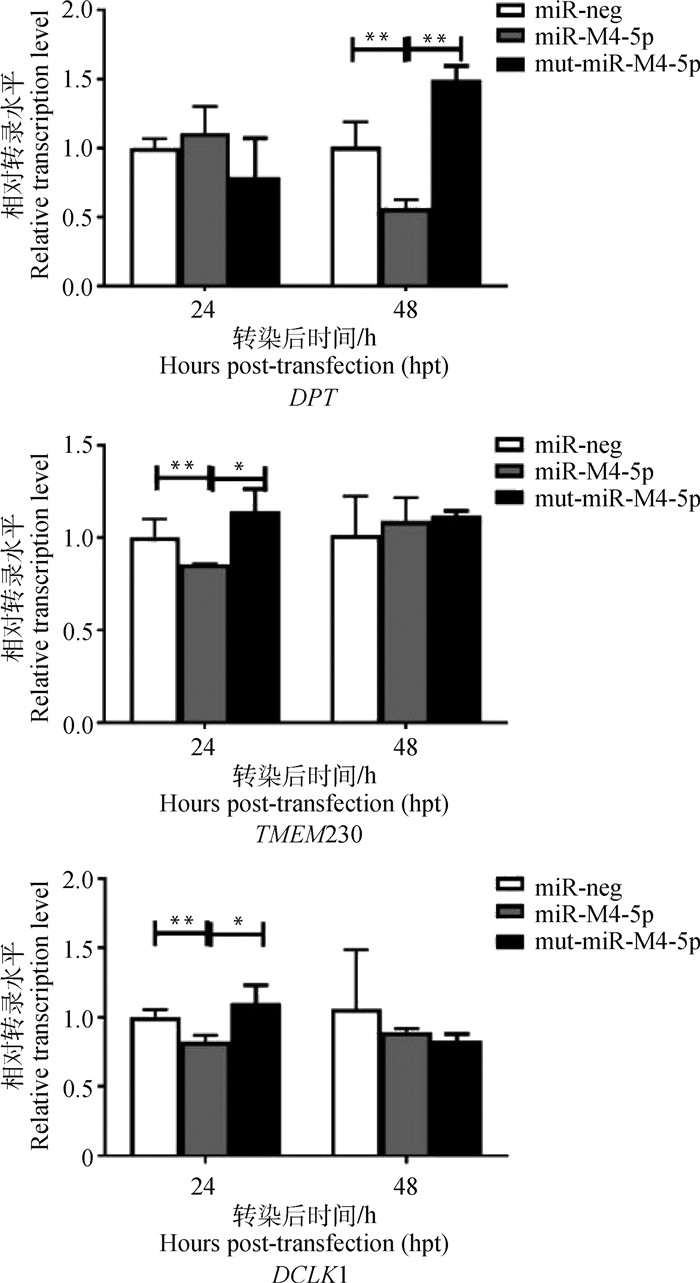
|
图 5 qRT-PCR分析miR-M4-5p过表达CEF中的候选靶基因的mRNA表达水平 Fig. 5 Expression levels of target mRNA genes in miR-M4-5p over-expressed CEFs analyzed by qRT-PCR |
miR-M4-5p是MDV-1编码的表达丰度最高的病毒miRNA之一,其种子序列与宿主miR-155完全保守,已被鉴定为病毒编码的重要miR-155同源物[20]。研究表明,miR-155是一个重要的宿主癌基因(oncomiR)[27],在维持细胞增殖、逃避生长抑制、抵抗细胞死亡、复制永生化、诱导血管生成和促进肿瘤的入侵、迁移等方面都发挥着调控作用。在此前的研究中,本课题组已鉴定LTBP1[25]和hnRNPAB[28]是miR-M4-5p调控的重要宿主靶基因。在MDV感染过程中,miR-M4-5p可通过靶向下调LTBP1的表达、抑制TGF-β信号通路的信号传导,这一作用过程随后激活宿主癌蛋白c-Myc的过表达,有助于促进诱导MD肿瘤的发生[25]。在CEF或DF-1细胞中过表达miR-M4-5p,或者用抑制剂基因沉默hnRNPAB的表达,可显著增强CEF与DF-1的细胞活性[28]。这些研究表明,miR-M4-5p在MDV-1的致病及致瘤过程中发挥重要的调控功能。本研究中,笔者利用hybrid-PCR法建立了miR-M4-5p的候选宿主靶基因cDNA文库,从中筛选获得23个与miR-M4-5p种子序列完全匹配的宿主基因3′-UTR。优先针对这些宿主基因3′-UTR,利用DLRA初筛、突变验证以及qRT-PCR分析,对miR-M4-5p与这些候选基因的体内外相互作用进行了探讨和验证。笔者发现,在CEF细胞中过表达miR-M4-5p后,与阴性对照组miR-neg、突变组mut-miR-M4-5p相比,DPT在转染后24 h的转录水平没有发生明显变化,但转染后48 h的转录水平出现了极显著差异;TMEM230和DCLK1在转染后24 h转录水平即出现显著差异。上述结果与DLRA分析结果基本一致,证实了miR-M4-5p与相应基因3′-UTR之间的特异性相互作用,可将DPT、TMEM230和DCLK1最终鉴定为miR-M4-5p的宿主靶基因。miRNA过表达后,在不同的时间点对不同的宿主靶基因产生的抑制效率可能不同,同时也可能与靶基因参与调控的复杂的信号通路有关。随着转染时间增加,miRNA的过表达达到峰值后逐渐降低,对靶基因的调控程度也随之发生变化。对于DPT而言,转染后24 h细胞中的miR-M4-5p表达量对DPT调控尚不明显,但之后逐渐对DPT产生更强抑制作用直至48 h产生显著抑制效果。miR-M4-5p对TMEM230和DCLK1的识别可能更为特异和敏感,足以在转染后24 h发生显著抑制,但同时细胞中也可能存在某些机制来避免这两个基因的大幅度减少,从而维持细胞活性,其中涉及的复杂机制仍有待深入研究。
DPT(皮肤桥蛋白)是一种非胶原细胞外基质成分[29-30],已被证明可调节多种肿瘤中的细胞增殖和侵袭性[31-32]。已知的功能包括与细胞表面受体(整合素α3β1)结合并介导黏附,连接真皮成纤维细胞的细胞表面与细胞外基质(ECM)环境之间的通讯,增加转化生长因子β1(TGF-β1)的活性以及抑制细胞增殖[29, 31]。研究表明,DPT还可以通过TGF-β1、核心蛋白聚糖和纤连蛋白(FN)介导伤口愈合过程中ECM环境之间的通信,并与FN相互作用,增加原纤维形成和细胞黏附[33]。这些数据表明,DPT在肿瘤形成过程中有抑制作用,miR-M4-5p可能通过负调控DPT的表达,从而抑制TGF-β1的活性,从而促进肿瘤的发生。TMEM230也称为C20orf30,编码跨膜蛋白230,目前,有关该基因功能的报道不多,可能与家族性帕金森有关[34-37],也有研究发现,突变的TMEM230可损害突触小泡运输[38]。DCLK1编码双皮质激素样激酶1,主要在中枢神经系统中表达丝氨酸-苏氨酸激酶[39],可以直接或间接调节基于微管的囊泡运输,这对神经元迁移和轴突生长均至关重要[40]。研究表明,DCLK1调节双极有丝分裂纺锤体的形成以及有丝分裂期间从前中期到中期的适当过渡[41]。本研究初步证实,miR-M4-5p可调控TMEM230和DCLK1的表达,这可能与MD病鸡临床表现出的神经性肿瘤有关。综上所述,本研究进一步筛选并鉴定了3个新的miR-M4-5p宿主靶基因,它们可能在MDV-1致病过程中的肿瘤发生和发展具有重要促进作用,但具体的分子调控机制仍有待进一步深入研究。
4 结论通过hybrid-PCR构建miR-M4-5p的宿主靶基因cDNA文库,经基因克隆、PCR鉴定和序列测定分析,筛选获得128个单一候选基因。利用DLRA初筛、突变验证以及qRT-PCR分析,对3′-UTR结合靶点与miR-M4-5p种子序列完全匹配的23个候选基因优先进行了分析和验证,最终鉴定DPT、TMEM230和DCLK1为miR-M4-5p的宿主靶基因,为后续深入研究miR-M4-5p介导的MD肿瘤发生调控机制奠定了重要基础。
| [1] |
SAIF Y M.禽病学[M].苏敬良, 高福, 索勋, 译. 12版.北京: 中国农业出版社, 2012: 129-151.
SAIF Y M. Diseases of poultry[M]. SU J L, GAO F, SUO X, trans. 12th ed. Beijing: China Agriculture Press, 2012: 129-151. (in Chinese) |
| [2] | DAVISON A J, EBERLE R, EHLERS B, et al. The order Herpesvirales[J]. Arch Virol, 2009, 154(1): 171–177. DOI: 10.1007/s00705-008-0278-4 |
| [3] | OSTERRIEDER N, KAMIL J P, SCHUMACHER D, et al. Marek's disease virus:from miasma to model[J]. Nat Rev Microbiol, 2006, 4(4): 283–294. DOI: 10.1038/nrmicro1382 |
| [4] | BARTEL D P. MicroRNAs:genomics, biogenesis, mechanism, and function[J]. Cell, 2004, 166(2): 281–297. |
| [5] | FILIPOWICZ W, BHATTACHARYYA S N, SONENBERG N. Mechanisms of post-transcriptional regulation by microRNAs:are the answers in sight?[J]. Nat Rev Genet, 2008, 9(2): 102–114. DOI: 10.1038/nrg2290 |
| [6] | LEE R C, FEINBAUM R L, AMBROS V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14[J]. Cell, 1993, 75(5): 843–854. DOI: 10.1016/0092-8674(93)90529-Y |
| [7] | PFEFFER S, ZAVOLAN M, GRÄSSER F A, et al. Identification of virus-encoded microRNAs[J]. Science, 2004, 304(5671): 734–736. DOI: 10.1126/science.1096781 |
| [8] | BURNSIDE J, BERNBERG E, ANDERSON A, et al. Marek's disease virus encodes microRNAs that map to meq and the latency-associated transcript[J]. J Virol, 2006, 80(17): 8778–8786. DOI: 10.1128/JVI.00831-06 |
| [9] |
崔治中, 苏帅, 罗俊, 等. 鸡马立克病毒的研究进展[J]. 微生物学通报, 2019, 46(7): 1812–1826.
CUI Z Z, SU S, LUO J, et al. Progress in Marek's disease virus[J]. Microbiology China, 2019, 46(7): 1812–1826. (in Chinese) |
| [10] | ZHAO P, LI X J, TENG M, et al. In vivo expression patterns of microRNAs of Gallid herpesvirus 2(GaHV-2) during the virus life cycle and development of Marek's disease lymphomas[J]. Virus Genes, 2015, 50(2): 245–252. DOI: 10.1007/s11262-015-1167-z |
| [11] |
张雅, 滕蔓, 李会珍, 等. 与马立克病病毒编码的miR-M31-3p互作的宿主靶基因筛选与鉴定[J]. 中国预防兽医学报, 2019, 41(5): 441–448.
ZHANG Y, TENG M, LI H Z, et al. Screening and Identification of host target genes recognized by miR-M31-3p encoded by Marek's disease virus[J]. Chinese Journal of Preventive Veterinary Medicine, 2019, 41(5): 441–448. (in Chinese) |
| [12] |
刘豪丽, 滕蔓, 李会珍, 等. 马立克病病毒编码的miR-M11-5p宿主靶基因的筛选与鉴定[J]. 畜牧兽医学报, 2019, 50(5): 1026–1038.
LIU H L, TENG M, LI H Z, et al. Screening and Identification of host mRNA targets for the viral microRNA miR-M11-5p encoded by Marek's disease virus[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(5): 1026–1038. (in Chinese) |
| [13] |
薛正飞, 滕蔓, 李会珍, 等. 马立克病病毒miR-M4-5p宿主靶基因cDNA文库的构建及鉴定[J]. 畜牧兽医学报, 2018, 49(2): 348–359.
XUE Z F, TENG M, LI H Z, et al. Construction and identification of the cDNA library of host mRNA targets recognized by miR-M4-5p encoded by Marek's disease virus[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(2): 348–359. (in Chinese) |
| [14] | LUO J, SUN A J, TENG M, et al. Expression profiles of microRNAs encoded by the oncogenic Marek's disease virus reveal two distinct expression patterns in vivo during different phases of disease[J]. J Gen Virol, 2011, 92(3): 608–620. DOI: 10.1099/vir.0.024158-0 |
| [15] |
李会珍, 滕蔓, 党露, 等. 马立克氏病病毒编码的miR-M12-5p对鸡HVCN1基因表达的靶向调控[J]. 河南农业科学, 2016, 45(6): 121–125, 129.
LI H Z, TENG M, DANG L, et al. Targeted regulation of host gene HVCN1 by viral microRNA miR-M12-5p encoded by Marek's disease virus[J]. Journal of Henan Agricultural Sciences, 2016, 45(6): 121–125, 129. (in Chinese) |
| [16] | TENG M, YU Z H, ZHAO P, et al. Putative roles as oncogene or tumour suppressor of the Mid-clustered microRNAs in Gallid alphaherpesvirus 2(GaHV2) induced Marek's disease lymphomagenesis[J]. J Gen Virol, 2017, 98(5): 1097–1112. DOI: 10.1099/jgv.0.000786 |
| [17] |
赵朴, 滕蔓, 罗俊, 等. miR-M7-5p靶向调控马立克病病毒原癌基因meq的表达[J]. 畜牧兽医学报, 2014, 45(9): 1518–1525.
ZHAO P, TENG M, LUO J, et al. Marek's disease virus oncogene meq is targeted by the viral microRNA miR-M7-5p[J]. Acta Veterinaria et Zootechnica Sinica, 2014, 45(9): 1518–1525. (in Chinese) |
| [18] |
罗俊, 滕蔓, 樊剑鸣, 等. 马立克氏病病毒编码的microRNA:从基因组学到功能研究[J]. 中国科学:生命科学, 2010, 53(10): 1174–1180.
LUO J, TENG M, FAN J M, et al. Marek's disease virus-encoded microRNAs:genomics, expression and function[J]. Science China Life Sciences, 2010, 53(10): 1174–1180. (in Chinese) |
| [19] | TENG M, YU Z H, SUN A J, et al. The significance of the individual Meq-clustered miRNAs of Marek's disease virus in oncogenesis[J]. J Gen Virol, 2015, 96(3): 637–649. DOI: 10.1099/jgv.0.000013 |
| [20] | ZHAO Y G, YAO Y X, XU H T, et al. A functional microRNA-155 ortholog encoded by the oncogenic Marek's disease virus[J]. J Virol, 2009, 83(1): 489–492. DOI: 10.1128/JVI.01166-08 |
| [21] | ZHUANG G Q, SUN A J, TENG M, et al. A tiny RNA that packs a big punch:the critical role of a viral miR-155 ortholog in lymphomagenesis in Marek's disease[J]. Front Microbiol, 2017, 8: 1169. DOI: 10.3389/fmicb.2017.01169 |
| [22] | YU Z H, TENG M, SUN A J, et al. Virus-encoded miR-155 ortholog is an important potential regulator but not essential for the development of lymphomas induced by very virulent Marek's disease virus[J]. Virology, 2014, 448: 55–64. DOI: 10.1016/j.virol.2013.09.017 |
| [23] | XU H T, YAO Y X, ZHAO Y G, et al. Analysis of the expression profiles of Marek's disease virus-encoded microRNAs by real-time quantitative PCR[J]. J Virol Methods, 2008, 149(2): 201–208. DOI: 10.1016/j.jviromet.2008.02.005 |
| [24] | ZHANG Y Y, TANG N, LUO J, et al. Marek's disease virus-encoded microRNA 155 ortholog critical for the induction of lymphomas is not essential for the proliferation of transformed cell lines[J]. J Virol, 2019, 93(17): e00713–19. |
| [25] | CHI J Q, TENG M, YU Z H, et al. Marek's disease virus-encoded analog of microRNA-155 activates the oncogene c-Myc by targeting LTBP1 and suppressing the TGF-β signaling pathway[J]. Virology, 2015, 476: 72–84. DOI: 10.1016/j.virol.2014.11.027 |
| [26] | HUANG Y J, QI Y, RUAN Q, et al. A rapid method to screen putative mRNA targets of any known microRNA[J]. Virol J, 2011, 8: 8. DOI: 10.1186/1743-422X-8-8 |
| [27] | ZHANG G J, XIAO H X, TIAN H P, et al. Upregulation of microRNA-155 promotes the migration and invasion of colorectal cancer cells through the regulation of claudin-1 expression[J]. Int J Mol Med, 2013, 31(6): 1375–1380. DOI: 10.3892/ijmm.2013.1348 |
| [28] | DANG L, TENG M, LI H Z, et al. Marek's disease virus type 1 encoded analog of miR-155 promotes proliferation of chicken embryo fibroblast and DF-1 cells by targeting hnRNPAB[J]. Vet Microbiol, 2017, 207: 210–218. DOI: 10.1016/j.vetmic.2017.06.015 |
| [29] | LIU X Y, MENG L K, SHI Q, et al. Dermatopontin promotes adhesion, spreading and migration of cardiac fibroblasts in vitro[J]. Matrix Biol, 2013, 32(1): 23–31. |
| [30] | KIM T, AHMAD K, SHAIKH S, et al. Dermatopontin in skeletal muscle extracellular matrix regulates myogenesis[J]. Cells, 2019, 8(4): 332. DOI: 10.3390/cells8040332 |
| [31] | GUO Y, LI H, GUAN H Y, et al. Dermatopontin inhibits papillary thyroid cancer cell proliferation through MYC repression[J]. Mol Cell Endocrinol, 2019, 480: 122–132. DOI: 10.1016/j.mce.2018.10.021 |
| [32] | PATEL A, MALIK M, BRITTEN J, et al. Mifepristone inhibits extracellular matrix formation in uterine leiomyoma[J]. Fertil Steril, 2016, 105(4): 1102–1110. DOI: 10.1016/j.fertnstert.2015.12.021 |
| [33] | KATO A, OKAMOTO O, ISHIKAWA K, et al. Dermatopontin interacts with fibronectin, promotes fibronectin fibril formation, and enhances cell adhesion[J]. J Biol Chem, 2011, 286(17): 14861–14869. DOI: 10.1074/jbc.M110.179762 |
| [34] | KALINDERI K, BOSTANTJOPOULOU S, FIDANI L. The genetic background of Parkinson's disease:current progress and future prospects[J]. Acta Neurol Scand, 2016, 134(5): 314–326. DOI: 10.1111/ane.12563 |
| [35] | IBANEZ L, DUBE U, BUDDE J, et al. TMEM230 in Parkinson's disease[J]. Neurobiol Aging, 2017, 56: 212. e1–212. e3. DOI: 10.1016/j.neurobiolaging.2017.03.014 |
| [36] | WEINTRAUB D, PAPAY K, SIDEROWF A. Screening for impulse control symptoms in patients with de novo Parkinson disease:a case-control study[J]. Neurology, 2013, 80(2): 176–180. DOI: 10.1212/WNL.0b013e31827b915c |
| [37] | BAUMANN H, WOLFF S, MUNCHAU A, et al. Evaluating the role of TMEM230 variants in Parkinson's disease[J]. Parkinsonism Relat Disord, 2017, 35: 100–101. DOI: 10.1016/j.parkreldis.2016.12.015 |
| [38] | DENG H X, SHI Y, YANG Y, et al. Identification of TMEM230 mutations in familial Parkinson's disease[J]. Nat Genet, 2016, 48(7): 733–739. DOI: 10.1038/ng.3589 |
| [39] | SAQUI-SALCES M, KEELEY T M, GROSSE A S, et al. Gastric tuft cells express DCLK1 and are expanded in hyperplasia[J]. Histochem Cell Biol, 2011, 136(2): 191–204. DOI: 10.1007/s00418-011-0831-1 |
| [40] | SUREBAN S M, MAY R, QU D F, et al. DCLK1 regulates pluripotency and angiogenic factors via microRNA-dependent mechanisms in pancreatic cancer[J]. PLoS One, 2013, 8(9): e73940. DOI: 10.1371/journal.pone.0073940 |
| [41] | LIN P T, GLEESON J G, CORBO J C, et al. DCAMKL1 encodes a protein kinase with homology to doublecortin that regulates microtubule polymerization[J]. J Neurosci, 2000, 20(24): 9152–9161. DOI: 10.1523/JNEUROSCI.20-24-09152.2000 |



