2. 上海交通大学系统生物医学研究院, 上海 200240
2. Shanghai Center for Systems Biomedicine, Shanghai Jiao Tong University, Shanghai 200240, China
CRISPR/Cas9技术作为第三代基因编辑技术,其原理主要是:在sgRNA(small guide RNA)的引导下,Cas9酶特异性切割与gRNA(guide RNA)靶向的双连DNA,细胞通过非同源末端链接(NHEJ)或同源重组修复(HDR)实现目的基因编辑的技术。该技术与传统的基因编辑技术相比具有操作简单、效率高的特点。CRISPR/Cas9基因编辑技术自2013年首次应用于哺乳动物细胞以来[1-2],该技术随后在小鼠[3-5]、斑马鱼[6]、猪[7]和猴[8]等不同动物中广泛应用。近来,该技术作为一种基因治疗的潜在手段也被广泛应用于疾病治疗性研究[9-11]。也有研究者将该技术应用于人类早期胚胎的基因编辑[12-13]、甚至诞生出世界首例基因编辑婴儿。然而,基因编辑的效率及精准性一直是限制该技术临床应用的两大核心问题。近期,研究人员针对CRISPR/Cas9技术做了多项改进,包括对Cas9酶进行定点突变[14],优化sgRNA设计策略[15]、模板DNA引入[16],以及单碱基编辑器的开发[17-18]等,上述所有改进其宗旨均在提高CRISPR/Cas9技术的效率及精准性。
传统观点认为,CRISPR/Cas9基因编辑过程中,Cas9酶的切割及切割后细胞的修复过程完全随机,且不可预见[19-20]。而近期多项研究通过大规模的数据分析发现,CRISPR/Cas9基因切割并不完全随机,其存在一定的规律性,不同的靶区特异性决定其切割的偏好性[21-24]。2018年,Chakrabarti等[25]发现,切割后的DNA修复同样遵循特定的规律,不同的靶区序列其基因修复的结果有其特有的偏好性。至此,如何选择合适的sgRNA靶区即成为基因编辑前尤为重要的关键步骤。本课题以具体的Pyk2基因为研究对象,针对其第一外显子区设计3个不同靶区,通过二代靶向测序方法,比较了不同靶区的CRISPR/Cas9基因编辑结局结果,为CRISPR/Cas9基因编辑技术应用过程中的靶区筛选提供一定的技术参考。
1 材料与方法 1.1 材料 1.1.1 试验材料、细胞及载体本试验使用的小鼠均购自于上海交通大学医学院附属第九人民医院实验动物中心,并在该中心饲养。饲养环境维持在恒温22 ℃,相对湿度50%~60%,日照交替12 h白天/12 h黑夜;动物摄食自由、饮水自由、通风良好;感受态细胞为DH5α菌株,购自于北京全式金公司;细胞系采用BALB/c-3T细胞,由上海交通大学实验室赠予;sgRNA质粒载体为pgl3-u6-sgrna-pgk-puromycin (# 51133);Cas9表达载体为pcDNA3.1+(# V790-20),上述载体均购自于Addgene。
1.1.2 试剂与试剂盒本试验分子克隆所需的T4连接酶、内切酶及体外切割所需的Cas9重组蛋白均购自NEB公司;质粒小量抽提试剂盒购自于Xygene公司;质粒中量抽提试剂盒购自Qiagen公司;体外转录试剂盒购自Life Technology公司;细胞培养液DMEM、青链霉素、FBS均购自Invitrogen公司;细胞电转染试剂盒为AmaxaTM SF Cell Line 4D-NucleofectorTM X Kit L,购自Lonza公司;胚胎操作液为HTF,购自Irvine Scientific公司;胚胎培养液为KSOM,购自Millipore公司;DNA抽提试剂为DNeasy Blood & Tissure Kit,购自Qiagen公司;靶向测序构库所需试剂盒购自于诺禾致源科技有限公司。
1.1.3 仪器PCR仪购自Applied Biosystem;细胞电转染仪为4D-NucleofectorTM,购自LOZA公司;低温离心机购自于Eppendorf公司;核酸蛋白定量仪为Nanodrop-8000,购自Thermo Fisher Scientific;二代测序构库质控采用Angilent 2100生物分析仪,购自安捷伦公司。
1.1.4 生物合成及测序服务试验所需各种引物合成(包括sgRNA寡核苷酸引物以及测序相关引物, 表 1)及后续的Sanger测序均由上海生工生物工程有限公司完成;试验涉及的二代测序均由诺禾致源科技有限公司完成。
|
|
表 1 构建sgRNA的引物信息 Table 1 Primers for sgRNA construction |
本试验所用Pyk2相关sgRNA的克隆方法:1)将设计好的sgRNA寡核苷酸引物(表 1)退火后采用T4连接酶连接到pgl3-u6-sgrna-pgk-puromycin载体;2)通过热激法完成感受态细胞DH5a的转化并过夜培养;3)挑取克隆进行测序,并挑选测序正确的克隆进行扩增培养及质粒中量抽提后用于后续的细胞转染试验。
1.2.2 BALB/c -3T3细胞培养及电转染BALB/c-3T3细胞采用含有10% FBS、100 μg·mL-1青链霉素的高糖DMEM培养基培养。细胞转染则采用AmaxaTM SF Cell Line 4D-NucleofectorTM X Kit L(Lonza)在6孔板内完成。转染浓度为:pcDNA3.1(+)-Cas9:2~3 μg;Pyk2-sgRNA:1.5~2 μg;4D-NucleofectorTM设备的转染程序设置为EN-158。转染后细胞采用DNeasy Blood & Tissure Kit(Qiagen)提取基因组用于后续测序分析。
1.2.3 DNA测序文库构建、二代测序及数据分析针对编辑位点进行靶向建库,使用标准的Hi-TOM[26]流程:1)以Pyk2靶点位置为序列中心设计含有搭桥序列(小写字母部分)的特异性引物(Pyk2 target NGS F:5′-ggagtgagtacggtgtgcGT-CCTAGCTGCAGTCTGAGAGGAT-3′;Pyk2 target NGS R:5′-gagttggatgctggatggTTCTTCCCTG-GGTTGAAGCTGTTG-3′)以扩增片段大小为200 bp左右DNA模板,用于第二轮构库使用。PCR的组分和用量:8 μL Nuclease-Free Water,10 μL 2×PCR mix,0.5 μL Primer1(10 μmol·L-1),0.5 μL Primer2(10 μmol·L-1),1 μL gDNA模板;PCR参数:94 ℃预变性2 min; 94 ℃变性30 s,62 ℃退火30 s,72 ℃延伸30 s,重复33个循环; 72 ℃终延伸5 min后,4 ℃保持;2)第二轮PCR建库使用Hi-TOM试剂盒完成,向预混有PCR mix的反应孔中加入第一轮PCR反应液1 μL进行PCR,PCR结束后再次取5 μL进行琼脂糖电泳检测PCR产物;确定条带正确后将所有样品混合在一起,吸取200 μL进行琼脂糖电泳,并切胶回收纯化,将纯化产物送北京诺禾致源科技股份有限公司测序,二代测序的结果使用Cas-analyzer[27]在线分析平台进行分析。
1.2.4 sgRNA体外转录及胚胎注射以上述Pgl3-u6-sgrna-pgk-puromycin为模板,设计含有T7启动子的引物F: 5′-TAATACGACTCACTATAGGGGG-CACTTTACGCCGGCCTGA-3′,R: 5′-GTTTTAGA-GCTAGAAATAG-3′进行PCR扩增,然后以扩增产物为模板,利用MEGA shortscript T7 kit体外转录试剂盒进行体外转录生产Pyk2 site1对应的sgRNA;采用类似的方法将线性化的pcDNA3.1-Cas9质粒,利用mMESSAGE mMACHINE T7 ULTRA kit进行体外转录生产Cas9 mRNA;将上述2种RNA分别以100 ng·μL-1溶于RNase free的TE溶液中,注射到双原核期胚胎。注射后,胚胎移植到假孕受体鼠中,待产出后对子鼠基因组DNA通过引物F:5′-GGATGTGGCATGTGGCTTGCAAGAG-3′;R:5′-TACCTGGATCTCTGTCTGCACTGTG-3′扩增后进行Sanger测序。
1.2.5 sgRNA体外活性分析Pyk2不同sgRNA活性采用Cas9重组蛋白通过体外切割试验完成:首先,将体外转录的sgRNA纯化后稀释至300 nmol·L-1,Pyk2 PCR底物模板稀释至30 nmol·L-1,同时将Cas9酶用Diluent B(NEB#B8002S)稀释到1 μmol·L-1,按产品说明书配制反应体系;然后在PCR仪中37 ℃孵育15 min,进行琼脂糖凝胶电泳并拍照。凝胶电泳图采用Image J进行灰度分析,比较不同组别的切割效率。
2 结果 2.1 Pyk2相关sgRNA的设计及构建本试验针对Pyk2基因的第一个外显子区域选择3个含有NGG序列的靶区,合成含有ACCG -N20和AAAC-N20的特异性引物序列(表 1),通过退火、链接、克隆筛选、摇菌、质粒抽提后进行Sanger测序,结果发现3个Pyk2相关的sgRNA(Pyk2 site1 sgRNA、Pyk2 site2 sgRNA、Pyk2 site3 sgRNA)均序列正确(图 1),可满足后续试验的要求。
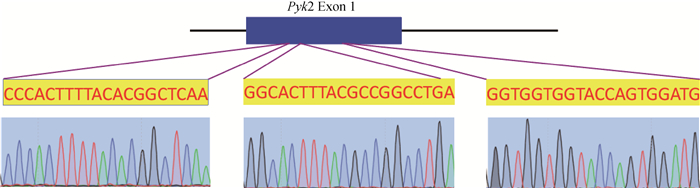
|
图 1 Pyk2相关sgRNA设计及构建 Fig. 1 Pyk2 related sgRNA design and construction |
将上述构建好的3种Pyk2相关的sgRNA, 分别与pcDNA3.1-Cas9质粒一同通过电转染技术转入BALB/C-3T3细胞,通过二代靶向测序技术对不同sgRNA对应的基因编辑测序数据分析后发现(表 2),其总体基因编辑效率(indels)在3个不同的靶区存在明显差异(site1:32.0%;site2:7.9%;site3:69.5%);进一步将上述数据按照随机插入(insertion)和随机缺失(deletion)分开统计发现,各靶区的基因编辑结局均以deletion为主(Pyk2 site1:26.3% vs 5.7%;Pyk2 site2:7.8% vs 0.1%;Pyk2 site3:66.5% vs 3.0%)。对于同一靶区,其sgRNA在不同重复间的基因编辑(indels)效率相对稳定,显示出较好的重复性(site1:31.8% vs 32.3%;site2:7.4% vs 8.4%;site3:71.3% vs 67.8%)。
|
|
表 2 Pyk2不同靶区对Pyk2基因编辑效率的影响 Table 2 Effect of different target regions of Pyk2 on the gene editing efficiency |
通过对上述基因编辑后测序结果的进一步分析发现,不同靶区对应sgRNA的基因编辑模式均显示出序列的特异性,在基因缺失(deletion)方面(图 2),Pyk2 site1发生缺失的位点较为集中,主要发生在PAM区上游的第4~5个碱基,且不同重复之间的结果也显示出较好的保守性(图 2A、2D);而Pyk2 site2靶区对应的sgRNA编辑后的修复则显示出较多类型(图 2B、2E),具有明显的不可预测性;而Pyk2 site3靶区对应的sgRNA切割后基因修复的模式显示(图 2C、2F),该靶区的基因编辑综合效果介于site1和site2之间。
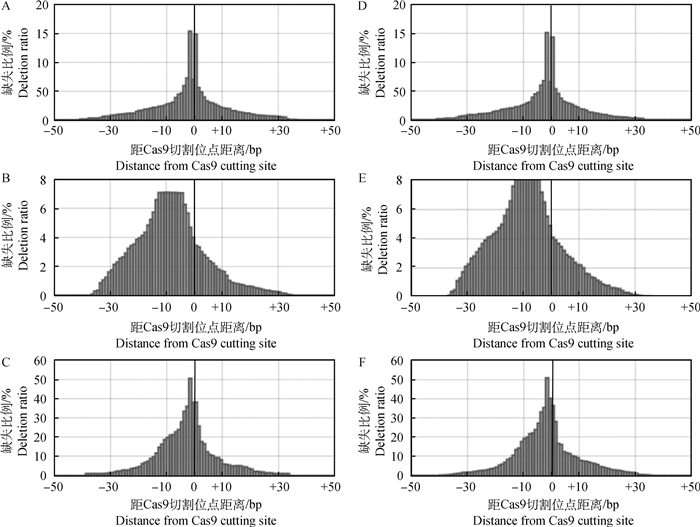
|
A、D. Pyk2 site1靶区对应sgRNA在第一重复和第二重复的随机缺失模式图;B、E. Pyk2 site2靶区对应sgRNA在第一重复和第二重复的随机缺失模式图;C、F. Pyk2 site3靶区对应sgRNA在第一重复和第二重复的随机缺失模式图 A, D. Deletion pattern of Pyk2 site1 sgRNA in 2 experimental replicates; B, E. Deletion pattern of Pyk2 site2 sgRNA in 2 experimental replicates; C, F. Deletion pattern of Pyk2 site3 sgRNA in 2 experimental replicates 图 2 不同靶区对Pyk2基因编辑后碱基缺失的影响 Fig. 2 Effect of different target regions on the base deletion efficiency after Pyk2 gene editing |
在基因插入方面,Pyk2 site1发生插入的位点同样较为集中,主要发生在PAM区上游的第4~5个碱基,且不同重复间显示出较好的保守性(图 3A、3D);而Pyk2 site2靶区对应的sgRNA编辑后的碱基插入则显示出多种类型(图 3B、3E),具有明显的随机性。此外,与碱基的缺失类似,Pyk2 site3靶区作为基因编辑效率最高的靶区,其对应sgRNA切割后碱基插入精准性介于site1与site2之间(图 3C、3F)。上述结果说明,对于CRISPR/Cas9基因编辑所选靶区,基因编辑的精确性与基因编辑效率无关,即基因编辑效率较高的靶区并非是最理想的靶区。
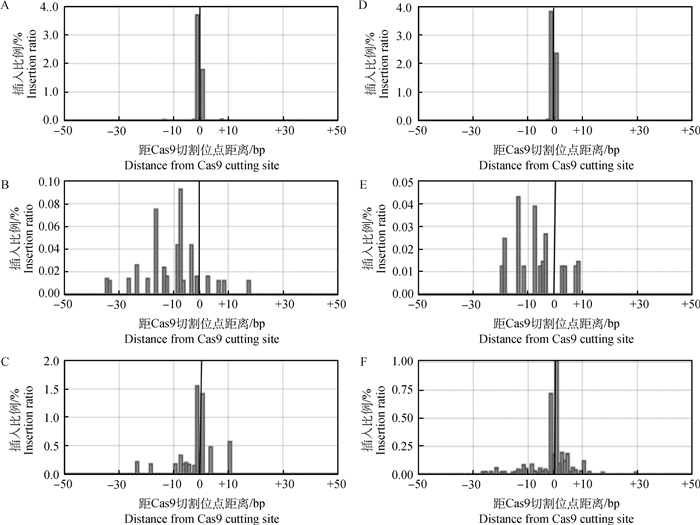
|
A、D. Pyk2 site1靶区对应sgRNA在第一重复和第二重复的随机插入模式图;B、E. Pyk2 site2靶区对应sgRNA在第一重复和第二重复的随机插入模式图;C、F. Pyk2 site3靶区对应sgRNA在第一重复和第二重复的随机插入模式图 A, D. Insertion pattern of Pyk2 site1 sgRNA in 2 experimental replicates; B, E. Insertion pattern of Pyk2 site2 sgRNA in 2 experimental replicates; C, F. Insertion pattern of Pyk2 site3 sgRNA in 2 experimental replicates 图 3 不同靶区对Pyk2基因编辑后碱基插入效率的影响 Fig. 3 Effect of different target regions on the base insertion efficiency after Pyk2 gene editing |
针对上述Pyk2 site1靶区,本试验通过RNA体外转录、原核注射等进行基因编辑动物生产(图 4A),结果表明,在出生的5只小鼠中鉴定出2种不同的基因编辑(indels)结果,分别为插入1 bp(Pyk2+1 bp) (图 4B)和缺失8 bp (Pyk2-8 bp) (图 4C),而该靶区在细胞学试验中同样表明,插入1 bp(图 4D)或者缺失10 bp以下(图 4E)占多数。因此,基因编辑动物中的结果与Pyk2 site1靶点在上述细胞中显示的基因编辑偏好性结果高度一致。这一结果表明,对于同一靶区,其对应sgRNA基因编辑的偏好性不受细胞类型的限制,在细胞中筛出的基因编辑靶区可应用于动物水平的基因编辑应用。
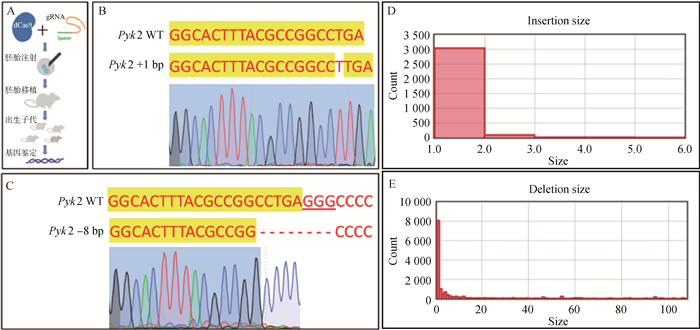
|
A.基因编辑小鼠生产流程图;B.基因编辑小鼠Pyk2 +1 bp基因型测序图;C.基因编辑小鼠Pyk2-8 bp基因型测序图;D、E. Pyk2 site1靶区在细胞基因编辑后的碱基插入和碱基缺失模式图 A. Gene editing mouse production flow chart; B. Pyk2 +1 bp genotype sequencing map; C. Pyk2-8 bp genotype sequencing map; D, E. The pattern of insertion and deletion in the target region of Pyk2 site1 after gene editing 图 4 不同细胞类型对基因编辑偏好性的影响 Fig. 4 Effect of different cell types on the gene editing preference |
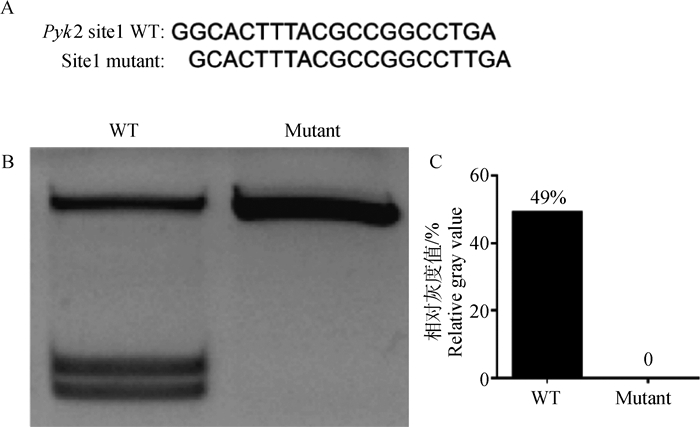
利用上述获得的+1 bp基因型小鼠基因组为模板,以Pyk2 site1原靶区设计1个+1 bp sgRNA,与site1靶区序列仅相差一个碱基(图 5A),通过Cas9酶体外切割分析发现(图 5B、5C),野生型Pyk2切割效率约为49.2%,而单碱基的插入改变可使其切割效率降低为0,这说明对于基因编辑效率较高的靶区,即使单个碱基发生改变,也可对其基因编辑的效率产生显著影响。
|
A.Pyk2 site1靶区及其单碱基插入序列信息图;B. Cas9体外切割琼脂糖凝胶电泳图;C.凝胶电泳图灰度分析柱形图 A. Graph of Pyk2 site1 target region and its single base insertion sequence information; B. Agarose gel electrophoresis of Cas9 in vitro dissected; C. Gel electrophoresis map grayscale analysis histogram 图 5 靶区单碱基改变对Cas9酶剪切效率的影响 Fig. 5 Effect of a single base change in target region on the cut efficiency of Cas9 |
基因编辑的靶区选择是基因编辑成败的关键。传统观点认为,基因编辑的结局是随机的,且不可预测。而近期研究发现,在基因编辑的过程中,有些靶区效率较高,容易发生精准编辑,但也有些靶区其切割效率较低,还表现出严重的脱靶效应[28]。因此,如何建立一种方法,进行靶区的筛查对于基因编辑的可预测性具有重要意义。为此,本研究以具体基因-Pyk2为研究对象,建立了一种基因编辑靶区的筛选办法,采用二代靶向测序方法发现该基因的3个靶区在基因编辑效率和偏好性均存在显著差异,且这种偏好性不受细胞类型的限制。
首先,本研究参考前人的方法快速实现Pyk2 sgRNA的载体构建[29],并通过电转染细胞后的DNA测序分析发现,不同的靶区在基因编辑效率及偏好性方面均存在较大差异;而对于同一靶区,不论其基因编辑的效率还是偏好性方面均具有较好的可重复性,这说明不同的靶区在基因切割后的修复并非完全随机,而是具有一定规律性的。此外,本研究还发现,Pyk2 site3靶区作为基因编辑效率最高的靶区,其对应sgRNA切割后的基因编辑精准性并不理想。上述结果说明,对于CRISPR/Cas9基因编辑所选靶区,基因编辑的精确性与基因编辑效率无关,即基因编辑效率较高的靶区并非是最理想的靶区。
众所周知,CRISPR-Cas9基因编辑过程分两步完成,首先是在sgRNA的指导下,完成DNA双连的断裂,然后在细胞DNA修复机制的作用下完成DNA的修复工作[30]。对于同一靶区,不同细胞类型之间其参与DNA修复的相关基因表达存在差异,但其不同细胞中DNA修复机制是否存在差异还不得而知。为此,本研究将上述Pyk2 site1靶区基因编辑动物生产进行验证,结果表明,基因编辑动物中的结果与Pyk2 site1靶点在上述细胞中显示的基因编辑偏好性结果高度一致。这说明对于同一靶区,其基因编辑的偏好性不受细胞类型的限制,在细胞中筛出的基因编辑靶区可应用于动物水平的基因编辑。
然而,究竟什么样的靶区其基因编辑的结果具有可预见性,该类靶区有何特点。本研究发现,当上述Pyk2 site1靶区的Cas9切割位点插入单个碱基后,Cas9酶体外切割分析显示单个碱基的改变对靶区效率即可产生显著影响。
4 结论综上所述,靶区选择对CRISPR/Cas9基因编辑的结局有显著影响,不同靶区切割效率不同,其基因修复的偏好性也存在较大差异;对于CRISPR/Cas9基因编辑所选靶区,基因编辑的精确性与基因编辑效率无关,即基因编辑效率较高的靶区并非是最理想的靶区;对于同一靶区,其基因编辑的偏好性不依赖于细胞类型,在细胞中筛出的基因编辑靶区可应用于动物水平的基因编辑;靶区单碱基的改变可对基因编辑的结局产生显著影响。
| [1] | CONG L, RAN F A, COX D, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339(6121): 819–823. DOI: 10.1126/science.1231143 |
| [2] | MALI P, YANG L H, ESVELT K M, et al. RNA-guided human genome engineering via Cas9[J]. Science, 2013, 339(6121): 823–826. DOI: 10.1126/science.1232033 |
| [3] | WANG H Y, YANG H, SHIVALILA C S, et al. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering[J]. Cell, 2013, 153(4): 910–918. DOI: 10.1016/j.cell.2013.04.025 |
| [4] | YANG H, WANG H Y, SHIVALILA C S, et al. One-step generation of mice carrying reporter and conditional alleles by CRISPR/Cas-mediated genome engineering[J]. Cell, 2013, 154(6): 1370–1379. DOI: 10.1016/j.cell.2013.08.022 |
| [5] |
舒磊磊, 甲芝莲, 吴勇浒, 等. CRISPR/Cas9技术的应用性研究[J]. 畜牧兽医学报, 2016, 47(7): 1316–1323.
SHU L L, JIA Z L, WU Y H, et al. The application research of CRISPR/Cas9[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(7): 1316–1323. (in Chinese) |
| [6] | CHANG N N, SUN C H, GAO L, et al. Genome editing with RNA-guided Cas9 nuclease in zebrafish embryos[J]. Cell Res, 2013, 23(4): 465–472. DOI: 10.1038/cr.2013.45 |
| [7] | NIU D, WEI H J, LIN L, et al. Inactivation of porcine endogenous retrovirus in pigs using CRISPR-Cas9[J]. Science, 2017, 357(6357): 1303–1307. DOI: 10.1126/science.aan4187 |
| [8] | YAO X, LIU Z, WANG X, et al. Generation of knock-in cynomolgus monkey via CRISPR/Cas9 editing[J]. Cell Res, 2018, 28(3): 379–382. |
| [9] | AMOASⅡ L, HILDYARD J C W, LI H, et al. Gene editing restores dystrophin expression in a canine model of Duchenne muscular dystrophy[J]. Science, 2018, 362(6410): 86–91. DOI: 10.1126/science.aau1549 |
| [10] | NELSON C E, WU Y Y, GEMBERLING M P, et al. Long-term evaluation of AAV-CRISPR genome editing for Duchenne muscular dystrophy[J]. Nat Med, 2019, 25(3): 427–432. DOI: 10.1038/s41591-019-0344-3 |
| [11] | BEYRET E, LIAO H K, YAMAMOTO M, et al. Single-dose CRISPR-Cas9 therapy extends lifespan of mice with Hutchinson-Gilford progeria syndrome[J]. Nat Med, 2019, 25(3): 419–422. DOI: 10.1038/s41591-019-0343-4 |
| [12] | LEDFORD H. CRISPR fixes disease gene in viable humanembryos[J]. Nature, 2017, 548(7665): 13–14. DOI: 10.1038/nature.2017.22382 |
| [13] | MA H, MARTI-GUTIERREZ N, PARK S W, et al. Correction of a pathogenic gene mutation in human embryos[J]. Nature, 2017, 548(7668): 413–419. DOI: 10.1038/nature23305 |
| [14] | ADLI M. The CRISPR tool kit for genome editing and beyond[J]. Nat Commun, 2018, 9(1): 1911. DOI: 10.1038/s41467-018-04252-2 |
| [15] | DOENCH J G, FUSI N, SULLENDER M, et al. Optimized sgRNA design to maximize activity and minimize off-target effects of CRISPR-Cas9[J]. Nat Biotechnol, 2016, 34(2): 184–191. |
| [16] | PAQUET D, KWART D, CHEN A, et al. Efficient introduction of specific homozygous and heterozygous mutations using CRISPR/Cas9[J]. Nature, 2016, 533(7601): 125–129. DOI: 10.1038/nature17664 |
| [17] | KOMOR A C, KIM Y B, PACKER M S, et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage[J]. Nature, 2016, 533(7603): 420–424. DOI: 10.1038/nature17946 |
| [18] | GAUDELLI N M, KOMOR A C, REES H A, et al. Publisher correction:Programmable base editing of A·T to G·C in genomic DNA without DNA cleavage[J]. Nature, 2017, 551(7681): 464–471. DOI: 10.1038/nature24644 |
| [19] | ZHANG F. CRISPR/Cas9:prospects and challenges[J]. Hum Gene Ther, 2015, 26(7): 409–410. DOI: 10.1089/hum.2015.29002.fzh |
| [20] | WILES M V, QIN W N, CHENG A W, et al. CRISPR-Cas9-mediated genome editing and guide RNA design[J]. Mamm Genome, 2015, 26(9-10): 501–510. DOI: 10.1007/s00335-015-9565-z |
| [21] | VAN OVERBEEK M, CAPURSO D, CARTER M M, et al. DNA repair profiling reveals nonrandom outcomes at Cas9-mediated breaks[J]. Mol Cell, 2016, 63(4): 633–646. DOI: 10.1016/j.molcel.2016.06.037 |
| [22] | SHEN M W, ARBAB M, HSU J Y, et al. Predictable and precise template-free CRISPR editing of pathogenic variants[J]. Nature, 2018, 563(7733): 646–651. DOI: 10.1038/s41586-018-0686-x |
| [23] | SHOU J, LI J H, LIU Y B, et al. Precise and predictable CRISPR chromosomal rearrangements reveal principles of Cas9-mediated nucleotide insertion[J]. Mol Cell, 2018, 71(4): 498–509. e4. DOI: 10.1016/j.molcel.2018.06.021 |
| [24] | ALLEN F, CREPALDI L, ALSINET C, et al. Predicting the mutations generated by repair of Cas9-induced double-strand breaks[J]. Nat Biotechnol, 2019, 37(1): 64–72. |
| [25] | CHAKRABARTI A M, HENSER-BROWNHILL T, MONSERRAT J, et al. Target-specific precision of CRISPR-mediated genome editing[J]. Mol Cell, 2019, 73(4): 699–713. e6. DOI: 10.1016/j.molcel.2018.11.031 |
| [26] | LIU Q, WANG C, JIAO X Z, et al. Hi-TOM:a platform for high-throughput tracking of mutations induced by CRISPR/Cas systems[J]. Sci China Life Sci, 2019, 62(1): 1–7. DOI: 10.1007/s11427-018-9402-9 |
| [27] | PARK J, LIM K, KIM J S, et al. Cas-analyzer:an online tool for assessing genome editing results using NGS data[J]. Bioinformatics, 2017, 33(2): 286–288. |
| [28] | TSAI S Q, ZHENG Z L, NGUYEN N T, et al. GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases[J]. Nat Biotechnol, 2015, 33(2): 187–197. |
| [29] | SHEN B, ZHANG W S, ZHANG J, et al. Efficient genome modification by CRISPR-Cas9 nickase with minimal off-target effects[J]. Nat Methods, 2014, 11(4): 399–402. DOI: 10.1038/nmeth.2857 |
| [30] | HILLE F, RICHTER H, WONG S P, et al. The biology of CRISPR-Cas:backward and forward[J]. Cell, 2018, 172(6): 1239–1259. DOI: 10.1016/j.cell.2017.11.032 |



