2. 西藏农牧学院西藏高原动物疫病研究自治区高校重点实验室, 林芝 860000;
3. 西藏自治区兽医生物药品制造厂, 拉萨 850000;
4. 中国农业大学动物科技学院, 北京 100193
2. Provincial Key Laboratory of Tibet Plateau Animal Epidemic Disease Research, Tibet Agriculture & Animal Husbandry University, Linzhi 860000, China;
3. Tibet Veterinary Biological Pharmaceutical Factory, Lhasa 850000, China;
4. College of Animal Science and Technology, China Agricultural University, Beijing 100193, China
天然抗性相关巨噬细胞蛋白(natural-resistance-associated marophage protein,Nramp)家族,又称为溶质载体家族11成员(solute carrier 11,SLC11)[1]。随着近年对Nramp基因的深入研究,发现该基因广泛存在于细菌、真菌、动植物中,其编码的天然抗性相关巨噬细胞蛋白(Nramp),在所有生命体中具有高度保守的序列,主要分为Nramp1(SLC11A1)和Nramp2(SLC11A2)两种,Nramp1基因主要在吞噬细胞和初级溶酶体中特异性表达[2],Nramp2基因在很多组织广泛表达[3]。目前对Nramp基因主要集中于Nramp1(SLC11A1)的相关研究上,Nramp1基因长度为12 kb,包含15个外显子,编码由550个氨基酸组成的的蛋白质,具有10~12个预测的跨膜结构域,位于吞噬体膜,参与二价阳离子的转运。在细胞感染期间,Nramp1蛋白转运必需元素(如Mn2+、Fe2等),对于病原微生物的存活至关重要,可饥饿并限制它们的生长[4]。很多研究已表明,Nramp基因在先天性免疫调节中具有重要作用,在红绸鱼受到致病弧菌侵袭时,Nramp基因mRNA的表达量显著升高[5],Nramp1在小鼠抵抗弗朗西斯菌中起着重要作用,且对沙门氏菌、结核分支杆菌及利什曼原虫也有着显著抗性[6],在团头鲂受脂多糖的刺激下,Nramp基因mRNA水平上调[7],在牛Nramp1基因的相关研究中也发现,使用脂多糖刺激嗜中性粒细胞可产生相应的转录产物[8],但迄今为止,鲜见有关于牦牛Nramp1基因及其mRNA表达情况的研究报道。
微小核糖核酸(micro RNA,miRNA)是一类真核生物内源性非编码RNA,大小一般是18~25个核苷酸,其通过靶基因转录水平的调控,从而间接影响蛋白质的翻译[9]。在哺乳动物中,miRNA通过互补碱基配对与靶mRNA中不完全匹配的结合序列结合,指导miRISC活性,导致翻译抑制,加速mRNA降解[10]。越来越多的科学研究和试验证明,miRNA与人类、动物疾病的发生发展有着密切的联系[11],如参与人系统性红斑狼疮[12]和人兽共患病流行性乙型脑炎[13]的免疫调节。在无脊椎动物中具有抗病毒先天免疫[14]以及抗寄生虫的重要作用[15]。在猪上发现,其参与猪圆环病毒2型[16]和猪甲型流感[17]免疫调控,在鸡上发现多种miRNAs参与禽流感病毒[18]、传染性法氏囊病毒的易感和抵抗作用[19],在对牛的研究也发现多种miRNAs参与免疫调控作用,如:miR-101可以抑制牛流行性热病毒在体内的复制[20];miR-29b能显著性降低牛病毒性腹泻病毒的复制水平及其感染诱导的细胞自噬等[21]。目前对牦牛相关miRNAs的研究主要集中于生殖细胞的增殖、分化及调控作用研究[22-26],但是关于miRNAs在牦牛免疫调控方面的研究未见报道。
本试验拟研究牦牛Nramp1基因mRNA组织表达谱,并探索研究可能靶向Nramp1基因的miRNAs,为研究牦牛宿主免疫的可能分子调控机制提供一定的理论基础。
1 材料与方法 1.1 试剂和材料mRNA反转录试剂盒购自Zomanbio公司,Trizol试剂购自Invitrogen公司,miRNA反转录试剂盒购自HaiGene公司,PCR Premix TaqTM、DEPC、DNA marker、Sample Protector for RNA均购自TaKaRa公司,其他试剂均为国产或进口分析纯。
选3头健康的2岁雌性牦牛,屠宰后立即采取牦牛肝、脾、肺、肾、后腿骨骼肌、颌下淋巴结、卵巢、小肠、大肠、肠系淋巴结10种不同组织,立刻置于含Sample Protector for RNA溶液的冻存管内,置于-80 ℃冰箱保存,以便提取总的RNA。
1.2 组织RNA提取首先取50 mg组织样置于无RNA酶的离心管中,随即加入1 mL Trizol并用Bullet Blender进行组织匀浆,然后用Trizol法提取总RNA,使用琼脂糖电泳检测其质量,利用Thermo公司的NanoDrop 2000超微量分光光度计检测RNA浓度和纯度。
1.3 反转录与RT-PCR反应 1.3.1 mRNA反转录和RT-PCR反应根据反转录试剂盒EX 5×RT Master MIX(Zomanbio公司)说明进行mRNA反转录。以20 μL的体系进行反转录,操作时置于冰盒上,向离心管中加入:总RNA 2 μL、EX 5×RT Master MIX 2 μL、RNase-Free ddH2O 16 μL;反应条件:25 ℃ 10 min,42 ℃ 50 min,85 ℃ 5 min,RT产物-20 ℃保存备用。
从GenBank中检索到Nramp1基因和β-actin基因参考序列信息,用Primer premier 5.0软件设计引物(表 1),由上海赛百盛基因技术有限公司合成。PCR反应体系:Premix TaqTM 4 μL、上、下游引物(10 μmol·L-1)各0.4 μL、RT产物0.4 μL、ddH2O 4.8 μL;滴加少许石蜡油,以防止挥发。PCR扩增条件:94 ℃预变性5 min;94 ℃ 45 s,60 ℃ 45 s,72 ℃ 45 s,扩增40个循环;72 ℃延伸10 min,4 ℃保存。PCR产物用2%琼脂糖凝胶电泳分离,紫外灯下割取目的片段,用V-gene DNA凝胶回收试剂盒回收纯化。纯化的PCR产物送上海英骏生物技术有限公司做克隆测序。组织表达谱RT-PCR检测产物用8%聚丙烯酰胺凝胶(Acr:Bis=29:1)电泳分离,银染后用凝胶成像系统(美国Bio-Rad)照相。
|
|
表 1 PCR引物参数 Table 1 Primer parameters of PCR |
运用加PloyA尾法设计Ploy(T)引物序列5′-GCGA-GCACAGAATTAATACGACTCACTATAGG(T)-12V(A\G\C) N-3′,再根据HaiGene公司反转录试剂盒One Step miRNA cDNA Synthesis Kit说明进行,具体操作:反转录体系为20 μL,在冰上操作,在离心管中加入总RNA 1 μL、4×One step miRNA RT solution 5 μL、Ploy(T)2 μL、RNase-Free ddH2O 12 μL;反应条件:37 ℃ 60 min,95 ℃ 5 min,4 ℃ 5 min,RT产物-20 ℃保存备用。
设计各特异miRNA引物(表 1),由上海赛百盛基因技术有限公司合成。PCR反应体系:TaKaRa Primer TaqTM 4 μL、正、反引物(10 μmol·L-1)各0.4 μL、反转录产物0.4 μL、ddH2O 4.8 μL;滴加少许石蜡油,以防止挥发。PCR扩增条件:94 ℃预变性5 min;94 ℃ 45 s,退火45 s,72 ℃ 45 s,40个循环;72 ℃延伸10 min,4 ℃保存,8%的聚丙烯酰胺凝胶电泳检测。
1.4 牦牛Nramp1基因靶标miRNA预测运用TargentScan(http://www.targetscan.org)和miRBase(http://www.mirbase.org)在线预测软件,参数为系统默认设置,以牛Nramp1基因作为靶基因,预测靶定此基因的miRNAs。
1.5 数据统计分析Nramp1基因和miRNAs表达量在各组织中的差异用SPSS 19.0软件包中One-way ANOVA进行分析, 试验结果均表示为“x±Sd”。
2 结果 2.1 牦牛Nramp1基因的组织表达谱分析以牦牛组织总RNA混合物为模板,用Primer premier 5.0所设计的Nramp1和β-actin基因引物分别进行RT-PCR扩增,结果见图 1。通过测序发现,Nramp1的扩增产物为179 bp,β-actin基因的扩增产物为148 bp。用DNAMAN 5.2.2对测序结果与引物设计源序列进行同源性比较,结果发现,Nramp1和β-actin扩增片段与引物设计源序列同源性分别为100%,表明PCR扩增片段为牦牛特异的Nramp1和β-actin基因cDNA片段。
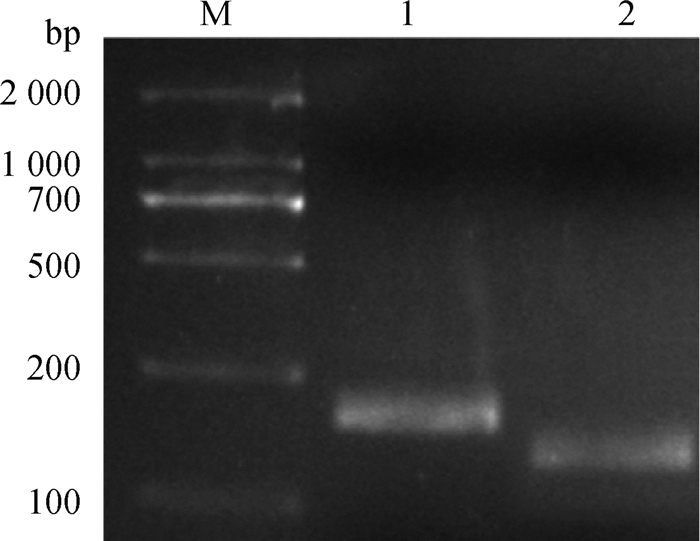
|
M. DNA相对分子质量标准;1.牦牛Nramp1基因扩增片段;2.牦牛β-actin基因扩增片段 M. DL2000 DNA marker; 1. Fragments amplified of Nramp1 in yak; 2. Fragments amplified of β-actin in yak 图 1 牦牛Nramp1和β-actin基因的RT-PCR电泳图 Fig. 1 RT-PCR electropherogram of yak Nramp1 and β-actin genes |
利用RT-PCR对牦牛肝、脾、肺、肾、后腿骨骼肌、卵巢、小肠、颌下淋巴结、大肠、肠系淋巴结10种组织中Nramp1和内参基因β-actin片段进行PCR扩增,扩增产物用8%聚丙烯酰胺凝胶电泳进行检测,结果见图 2。从图 2可以看出,Nramp1基因在所检测的牦牛各组织中均有表达。再利用Quantity One V4.52软件对Nramp1和β-actin的目的条带进行灰度体积分析,得到Nramp1基因mRNA在各种组织中的相对表达量,试验重复3次,结果见图 3。从图 3中可以看出,Nramp1基因mRNA在脾、肺、后腿骨骼肌、卵巢及颌下淋巴结5种组织中的表达量无显著差异(P>0.05);其中在脾、肺及颌下淋巴结组织中的表达量极显著高于在肝、肾、大肠、肠系淋巴结组织中的表达量(P<0.01),显著高于在小肠组织中的表达量(P<0.05)。
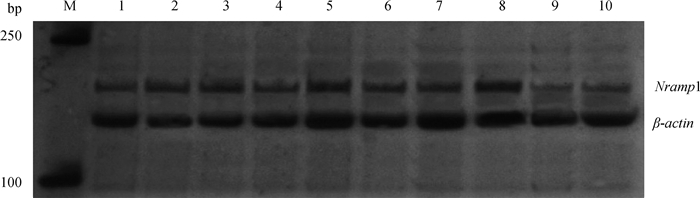
|
M. DNA相对分子质量标准;1.肝;2.脾;3.肺;4.肾;5.后腿骨骼肌;6.卵巢;7.小肠;8.颌下淋巴结;9.大肠;10.肠系淋巴结。图 4同 M. DL2000 DNA marker; 1.Liver; 2.Spleen; 3.Lung; 4.Kidney; 5. Hind leg skeletal muscle; 6. Ovary; 7.Small intestine; 8. Submandibular lymph node; 9. Large intestine; 10. Intestinal lymph node. The same as figure 4 图 2 牦牛β-actin与Nramp1基因的组织表达谱 Fig. 2 The tissue expression profiles of β-actin and Nramp1 in yak |
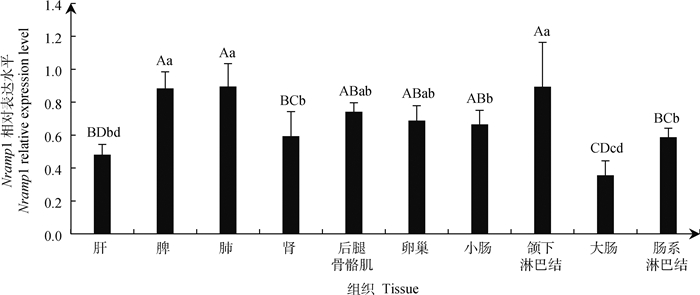
|
不同小写字母表示差异显著(P<0.05),不同大写字母差异极显著(P<0.01),图 5同 Different superscript lowercase and capital letters indicate significant difference(P < 0.05) and extremely significant difference(P < 0.01) respectively, the same as figure 5 图 3 牦牛Nramp1基因mRNA在不同组织的相对表达量 Fig. 3 The expression levels of Nramp1 mRNA in various tissues of yak |
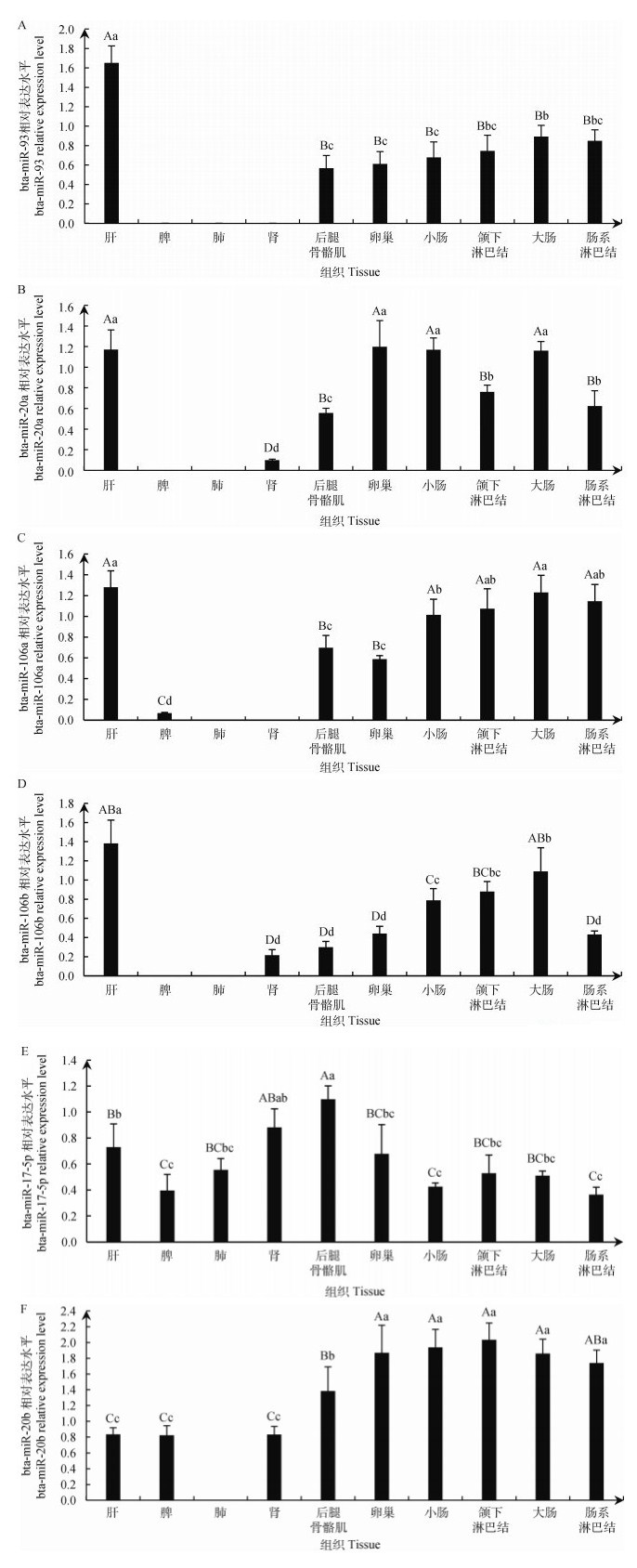
运用TargentScan(http://www.targetscan.org)和miRBase(http://www.mirbase.org)在线预测软件对可能靶向基因Nramp1的miRNAs进行预测,共预测到201个miRNAs,根据miRNA与靶基因结合位点的互补性、miRNA靶位点在不同物种之间的保守性、miRNA-mRNA结合的热稳定性等原则,筛选其中6个进行研究(表 2)。对这6个miRNAs分别设计特异性引物(表 1),利用Poly-A加尾法RT-PCR技术分别检测其在肝、脾、肺、肾、后腿骨骼肌、卵巢、小肠、颌下淋巴结、大肠、肠系淋巴结等组织中的表达情况,以U6基因为内参,将PCR扩增产物用8%聚丙烯酰胺凝胶电泳进行检测,结果见图 4。再利用Quantity One V4.52软件将筛选的6个miRNAs进行灰度体积分析,从而获得每个miRNA相对表达量,结果见图 5。可以看出,Nramp1基因mRNA与bta-miR-106a、bta-miR-20b、bta-miR-17-5p在牦牛免疫组织脾、颌下淋巴结、肠系淋巴结中共表达,与bta-miR-93、bta-miR-106b、bta-miR-20a在牦牛免疫组织颌下淋巴结、肠系淋巴结中共表达;并且发现,bta-miR-93在脾、肺、肾组织中无表达,且在颌下淋巴结、肠系淋巴结组织中的表达量与后腿骨骼肌、卵巢、小肠、大肠组织表达量无显著差异(P>0.05),见图 5A;bta-miR-20a在脾、肺无表达,在颌下淋巴结、肠系淋巴结组织中的表达量极显著低于卵巢、小肠、大肠组织表达量(P<0.01), 见图 5B;bta-miR-106a在肺、肾中无表达,在颌下淋巴结、肠系淋巴结中的表达量极显著高于脾、后腿骨骼肌、卵巢组织表达量(P<0.01), 见图 5C;bta-miR-106b在脾、肺无表达,在颌下淋巴结组织中的表达量极显著高于肠系淋巴结组织表达量(P<0.01), 见图 5D;bta-miR-17-5p在所检测的10种组织中均有表达,其中在脾、颌下淋巴结、肠系淋巴结组织中的表达量与肺、卵巢、小肠、大肠组织表达量无显著差异(P>0.05)见图 5E;bta-miR-20b在肺中无表达,在颌下淋巴结、肠系淋巴结组织中的表达量极显著高于肝、脾、肾、后腿骨骼肌组织表达量(P<0.01),与卵巢、小肠、大肠组织表达量无显著差异(P>0.05), 见图 5F。
|
|
表 2 预测的靶定牦牛Nramp1基因的部分miRNAs Table 2 The partial predicted miRNAs targeting to Nramp1 gene in yak |
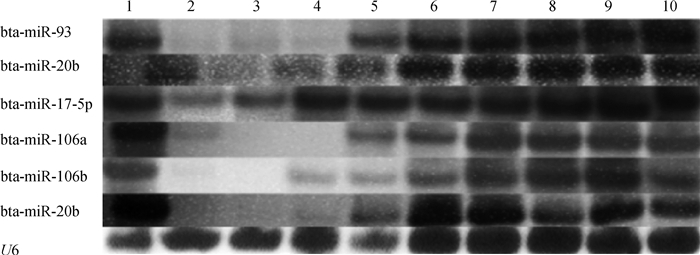
|
图 4 牦牛部分miRNAs组织表达谱 Fig. 4 The tissue expression profiles of partial miRNAs in yak |
|
图 5 牦牛部分miRNAs组织表达水平 Fig. 5 The relative expression level of partial miRNAs in various tissues of yak |
Nramp1基因在先天性免疫调节中具有重要作用[5, 7, 18-19],主要在骨髓单核细胞或巨噬细胞中特异性表达,其通过控制摄入微生物的重要微量营养物质而影响着动物的免疫能力[27],在巨噬细胞受到弗朗西斯菌侵染后,通过Nramp1蛋白对Fe2+转运限制而发挥抵抗作用[6]。对奶牛Nramp1基因的研究表明,Nramp1对金黄色葡萄球菌导致的乳腺炎具有显著抗性[8],在家禽上发现,Nramp1基因与沙门氏菌的定植显著关联[2, 28],在细菌早期感染期间其表达产物具有重要调控作用,且连锁分析表明,Nramp1基因与鸡、小鼠的鼠伤寒沙门氏菌感染抗性相关[2, 29]。对不同日龄梅山仔猪Nramp1基因mRNA表达谱研究发现,Nramp1基因在所检测的组织中均有不同程度的表达,且在脾中高度表达,并随着年龄增加而增加[30]。本试验在牦牛上的研究发现,Nramp1基因mRNA在所检测的肝、脾、肺、肾、后腿骨骼肌、卵巢、小肠、颌下淋巴、大肠和肠系淋巴结10种组织中均有表达,但在脾、肺、颌下淋巴结的表达量极显著高于肝、肾、大肠、肠系淋巴结组织表达量(P<0.01),显著高于小肠组织表达量(P<0.05)。这与Dai等[30]在梅山猪上的研究结果是基本一致的,同时在脾、颌下淋巴结的显著表达与应三成[31]在藏猪上的研究以及胡国顺[32]在鸡上的研究结果也基本是一致的。因此,表明Nramp1基因在牦牛体内可能与其他动物一样也是参与牦牛免疫调节的重要基因,而Nramp1基因mRNA在牦牛不同组织中表达量有差异,可能是由于单核细胞或吞噬细胞在不同组织分布不均所导致的。
微小核糖核酸(micro RNA,miRNA)是一类在真核生物中发现的内源性非编码RNA,其参与调节细胞的增殖与分化、疾病的发生发展,在机体中有着重要作用,miRNA大多数是在核内由RNA聚合酶转录形成的,最初被转录为数百个核苷酸长的miRNA转录物,称为初级miRNA(pri-miRNA),通过核和细胞质加工产生前体miRNA和成熟miRNA[33]。相关研究表明,miRNA具有重要的免疫调控作用, 如:在对猪圆环病毒2型(Porcine circovirus type 2 infection,PCV2)的研究中发现,PCV2通过抑制NF-κBp65与l12B启动子的结合以及miR-23a和miR-29b的上调来激活PI3K/Akt1和p38MAPK信号,从而抑制IL-12p40表达,致使IL-12p40表达和Th1细胞群受到抑制,降低宿主免疫力,增加感染其它病原风险[16];在使用猪甲型流感对未免疫猪进行攻毒试验发现,攻毒1~3 d,猪体内发生强烈干扰素反应,并差异表达27个miRNAs,攻毒后第14天,感染已经清除,此时差异表达11种miRNAs,miRNA在急性感染期以及之后均具有重要免疫作用[17]。赵冬雪等[18-19]对禽流感病毒、传染性法氏囊病毒侵染鸡胚细胞后miRNAs表达变化的研究发现,多种miRNAs参与病毒的易感和抵抗作用。本研究根据miRNA与靶基因结合位点的互补性、miRNA靶位点在不同物种之间的保守性等原则,预测可能靶定Nramp1基因的miRNAs有201个,并利用RT-PCR检测了bta-miR-93、bta-miR-20b、bta-miR-17-5p、bta-miR-106a、bta-miR-106b和bta-miR-20a在牦牛肝、脾、肺、肾、后腿骨骼肌、卵巢、小肠、颌下淋巴结、大肠和肠系淋巴结10种组织中的表达情况,发现bta-miR-93、bta-miR-20a、bta-miR-106a、bta-miR-17-5p和bta-miR-20b在颌下淋巴结、肠系淋巴结组织间的表达量无显著差异(P>0.05),bta-miR-106a、bta-miR-20b在颌下淋巴结、肠系淋巴结组织中的表达量极显著高于脾组织表达量(P<0.01), bta-miR-106b在颌下淋巴结组织中的表达量极显著高于肠系淋巴结组织表达量(P<0.01),已有的研究表明,这6个miRNAs中miR-93参与多种癌症免疫调节[34-36],miR-106a参与控制T辅助细胞的分化和功能表达[37],miR-93、miR-106a通过牛乳参与免疫反应[38],miR-106b直接靶向锌指和含BTB结构域的蛋白7A基因进而调节Hub-7细胞的凋亡,延缓裸鼠中异种移植肿瘤的生长[39],miR-20a和miR-106b通过靶向自噬激活激酶1基因调节成肌细胞中亮氨酸剥夺诱导的自噬[40],miR-20b通过靶向鸟嘌呤核苷酸交换因子1和细胞外调节蛋白激酶2基因显示出肿瘤抑制功能[41],骨髓间充质干细胞可以通过下调miR-17-5p的表达进一步上调PD-L1表达,从而减弱肝同种异体移植排斥反应[42],地塞米松诱导的细胞凋亡与下调的miR-17-5p水平有关[43],但未见这6个miRNAs在牦牛免疫方面研究的报道。
4 结论对牦牛Nramp1基因mRNA组织表达谱的研究显示,Nramp1基因mRNA在所检测的10种组织中均有表达,说明它可能具有广泛免疫调节作用。对筛选的6个miRNAs进行表达谱分析发现,Nramp1基因mRNA与bta-miR-106a、bta-miR-20b、bta-miR-17-5p在脾、颌下淋巴结和肠系淋巴结组织中共表达,与bta-miR-93、bta-miR-20a、bta-miR-106b在颌下淋巴结、肠系淋巴结组织中共表达,表明它们可能通过靶定Nramp1基因在牦牛免疫组织中参与调控作用,但二者是否存在真实的靶向调控以及其调控机制有待于深入研究。
| [1] | PERACINO B, BURACCO S, BOZZARO S. The Nramp (Slc11) proteins regulate development, resistance to pathogenic bacteria and iron homeostasis in Dictyostelium discoideum[J]. J Cell Sci, 2013, 126: 301–311. DOI: 10.1242/jcs.116210 |
| [2] | DAR M A, AHMED R, URWAT U, et al. Expression kinetics of natural resistance associated macrophage protein (NRAMP) genes in Salmonella Typhimurium-infected chicken[J]. BMC Vet Res, 2018, 14(1): 180. DOI: 10.1186/s12917-018-1510-4 |
| [3] | NEVES J V, WILSON J M, KUHL H, et al. Natural history of SLC11 genes in vertebrates:tales from the fish world[J]. BMC Evol Biol, 2011, 11: 106. DOI: 10.1186/1471-2148-11-106 |
| [4] | SOPHIE M, HAMEED A, MUNEER A, et al. SLC11A1 polymorphisms and host susceptibility to cutaneous leishmaniasis in Pakistan[J]. Parasit Vect, 2017, 10: 12. DOI: 10.1186/s13071-016-1934-2 |
| [5] | CHEN S L, XU M Y, JI X S, et al. Cloning and characterisation of natural resistance associated macrophage protein (Nramp) cDNA from red sea bream (Pagrus major)[J]. Fish Shellfish Immunol, 2004, 17(4): 305–313. DOI: 10.1016/j.fsi.2004.04.003 |
| [6] | POWELL D A, FRELINGER J A. Efficacy of resistance to Francisella imparted by ITY/NRAMP/SLC11A1 depends on route of infection[J]. Front Immunol, 2017, 8: 206. |
| [7] | JIANG Y H, MAO Y, LV Y N, et al. Natural resistance associated macrophage protein is involved in immune response of blunt snout bream, Megalobrama amblycephala[J]. Cells, 2018, 7(4): 27. DOI: 10.3390/cells7040027 |
| [8] | WORKU M, MORRIS A. Binding of different forms of lipopolysaccharide and gene expression in bovine blood neutrophils[J]. J Dairy Sci, 2009, 92(7): 3185–3193. DOI: 10.3168/jds.2008-1905 |
| [9] | ALI O S, SHABAYEK M I, SELEEM M M, et al. microRNAs 182 and 375 sera expression as prognostic biochemical markers in breast cancer[J]. Clin Breast Cancer, 2018, 18(6): e1373–e1379. DOI: 10.1016/j.clbc.2018.07.020 |
| [10] | BRONEVETSKY Y, ANSEL K M. Regulation of miRNA biogenesis and turnover in the immune system[J]. Immunol Rev, 2013, 253(1): 304–316. DOI: 10.1111/imr.12059 |
| [11] | CHEN X, YIN J, QU J, et al. MDHGI:matrix decomposition and heterogeneous graph inference for miRNA-disease association prediction[J]. PLoS Comput Biol, 2018, 14(8): e1006418. DOI: 10.1371/journal.pcbi.1006418 |
| [12] | DONG H, JIANG W, CHEN H Q, et al. microRNA-145 attenuates IL-6-induced enhancements of sensitivity to UVB irradiation by suppressing MyD88 in HaCaT cells[J]. Int J Immunopathol Pharmacol, 2018, 32: 2058738418795940. |
| [13] | BALUNI M, GHILDIYAL S, SINGHD, et al. Increased serum microRNA-29b expression and bad recovery in Japanese encephalitis virus infected patients; a new component to improve the disease recovery[J]. J Neuroimmunol, 2018, 323: 56–61. DOI: 10.1016/j.jneuroim.2018.07.014 |
| [14] | WANG K, SHEN X L, JIA J S, et al. High-throughput sequencing analysis of microRNAs in gills of red swamp crayfish, Procambarus clarkii infected with white spot syndrome virus[J]. Fish Shellfish Immunol, 2018, 83: 18–25. DOI: 10.1016/j.fsi.2018.09.007 |
| [15] | LI M, HUANG Q, WANG J F, et al. Differential expression of microRNAs in Portunus trituberculatus in response to Hematodinium parasites[J]. Fish Shellfish Immunol, 2018, 83: 134–139. DOI: 10.1016/j.fsi.2018.09.011 |
| [16] | DU Q, WU X C, WANG T T, et al. Porcine circovirus type 2 suppresses IL-12p40 induction via capsid/gC1qR-mediated microRNAs and signalings[J]. J Immunol, 2018, 201(2): 533–547. DOI: 10.4049/jimmunol.1800250 |
| [17] | BROGAARD L, LARSEN L E, HEEGAARD P M H, et al. IFN-λ and microRNAs are important modulators of the pulmonary innate immune response against influenza A (H1N2) infection in pigs[J]. PLoS One, 2018, 13(4): e0194765. DOI: 10.1371/journal.pone.0194765 |
| [18] |
赵冬雪, 穆卫涛, 王立砺, 等. 禽流感病毒与microRNA[J]. 中国兽医学报, 2017, 37(8): 1613–1616.
ZHAO D X, MU W T, WANG L L, et al. Avian influenza virus and microRNA[J]. Chinese Journal of Veterinary Science, 2017, 37(8): 1613–1616. (in Chinese) |
| [19] |
赵冬雪, 穆卫涛, 王立砺, 等. 传染性法氏囊病病毒与microRNA(miRNA)[J]. 中国兽医学报, 2017, 37(4): 762–767.
ZHAO D X, MU W T, WANG L L, et al. Infectious bursal disease virus and microRNA (miRNA)[J]. Chinese Journal of Veterinary Science, 2017, 37(4): 762–767. (in Chinese) |
| [20] |
江船.牛流行热病毒感染宿主细胞差异表达miRNA的筛选及其鉴定[D].济南: 山东大学, 2018.
JIANG C.The screening and identification of differentially expressed miRNAs of bovine ephemeral fever virus infected host cells[D]. Jinan: Shandong University, 2018.(in Chinese) |
| [21] |
付强.牛病毒性腹泻病毒持续性感染的分子机制研究及其定点突变株的构建[D].石河子: 石河子大学, 2015.
FU Q.Study on the molecular mechanisms of persistent replication of bovine viral diarrhea virus and construction of site-directed mutation of bovine viral diarrhea virus[D]. Shihezi: Shihezi University, 2015.(in Chinese) |
| [22] |
王军, 燕晓晓, 丁赫, 等. LPS诱导牛子宫内膜细胞miRNA差异表达分析[J]. 中国兽医杂志, 2017, 53(10): 10–12, 16.
WANG J, YAN X X, DING H, et al. Lipopolysaccharide-induced differential expression of miRNAs in bovine endometrial cells[J]. Chinese Journal of Veterinary Medicine, 2017, 53(10): 10–12, 16. (in Chinese) |
| [23] |
王玉恒, 徐业芬, 牛家强, 等. 牦牛Smad 4基因mRNA组织表达谱及其miRNAs初步研究[J]. 畜牧兽医学报, 2017, 48(10): 1882–1891.
WANG Y H, XU Y F, NIU J Q, et al. Preliminary studies on tissue expression profiles of Smad 4 mRNA and its miRNAs in yak[J]. Acta Veterinaria et Zootechnica Sinica, 2017, 48(10): 1882–1891. DOI: 10.11843/j.issn.0366-6964.2017.10.011 (in Chinese) |
| [24] |
廖珂.牦牛、普通牛和犏牛睾丸组织microRNA的比较研究[D].成都: 西南民族大学, 2016.
LIAO K.Comparative analysis of microRNA in yak, ordinary cattle and cattle-yak testis tissue[D]. Chengdu: Southwest Minzu University, 2016.(in Chinese) |
| [25] |
熊显荣, 兰道亮, 李键, 等. 牦牛卵巢小RNA高通量测序及生物信息学分析[J]. 畜牧兽医学报, 2016, 47(1): 55–63.
XIONG X R, LAN D L, LI J, et al. Solexa sequencing of small RNAs in yak (Bos grunniens) ovaries and bioinformatics analysis[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(1): 55–63. (in Chinese) |
| [26] |
李明霞, 潘和平, 阎萍, 等. 牦牛GDF9和BMP15基因的克隆、表达分析及miRNA研究[J]. 生物技术通报, 2016, 32(2): 100–108.
LI M X, PAN H P, YAN P, et al. Cloning, expression analysis and miRNA study of gene GDF9 and BMP15 in yak[J]. Biotechnology Bulletin, 2016, 32(2): 100–108. (in Chinese) |
| [27] | CELLIER M F M.Nutritional immunity: homology modeling of Nramp metal import[M]//LAMBRIS J D, HAJISHENGALLIS G.Current Topics in Innate Immunity Ⅱ.New York, NY: Springer, 2012: 335-351. |
| [28] | HU Y, SHAN Y J, ZHU C H, et al. Upregulation of NRAMP1 mRNA confirms its role in enhanced host immunity in post-artificial infections of Salmonella enteritidis in chicks[J]. Br Poult Sci, 2015, 56(4): 408–415. DOI: 10.1080/00071668.2015.1052371 |
| [29] | FRITSCHE G, NAIRZ M, LIBBY S J, et al. Slc11a1(Nramp1) impairs growth of Salmonella enterica serovar typhimurium in macrophages via stimulation of lipocalin-2 expression[J]. J Leukoc Biol, 2012, 92(2): 353–359. DOI: 10.1189/jlb.1111554 |
| [30] | DAI C H, WU J Y, ZHAO C X, et al. Nramp1 gene expression in different tissues of Meishan piglets from newborn to weaning[J]. Genet Mol Res, 2017, 16(1): gmr16019288. |
| [31] |
应三成.藏猪几个重要遗传特性基因的研究[D].成都: 四川大学, 2007.
YING S C.Study on genes related to several important genetic traits in Tibetan pig[D]. Chengdu: Sichuan University, 2007.(in Chinese) |
| [32] |
胡国顺.鸡Nramp1基因的遗传变异、表达以及与部分免疫性状的关联分析[D].扬州: 扬州大学, 2011.
HU G S.Genetic variation and expression of Nramp1 gene and its association with chicken immune qualities[D]. Yangzhou: Yangzhou University, 2011.(in Chinese) |
| [33] | OH S, PARK M R, SON S J, et al. Comparison of total RNA isolation methods for analysis of immune-related microRNAs in Market Milks[J]. Korean J Food Sci Anim Resour, 2015, 35(4): 459–465. DOI: 10.5851/kosfa.2015.35.4.459 |
| [34] | SHYAMASUNDAR S, LIM J P, BAY B H. miR-93 inhibits the invasive potential of triple-negative breast cancer cells in vitro via protein kinase WNK1[J]. Int J Oncol, 2016, 49(6): 2629–2636. DOI: 10.3892/ijo.2016.3761 |
| [35] | WU M Z, CHENG W C, CHEN S F, et al. miR-25/93 mediates hypoxia-induced immunosuppression by repressing cGAS[J]. Nat Cell Biol, 2017, 19(10): 1286–1296. DOI: 10.1038/ncb3615 |
| [36] | NI Q T, STEVIC I, PAN C, et al. Different signatures of miR-16, miR-30b and miR-93 in exosomes from breast cancer and DCIS patients[J]. Sci Rep, 2018, 8(1): 12974. DOI: 10.1038/s41598-018-31108-y |
| [37] | KÄSTLE M, BARTEL S, GEILLINGER-KÄSTLE K, et al. microRNA cluster 106a-363 is involved in T helper 17 cell differentiation[J]. Immunology, 2017, 152(3): 402–413. DOI: 10.1111/imm.12775 |
| [38] | OH S, PARK M R, RYU S, et al. Quantitative analysis of milk-derived micrornas and microbiota during the manufacturing and ripening of soft cheese[J]. J Microbiol Biotechnol, 2017, 27(9): 1566–1575. |
| [39] | LIANG X, ZHAO Q H, GENG T T, et al. miR-106b regulates the apoptosis and tumorigenesis of hepatocellular carcinoma via targeting Zinc finger and BTB domain-containing protein 7A (Zbtb7a)[J]. J Biochem Mol Toxicol, 2018, 32(8): e22169. DOI: 10.1002/jbt.22169 |
| [40] | WU H, WANG F L, HU S L, et al. miR-20a and miR-106b negatively regulate autophagy induced by leucine deprivation via suppression of ULK1 expression in C2C12 myoblasts[J]. Cell Signal, 2012, 24(11): 2179–2186. DOI: 10.1016/j.cellsig.2012.07.001 |
| [41] | HONG S B, YU S, LI J, et al. miR-20b displays tumor-suppressor functions in papillary thyroid carcinoma by regulating the MAPK/ERK signaling pathway[J]. Thyroid, 2016, 26(12): 1733–1743. DOI: 10.1089/thy.2015.0578 |
| [42] | CHEN Q X, ZHOU R, ZHANG Y C, et al. Bone marrow mesenchymal stromal cells attenuate liver allograft rejection may via upregulation PD-L1 expression through downregulation of miR-17-5p[J]. Transpl Immunol, 2018, 51: 21–29. DOI: 10.1016/j.trim.2018.08.004 |
| [43] | CHEN H Q, ZHANG D H, ZHANG G P, et al. A semantics-oriented computational approach to investigate microRNA regulation on glucocorticoid resistance in pediatric acute lymphoblastic leukemia[J]. BMC Med Inform Decis Mak, 2018, 18(Suppl 2): 57. |



