长纯合片段(runs of homozygosity,ROH)是二倍体生物基因组中纯合基因型的连续片段,它是由子代从亲代继承了相同单倍型而形成[1]。连锁不平衡、遗传漂变、群体结构、人工选择、近亲交配等都会影响ROH的产生,但是近交被认为是最重要的影响因素[1-2]。动物群体不同的交配体制、选择方向、选择强度、群体大小及群体结构会在基因组上形成独特的ROH模式,因此说ROH蕴藏着动物群体宝贵的遗传背景信息。ROH可以用于估计动物的近交情况[3-4]、推测近交的历史[5-6]、鉴定受到选择的基因[7-8]和有害突变[9-11]、评估遗传多样性和遗传资源保护[3, 12-14],以及优化动物育种规划[9, 15]。
目前,在家畜群体中已有多篇基于ROH的近交系数和其他方法比较的报道。Ferencakovic等[16]首次利用ROH对奥地利弗莱维赫牛进行近交评估,并发现基于ROH计算的基因组近交系数(FROH)和系谱近交系数(FPED)高度相关。Keller等[5]利用模拟数据比较了FPED、FROH、基于基因组杂合度的近交系数(Fh)和基于基因组关系矩阵的近交系数(FGRM)4种计算近交系数的方法,发现FROH是估计近交最有效的方法。Forutan等[17]也基于北美荷斯坦奶牛群体结构模拟比较了FPED、FGRM、FROH和真实的近交系数的关系,发现FROH最接近真实的近交系数,而FPED值低于真实值。因此,基于ROH计算近交系数具有较高的应用价值,特别是对绵山羊系谱记录不完善的情况。
ROH片段的长短还可以推断近交历史。长的ROH片段反映最近世代发生过近交,而短的ROH说明较远世代产生近交,因为世代数越短,ROH片段被重组打断的可能性就越小[5, 18]。在动物育种中,种畜按照特定的生产方向进行选留,如果所选个体之间存在亲缘关系,就必然会造成基因组上受选择区域纯合度增加[19],进而产生ROH片段。因此,基于基因组中高频ROH区域可以鉴定受到正向选择的基因。Kim等[20]利用50K芯片数据对北美荷斯坦奶牛基因组中的ROH进行检测,基于高频ROH区域检索到大量与产奶量、乳脂量及乳蛋白量相关的数量性状基因座。Purfield等[7]利用芯片数据对6个商业化肉羊品种进行ROH检测,在高频ROH区域检索到了大量与绵羊生长发育、体尺等肉用性状相关的基因。
本研究旨在利用绵羊50K SNP芯片数据对10个中外绵羊群体进行全基因组ROH检测。统计ROH的数目、长度、分布及频率,计算基因组近交系数FROH,并在高频ROH区域注释与绵羊经济性状相关的候选基因。该研究结果不仅能够反映不同绵羊群体的近交情况,还能鉴定一些与绵羊经济性状相关的基因,为绵羊标记辅助选择提供候选基因,同时也能为改进育种规划和畜禽遗传资源保种提供参考依据。
1 材料与方法 1.1 样本及数据来源本研究基于Illumina Ovine SNP50芯片数据对中外10个绵羊群体共440个个体进行ROH检测,其中9个群体来自于天津澳群、锡林格勒盟等羊场,以及各地的原保种场。有关样本的详细说明可以参考本实验室前期的研究[21-24],另外从国际绵羊协会网站(http://www.sheephapmap.org)下载了西藏藏羊的芯片数据。各群体名称、群体名称缩写及样本数分别为杜泊羊(DOR,93)、德国肉用美利奴羊(也叫德美羊,GMM,159)、苏尼特羊(SUN,66)、湖羊(HUS,12)、大尾寒羊(LTH,15)、罗布羊(LOP,15)、同羊(TON,15)、青海藏羊(TIBQ,14)、四川藏羊(TIBS,14)和西藏藏羊(TIBT,37),其中杜泊羊和德美羊为国外专门化品种,其他群体属于中国地方绵羊群体。本研究中所有的SNP芯片数据都是基于Ovis_aries_v1.0版本,相关数据可以从https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE46231和http://dx.doi.org/10.5061/dryad.g7rk5下载。因为藏系绵羊分布地域大,各地生态条件差异较大,依据不同特征分为3种类型:高原型(草地型)、山谷型和欧拉型[25]。本研究中的西藏藏羊属于高原型,四川藏羊属于山谷型,青海藏羊属于欧拉型。为了研究不同亚群的情况,将3个类型的藏羊当作不同的群体进行研究。
1.2 基因型数据处理通过PLINK软件[26]将440个样本的Illumina Ovine SNP50K芯片数据合并,并进行质控。其质控标准:1)最小等位基因频率大于0.01;2)哈迪温伯格平衡P值大于10-6;3)剔除0号和性染色体。
因为样本数据是Ovis_aries_v1.0版本,为了能更好的与现有的注释文件相对应,将SNP位置信息按照Ovis_aries_v4.0的位置重新排序,最后,利用Beagle软件[27]对缺失数据进行填充。将剩余46 827个SNPs用于全基因组ROH检测。
1.3 绵羊群体结构分析为了解所选群体的遗传结构,对10个绵羊群体进行主成分分析(principal component analysis, PCA),PCA分析使用gcta软件中的make-grm和pca命令[28]。
1.4 绵羊全基因组ROH检测及基因组近交系数(FROH)的计算使用R软件detectRUNS包对10个绵羊群体进行ROH检测。检测参数:1)每一个滑动窗口允许的杂合子不超过1个;2)组成单个ROH的SNP个数不少于30个;3)ROH最小长度为1 Mb;4)组成ROH的SNP最大间隔小于250 kb;5)SNP的最低密度为每100 kb内至少有1个SNP;其余参数使用detectRUNS包的默认参数。
基于所检测的ROH计算基因组近交系数FROH:
| $ {{\text{F}}_{{\text{ROH}}}} = \frac{{\sum {{{\text{L}}_{{\text{ROH}}}}} }}{{{{\text{L}}_{{\text{auto}}}}}} $ |
其中,∑LROH为常染色体上ROH片段的长度之和,Lauto为常染色体的物理总长度。
1.5 高频ROH区域的检测及候选基因鉴定统计每个SNP在各自绵羊群体内参与组成ROH的次数占样本数的比例,并将高于45%的SNP区域作为高频ROH区域,并利用RCircos[29]包绘制10个绵羊群体ROH频率在染色体上分布的Circos图。基于高频ROH区段的物理位置在绵羊基因组注释文件(ftp://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/000/298/735/GCF_000298735.2_Oar_v4.0/GCF_000298735.2_Oar_v4.0_genomic.gff.gz)中检索基因,然后依据NCBI(https://www.ncbi.nlm.nih.gov/)、GeneCards(https://www.genecards.org/)网站及文献查询基因功能。
2 结果 2.1 10个绵羊群体遗传结构分析图 1显示了10个绵羊群体的PCA分析结果。从图 1可以看出,所有群体分为了3大类,国内外品种能够清楚的分开,其中国内地方品种聚在同一类,但藏系绵羊出现在最右端,且西藏藏羊大致能与其他两个藏羊群体分开。
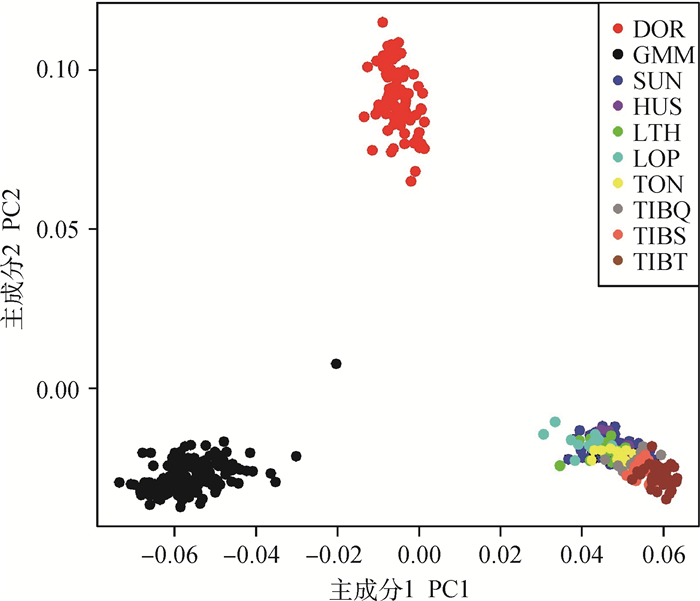
|
图 1 10个绵羊群体的主成分分析图 Fig. 1 Principal component diagram of 10 sheep populations |
在10个群体中共检测到25 920个ROH。图 2展示了10个绵羊群体ROH的数目在1~5、5~10、10~20、20~40和40 Mb以上5个分类中的比例,可以看出ROH的长度主要集中在10 Mb以内。表 1统计了不同绵羊群体ROH的平均长度、平均数目以及变化范围。由表 1可知,在10个群体中个体平均ROH数目为58.91个,ROH片段的平均长度为4.00 Mb。图 3展示了不同绵羊群体中绵羊个体ROH总长度和总数目的分布。从图 3可以看出,杜泊羊个体(红色)的ROH总长度最长且ROH数目最多,苏尼特羊个体(蓝色)的ROH总长度最短且ROH数目最少,其余群体介于中间,同时也可以看出一些极端个体ROH的总长度超过了600 Mb。
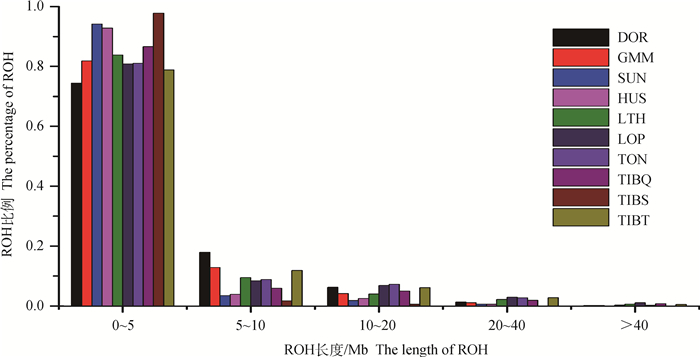
|
图 2 ROH在不同长度分类中的比例 Fig. 2 The percentage of ROH in different length categories |
|
|
表 1 10个绵羊群体的样本数、ROH的平均长度、数目及基因组近交系数(FROH) Table 1 Sample size of 10 sheep populations, average length, number of ROH, genomic inbreeding coefficient(FROH) in 10 sheep populations |
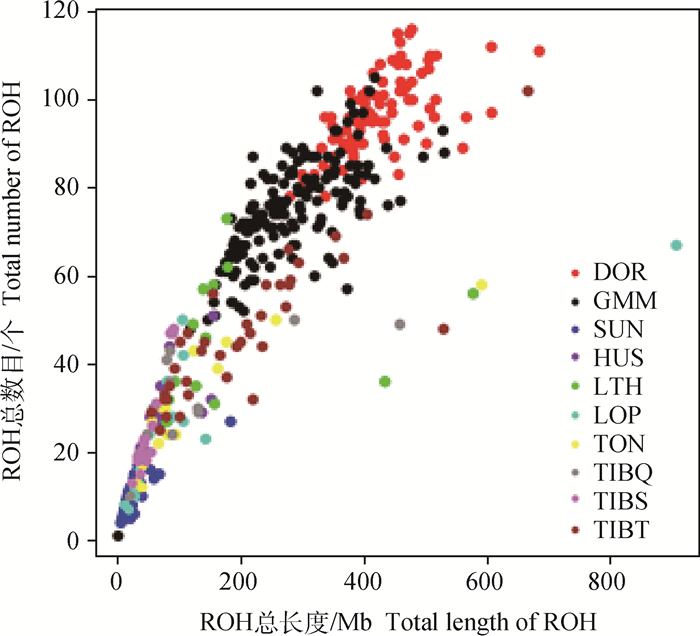
|
图 3 不同个体ROH总长度和总数目的分布 Fig. 3 The distribution of the total length and total number of ROH of different individuals |
不同绵羊群体的平均基因组近交系数及变化范围见表 1。杜泊羊的平均基因组近交系数最高,达到了0.172,苏尼特羊的平均基因组近交系数最低,仅为0.010,国外绵羊品种(杜泊羊和德美羊)的基因组近交系数明显高于国内品种。在个体层面,基因组近交系数最高的个体出现在罗布羊群体中(0.371),基因组近交系数最低的个体出现在德美羊群体中(0.001)。
2.4 高频ROH区域与候选基因鉴定图 4展示了不同绵羊群体ROH在1~26号染色体上出现的频率。本研究以45%作为高频ROH的阈值。一共检索到44个高频ROH区域,其中37个区域共注释到301个基因,剩余的7个区域未注释到基因,见表 2。其中已报道的与动物经济性状相关的基因有26个,见表 3。
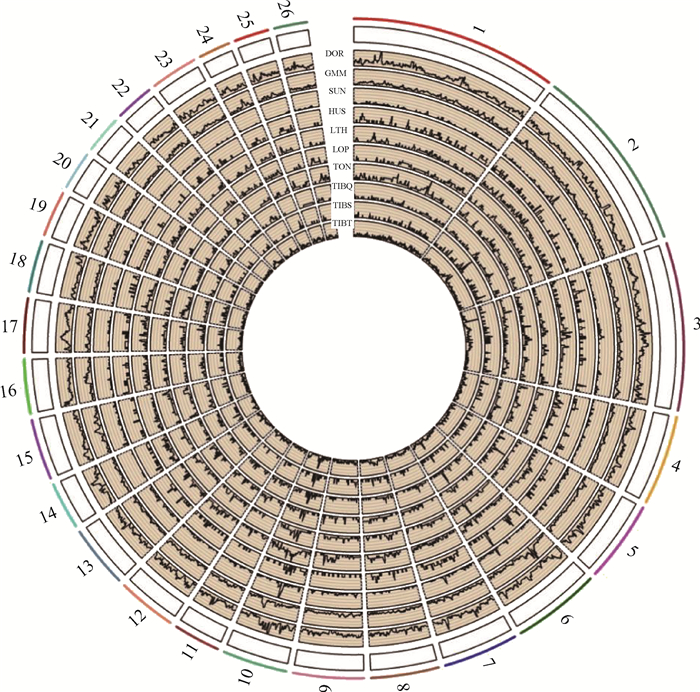
|
1~26表示绵羊染色体号,每一环代表一个绵羊群体,波浪线代表ROH频率 1-26 stand for the chromosome number of sheep, each ring stands for a sheep population, the wavy lines stand for ROH frequency 图 4 不同绵羊群体ROH分布频率的Rcircos图 Fig. 4 Rcircos diagram of the ROH frequencies distribution in different sheep populations |
|
|
表 2 绵羊基因组部分高频ROH区域及候选基因 Table 2 The partial high frequency ROH regions and candidate genes of sheep |
|
|
表 3 与动物经济性状相关的候选基因 Table 3 Candidate genes related to economic traits of animals |
强连锁通常能够在绵羊基因组上延伸100 kb[30],为了排除普遍存在的短ROH,本研究将ROH检测的最短长度设为1 Mb,另外所有绵羊群体ROH的检测使用统一标准,以便能在同一水平上反映不同绵羊群体实际的遗传差异。本研究中,ROH数目、长度及分布在不同群体间存在明显差异。不同群体ROH平均数目的变化为10.17(苏尼特羊)~95.99个(杜泊羊),国外商业化品种的ROH平均数目明显高于地方种群,这在一定程度上反映了人工选择对ROH的影响。据Kim等[20]对北美荷斯坦奶牛群体的研究,受到选择的荷斯坦奶牛群体的平均ROH数目明显多于未受选择的群体。若仅考虑中国地方绵羊群体,ROH平均数目在10.17(苏尼特羊)~47.27(西藏藏羊)个之间,所有个体ROH平均数目为26.90个。Mastrangelo等[31]研究发现,21个意大利本地绵羊品种ROH平均数目的变化范围为10.58~44.54个,所有个体ROH平均数目为24.51个,对于两国的地方绵羊品种,两项研究的结果基本一致。而对于ROH长度的分布而言,在0~5 Mb的长度分类中,四川藏羊、苏尼特羊和湖羊具有较高的比例,而杜泊羊、西藏藏羊和罗布羊具有较低的比例。相反,在长度大于5 Mb的分类中,杜泊羊、西藏藏羊和罗布羊具有较高的比例。这一结果与ROH平均长度的结果基本一致,ROH平均长度较短的群体为四川藏羊、苏尼特羊和湖羊,ROH平均长度较长的群体分别是罗布羊、杜泊羊和西藏藏羊。据此可推断,杜泊羊、西藏藏羊和罗布羊在最近世代经历了高强程度的近交,导致其基因组中出现了较高比例的长ROH片段。这一结果与这3个种群的实际情况相符,杜泊羊为引进的专门化肉羊品种,自身选育程度较高,加之国内引进数量有限,种公羊的高强度利用可能导致了杜泊羊近世代的较强的近交。西藏藏羊基因组中出现较高比例的长ROH片段,这可能与样本来源有关,西藏藏羊样本采集于保种场,因数量有限,其近交就难以避免。据中国畜禽遗传资源志羊志中的数据[25],2008年末罗布羊仅为8 800只,较小的群体可能造成了其近世代较强程度的近交。
3.2 绵羊群体间基于ROH的基因组近交系数本研究发现,国外绵羊群体的平均基因组近交系数明显高于国内地方绵羊群体,这一结果符合商业化品种和地方品种各自的选择强度。国内地方绵羊群体中,平均基因组近交系数较高的群体为西藏藏羊、大尾寒羊和同羊。近交水平能够一定程度反映群体大小及选配管理状况,本研究中,西藏藏羊和大尾寒羊样本均采集于保种场,另外据《中国畜禽遗传资源志羊志》的数据[25],2006年同羊的存栏量约3 000只,处于濒危/维持状态。平均基因组近交系数较低的3个群体分别是苏尼特羊、四川藏羊和湖羊,据《中国畜禽遗传资源志羊志》的统计[25],这3个群体是中国分布范围较广、存栏量较大的地方绵羊群体。
目前,很多羊场没有完善的系谱信息,不能准确的估计系谱近交系数,但已有研究表明,通过ROH计算的近交系数与基于系谱的近交系数具有较高的相关性[16],且基于ROH计算基因组近交系数的方法比其他多种近交系数计算的方法更能反映群体真实的近交水平[5, 17]。因此,本研究计算了基于ROH的基因组近交系数(FROH),但未与系谱近交系数进行比较。
3.3 绵羊基因组高频ROH区域的候选基因本研究在绵羊基因组中高频ROH区域一共鉴定到26个已报道的与动物经济性状相关的基因。26个基因主要分布在杜泊羊、德美羊和大尾寒羊基因组中高频ROH区域。部分鉴定到的基因也验证了前期的研究结果,与体尺及生长发育相关的基因NCAPG[21],与肉质、胴体性状及繁殖性状相关的基因CAPN3[32],与绵羊断奶后日增重、胸围、管围、6月龄重及断奶重相关的基因HYDIN[23]。
除了上述3个基因之外,在杜泊羊群体中也鉴定了与肌肉、骨骼发育及脂肪沉积等相关的基因。PRKAA2是影响猪肌肉生长和脂肪沉积的一个重要候选基因[33-34]。FAIM2在秦川牛的肌肉组织中高表达,可作为影响秦川牛生长和胴体性状的一个候选基因[35]。SREBF2在二郎山山地鸡群体中与屠体重、活重、全净膛重、胸肌重等屠体性状存在显著关联[36]。DLEU7是淋巴细胞白血病缺失7基因,该基因与成年人身高存在显著关联[37]。王文浩[38]利用简化基因组测序数据,采用全基因组关联分析方法研究发现,FAM124A和INTS6基因与京海黄鸡的生长性状显著相关。DACH1与北京油鸡的80日龄体重、屠体重、全净膛重显著相关[39]。NKX2-5与鸡的胫长和胫围存在显著关联[40]。LEPR影响猪的繁殖[41]、脂肪沉积[42],并且与牛的生长发育性状显著相关[43]。KLHL1可显著影响多浪羊的产羔数[44]。FZD3显著影响中国美利奴羊羊毛细度[45]。LALBA与内蒙古白绒山羊产绒量存在显著相关[46]。EXTL3突变可引起骨骼发育不良、免疫缺陷和发育迟缓[47]。DAZAP2在多发性骨髓瘤发生过程中具有重要作用[48]。NCKAP5L是影响Ⅱ型糖尿病和肥胖新的候选基因[49]。PRKAG1参与调控家畜的能量代谢,可作为与动物能量代谢相关性状的候选基因[50]。WNT10B在山羊的脂肪组织中表达量最高,可能在脂肪代谢过程中发挥重要作用[51]。
在德美羊群体中鉴定的基因主要与生长发育及体尺性状相关。NCAPG-LCORL位点与牛的日增重、胴体重、眼肌面积等性状存在显著关联[52]。NCAPG和LCORL也可显著影响马的体尺[53]。SH2B1与南阳黄牛的体高、胸围、体斜长、体重及日增重显著相关[54]。CDIPT与猪的肩部背膘厚、股二头肌色值、肌内脂肪含量及肌内水分含量等肉质和胴体性状显著相关[55]。
基于大尾寒羊群体,鉴定到2个与生产性状相关的基因。FGF9与肉牛的生长发育及胴体性状显著相关[56]。LATS2可能与湖羊肌肉的生长发育存在一定的联系[57]。
4 结论本研究针对中外10个绵羊群体进行全基因组ROH检测,发现ROH的数目、长度、频率及分布在不同绵羊品种中存在明显差异,国外专门化绵羊品种的平均基因组近交系数明显高于国内地方绵羊品种。在国内地方绵羊种群中,西藏藏羊具有最高的平均基因组近交系数,苏尼特羊具有最低的平均基因组近交系数。杜泊羊、西藏藏羊及罗布羊基因组中具有较高比例的长ROH片段,说明其最近世代经历了较强程度的近交。同时,本研究在不同绵羊种群基因组中高频ROH区段鉴定到了26个与动物经济性状相关的候选基因,这些基因可作为绵羊标记辅助选择的候选基因。
| [1] | CEBALLOS F C, JOSHI P K, CLARK D K, et al. Runs of homozygosity: windows into population history and trait architecture[J]. Nat Rev Genet, 2018, 19(4): 220–234. DOI: 10.1038/nrg.2017.109 |
| [2] | BROMAN K W, WEBER J L. Long homozygous chromosomal segments in reference families from the centre d'Étude du polymorphisme humain[J]. Am J Hum Genet, 1999, 65(6): 1493–1500. DOI: 10.1086/302661 |
| [3] | PURFIELD D C, BERRY D P, MCPARLAND S, et al. Runs of homozygosity and population history in cattle[J]. BMC Genet, 2012, 13: 70. |
| [4] | MASTRANGELO S, TOLONE M, DI GERLANDO R, et al. Genomic inbreeding estimation in small populations: evaluation of runs of homozygosity in three local dairy cattle breeds[J]. Animal, 2016, 10(5): 746–754. DOI: 10.1017/S1751731115002943 |
| [5] | KELLER M C, VISSCHER P M, GODDARD M E. Quantification of inbreeding due to distant ancestors and its detection using dense single nucleotide polymorphism data[J]. Genetics, 2011, 189(1): 237–249. DOI: 10.1534/genetics.111.130922 |
| [6] | BROWNING S R, BROWNING B L. Identity by descent between distant relatives: detection and applications[J]. Annu Rev Genet, 2012, 46: 617–633. DOI: 10.1146/annurev-genet-110711-155534 |
| [7] | PURFIELD D C, MCPARLAND S, WALL E, et al. The distribution of runs of homozygosity and selection signatures in six commercial meat sheep breeds[J]. PLoS One, 2017, 12(5): e0176780. DOI: 10.1371/journal.pone.0176780 |
| [8] | SZMATOŁA T, GURGUL A, ROPKA-MOLIK K, et al. Characteristics of runs of homozygosity in selected cattle breeds maintained in Poland[J]. Livest Sci, 2016, 188: 72–80. DOI: 10.1016/j.livsci.2016.04.006 |
| [9] | ZHANG Q Q, GULDBRANDTSEN B, BOSSE M, et al. Runs of homozygosity and distribution of functional variants in the cattle genome[J]. BMC Genomics, 2015, 16(1): 542. DOI: 10.1186/s12864-015-1715-x |
| [10] | SZPIECH Z A, XU J S, PEMBERTON T J, et al. Long runs of homozygosity are enriched for deleterious variation[J]. Am J Hum Genet, 2013, 93(1): 90–102. |
| [11] | KIM E S, SONSTEGARD T S, VAN TASSELL C P, et al. The relationship between runs of homozygosity and inbreeding in jersey cattle under selection[J]. PLoS One, 2015, 10(7): e0129967. DOI: 10.1371/journal.pone.0129967 |
| [12] | AL-MAMUN H A, CLARK S A, KWAN P, et al. Genome-wide linkage disequilibrium and genetic diversity in five populations of Australian domestic sheep[J]. Genet Sel Evol, 2015, 47: 90. DOI: 10.1186/s12711-015-0169-6 |
| [13] | HERRERO-MEDRANO J M, MEGENS H J, GROENEN M A M, et al. Conservation genomic analysis of domestic and wild pig populations from the Iberian Peninsula[J]. BMC Genet, 2013, 14: 106. |
| [14] | BISCARINI F, NICOLAZZI E L, STELLA A, et al. Challenges and opportunities in genetic improvement of local livestock breeds[J]. Front Genet, 2015, 6: 33. |
| [15] | MARRAS G, GASPA G, SORBOLINI S, et al. Analysis of runs of homozygosity and their relationship with inbreeding in five cattle breeds farmed in Italy[J]. Anim Genet, 2015, 46(2): 110–121. |
| [16] | FERENCAKOVIC M, HAMZIC E, GREDLER B, et al. Runs of homozygosity reveal genome-wide autozygosity in the Austrianfleckvieh cattle[J]. Agric Conspec Sci, 2011, 76(4): 325–328. |
| [17] | FORUTAN M, MAHYARI S A, BAES C, et al. Inbreeding and runs of homozygosity before and after genomic selection in North American Holstein cattle[J]. BMC Genomics, 2018, 19: 98. DOI: 10.1186/s12864-018-4453-z |
| [18] | MCQUILLAN R, LEUTENEGGER A L, ABDEL-RAHMAN R, et al. Runs of homozygosity in European populations[J]. Am J Hum Genet, 2008, 83(3): 359–372. DOI: 10.1016/j.ajhg.2008.08.007 |
| [19] | PEMBERTON T J, ABSHER D, FELDMAN M W, et al. Genomic patterns of homozygosity in worldwide human populations[J]. Am J Hum Genet, 2012, 91(2): 275–292. |
| [20] | KIM E S, COLE J B, HUSON H, et al. Effect of artificial selection on runs of homozygosity in U.S. Holstein cattle[J]. PLoS One, 2013, 8(11): e80813. DOI: 10.1371/journal.pone.0080813 |
| [21] | ZHAO F P, WEI C H, ZHANG L, et al. A genome scan of recent positive selection signatures in three sheep populations[J]. J Integr Agric, 2016, 15(1): 162–174. DOI: 10.1016/S2095-3119(15)61080-2 |
| [22] | YUAN Z, LIU E, LIU Z, et al. Selection signature analysis reveals genes associated with tail type in Chinese indigenous sheep[J]. Anim Genet, 2017, 48(1): 55–66. |
| [23] | ZHANG L, LIU J S, ZHAO F P, et al. Genome-wide association studies for growth and meat production traits in sheep[J]. PLoS One, 2013, 8(6): e66569. DOI: 10.1371/journal.pone.0066569 |
| [24] |
王光凯, 曾淘, 王慧华, 等. 苏尼特羊全基因组选择信号检测[J]. 中国农业科学, 2014, 47(6): 1190–1199.
WANG G K, ZENG T, WANG H H, et al. Genome-wide detection of selection signature on Sunite sheep[J]. Scientia Agricultura Sinica, 2014, 47(6): 1190–1199. DOI: 10.3864/j.issn.0578-1752.2014.06.016 (in Chinese) |
| [25] |
国家畜禽遗传资源委员会.
中国畜禽遗传资源志羊志[M]. 北京: 中国农业出版社, 2011.
China National Commission of Animal Genetic Resources. Animal genetic resources in China sheep and goat[M]. Beijing: China Agriculture Press, 2011. (in Chinese) |
| [26] | PURCELL S, NEALE B, TODD-BROWN K, et al. PLINK: a tool set for whole-genome association and population-based linkage analyses[J]. Am J Hum Genet, 2007, 81(3): 559–575. DOI: 10.1086/519795 |
| [27] | BROWNING B L, ZHOU Y, BROWNING S R. A one-penny imputed genome from next-generation reference panels[J]. Am J Hum Genet, 2018, 103(3): 338–348. |
| [28] | YANG J, LEE S H, GODDARD M E, et al. GCTA: A tool for genome-wide complex trait analysis[J]. Am J Hum Genet, 2011, 88(1): 76–82. |
| [29] | ZHANG H G, MELTZER P, DAVIS S. RCircos: an R package for Circos 2D track plots[J]. BMC Bioinformatics, 2013, 14: 244. DOI: 10.1186/1471-2105-14-244 |
| [30] | MASTRANGELO S, DI GERLANDO R, TOLONE M, et al. Genome wide linkage disequilibrium and genetic structure in Sicilian dairy sheep breeds[J]. BMC Genet, 2014, 15: 108. |
| [31] | MASTRANGELO S, CIANI E, SARDINA M T, et al. Runs of homozygosity reveal genome-wide autozygosity in Italian sheep breeds[J]. Anim Genet, 2018, 49(1): 71–81. |
| [32] |
曾滔, 赵福平, 王光凯, 等. 基于群体分化指数FST的绵羊全基因组选择信号检测[J]. 畜牧兽医学报, 2013, 44(12): 1891–1899.
ZENG T, ZHAO F P, WANG G K, et al. Genome-wide detection of selection signatures in sheep populations with use of population differentiation index FST[J]. Acta Veterinaria et Zootechnica Sinica, 2013, 44(12): 1891–1899. (in Chinese) |
| [33] | LEE H Y, CHOI B H, LEE J S, et al. Molecular characterization and chromosomal mapping of the porcine AMP-activated protein kinase α2(PRKAA2) gene[J]. Asian Aust J Anim Sci, 2007, 20(5): 615–621. DOI: 10.5713/ajas.2007.615 |
| [34] | LIN L, FLISIKOWSKI K, SCHWARZENBACHER H, et al. Characterization of the porcine AMPK alpha 2 catalytic subunitgene (PRKAA2): genomic structure, polymorphism detection and association study[J]. Anim Genet, 2010, 41(2): 203–207. |
| [35] |
王丽君.秦川牛生长与胴体性状相关候选基因的表达谱、遗传多样性及其关联分析[D].杨凌: 西北农林科技大学, 2013.
WANG L J.Expression patterns, genetic diversity and correlation analysis with related traits of candidate genes in Qinchuan cattle[D]. Yangling: Northwest A&F University, 2013.(in Chinese) |
| [36] |
邱莫寒.鸡SREBF1、SREBF2基因的克隆、表达及其与屠体性状的关联分析[D].雅安: 四川农业大学, 2011.
QIU M H.Cloning, expression of SREBF1, SREBF2 genes and association analysis between polymorphisms and carcass traits in chicken[D]. Ya'an: Sichuan Agricultural University, 2011.(in Chinese) |
| [37] | KANG S J, CHIANG C W, PALMER C D, et al. Genome-wide association of anthropometric traits in African- and African-derived populations[J]. Hum Mol Genet, 2010, 19(13): 2725–2738. DOI: 10.1093/hmg/ddq154 |
| [38] |
王文浩.基于SLAF-seq技术对京海黄鸡生长、屠宰及部分抗病性状的全基因组关联分析[D].扬州: 扬州大学, 2015.
WANG W H.A Genome-wide association study of growth, carcass and some disease-resistance traits on Jinghai Yellow Chicken[D]. Yangzhou: Yangzhou University, 2015.(in Chinese) |
| [39] |
吴丹.北京油鸡体重和屠体性状的全基因组关联研究[D].北京: 中国农业科学院, 2012.
WU D.Genome-wide association study of loci affecting body weight and carcass traits in Beijing-You chicken[D]. Beijing: Chinese Academy of Agricultural Sciences, 2012.(in Chinese) |
| [40] |
孙艳发, 刘冉冉, 郑麦青, 等. 鸡胫长和胫围的全基因组关联分析[J]. 畜牧兽医学报, 2013, 44(3): 358–365.
SUN Y F, LIU R R, ZHENG M Q, et al. Genome-wide association study on shank length and shank girth in chicken[J]. Acta Veterinaria et Zootechnica Sinica, 2013, 44(3): 358–365. (in Chinese) |
| [41] |
张冰, 潘天彪, 蓝海恩, 等. 陆川猪和大白猪LEPR基因对产仔数的效应分析[J]. 中国畜牧兽医, 2009, 36(11): 105–107.
ZHANG B, PAN T B, LAN H E, et al. Effects of LEPR gene on litter size in Luchuan and Large White pig[J]. China Animal Husbandry & Veterinary Medicine, 2009, 36(11): 105–107. (in Chinese) |
| [42] |
付言峰, 李兰, 周艳红, 等. 瘦素受体对苏钟猪脂肪沉积调控的影响[J]. 南京农业大学学报, 2015, 38(6): 986–992.
FU Y F, LI L, ZHOU Y H, et al. Role of leptin receptor in the regulation of fat deposition in Suzhong pigs[J]. Journal of Nanjing Agricultural University, 2015, 38(6): 986–992. (in Chinese) |
| [43] | GUO Y K, CHEN H, LAN X Y, et al. Novel SNPs of the bovine LEPR gene and their association with growth traits[J]. Biochem Genet, 2008, 46(11-12): 828–834. DOI: 10.1007/s10528-008-9197-z |
| [44] |
王珊, 李晓林, 牛志刚, 等. 多浪羊多胎基因全基因组关联分析研究[J]. 江西农业学报, 2017, 29(5): 77–81.
WANG S, LI X L, NIU Z G, et al. Whole genomic association analysis of polyembryonic gene in Duolang sheep[J]. Acta Agriculturae Jiangxi, 2017, 29(5): 77–81. (in Chinese) |
| [45] |
柏妍.细毛羊毛性状候选基因遗传效应的研究[D].乌鲁木齐: 新疆农业大学, 2015.
BAI Y.Study on genetic effects of candidate gene on wool traits in fine wool sheep[D]. Urumqi: Xinjiang Agricultural University, 2015.(in Chinese) |
| [46] |
蓝贤勇, 陈宏, 田智泉, 等. LALBA基因SNP与内蒙古白绒山羊经济性状的关联[J]. 遗传, 2008, 30(2): 169–174.
LAN X Y, CHEN H, TIAN Z Q, et al. Correlations between SNP of LALBA gene and economic traits in Inner Mongolian white cashmere goat[J]. Hereditas, 2008, 30(2): 169–174. DOI: 10.3321/j.issn:0253-9772.2008.02.008 (in Chinese) |
| [47] | VOLPI S, YAMAZAKI Y, BRAUER P M, et al. EXTL3 mutations cause skeletal dysplasia, immune deficiency, and developmental delay[J]. J Exp Med, 2017, 214(3): 623–637. |
| [48] | SHI Y W, SHEN R, REN W, et al. Molecular features and expression of DAZAP2 in human multiple myeloma[J]. Chin Med J (Engl), 2007, 120(19): 1659–1665. DOI: 10.1097/00029330-200710010-00003 |
| [49] | CHEN J H, MENG Y H, ZHOU J H, et al. Identifying candidate genes for Type 2 Diabetes Mellitus and obesity through gene expression profiling in multiple tissues or cells[J]. J Diabetes Res, 2013, 2013: 970435. |
| [50] | BENKEL B, KOLLERS S, FRIES R, et al. Characterization of the bovine ampk γ1 gene[J]. Mamm Genome, 2005, 16(3): 194–200. DOI: 10.1007/s00335-004-2426-9 |
| [51] |
林森, 林亚秋, 朱江江, 等. 山羊Wnt10b基因生物学特征及组织细胞表达模式分析[J]. 基因组学与应用生物学, 2017, 36(10): 4070–4078.
LIN S, LIN Y Q, ZHU J J, et al. Analysis of biological characteristics and tissue cell expression pattern of Wnt10b gene in goat[J]. Genomics and Applied Biology, 2017, 36(10): 4070–4078. (in Chinese) |
| [52] | LINDHOLM-PERRY A K, SEXTEN A K, KUEHN L A, et al. Association, effects and validation of polymorphisms within the NCAPG - LCORL locus located on BTA6 with feed intake, gain, meat and carcass traits in beef cattle[J]. BMC Genet, 2011, 12: 103. |
| [53] | MAKVANDI-NEJAD S, HOFFMAN G E, ALLEN J J, et al. Four loci explain 83% of size variation in the horse[J]. PLoS One, 2012, 7(7): e39929. DOI: 10.1371/journal.pone.0039929 |
| [54] | YANG M J, QU L, LIU J X, et al. Polymorphisms and effects on growth traits of the SH2B1 gene in Chinese cattle[J]. Livest Sci, 2012, 143(2-3): 283–288. DOI: 10.1016/j.livsci.2011.09.020 |
| [55] |
符亚原, 熊远著, 潘刚, 等. 猪CDIPT基因对肉质和胴体性状的影响[J]. 畜牧兽医学报, 2009, 40(6): 787–791.
FU Y Y, XIONG Y Z, PAN G, et al. Association of the polymorphism of porcine CDIPT gene with carcass and meat quality traits[J]. Acta Veterinaria et Zootechnica Sinica, 2009, 40(6): 787–791. DOI: 10.3321/j.issn:0366-6964.2009.06.001 (in Chinese) |
| [56] |
张文刚.肉牛生长发育与胴体性状全基因组关联分析及目标区域测序捕获功能基因的研究[D].北京: 中国农业科学院, 2017.
ZHANG W G.Genome-wide association study of growth and carcass traits and target sequencing analysis for detecting function genes in beef cattle[D] Beijing: Chinese Academy of Agricultural Sciences, 2017.(in Chinese) |
| [57] |
王庆增.Lats2基因在湖羊骨骼肌生长发育中功能的研究[D].扬州: 扬州大学, 2016.
WANG Q Z.Study of the function of Lats2 gene in Hippo signal pathway in skeletal muscle growth and development in Hu sheep[D] Yangzhou: Yangzhou University, 2016.(in Chinese) |



