2. 河南省肉羊繁育工程技术研究中心, 洛阳 471023
2. Henan Province Mutton Sheep Breeding Engineering Technology Research Center, Luoyang 471023, China
在动物界,一般来讲,随着季节环境的改变,野生动物一年四季都会进行着一些规律性活动,如鸟类的迁徙,动物的冬眠[1]等。研究发现,动物在各个季节中产生的规律性活动,不仅受环境中温度、光照、降水量等的影响,更重要的是来自于自身的内源性节律调控[2]。此外,还有研究表明,哺乳动物在长期的进化过程中,参与繁殖调控的相关基因在年度周期中有着节律性的表达变化,并且在调节季节性活动变化中发挥重要功能[3]。
众所周知,在自然界中,马、驴等季节性繁殖动物在春季白天光照逐渐延长时开始发情交配,被称为长日照哺乳动物。绵羊和山羊在秋天白天光照缩短的时候发情交配,被称为短日照动物[4]。日照长度的变化诱导季节性繁殖哺乳动物发生强烈的生理变化,如绵羊的发情、交配和妊娠等[5-6]。
后代的有利繁殖对于物种的延续是不可或缺的。哺乳动物的繁殖主要由下丘脑-垂体-性腺(H-P-G)轴调控网络控制。下丘脑不仅是中枢神经系统的重要部位,亦是调节动物内脏器官及内分泌活动的中枢。研究发现,下丘脑视交叉上核可通过视网膜-视交叉上核束来感受外界环境光暗信号的变化,使机体的生物节律与环境的光暗变化同步起来,如果这条神经通路被切断,视交叉上核的节律活动就不再能与外界环境的光暗变化发生同步[7]。
山羊的季节性发情受到光照周期的严格调控,短光照周期是诱导山羊开始季节性发情的主要外界环境因素,下丘脑在山羊季节性发情调控过程中发挥着重要的作用。因此,本研究采用人工短光照诱导山羊进入季节性发情状态,进一步采用RNA-Seq技术筛选参与山羊季节性发情调控的相关基因及信号传导通路,为今后阐明山羊季节性发情调控机制提供更多的理论依据。
1 材料与方法 1.1 试验动物模型及组织样本的采集以饲养在河南科技大学周山校区牧场的16只河南槐山羊空怀母羊为研究对象,母羊平均年龄1周岁,平均体重37.75 kg。试验开始时间为2016年3月21日(春分),试验初期所有羊只饲养在同一特定的羊舍内。羊只给光分为自然光照和人工光照,自然光照时长不足时,采用白织灯补充光照时长。自然光照的给光时间为:8:00-16:00,共计8 h,黑暗时间16 h。首先,所有羊只给予自然光照。在自然光照第42天中午12:00时刻(羊只由黑暗进入光照),随机挑选2只母羊(编号分别为E2、F2)进行屠宰,迅速取出下丘脑组织,放入经DEPC水处理过的无菌冻存管中置于液氮中保存;随后至晚上20:00时刻(羊只由光照进入黑暗),随机挑选2只母羊(编号分别为G2、H2)进行屠宰,并迅速取出下丘脑组织,放入经DEPC水处理过的无菌冻存管中置于液氮中保存。随后,剩余羊只给予人工光照。人工光照的给光时间为:4:00-20:00,共计16 h,黑暗时间8 h。在人工光照第28天上午8:00时刻(羊只由黑暗进入光照),随机挑选2只母羊(编号分别为L1、M1)进行屠宰,迅速取出下丘脑组织,放入经DEPC水处理过的无菌冻存管中置于液氮中保存;随后至晚上00:00时刻(羊只由光照进入黑暗),随机挑选2只母羊(编号分别为O1、P1)进行屠宰,并迅速取出下丘脑组织,放入经DEPC水处理过的无菌冻存管中置于液氮中保存[8]。
1.2 试验方法 1.2.1 下丘脑总RNA的提取按照Trizol总RNA提取试剂盒(购自上海生工生物工程股份有限公司)操作说明,提取8只羊下丘脑组织的总RNA,经Nanodrop-2000核酸蛋白测定仪(购自基因有限公司)测定总RNA的浓度和纯度,并于-80 ℃冰箱保存。
1.2.2 转录组cDNA文库构建样品检测合格后,把同一时间屠宰的2个羊只总RNA等量混合,组成4个RNA混合池。使用带有Oligo(dT)的磁珠富集mRNA;富集完成后,加入fragmentation buffer将mRNA打断成短片段。随后,用六碱基随机引物(random hexamers)以mRNA为模板进行反转录合成一链cDNA,再加入缓冲液、dNTPs和DNA聚合酶Ⅰ合成二链cDNA。接着,利用AMPure XP beads纯化双链cDNA。对纯化后的双链cDNA进行末端修复、加A、加接头。通过AMPure XP beads对双链cDNA进行片段大小选择,最后进行PCR扩增以构建cDNA文库。
1.2.3 下丘脑转录组测序利用Illumina二代高通量测序平台(HiSeqTM2500/4000),采用PE150测序策略进行文库的测序,由安诺优达基因科技(北京)有限公司来完成。
1.2.4 数据处理与参考基因组的比对分析用组装软件Trinity对下丘脑转录组从头组装[9]。首先使用SOAPdenovo程序将reads(芯片上每个反应读出的短序列)通过片段重叠连成更长的片段Contig[10],再将Contig连在一起得到两端不能再延长的序列Unigene,再对其做去冗余处理,进一步拼接(Tophat2软件[11])。利用Cufflinks[12]对TopHat2的比对结果进行组装,并将处理后的高质量reads与参考基因组(GCA_001704415.1,包含28 404个注释基因)进行比对,获得各样品reads的比对效率和reads在基因组上的位置信息。
1.2.5 下丘脑基因差异表达分析通过Cuffcompare软件将测序结果与已知的基因序列进行比较和同源转录本聚类。根据Cufflinks预测出基因模型,用ASprofile软件[13]对每个样品的可变剪接事件进行分类统计,得到最终的Unigene。Unigene表达量的计算使用FPKM法(fragments per kb per million fragments)[14],采用HTSeq软件[15]对各样品进行基因表达水平分析,模型为union。结果文件分别统计了不同表达水平下基因的数量以及单个基因的表达水平。根据基因表达水平分析中得到的readcount数据来进行基因差异表达分析。对于有生物学重复的样品,使用DESeq[16]进行差异表达分析。
1.2.6 差异表达基因的GO分类与富集分析用DAVID在线软件[17]和GOseq软件[18]对长短光照下山羊下丘脑组织的差异表达基因进行GO注释分类。
1.2.7 差异表达基因的KEGG富集分析利用KEGG注释系统[19]将长短光照下山羊下丘脑组织的差异表达基因注释到KEGG数据库中,得到所有可能的代谢途径(pathway)。
1.2.8 测序结果可靠性分析将保存在液氮中的下丘脑组织样品提取总RNA,利用Nanodrop 2000测定总RNA的纯度和浓度,用经DEPC处理的双蒸水稀释至1 μg·μL-1。用TaKaRa公司的PrimeScriptTM 1st strand cDNA Synthesis Kit试剂盒制备cDNA第一链。本研究选择了15个差异表达基因为候选基因并设计了特异性引物,并以在各组织中可高水平、恒定表达的GAPDH基因为内参基因[20],见表 1。以合成的cDNA第一链为模板,用TaKaRa公司生产的SYBR® Premix Ex TaqTM Ⅱ试剂盒对8个下丘脑组织进行荧光定量分析(CFX ConnectTM Real Time PCR Detection System)。将RT-qPCR的结果与RNA-Seq的结果进行相关性分析,以检验RNA-Seq的可靠性。
|
|
表 1 RT-qPCR的引物信息 Table 1 The primer information of RT-qPCR |
提取8只羊下丘脑组织的总RNA,经Nanodrop-2000核酸蛋白测定仪测定,各下丘脑组织样本的总RNA浓度均≥10 ng·μL-1,纯度(260 nm/280 nm)均在2.0以上。RNA提取后经1%琼脂糖电泳检测后可见3条带(图略),说明RNA完整性较好未被降解,可用于下一步试验。
2.2 山羊下丘脑cDNA文库质量检测使用Agilent 2100对文库的插入片段大小进行检测;片段大小符合预期,通过Q-PCR方法对文库的有效浓度进行精确定量(文库有效浓度>4 nmol·L-1),结果表明获得了高质量的cDNA文库,文库构建成功。
2.3 山羊下丘脑转录组测序质量评估通过双端测序法共得到34.9 G的碱基总数,经拼接后得到220 071 622个raw read pairs(约4.4亿条reads), 经过去除带接头的、低质量的reads后得到209 941 031个clean read pairs,平均每个样本的reads数约为5 249万条。其中样品左端测序的碱基错误率为0.01%,碱基质量值大于20的占98.18%,大于30的占94.91%,GC含量为48.13%;样品右端测序的碱基错误率为0.02%,碱基质量值大于20的占95.54%,大于30的占89.52%,GC含量为48.25%。上述结果表明得到了高质量的转录组测序结果。
2.4 山羊转录组测序数据拼接和组装将Clean Reads与参考基因组进行序列比对,获取其在参考基因组或基因上的位置信息(图 1),定位区域分为Exon(外显子)、Intron(内含子)和Intergenic(基因间区)。由比对到参考基因组上的Reads(Mapped Reads)占Clean Reads的百分比表明,所选参考基因组组装可以满足信息分析的需求(表 2)。
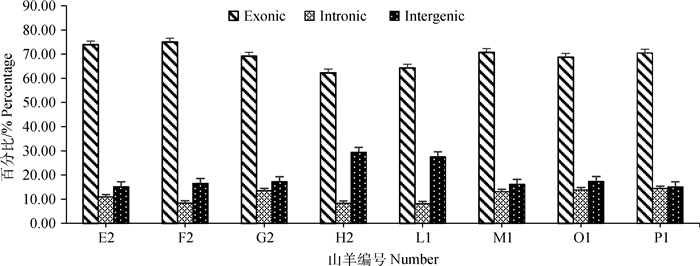
|
纵坐标表示Clean Reads比对到参考基因组Exon(外显子)、Intron(内含子)和Intergenic(基因间区)位置上的百分比 The vertical coordinate indicates the percentage of clean reads to the reference genome exon, intron and intergenic 图 1 不同样本测序序列与参考基因组序列各位置比对百分比 Fig. 1 Percentage of different sample sequences compared with reference genome sequence |
|
|
表 2 8个转录组样本测序序列与参考序列比对结果 Table 2 The comparison of sequencing sequence of 8 transcriptome samples to reference sequence |
不同试验条件下基因表达水平分布情况见图 2。结果表明,各试验组组间基因表达水平存在差异,G2H2与L1M1试验组整体基因表达量以及基因密度高于E2F2和O1P1组。
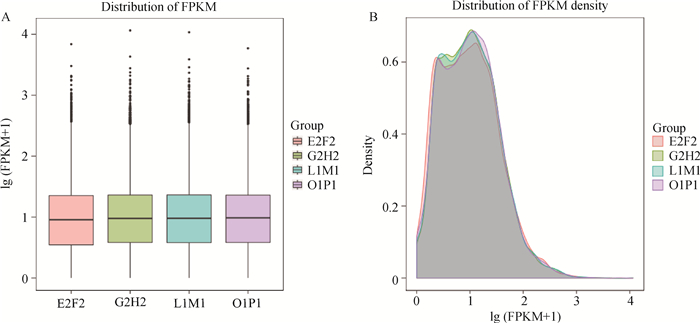
|
A.基因表达水平分布箱线图横坐标为样品(组)名称,纵坐标为lg(FPKM+1),且展示了最大值、上四分位数、中值、下四分位数和最小值。B.基因密度分布图,横坐标为lg(FPKM+1), 纵坐标为基因的密度 A. The abscissa is the sample(group) name, ordinate is lg(FPKM+1) in the gene expression level distribution box plot, and shows the maximum, upper quartile, median, lower quartile and minimum. B. The abscissa is lg(FPKM+1), and the ordinate is the density of the gene in the gene density distribution map 图 2 不同试验条件下基因表达水平分布箱线图和基因密度分布图 Fig. 2 The gene expression level distribution box plot and gene density distribution chart under different experimental conditions |
此外,RNA-Seq相关性检查结果表明,每组样品间基因表达水平皮尔逊相关系数的平方(R2)均大于0.92(图 3),即各组样品之间表达模式的相似度较高,增加了试验的可靠性。
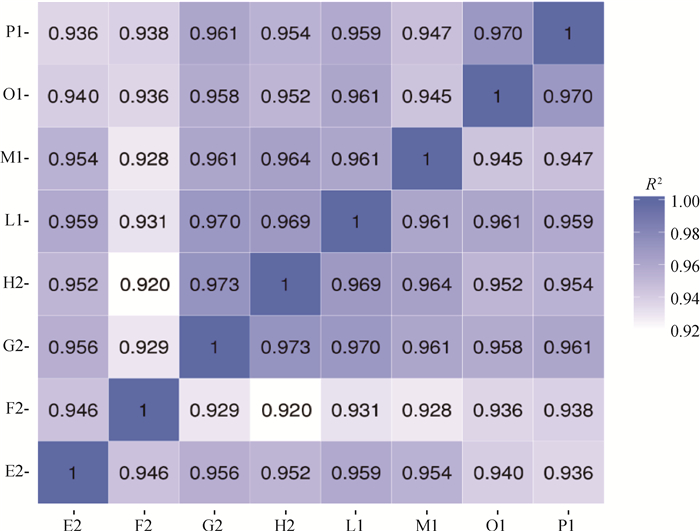
|
Encode计划建议皮尔逊相关系数的平方(R2)大于0.92(理想的取样和试验条件下)。具体的项目操作中,要求R2大于0.8,否则需要对样品做出合适的解释,或者重新进行试验 The Encode plan recommends that the square of Pearson correlation coefficient (R2) is greater than 0.92 (under ideal sampling and experimental conditions). In specific project operations, R2 is required to be greater than 0.8, otherwise appropriate interpretation of the sample or retesting is required 图 3 8个转录组RNA-Seq相关性检查分布图 Fig. 3 Distribution of RNA-Seq correlation examination in 8 transcriptional groups |
将4个试验组基因表达水平分析中得到的readcount数据采用DESeq进行分析,结果显示,E2F2与G2H2组中共筛选到202个差异表达基因,其中包含114个上调基因,88个下调基因;E2F2与L1M1组中共筛选到27个差异表达基因,其中包含16个上调基因,11个下调基因;G2H2与O1P1组中共筛选到1 067个差异表达基因,其中包含444个上调基因,623个下调基因;L1M1与O1P1组中共筛选到298个差异表达基因,其中包含80个上调基因,218个下调基因(图 4)。
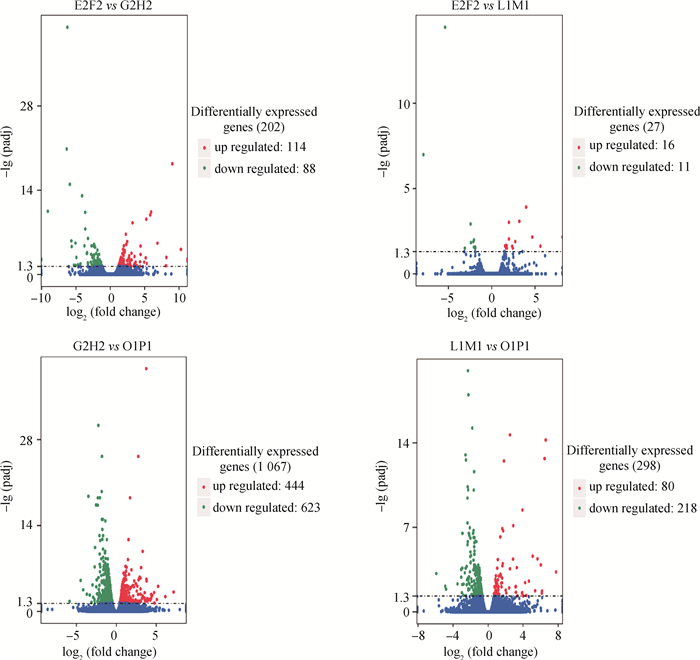
|
横坐标代表基因在不同样本中的表达倍数变化,纵坐标代表基因表达量变化差异的统计学显著性;差异表达显著的基因用红点(上调)和绿点(下调)表示。差异表达不显著的基因用蓝点表示 The abscissa represents the fold change of gene expression in different samples, and the ordinate represents the statistical significance of difference of the gene expression change; the genes with significant differential expression are represented by red dots (up-regulation) and green dots (down-regulation). Genes with insignificant differential expression are represented by blue dots 图 4 不同试验组差异基因火山图 Fig. 4 Differential gene volcano maps from different experimental groups |
差异基因聚类分析结果表明,E2F2与O1P1两个试验组的差异基因表达谱相近,而G2H2与L1M1两个试验组的差异基因表达谱相近(图 5)。
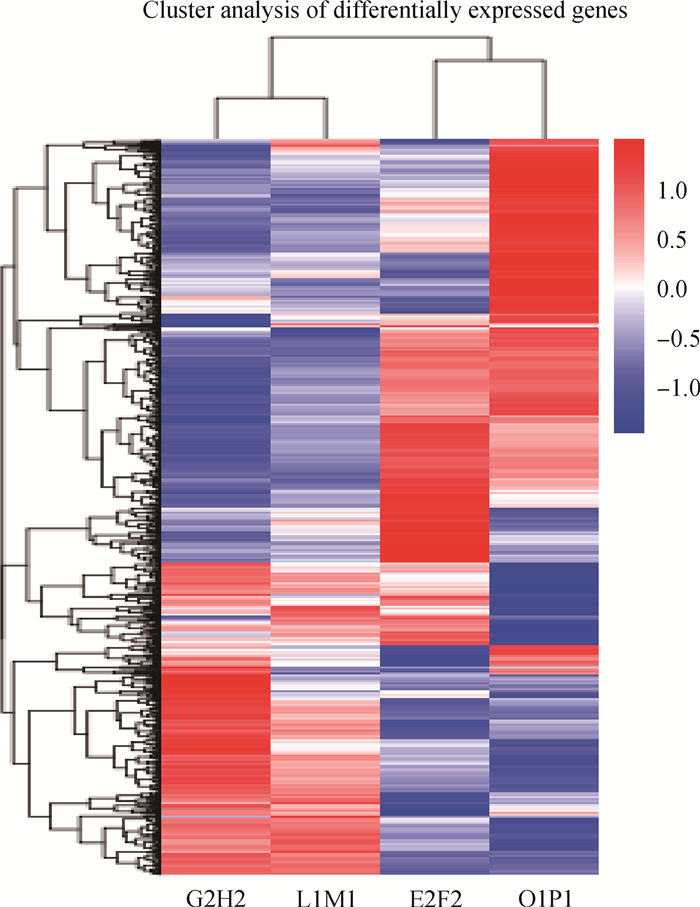
|
每一行表示一个基因,每一列表示一个样本。颜色从红到蓝,表示lg(FPKM+1)从大到小 Each row represents a gene and each column represents a sample. Color from red to blue, for lg(FPKM+1) from big to small 图 5 不同试验组差异基因表达量层次聚类图 Fig. 5 Hierarchical cluster diagram of differentially expressed genes in different experimental groups |
将E2F2与G2H2组(自然光照组)同L1M1与O1P1组(人工光照组)的差异基因列表进行比较,共筛选出448个差异表达基因(DEGs),包含26个共有差异基因(图 6)。
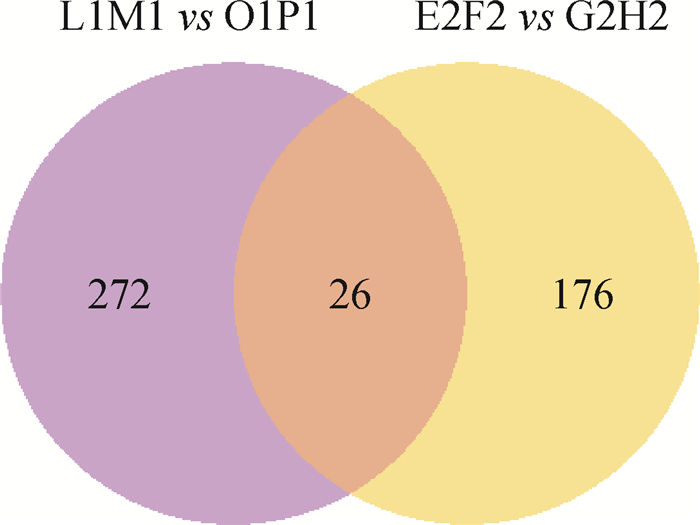
|
其中圆圈代表比较组合,每个圆圈中的数字之和代表相互比较样品的差异基因总个数,圆圈重叠的部分表示其共有的差异基因个数 The circles represent the comparison combination, the sum of the numbers in each circle represents the total number of differential genes in the sample compared with each other, and the overlapping part of the circle indicates the number of differential genes shared by the two circles 图 6 不同光照条件下差异基因维恩图 Fig. 6 Vine map of differentially expressed genes under different illumination conditions |
利用GOseq软件对Unigene富集分析,共得到16 282条注释基因。按照GO分类方式,将注释基因分为分子功能(molecular Function)、生物过程(biological process)和细胞组成(cellular component)3大类,以及富集结果最显著的30个亚类。各试验组对比差异基因注释结果见图 7。E2F2与G2H2组中有138个差异表达注释基因,E2F2与L1M1组中有23个差异表达注释基因,G2H2与O1P1组中有716个差异表达注释基因,L1M1与O1P1组中有216个差异表达注释基因。

|
纵坐标为富集的GO条目,横坐标为该条目中差异表达基因个数。不同颜色用来区分生物过程、细胞组分和分子功能,带“*”为显著富集的GO条目 The ordinate is the enriched GO term, and the abscissa is the number of differentially expressed genes in the term. Different colors are used to distinguish biological processes, cellular components and molecular functions, the items with "*" as a significantly enriched GO term 图 7 不同试验组差异基因GO富集柱状图 Fig. 7 Go enrichment histogram of differentially expressed genes in different experimental groups |
将448个差异表达基因(DEGs)标注到KEGG数据库中,自然光照组与人工光照组共富集到241个信号通路。在差异基因KEGG富集结果中,挑选了富集最显著的20条通路在图形中进行展示,若富集的通路不足20条,则全部展示(图 8)。在各试验组所有KEGG通路中,对参与山羊繁殖相关的代谢通路及候选基因进行筛选,共得到5个信号通路,包含165个差异表达基因,各信号通路与差异基因表达水平见表 3。其中涉及差异表达基因数量最多的是包括速激肽家族中TACR1、TACR2和TACR3基因在内的Calcium signaling pathway(chx04020)信号通路(图 9)。
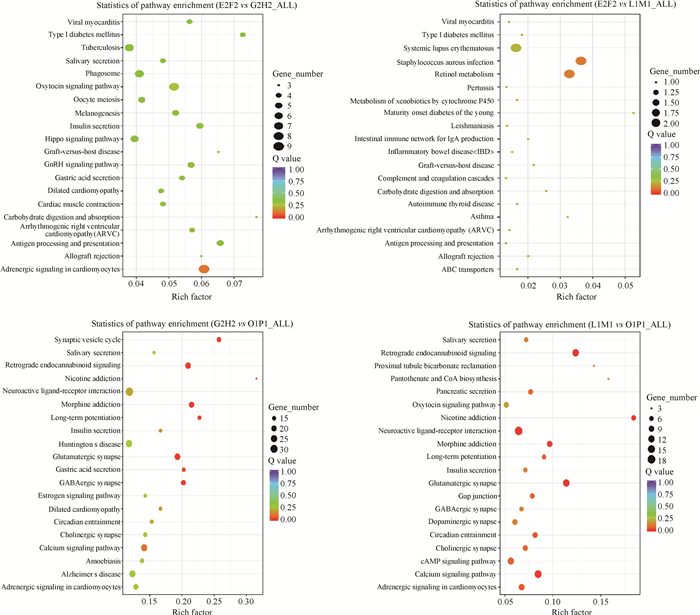
|
纵坐标表示通路名称,横坐标表示富集因子,点的大小表示此通路中差异表达基因个数多少,而点的颜色对应于不同的Q value范围上调差异基因KEGG富集散点图 The ordinate indicates the pathway name, the abscissa indicates the enrichment factor, the size of the point indicates the number of differentially expressed genes in this pathway, and the color of the points correspond to the different Q value range up-regulated differential gene KEGG enrichment scatter plot 图 8 不同试验组所有差异基因KEGG富集散点图 Fig. 8 Scattered plot of KEGG enrichment of all differentially expressed genes in different experimental groups |
|
|
表 3 速激肽受体与繁殖相关差异表达基因富集的通路 Table 3 The enrichment pathway of tachykinin receptor and reproduction-related differentially expressed genes |
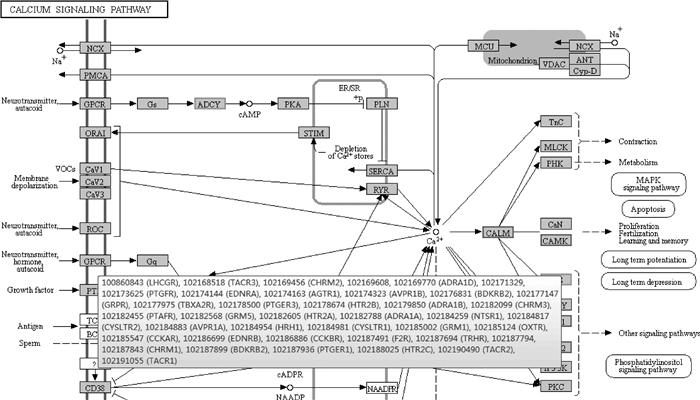
|
图 9 Calcium signaling pathway结构示意图 Fig. 9 Calcium signaling pathway structure diagram |
RT-qPCR的结果用相对表达量表示,本研究用到的内参基因是GAPDH;RNA-Seq结果用FPKM值表示。通过计算两者间的相关系数分析RT-qPCR和RNA-Seq结果的一致性。本研究发现,RT-qPCR和RNA-Seq的结果高度一致,两者之间存在显著的相关关系,见表 4。
|
|
表 4 RT-qPCR和RNA-Seq结果的相关性分析 Table 4 The correlation analysis between RT-qPCR and RNA-Seq results |
有关下丘脑转录组测序技术与动物繁殖相关的研究在禽类[21-23]、鱼类[24]和哺乳动物绵羊[25]中均有报道。本试验以河南槐山羊为研究对象,通过建立长短光照试验动物模型,取不同光照时长处理后的山羊下丘脑组织进行RNA-Seq和生物信息学分析。
筛选短光照诱导的山羊季节性发情相关的差异表达基因是本研究的重点。在分析差异表达基因时设定了4组对比,分别是自然光照条件下白天与夜晚(E2F2 vs G2H2)、人工光照条件下白天与夜晚(L1M1 vs O1P1)、自然光照白天与人工光照白天(E2F2 vs L1M1)、自然光照夜晚与人工光照夜晚(G2H2 vs O1P1)。在自然光照条件下的白天与夜晚对比组中筛选到了202个差异表达基因,其中包括114个在夜间表达水平上调的基因和88个在夜间表达水平下调的基因。当山羊转入人工光照条件后,再次比较白天与夜晚下丘脑组织的差异表达基因,共筛选到1 067个差异表达基因,其中包括444个在夜间表达水平上调的基因和623个在夜间表达水平下调的基因(图 4)。
为揭示差异表达基因的生理功能,本研究对筛选到的差异表达基因进行了GO分析。在自然光照条件下白天与夜晚(E2F2 vs G2H2)、人工光照条件下白天与夜晚(L1M1 vs O1P1)、自然光照白天与人工光照白天(E2F2 vs L1M1)和自然光照夜晚与人工光照夜晚(G2H2 vs O1P1)4个对比组中富集到的GO条目分别为297、96、658和350。在GO的分子功能(molecular function)中富集最多的是Transporter activity(蛋白转运)、Ion transmembrane transporter(蛋白跨膜转运离子)和Receptor activity(受体活性),它们都与哺乳动物海马神经元信号传导[26],Ca2+转运[27]等中枢神经调节机制相关,在4个对比组中分别富集到246、55、496和320条。GO亚细胞组分(cellular component)富集到的条目比较分散,各对比组富集到的代表性GO条目见图 7。
一般来说,生物体内重要生理过程的完成往往需要多个基因的共同参与,各自行使相关的功能,发生一系列调控。在对生物体内某一现象的产生进行研究时,阐明重要生理功能涉及的信号通路至关重要。因此,本研究在GO分析的基础上又进行了KEGG信号通路分析。富集到差异表达基因数量最多的是Calcium signaling pathway(钙离子信号通路,chx04020),共有40个差异表达基因参与了该信号通路(图 9)。已有的研究表明,该信号通路在神经冲动传递[28]和情绪控制[29]上有重要的调控作用。钙离子作为第二细胞内信使,不仅调节着细胞中许多重要的生理和病理过程,而且与哺乳动物卵母细胞的增长和减数分裂,以及激素和生长因子分泌有着密切的联系[30-31]。尤其值得注意的是,本研究还富集到了许多与动物生殖调控或神经内分泌活动有关的信号通路,如GnRH signaling pathway(促性腺激素释放激素信号通路,chx04912)、Neuroactive ligand-receptor interaction(神经活性配体-受体相互作用信号通路,chx04080)、Oxytocin signaling pathway(催产素信号通路,chx04921)、Estrogen signaling pathway(雌激素信号通路,chx04915)等,这表明长短光照处理后山羊下丘脑对机体的内分泌调节水平可能存在差异,影响了下丘脑的神经活动规律,改变了山羊的生殖内分泌活动。
光照调节哺乳动物生殖生理的过程主要包括:光照→视网膜→下视丘→视交叉上核→下丘脑室周区和结节区→脊髓的胸段→脊髓节前交感神经束→颈上神经节→节后交感神经纤维→松果体→褪黑激素→下丘脑→垂体→性腺轴[32-33]。有研究表明,速激肽通过下丘脑-垂体-性腺轴在人、鼠等哺乳动物繁殖功能调节方面发挥重要作用[34]。尤其是在下丘脑-垂体-性腺轴方面的研究已表明,速激肽通过下丘脑神经元影响GnRH的释放[35],并在调节动物发情、排卵和妊娠等生殖过程中发挥重要功能。
速激肽是一种激肽类家族,由一组羧基末端为苯丙氨酸-X-甘氨酸-亮氨酸-蛋氨酸-NH2序列的肽类组成。现已知在哺乳动物中存在速激肽类物质,如P物质(substance P,SP)、神经激肽(neurokinin A,NKA)和神经激肽B(neurokinin B,NKB)及血细胞激肽(hemokinin-1,HK)。对鼠的相关研究表明,SP和NKA由TAC1基因编码[36],NKB由TAC2和TAC3基因编码,HK由TAC4基因编码[37]。由于它们的氨基酸分子序列中有着共同的羧基末端,从而有着共同的生物活性,受G蛋白偶联受体调控。根据其对酪氨酸激酶(TK)敏感性的不同,TK受体(NKR)分为NK1R、NK2R、NK3R 3类。在鼠上已有报道,3种受体类型分别由不同基因编码:TACR1、TACR2和TACR3基因[38]。
人们利用体外卵母细胞培养技术研究发现,卵泡细胞及颗粒细胞均能分泌速激肽P物质于卵泡液中[39]。在有关男性生殖生理方面的研究亦有报道,如男性精子质量受TAC1编码的SP的影响[40],以及在绵羊垂体中含有由TAC1编码的速激肽,包括P物质和神经激肽A(NKA)在内的生物活性肽,能够调节体内PRL的释放[41]。近几年来有关TACR1基因的研究进展在现有的人体医学领域中研究众多,TACR1基因在某些疾病的发生和调控方面发挥着重要的作用,如糖尿病[42]、头颈等细胞癌症[43]及一些神经细胞的病变[44]。在动物上也有该基因相关疾病的研究报道,如基因缺陷小鼠动脉压升高[45]、疼痛焦虑[46]及运动障碍[47]和行为异常[48]。通过蛋白序列分析图发现,不同种属动物的TACR1基因序列具有生物多样性,如人体结肠炎症[49]与猪的结肠疾病[50]均与TACR1基因受体有关。NKB的mRNA被发现在人类胎盘中有表达,并与NK1和NK2受体结合,参与母体妊娠调节[51]。而NK3R受体主要在中枢神经系统中表达,在人、大鼠胎盘和子宫,以及骨骼肌、肺、肝、大鼠门静脉和肠系膜静脉等不同物种肠道的某些肠神经元中也可检测到[52]。在鸟类中已经分离出了第一个由长光照可以激活的转录因子Eya3[53],同时人们也通过研究认为,速激肽基因很可能是这个“神秘因子”中的一员。Dupré等[8]对Eya3和TAC1作为长日照信号基因调节绵羊垂体结节释放促乳素机理的研究发现,当从短光照(SP冬天日照)转入长光照(LP夏天光照)的第一天,绵羊垂体结节部Eya3和TAC1的表达就迅速发生变化。这也就证实了光照对速激肽家族基因在哺乳动物体内的表达变化起着十分重要的作用。
本研究在富集到差异表达基因数量最多的Calcium signaling pathway(钙离子信号通路,chx04020)通路中,发现了速激肽受体基因TACR1、TACR2和TACR3(图 9)的存在。那么可以肯定,速激肽受体基因受长短光照条件的影响,亦会参与调节山羊繁殖的相关生理活动。
4 结论本研究利用Illumina二代高通量测序技术对经过不同长短光照处理的山羊下丘脑组织进行转录组测序,经过分析后得出初步结论:Calcium signaling pathway(钙离子信号通路,chx04020)及候选基因TACR1、TACR2、TACR3可能与山羊繁殖过程有关,并发挥着重要作用。
| [1] | KONDO N, SEKIJIMA T, KONDO J, et al. Circannual control of hibernation by HP complex in the brain[J]. Cell, 2006, 125(1): 161–172. DOI: 10.1016/j.cell.2006.03.017 |
| [2] | DE MIERA C S, MONECKE S, BARTZEN-SPRAUER J, et al. A circannual clock drives expression of genes central for seasonal reproduction[J]. Curr Biol, 2014, 24(13): 1500–1506. DOI: 10.1016/j.cub.2014.05.024 |
| [3] |
姚蔚, 王德华, 张学英. 哺乳动物季节性繁殖的内源年生物钟及光敏神经环路研究进展[J]. 动物学杂志, 2017, 52(4): 717–725.
YAO W, WANG D H, ZHANG X Y. Molecular basis of annual clock and photosensitive neural circuits for regulating seasonal reproduction in mammals[J]. Chinese Journal of Zoology, 2017, 52(4): 717–725. (in Chinese) |
| [4] | LEWIS J E, EBLING F J P. Tanycytes as regulators of seasonal cycles in neuroendocrine function[J]. Front Neurol, 2017, 8: 79. |
| [5] |
王世银, 石国庆, 甘尚权, 等. 不同繁殖状态阿勒泰羊下丘脑MTNR1A基因表达变化与其季节性繁殖的关系[J]. 南方农业学报, 2015, 46(10): 1887–1892.
WANG S Y, SHI G Q, GAN S Q, et al. Expression change of MTNR1A gene in hypothalamus of Altay sheep(Ovis aries) at different reproductive stages and its relationship with seasonal reproduction[J]. Journal of Southern Agriculture, 2015, 46(10): 1887–1892. DOI: 10.3969/j:issn.2095-1191.2015.10.1887 (in Chinese) |
| [6] | HANON E A, LINCOLN G A, FUSTIN J M, et al. Ancestral TSH mechanism signals summer in a photoperiodic mammal[J]. Curr Biol, 2008, 18(15): 1147–1152. DOI: 10.1016/j.cub.2008.06.076 |
| [7] | HERBISON A E. Control of puberty onset and fertility by gonadotropin-releasing hormone neurons[J]. Nat Rev Endocrinol, 2016, 12(8): 452–466. DOI: 10.1038/nrendo.2016.70 |
| [8] | DUPRÉ S M, MIEDZINSKA K, DUVAL C V, et al. Identification of Eya3 and TAC1 as long-day signals in the sheep pituitary[J]. Curr Biol, 2010, 20(9): 829–835. DOI: 10.1016/j.cub.2010.02.066 |
| [9] | GRABHERR M G, HAAS B J, YASSOUR M, et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome[J]. Nat Biotechnol, 2011, 29(7): 644–652. DOI: 10.1038/nbt.1883 |
| [10] | LI R Q, YU C, LI Y R, et al. SOAP2:an improved ultrafast tool for short read alignment[J]. Bioinformatics, 2009, 25(15): 1966–1967. DOI: 10.1093/bioinformatics/btp336 |
| [11] | KIM D, PERTEA G, TRAPNELL C, et al. TopHat2:accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions[J]. Genome Biol, 2013, 14(4): R36. DOI: 10.1186/gb-2013-14-4-r36 |
| [12] | TRAPNELL C, ROBERTS A, GOFF L, et al. Corrigendum:differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks[J]. Nat Protoc, 2014, 9(10): 2513. |
| [13] | FLOREA L, SONG L, SALZBERG S L. Thousands of exon skipping events differentiate among splicing patterns in sixteen human tissues[J]. F1000Res, 2013, 2: 188. DOI: 10.12688/f1000research |
| [14] | MORTAZAVI A, WILLIAMS B A, MCCUE K, et al. Mapping and quantifying mammalian transcriptomes by RNA-Seq[J]. Nat Methods, 2008, 5(7): 621–628. DOI: 10.1038/nmeth.1226 |
| [15] | ANDERS S, PYL P T, HUBER W. HTSeq-a Python framework to work with high-throughput sequencing data[J]. Bioinformatics, 2015, 31(2): 166–169. DOI: 10.1093/bioinformatics/btu638 |
| [16] | ANDERS S, HUBER W. Differential expression analysis for sequence count data[J]. Genome Biol, 2010, 11(10): R106. DOI: 10.1186/gb-2010-11-10-r106 |
| [17] | HUANG D W, SHERMAN B T, LEMPICKI R A. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources[J]. Nat Protoc, 2009, 4(1): 44–57. DOI: 10.1038/nprot.2008.211 |
| [18] | YOUNG M D, WAKEFIELD M J, SMYTH G K, et al. Gene ontology analysis for RNA-seq:accounting for selection bias[J]. Genome Biol, 2010, 11(2): R14. DOI: 10.1186/gb-2010-11-2-r14 |
| [19] | KANEHISA M, GOTO S. KEGG:kyoto encyclopedia of genes and genomes[J]. Nucleic Acids Res, 2000, 28(1): 27–30. DOI: 10.1093/nar/28.1.27 |
| [20] |
郑琳, 郑慧玲, 刘新光. 管家基因GAPDH在肿瘤与衰老组织中作为内参应慎重[J]. 中国生物化学与分子生物学报, 2018, 34(4): 385–389.
ZHENG L, ZHENG H L, LIU X G. GAPDH should be cautiously used as an internal reference in tumor and aging tissues[J]. Chinese Journal of Biochemistry and Molecular Biology, 2018, 34(4): 385–389. (in Chinese) |
| [21] | WU N, ZHU Q, CHEN B L, et al. High-throughput sequencing of pituitary and hypothalamic microRNA transcriptome associated with high rate of egg production[J]. BMC Genomics, 2017, 18: 255. DOI: 10.1186/s12864-017-3644-3 |
| [22] | SHARMA A, SINGH D, DAS S, et al. Hypothalamic and liver transcriptome from two crucial life-history stages in a migratory songbird[J]. Exp Physiol, 2018, 103(4): 559–569. DOI: 10.1113/eph.2018.103.issue-4 |
| [23] | LIU H H, WANG J W, LI L, et al. Transcriptome analysis revealed the possible regulatory pathways initiating female geese broodiness within the hypothalamic-pituitary-gonadal axis[J]. PLoS One, 2018, 13(2): e0191213. DOI: 10.1371/journal.pone.0191213 |
| [24] | POPESKU J T, MARTYNIUK C J, DENSLOW N D, et al. Rapid dopaminergic modulation of the fish hypothalamic transcriptome and proteome[J]. PLoS One, 2010, 5(8): e12338. DOI: 10.1371/journal.pone.0012338 |
| [25] |
路婉茹, 徐红伟, 臧荣鑫, 等. 转录组测序技术在绵山羊育种中的应用研究进展[J]. 西北民族大学学报:自然科学版, 2017, 38(3): 48–52.
LU W R, XU H W, ZANG R X, et al. Application of transcriptome sequencing technology in ovine breeding[J]. Journal of Northwest University for Nationalities:Natural Science, 2017, 38(3): 48–52. (in Chinese) |
| [26] | WOODIN M A, GANGULY K, POO M M. Coincident pre- and postsynaptic activity modifies GABAergic synapses by postsynaptic changes in Cl- transporter activity[J]. Neuron, 2003, 39(5): 807–820. DOI: 10.1016/S0896-6273(03)00507-5 |
| [27] | MOBASHERI A, MOBASHERI R, FRANCIS M J, et al. Ion transport in chondrocytes:membrane transporters involved in intracellular ion homeostasis and the regulation of cell volume, free[Ca2+] and pH[J]. Histol Histopathol, 1998, 13(3): 893–910. |
| [28] | AGULHONC, SUN M Y, MURPHY T, et al. Calcium signaling and gliotransmission in normal vs. reactive astrocytes[J]. Front Pharmacol, 2012, 3: 139. |
| [29] | XIA L, DELOMÉNIE C, DAVID I, et al. Ventral hippocampal molecular pathways and impaired neurogenesis associated with 5-HT1A and 5-HT1B receptors disruption in mice[J]. Neurosci Lett, 2012, 521(1): 20–25. DOI: 10.1016/j.neulet.2012.05.046 |
| [30] | MAKKI M, SABOORI E, SABBAGHI M A, et al. Effects of selenium, calcium and calcium ionophore on human oocytes in vitro maturation in a chemically defined medium[J]. Iran J Reprod Med, 2012, 10(4): 343–348. |
| [31] |
兰道亮, 熊显荣, 柴志欣, 等. 牦牛发情期卵巢比较转录组学研究[J]. 畜牧兽医学报, 2016, 47(9): 1830–1839.
LAN D L, XIONG X R, CHAI Z X, et al. Comparative transcriptome analysis between yak and cattle estrus ovary[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(9): 1830–1839. (in Chinese) |
| [32] | SHI L, LI N, BO L, et al. Melatonin and hypothalamic-pituitary-gonadal axis[J]. Curr Med Chem, 2013, 20(15): 2017–2031. DOI: 10.2174/09298673113209990114 |
| [33] |
王淑娟, 刘文举, 王立克, 等. 褪黑激素对雌性动物生殖系统调节作用的研究进展[J]. 江苏农业科学, 2016, 44(6): 15–20.
WANG S J, LIU W J, WANG L K, et al. Advances in the Regulation of melatonin on the reproductive system of female animals[J]. Jiangsu Agricultural Sciences, 2016, 44(6): 15–20. (in Chinese) |
| [34] |
于永梅, 王生存. 季节性繁殖动物生殖的影响研究[J]. 上海畜牧兽医通讯, 2002(3): 16–17.
YU Y M, WANG S C. Effects of seasonal reproduction on reproduction of animals[J]. Shanghai Journal of Animal Husbandry and Veterinary Medicine, 2002(3): 16–17. DOI: 10.3969/j.issn.1000-7725.2002.03.005 (in Chinese) |
| [35] | LASAGA M, DEBELJUK L. Tachykinins and the hypothalamo-pituitary-gonadal axis:an update[J]. Peptides, 2011, 32(9): 1972–1978. DOI: 10.1016/j.peptides.2011.07.009 |
| [36] | BOTZ B, IMREH A, SÁNDOR K, et al. Role of pituitary adenylate-cyclase activating polypeptide and tac1 gene derived tachykinins in sensory, motor and vascular functions under normal and neuropathic conditions[J]. Peptides, 2013, 43: 105–112. DOI: 10.1016/j.peptides.2013.03.003 |
| [37] | ZHOU W Y, LI S S, LIU Y, et al. The evolution of tachykinin/tachykinin receptor (TAC/TACR) in vertebrates and molecular identification of the TAC3/TACR3 system in zebrafish (Danio rerio)[J]. Mol Cell Endocrinol, 2012, 361(1-2): 202–212. DOI: 10.1016/j.mce.2012.04.007 |
| [38] |
田志刚, 陈永艳. NK细胞的发育、分化与识别机制[J]. 中国免疫学杂志, 2009, 25(1): 31–34.
TIAN Z G, CHEN Y Y. Development, differentiation and immune recognition of natural killer cells[J]. Chinese Journal of Immunology, 2009, 25(1): 31–34. (in Chinese) |
| [39] | WANG Y Q, SHI H B, TIAN Z L, et al. Expression of TAC1 and its receptors TACR1, TACR2, TACR3 genes in different goat tissues under different photoperiods[J]. Agric Biotechnol, 2019, 8(1): 101–107. |
| [40] |
闵洁, 李梦颖, 柳赟昊, 等. 精子相关抗原6与速激肽1相互作用关系的探讨[J]. 中华男科学杂志, 2016, 22(12): 1059–1064.
MIN J, LI M Y, LIU Y H, et al. Interaction between SPAG6 and TAC1 proteins[J]. National Journal of Andrology, 2016, 22(12): 1059–1064. (in Chinese) |
| [41] | NAVARRO V M, BOSCH M A, LEÓN S, et al. The integrated hypothalamic tachykinin-kisspeptin system as a central coordinator for reproduction[J]. Endocrinology, 2015, 156(2): 627–637. DOI: 10.1210/en.2014-1651 |
| [42] | LEAL E C, CARVALHO E, TELLECHEA A, et al. Substance p promotes wound healing in diabetes by modulating inflammation and macrophage phenotype[J]. Am J Pathol, 2015, 185(6): 1638–1648. DOI: 10.1016/j.ajpath.2015.02.011 |
| [43] | MINETA H. Epigenetics and head and neck cancer[J]. Pract Oto-Rhino-Laryngol, 2016, 109(9): 599–607. DOI: 10.5631/jibirin.109.599 |
| [44] | KANDEL E. Compositions and methods for inhibiting hypoxia induced damage[J]. New Patents and Technology, 2017, 6(3): 779. |
| [45] | MOYES A J, STANFORD S C, HOSFORD P S, et al. Raised arterial blood pressure in neurokinin-1 receptor-deficient mice (NK1R-/-):evidence for a neural rather than a vascular mechanism[J]. Exp Physiol, 2016, 101(5): 588–598. DOI: 10.1113/EP085347 |
| [46] | HUANG H Z, KUZIRIAN M S, CAI X Y, et al. Generation of a NK1R-CreER knockin mouse strain to study cells involved in neurokinin 1 receptor signaling[J]. Genesis, 2016, 54(11): 593–601. DOI: 10.1002/dvg.v54.11 |
| [47] | PILLIDGE K, PORTER A J, YOUNG J W, et al. Perseveration by NK1R-/- ('knockout') mice is blunted by doses of methylphenidate that affect neither other aspects of their cognitive performance nor the behaviour of wild-type mice in the 5-Choice Continuous Performance Test[J]. J Psychopharmacol, 2016, 30(9): 837–847. DOI: 10.1177/0269881116642541 |
| [48] | PORTER A J, PILLIDGE K, STANFORD S C, et al. Differences in the performance of NK1R-/- ('knockout') and wildtype mice in the 5-choice continuous performance test[J]. Behav Brain Res, 2016, 298: 268–277. DOI: 10.1016/j.bbr.2015.10.045 |
| [49] | SIDERI A, BAKIRTZI K, SHIH D Q, et al. Substance P mediates proinflammatory cytokine release from mesenteric adipocytes in inflammatory bowel disease patients[J]. Cell Mol Gastroenterol Hepatol, 2015, 1(4): 420–432. DOI: 10.1016/j.jcmgh.2015.03.003 |
| [50] | WASOWICZ K, PODLASZ P, JAKIMIUK A, et al. Changes in the expression of NFκB and Nrf2 genes in the colon of pigs suffering from swine dysentery[J]. J Comp Pathol, 2018, 158: 139. |
| [51] | SHI H B, TIAN Z L, WANG Y Q, et al. Assessment of serum PRL, MLT, TSH, FSH and LH concentrations in the goat (Capra hircus) exposing to different photoperiod regimes[J]. Agric Biotechnol, 2018, 7(4): 110–114, 117. |
| [52] |
王玉琴, 吴至博, 张小辉, 等. 速激肽及其受体基因在哺乳动物生殖领域的研究[J]. 黑龙江畜牧兽医, 2018(2): 59–62.
WANG Y Q, WU Z B, ZHANG X H, et al. Study on the role of tachykinin and its receptor genes in mammalian reproduction[J]. Heilongjiang Animal Science and Veterinary Medicine, 2018(2): 59–62. (in Chinese) |
| [53] | NAKAO N, ONO H, YAMAMURA T, et al. Thyrotrophin in the pars tuberalis triggers photoperiodic response[J]. Nature, 2008, 452(7185): 317–322. DOI: 10.1038/nature06738 |



