2. 中国农业科学院北京畜牧兽医研究所, 北京 100193
2. Institute of Animal Science, Chinese Academy of Agricultural Sciences, Beijing 100193, China
猪肉在我国的肉类产品中占据了核心地位,是人们生活中必不可少的一部分[1]。随着人民生活水平的不断提高和饮食习惯的改变,对猪肉品质的要求也不断提高,使育种者更加注重肉质性状的改良。由于背膘厚与猪的生长性状密切相关,并与瘦肉率呈很强的负相关,一直以来是猪育种工作研究的重要性状之一[2]。因此在不影响生长速率的前提下减少背膘厚度,在猪的遗传改良中尤为重要。
高迁移率族蛋白(high mobility group protein, HMG)最早由Goodwin等[3]于1973年在牛胸腺细胞中发现,其特点为水溶性强并在聚丙烯酰胺凝胶电泳中呈现很高的迁移率。HMGA1是HMG家族的一员, 其在猪基因组上定位在7号染色体。目前大多数研究报道了HMGA1参与肿瘤的发生与发展、炎症反应、代谢等多种生物学过程[4-7]。有研究证明,在快速增殖的胚胎组织和肿瘤细胞中HMGA1呈现高的表达水平,而在正常组织、分化后细胞中的表达水平较低[8-10]。近年来,有研究指出,HMGA1对白色脂肪和棕色脂肪生成的调节起重要作用[11]。本研究团队前期对大白猪和民猪杂交F2代资源群体的背膘厚性状进行全基因组关联分析(genome-wide association study,GWAS),并对GWAS显著区间单倍型分析和选择性清除分析后,确定HMGA1为猪背膘厚的主效候选基因[12]。2015年,Qiao等[13]对白色杜洛克×二花脸猪杂交F2代资源家系的脂肪和生长性状进行GWAS分析,定位到HMGA1是猪背膘厚的重要候选基因。目前研究显示,HMGA1是猪背膘厚研究的重要候选基因,但对该基因的具体功能及对猪背膘厚的作用方式尚未见研究报道。
本研究探讨了HMGA1在猪不同组织以及生长发育各阶段不同猪种背部脂肪组织的表达谱。检测及筛选出该基因编码区和3′UTR区的SNPs,并将多态位点与猪背膘厚进行了关联分析。
1 材料与方法 1.1 试验材料本试验组织表达分析所用的3头180日龄大白猪均来自中国农业科学院北京畜牧兽医研究所昌平示范基地种猪场,屠宰后采集心、肝、肺、背最长肌、背部脂肪、卵巢、大脑组织样本,并迅速置于液氮保存,-80 ℃低温储存备用。不同日龄大白猪和民猪基因组织表达试验所用猪只(60、150、210日龄大白猪、民猪各5头)来自吉林省农业科学院畜牧分院,在各试验阶段屠宰后迅速采集猪只背部脂肪组织,将样品迅速置于液氮保存,并于-80 ℃低温储存备用。基因多态性分析所用576头大白猪×民猪F2代资源群体来自中国农业科学院北京畜牧兽医研究所昌平示范基地种猪场,采集耳组织样,提取DNA,且于240日龄屠宰,测定并记录猪6~7肋背膘厚数据。
1.2 主要试剂动物组织总RNA提取试剂盒、DNA MarkerⅠ购自北京天根生化科技有限公司。琼脂糖、反转录试剂盒(PrimeScriptTM RT Master Mix)和荧光定量试剂盒(TB GreenTM Premix Ex TaqTM)购自大连TaKaRa公司。
1.3 组织总RNA提取及cDNA的获得将采集的样本在液氮中磨碎后,按照动物组织总RNA提取试剂盒(TIANGEN)说明书进行组织总RNA提取。通过IMPLEN超微量分光光度计上260和280 nm处的吸光光度值来检测分离的总RNA浓度和纯度。对合格的RNA应用反转录试剂盒进行反转录,反应体系为10 μL:5× PrimeScript RT Master Mix 2 μL,Total RNA 500 ng,RNase-Free ddH2O补至10 μL。反应程序:37 ℃ 15 min;85 ℃ 5 s;4 ℃ ∞。
1.4 引物合成参考HMGA1基因mRNA序列(NM_001185-154.1),应用Primer Express 3.0软件设计荧光定量RCR引物,GAPDH作为内参基因,引物序列见表 1,且引物均由英潍捷基(上海)贸易有限公司合成。
|
|
表 1 荧光定量PCR引物 Table 1 Real-time PCR primer |
应用SYBR Green荧光定量PCR分别检测HMGA1基因在大白猪不同组织以及不同日龄大白、民猪背部脂肪中的相对表达量。定量检测使用ABI QuantStudioTM7 Flex实时荧光定量PCR仪和荧光定量试剂盒。反应体系为20 μL:2×TB Green Premix Ex Taq 10 μL,上下游引物(10 μmol·L-1)各0.4 μL,50×ROX Reference DyeⅡ 0.4 μL,cDNA 2 μL,ddH2O 6.8 μL。反应程序:95 ℃预变性30 s;95 ℃变性5 s, 60 ℃ 34 s,共40个循环;最后分析熔解曲线。
1.6 统计分析采用2-ΔΔCt法计算HMGA1基因的相对表达量(以GAPDH为内参基因)。使用SAS 9.4软件,通过单因素方差分析(ANOVA)进行统计学分析。
分别计算各突变位点的基因型频率、等位基因频率、多态信息含量、杂合度以及哈代-温伯格平衡检验。并以体重为协变量,使用SAS 9.4软件中的协方差分析法,并配合混合线性模型计算背膘厚在各基因型间的显著性差异。使用的模型为:Yij=μ+Gi+bW+eij。
其中,Yij表示性状测定值;μ表示群体均值;Gi表示基因型效应;W表示体重协变量效应;b为协变量的回归系数;eij为随机误差。
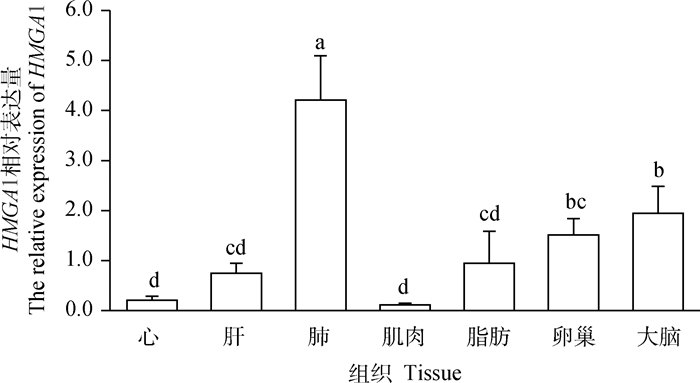
2 结果 2.1 猪HMGA1基因不同组织表达谱分析荧光定量PCR结果显示(图 1),HMGA1基因mRNA在大白猪不同组织(心、肝、肺、肌肉、脂肪、卵巢和大脑)中均有表达,且表达量在组织间存在显著差异(P < 0.05)。HMGA1在肺中的表达量最高,并显著高于其他组织(P < 0.05);其次为大脑组织,显著高于心、肝、肌肉和脂肪组织(P < 0.05),而在心和肌肉中HMGA1基因则微量表达,表达量显著低于肺、卵巢和大脑(P < 0.05)。
|
所标字母相异表示差异显著(P < 0.05),下同 Different letters indicate significant difference(P < 0.05), the same as below 图 1 HMGA1基因在大白猪不同组织中的表达水平 Fig. 1 The relative expression of HMGA1 in different tissues of Large White pigs |
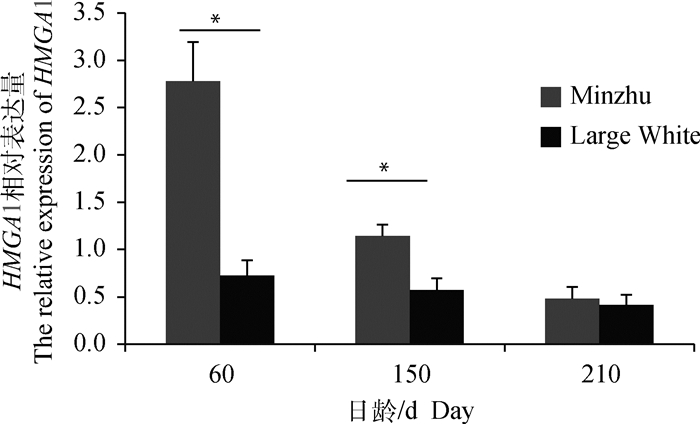
由图 2可见,HMGA1基因在猪生长发育各阶段(60、150、210日龄)均有不同程度的表达,且在前期表达量较高,随着猪的生长发育,表达量逐渐降低。在民猪和大白猪背部脂肪中HMGA1基因表达存在差异,且在60和150日龄时,HMGA1在民猪的表达量显著高于大白猪(P < 0.05),210日龄时该基因在2个猪种中的表达量差异不显著(P>0.05)。
|
*.P < 0.05 图 2 HMGA1基因在不同日龄民猪和大白猪背部脂肪中的表达 Fig. 2 The relative expression of HMGA1 in backfat of Minzhu and Large White at different ages |
对本实验室前期已获得的大白×民猪的重测序结果,利用UCSC BLAT和DNAMAN筛选HMGA1的SNPs并进行分析,结果在该基因编码区发现了13个SNPs:g.3218C>T、g.3236T>C、g.3261G>A、g.3284A>G、g.3287G>C、g.5072A>G、g.5096G>A、g.5111C>T、g.5114C>T、g.5818G>A、g.5826G>A、g.7134A>G、g.7158A>G。其中2个为错义突变位点:g.3261G>A位于第一外显子,使谷氨酸转变为赖氨酸;g.5818G>A位于外显子3,使苏氨酸转变为丙氨酸。
使用miRnada数据库分析全长1 347 bp的HMGA1基因3′UTR区潜在的miRNA结合位点,发现有3个SNPs为miRNA结合的靶点(表 2):g.7217G>C、g.7280T>C、g.8266A>G。
|
|
表 2 HMGA1基因3′UTR miRNA的结合靶点 Table 2 The miRNA binding sites in 3′UTR of HMGA1 gene |
对576头民猪×大白猪F2代资源群体的多态性位点进行基因型分型,HMGA1基因编码区及3′UTR多态位点的基因型频率和群体遗传特性见表 3。由表 3可知,该群体g.3261G>A、g.5818G>A位点显示了较低的PIC(PIC < 0.25),并且处于哈代-温伯格平衡(P>0.05)。g.7217G>C、g.7280T>C、g.8266A>G位点均属于中等多态位点(0.25 < PIC < 0.5),遗传变异处于中等水平,因此选择潜力较大。
|
|
表 3 HMGA1基因多态位点群体遗传特性 Table 3 Population genetic characteristics of HMGA1 SNPs |
以体重为协变量对HMGA1基因编码区及3′UTR的突变位点与背膘厚性状进行关联分析,结果如表 4所示,在编码区g.3261 G>A位点上,基因型与背膘厚出现了显著性关联,GG基因型个体的背膘厚显著高于GA型(P < 0.05);在g.5818G>A位点上未发现有显著关联(P>0.05)。在3′UTR区,共发现2个位点与背膘厚显著关联:g.7217G>C和g.7280T>C。g.7217G>C位点处CC基因型个体的背膘厚显著低于GG与GC型(P < 0.05),GC型个体的背膘厚显著低于GG型(P < 0.05)。g.7280T>C位点TT基因型个体的背膘厚显著低于CC型(P < 0.05),TC基因型与其他两种基因型个体的背膘厚差异不显著(P>0.05)。
|
|
表 4 HMGA1基因多态性与背膘厚的关联分析 Table 4 Association analysis between HMGA1 gene polymorphism and backfat thickness |
高迁移率族A1(HMGA1)蛋白是结合富含腺嘌呤-胸腺嘧啶(A-T)的DNA区域的核结构因子,属于非组蛋白染色质结合蛋白超家族[14]。目前,大量对HMGA1的研究集中在其对癌症细胞增殖和分化的调节作用上[15-17]。许多研究已经证实,HMGA1与多种类型肿瘤的发生和发展有关[18-19],其在多种癌症细胞中高表达。但近年来,HMGA1在脂肪生成方面的研究表明,其对脂肪细胞的生长和分化是至关重要的。有研究在血管平滑肌细胞中证明了HMGA1为PPARγ介导的转录抑制的新调节因子[20]。另有研究使用GST融合蛋白沉降技术和免疫共沉淀技术证明了HMGI-Y与Sp1和C/EBPβ相互作用[21]。此外,有研究在小鼠体内超表达HMGA1后,发现其白色脂肪及棕色脂肪含量显著下降[11],说明HMGA1与脂肪的生成和发育密切相关。类似的研究发现,HMGA1在体外脂肪生成过程中差异表达,其在未分化的3T3-L1前脂肪细胞中早期表达,在脂肪细胞成熟过程中逐渐减少[5]。本研究中,HMGA1在多组织中均有表达,在肺组织中高表达,其次是大脑组织,在心和肌肉组织中仅微量表达。同时,本研究检测了60、150和210日龄两个猪种背部脂肪中HMGA1的mRNA表达情况,结果发现,其在60和150日龄民猪中的表达量显著高于大白猪的表达量(P < 0.05),其中在60日龄时显示较高的表达和较大差异。有研究报道,在1~2月龄仔猪的脂肪发育过程中主要依靠脂肪细胞增生,而大日龄猪主要表现为脂肪细胞的增大[22]。因此,可以推测HMGA1可以调控脂肪的发育,与猪背部脂肪的生成有关。
本研究团队早期已经分析了HMGA1基因的2个SNPs对杜洛克猪的生长和饲料利用率有正向作用[23]。此外,Kim等[24-25]的研究结果表明,HMGA1基因的单核苷酸多态性(SNPs)与猪脂肪沉积性状显著相关。本研究通过对576头大白猪×民猪F2代资源群体的基因组DNA重测序结果分析,在HMGA1基因编码区发现13个SNPs位点,其中有2个为错义突变位点:g.3261G>A,g.5818G>A,两位点优势等位基因均为G。将两位点以体重为协变量与背膘厚进行关联分析,结果显示,g.3261G>A位点与背膘厚显著关联,且GG基因型个体的背膘厚显著高于GA基因型(P < 0.05)。因此,g.3261G>A位点可以作为猪背膘性状选育的潜在分子标记,但是由于本试验群体中该位点GA基因型个体数较少,只有8个,因此后续还需在其他群体中对该位点进行验证。
microRNA是一类具有调控功能的内源性非编码RNA(~22个核苷酸),其通过3′UTR区域中碱基互补配对的方式识别它们的靶mRNA,miRNA的结合通常导致翻译抑制或降解靶mRNA[26-27]。近年来大量的研究数据证实,miRNAs参与调控脂肪相关的多种生命过程,但是与人和小鼠相比,对参与猪脂肪发育的miRNAs的研究仍相对欠缺[28]。已报道的与调控猪脂肪沉积相关的miRNAs的研究为猪脂肪发育规律的研究提供了理论基础。例如,Dong等[29]的研究结果证实,miR-15a/b可能在猪脂肪形成的早期阶段起作用,且在猪前脂肪细胞分化过程中是正调控因子。Wu等[30]的研究表明,miR-146a-5p通过靶向猪原代脂肪细胞中的胰岛素受体抑制TNF-α诱导的脂肪形成。可见,研究者们越来越注重3′UTR对基因表达的调控作用。目前,对猪HMGA1基因3′调控区的研究还未曾报道。因此,本研究进一步分析了HMGA1 3′UTR区与miRNA结合的靶点。结果发现,HMGA1基因3′UTR区存在3个SNPs位点是miRNA的靶位点。对该3个位点的基因型频率和群体遗传特性分析,结果发现,g.7217G>C、g.7280T>C和g.8266A>G位点均属于中度多态位点,选择潜力较大。以体重为协变量将3个位点处的基因型与背膘厚进行关联分析,发现g.7217G>C和g.7280T>C位点与猪背膘厚显著关联,其中g.7217G>C位点CC基因型个体背膘厚显著低于GG与GC型(P < 0.05),GC型个体背膘厚显著低于GG型(P < 0.05)。g.7280T>C位点TT基因型个体的背膘厚显著低于CC型(P < 0.05),TC基因型与其他两种基因型个体的背膘厚差异不显著(P>0.05)。两位点对开发猪背膘厚的遗传标记及利用分子标记实现低背膘厚猪的选育有很大意义。
4 结论本研究结果表明,HMGA1基因与猪的脂肪发育相关。通过对HMGA1基因编码区及3′UTR的多态性与猪背膘厚关联分析,发现g.3261G>A、g.7217G>C和g.7280T>C位点的多态性与猪的背膘厚显著相关,它们对开发猪背膘厚的遗传标记及利用分子标记实现低背膘厚猪的选育有很大意义。
| [1] | ZHANG G H, GAO W L, TAO S, et al. Online ultrasonic terminal for measuring pig backfat thickness[J]. Int J Agric Biol Eng, 2018, 11(2): 190–195. DOI: 10.25165/j.ijabe.20181102.3278 |
| [2] | ZAMBONELLI P, GAFFO E, ZAPPATERRA M, et al. Transcriptional profiling of subcutaneous adipose tissue in Italian Large White pigs divergent for backfat thickness[J]. Anim Genet, 2016, 47(3): 306–323. DOI: 10.1111/age.2016.47.issue-3 |
| [3] | GOODWIN G H, SANDERS C, JOHNS E W. A new group of chromatin-associated proteins with a high content of acidic and basic amino acids[J]. Eur J Biochem, 1973, 38(1): 14–19. |
| [4] | LIU F, CHAU K Y, ARLOTTA P, et al. The HMG I proteins:dynamic roles in gene activation, development, and tumorigenesis[J]. Immunol Res, 2001, 24(1): 13–29. DOI: 10.1385/IR:24:1 |
| [5] | MELILLO R M, PIERANTONI G M, SCALA S, et al. Critical role of the HMGI(Y) proteins in adipocytic cell growth and differentiation[J]. Mol Cell Biol, 2001, 21(7): 2485–2495. DOI: 10.1128/MCB.21.7.2485-2495.2001 |
| [6] | SCHULDENFREI A, BELTON A, KOWALSKI J, et al. HMGA1 drives stem cell, inflammatory pathway, and cell cycle progression genes during lymphoid tumorigenesis[J]. BMC Genomics, 2011, 12: 549. DOI: 10.1186/1471-2164-12-549 |
| [7] | CHIEFARI E, ARCIDIACONO B, PALMIERI C, et al. Cross-talk among HMGA1 and FoxO1 in control of nuclear insulin signaling[J]. Sci Rep, 2018, 8: 8540. DOI: 10.1038/s41598-018-26968-3 |
| [8] | GIANCOTTI V, BANDIERA A, CIANI L, et al. High-mobility-group (HMG) proteins and histone H1 subtypes expression in normal and tumor tissues of mouse[J]. Eur J Biochem, 1993, 213(2): 825–832. DOI: 10.1111/ejb.1993.213.issue-2 |
| [9] | CHIAPPETTA G, BANDIERA A, BERLINGIERI M T, et al. The expression of the high mobility group HMGI (Y) proteins correlates with the malignant phenotype of human thyroid neoplasias[J]. Oncogene, 1995, 10(7): 1307–1314. |
| [10] | PIERANTONI G M, BATTISTA S, PENTIMALLI F, et al. A truncated HMGA1 gene induces proliferation of the 3T3-L1 pre-adipocytic cells:a model of human lipomas[J]. Carcinogenesis, 2003, 24(12): 1861–1869. DOI: 10.1093/carcin/bgg149 |
| [11] | ARCE-CEREZO A, GARCÍA M, RODRÍGUEZ-NUEVO A, et al. HMGA1 overexpression in adipose tissue impairs adipogenesis and prevents diet-induced obesity and insulin resistance[J]. Sci Rep, 2015, 5: 14487. DOI: 10.1038/srep14487 |
| [12] | LIU X, WANG L G, LIANG J, et al. Genome-wide association study for certain carcass traits and organ weights in a Large White×Minzhu intercross porcine population[J]. J Integr Agric, 2014, 13(12): 2721–2730. DOI: 10.1016/S2095-3119(14)60787-5 |
| [13] | QIAO R M, GAO J, ZHANG Z Y, et al. Genome-wide association analyses reveal significant loci and strong candidate genes for growth and fatness traits in two pig populations[J]. Genet Sel Evol, 2015, 47: 17. DOI: 10.1186/s12711-015-0089-5 |
| [14] | BUSTIN M, REEVES R. High-mobility-group chromosomal proteins:architectural components that facilitate chromatin function[J]. Prog Nucleic Acid Res Mol Biol, 1996, 54: 35–100, 100a, 100b. DOI: 10.1016/S0079-6603(08)60360-8 |
| [15] | ZHONG J, LIU C, CHEN Y J, et al. The association between S100A13 and HMGA1 in the modulation of thyroid cancer proliferation and invasion[J]. J Transl Med, 2016, 14: 80. DOI: 10.1186/s12967-016-0824-x |
| [16] | HUSO T H, RESAR L M. The high mobility group A1 molecular switch:turning on cancer-can we turn it off?[J]. Expert Opin Ther Targets, 2014, 18(5): 541–553. DOI: 10.1517/14728222.2014.900045 |
| [17] | ZHOU W B, ZHONG C N, LUO X P, et al. miR-625 suppresses cell proliferation and migration by targeting HMGA1 in breast cancer[J]. Biochem Biophys Res Commun, 2016, 470(4): 838–844. DOI: 10.1016/j.bbrc.2016.01.122 |
| [18] | PUCA F, COLAMAIO M, FEDERICO A, et al. HMGA1 silencing restores normal stem cell characteristics in colon cancer stem cells by increasing p53 levels[J]. Oncotarget, 2014, 5(10): 3234–3245. |
| [19] | PEGORARO S, ROS G, PIAZZA S, et al. HMGA1 promotes metastatic processes in basal-like breast cancer regulating EMT and stemness[J]. Oncotarget, 2013, 4(8): 1293–1308. |
| [20] | BLOCH M, PROCK A, PAONESSA F, et al. High-mobility group A1 protein:a new coregulator of peroxisome proliferator-activated receptor-γ-mediated transrepression in the vasculature[J]. Circ Res, 2012, 110(3): 394–405. DOI: 10.1161/CIRCRESAHA.111.253658 |
| [21] | FOTI D, IULIANO R, CHIEFARI E, et al. A nucleoprotein complex containing Sp1, C/EBPβ, and HMGI-Y controls human insulin receptor gene transcription[J]. Mol Cell Biol, 2003, 23(8): 2720–2732. DOI: 10.1128/MCB.23.8.2720-2732.2003 |
| [22] | ANDERSON D B, KAUFFMAN R G. Cellular and enzymatic changes in porcine adipose tissue during growth[J]. J Lipid Res, 1973, 14(2): 160–168. |
| [23] |
蒲蕾, 刘欣, 岳静伟, 等. 杜洛克猪HMGA1基因多态位点与生长、饲料利用性状的关联分析[J]. 中国畜牧兽医, 2016, 43(12): 3268–3274.
PU L, LIU X, YUE J W, et al. Study on HMGA1 gene polymorphism site and its association with feed efficiency related traits in Duroc pigs[J]. China Animal Husbandry & Veterinary Medicine, 2016, 43(12): 3268–3274. (in Chinese) |
| [24] | KIM K S, THOMSEN H, BASTIAANSEN J, et al. Investigation of obesity candidate genes on porcine fat deposition quantitative trait loci regions[J]. Obes Res, 2004, 12(12): 1981–1994. DOI: 10.1038/oby.2004.249 |
| [25] | KIM K S, LEE J J, SHIN H Y, et al. Association of melanocortin 4 receptor (MC4R) and high mobility group AT-hook 1 (HMGA1) polymorphisms with pig growth and fat deposition traits[J]. Anim Genet, 2006, 37(4): 419–421. DOI: 10.1111/age.2006.37.issue-4 |
| [26] | ROULEAU S, GLOUZON J P S, BRUMWELL A, et al. 3'UTR G-quadruplexes regulate miRNA binding[J]. RNA, 2017, 23(8): 1172–1179. DOI: 10.1261/rna.060962.117 |
| [27] | LINK J C, HASIN-BRUMSHTEIN Y, CANTOR R M, et al. Diet, gonadal sex, and sex chromosome complement influence white adipose tissue miRNA expression[J]. BMC Genomics, 2017, 18: 89. DOI: 10.1186/s12864-017-3484-1 |
| [28] |
陈晨, 胡雄贵, 朱吉, 等. 猪脂肪发育相关miRNAs的功能研究进展[J]. 畜牧兽医学报, 2015, 46(12): 2117–2126.
CHEN C, HU X G, ZHU J, et al. Progress on the research of miRNAs associated with fat development in pigs[J]. Acta Veterinaria et Zootechnica Sinica, 2015, 46(12): 2117–2126. (in Chinese) |
| [29] | DONG P Y, MAI Y, ZHANG Z Y, et al. MiR-15a/b promote adipogenesis in porcine pre-adipocyte via repressing FoxO1[J]. Acta Biochim Biophys Sin (Shanghai), 2014, 46(7): 565–571. DOI: 10.1093/abbs/gmu043 |
| [30] | WU D, XI Q Y, CHENG X, et al. MiR-146a-5p inhibits TNF-α-induced adipogenesis via targeting insulin receptor in primary porcine adipocytes[J]. J Lipid Res, 2016, 57(8): 1360–1372. DOI: 10.1194/jlr.M062497 |

 图 1(Fig. 1)
图 1(Fig. 1)


