2. 山东省动物生物工程与疾病防治重点实验室, 泰安 271018;
3. 潍坊科技学院, 潍坊 262700
2. Shandong Provincial Key Laboratory of Animal Biotechnology and Disease Control and Prevention, Tai'an 271018, China;
3. Weifang University of Science and Technology, Weifang 262700, China
随着生活水平的提高,人们对于猪肉的品质有了更高的要求。在生产过程中,各种应激对于猪肉品质具有重要的影响,机体受到的各种应激最终都会引起细胞的氧化应激。研究表明,氧化应激会导致体内活性氧自由基(ROS)产生过多[1],而过多的ROS如果不能被及时清除,会导致动物体内的氧化和抗氧化系统失衡,就会直接或间接地损伤细胞内蛋白、脂质、核酸,对细胞产生氧化损伤,表现为PSE肉的产生[2-3]。
为了应对氧化应激造成的损伤,生物体进化形成了完整的保护体系——抗氧化系统来清除体内多余的活性氧。包括酶促反应以及非酶促反应系统。非酶促反应主要是一些具有还原性的有机小分子,如谷胱甘肽(GSH)、维生素C等与体内的过氧化物、活性氧自由基等发生反应,从而起到抗氧化的作用;动物体内最主要也是最根本的抗氧化系统是酶促反应系统,主要抗氧化酶有超氧化物歧化酶(SOD)、谷胱甘肽过氧化物酶(GSH-Px)和过氧化氢酶(CAT)等以及Ⅱ相解毒酶, 如醌氧化还原酶-1(NQO1)等,它们在Keap1-Nrf2-ARE途径的调控下协同完成动物机体内抗氧化功能[4-5]。抗氧化酶和Ⅱ相解毒酶在动物机体内普遍都受到Kelch样环氧氯丙烷相关蛋白-1(Kelch-like ECH-associated protein 1, Keap1)-核因E2相关因子2(nuclear factor erythroid 2-related factor, Nrf2)-抗氧化反应元件(antioxidant response element, ARE)途径调控。而且Keap1-Nrf2-ARE途径是细胞应激保护中最主要的途径[6-7]。核因子Nrf2(nuclear factor erythroid 2-related factor)作为重要的转录调控因子,在调节体内氧化、抗氧化、维护机体正常生理功能方面具有重要作用,且已成为抗氧化应激的重要调控因子[8-9]。启动子是位于功能基因5′端上游的一段DNA序列,能特异性识别和结合RNA聚合酶并使之活化,从而起始转录[10-11]。对于Nrf2基因启动子的研究对探究该转录调控模式具有重要意义。
本研究通过克隆猪抗氧化应激关键基因Nrf2,并利用生物信息学方法分析Nrf2基因的结构。此外通过双荧光素酶报告系统分析Nrf2基因启动子区活性,寻找Nrf2基因的核心启动子,预测转录因子结合位点。本研究分析了Nrf2基因功能及其表达调控模式,为猪抗氧化应激的遗传改良提供了理论依据。
1 材料与方法 1.1 猪组织样品采集本研究所用大蒲莲猪试验样品采自济宁大蒲莲猪原种场,所用试验样品总数为6头,体重90 kg左右时屠宰,并采集肌肉和耳组织样,置于-80 ℃冻存备用。
1.2 主要试验试剂组织基因组DNA提取试剂盒、普通琼脂糖凝胶DNA回收试剂盒、无内毒素质粒大提试剂盒(DP117)、感受态细胞DH5α、2×Taq MasterMix购自天根生化科技有限公司;Taq DNA聚合酶、DNA Marker、Genome Walking Kit、Oligo d(T)18Primer、pMDTM18-T Vector Cloning Kit、Terminal Deoxynucleotidyl Trandferase(TdT)、dCTP、PrimeScript RT reagent Kit with gDNA Eraser反转录试剂盒、限制性内切酶Kpn Ⅰ和Xho Ⅰ、T4 DNA连接酶购自TaKaRa有限公司;Dual-luciferase Assay System试剂盒购自Promega公司;胰蛋白酶、PBS、DMEM、Opti-MEM®培养基、胎牛血清购自Gibco公司;TRIZOL Reagent、Lipofectamine® 3000转染试剂盒购自Invitrogen公司;氨苄青霉素(AMP)购自索莱宝科技有限公司;pGL3-Basic载体、pGL4-Basic载体人肾上皮细胞(293T)由实验室保存。引物合成与产物测序由生工生物工程(上海)股份有限公司完成。
1.3 试验方法 1.3.1 猪肌肉组织总RNA的提取采用Trizol法提取肌肉组织的总RNA,将提取的总RNA应用核酸分析仪测定其纯度和浓度,选用OD值为1.8~2.0的RNA作为模板。
1.3.2 猪Nrf2基因中间片段的克隆按照PrimeScript RT reagent Kit with gDNA Eraser试剂盒说明进行反转录得到cDNA。以此链为模板。根据NCBI(https://www.ncbi.nlm.nih.gov/)中预测的猪Nrf2基因的cDNA序列(XM_003133500.5),使用Primer Primer5.0软件设计所需引物,引物序列见表 1。以Nrf2-F和Nrf2-R为上下游引物扩增中间片段。
|
|
表 1 RACE引物序列 Table 1 Primer sequences for RACE |
使用引物3P2代替反转录试剂盒中的RT Primer Mix,按照PrimeScript RT reagent Kit with gDNA Eraser试剂盒说明得到用于3′RACE反应的模板链cDNA。利用Nrf2-3P1和3P2分别作为上下游引物进行第一次PCR扩增。反应体系:cDNA 1.0 μL,2×Taq MasterMix 12.5 μL,上下游引物(10 μmol·L-1)各1.0 μL,灭菌ddH2O补至20 μL。反应条件:94 ℃ 5 min;94 ℃ 30 s,55.2 ℃ 30 s,72 ℃ 1 min,35个循环;72 ℃ 10 min;4 ℃保存。然后将反应产物稀释100倍作为模板DNA,5P3和Nrf2-5P4作为上下游引物,进行第二次PCR,反应体系与第一次PCR相同,反应条件:94 ℃ 5 min;94 ℃ 30 s,50.4 ℃ 30 s,72 ℃ 1 min,35个循环;72 ℃ 10 min;4 ℃保存。2次PCR扩增3′UTR序列,产物通过1%琼脂糖凝胶电泳检测。
1.3.4 猪Nrf2基因的5′ RACE使用Oligo-d(T)18代替反转录试剂盒中的RT Primer Mix,按照试剂盒操作步骤得到用于5′RACE反应的模板链cDNA。cDNA的5′端加尾体系:5×TdT Buffer 2.5 μL,cDNA 9.0 μL,10 mmol·L-1 dCTP 0.5 μL。反应步骤:98 ℃反应1 min,冰上骤冷;加入0.5 μL末端转移酶TdT,37 ℃反应10 min;70 ℃反应10 min使TdT失活;将所获得产物使用ddH2O稀释至200 μL。以加尾后的cDNA为模板,5P1和Nrf2-5P2为上下游引物进行第一次PCR扩增。采用降落PCR:94 ℃ 5 min;94 ℃ 30 s,63 ℃ 1 min,72 ℃ 1 min,4个循环;94 ℃,30 s,61 ℃ 1 min,72 ℃ 1 min,5个循环;94 ℃ 30 s,59 ℃ 1 min,72 ℃ 1 min,5个循环;94 ℃ 30 s,57 ℃ 1 min,72 ℃ 1 min,5个循环;94 ℃ 30 s,55 ℃ 1 min,72 ℃ 1 min,5个循环;94 ℃ 30 s,54 ℃ 1 min,72 ℃ 1 min,5个循环;94 ℃ 30 s,53 ℃ 1 min,72 ℃ 1 min,5个循环;94 ℃ 30 s,52 ℃ 1 min,72 ℃ 1 min,5个循环;94 ℃ 30 s,63 ℃ 1 min,51 ℃ 1 min,4个循环;72 ℃ 10 min。4 ℃保存反应体系:cDNA 1.0 μL,2×Taq MasterMix 12.5 μL,上下游引物(10 μmol·L-1)各1.0 μL,灭菌ddH2O补至20 μL。将反应产物稀释100倍作为模板DNA,Nrf2-3P3和3P4作为上下游引物,进行第二次PCR,反应体系和条件同第一次PCR一致。将所得的PCR产物经1%琼脂糖凝胶电泳检测。
1.3.5 猪Nrf2基因的cDNA全长测序扩增使用DNA回收试剂盒回收目的DNA片段,连接pMD18-T Vector,转化至感受态细胞DH5α中,挑菌、摇菌,AMP抗性筛选阳性克隆,确定阳性产物,进行菌液测序。
1.3.6 猪Nrf2基因序列的生物信息学分析通过多种生物信息学软件对克隆得到的Nrf2基因及其5′侧翼区进行生物信息学分析,软件功能及其它具体信息见表 2。
|
|
表 2 生物信息学分析用软件 Table 2 Softwares for bioinformatics analysis |
根据所得的cDNA全长序列设计染色体步移所需的特异性引物,SP1:5′-CCACTGGTTTCTGACGGGATGTG-3′,SP2:5′-TTGTGCTGGCTGAATTGGGAGGAA-3′,SP3:5′-TCCAAAGTATGTCAATCAAATCCATG-3′。使用DNA提取试剂盒提取耳组织总基因组DNA,测定OD值定量DNA后,取适量作为模板,后续操作按Genome Walking Kit说明操作。使用兼并引物AP1为上游引物,SP1、SP2、SP3分别为下游引物进行5′侧翼序列的扩增。将特异性扩增的序列使用AliBaba和Transcription Element Search System预测转录因子结合位点,使用MethPrimer预测CpG岛,利用生物信息学软件预测启动子位点(表 2)。
1.3.8 猪Nrf2基因启动子区的PCR扩增和载体构建将PCR产物经1%琼脂糖凝胶电泳检测,确定条带后切胶回收,连接pMD18-T Vector,转化,构建重组质粒,菌液PCR筛选阳性克隆,将阳性菌液测序。以测序鉴定正确的重组质粒为模板,设计上游引物P1-F、P2-F、P3-F、P4-F、P5-F、P6-F、P7-F和下游引物P-R,并在引物上下游的5′端各引入KpnⅠ和XhoⅠ酶切位点,以及在酶切位点前加2个保护性碱基(表 3)。使用KpnⅠ和XhoⅠ分别对重组质粒和pGL3-basic双酶切,回收目的片段,用T4 DNA连接酶连接,转化,菌液酶切验证和测序,选择阳性克隆使用无内毒素质粒提取试剂盒提取质粒。依次命名为:pGL3-A、pGL3-B、pGL3-C、pGL3-D、pGL3-E、pGL3-F、pGL3-G。
|
|
表 3 引物序列 Table 3 Primer sequence |
将处于指数生长期的人肾上皮细胞(293T)均匀等量接种于24孔板中,待细胞融合度达70%~90%左右时,使用Lipofectamine®3000转染试剂盒进行转染。将构建好的报告基因质粒pGL3及内参质粒pGL 4.74按照说明书上的操作步骤转染到293T细胞,连续培养48 h后,利用双荧光素酶报告基因试剂盒检测萤火虫荧光素酶和海参荧光素酶荧光强度。每个试验组设置4个重复,pGL3-basic作为阴性对照。
1.3.10 试验设计与数据处理所有试验均设置不少于3次重复,所有数据均以“平均值±标准差”表示,采用SPSS 20统计分析软件中的One-way ANOVA进行方差分析,通过Duncan氏方法进行多重比较,显著性水平定为P < 0.05。
2 结果 2.1 大蒲莲猪Nrf2基因cDNA全长的扩增经过RT-PCR扩增,以及琼脂糖凝胶电泳检测,分别得到中间片段、5′RACE产物和3′RACE产物,所获得的序列与预期片段大小相符(图 1)。将图 1A中1~3,图 1B中2、3、5~9、12、13,图 1C中2~10条带所对应的菌液进行测序,得到Nrf2基因的部分序列信息。
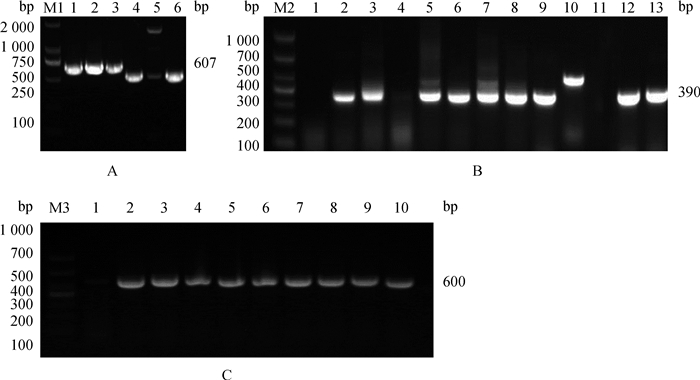
|
M1、M2、M3. DNA相对分子质量标准;图A中泳道1~6分别为Nrf2基因的部分CDS区;图B中1~13泳道为Nrf2基因的5′UTR;图C中泳道1~10分别为Nrf2基因的3′UTR M1, M2, M3. The 2 000, 1 000 and 1 000 bp DNA marker, respectively; In figureA, lane 1-6 are partial CDS region of Nrf2 gene, respectively; In figureB, lane 1-13 are 5′UTR of Nrf2 gene; In figureC, lane 1-10 are 3′UTR of Nrf2 gene 图 1 部分CDS(A)、5′RACE(B)和3′RACE(C)菌液PCR鉴定 Fig. 1 Microbial PCR identification of partial CDS(A), 5′RACE(B) and 3′RACE(C) regions |
利用DNAMAN软件对图 1中所有片段的测序结果进行序列拼接,最终获得Nrf2基因的全长cDNA,为2 358 bp,其中CDS区为1 776 bp,5′UTR为87 bp,3′UTR为495 bp(图 2)。

|
ATG为起始密码子;TAG为终止密码子 ATG is the starting codon; TAG is the termination codon 图 2 猪Nrf2基因cDNA序列 Fig. 2 cDNA sequence of Nrf2 gene of pig |
利用NCBI中的ORF Finder程序进行分析,在88 bp处有符合Kozak规则的完整开放阅读框,位于88~1 863 bp核苷酸之间,编码591个氨基酸(图 3)。
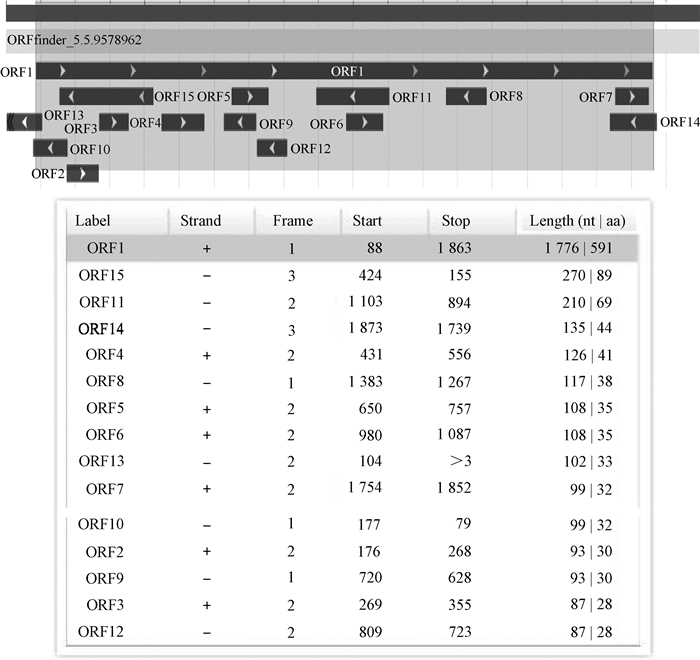
|
图 3 猪Nrf2基因ORF预测 Fig. 3 ORF finder of Nrf2 gene of pig |
使用NCBI、DNAMAN和GENSCAN对Nrf2基因核苷酸编码的氨基酸序列进行分析。ExPASy中Compute pI/Mw分析发现,Nrf2蛋白分子量为66.402 7 ku,理论等电点:pI=4.66。ProtParam分析得到,氨基酸序列中带负电氨基酸残基(Asp+Glu)101个,带正电氨基酸残基(Arg+Lys)57个。不稳定系数(Ⅱ):60.91,蛋白的亲水性平均系数(GRAVY)为-0.703。
2.2.4 Nrf2蛋白质疏水性与二级结构分析使用ProtScale程序得到猪Nrf2的蛋白质疏水性图谱(图 4)。纵坐标代表的是蛋白质疏水性分值,0值以上区域为疏水区,0值以下为亲水区。分值越高,说明此区域的疏水性越强;分值越低,说明此区域的疏水性越低;横坐标代表氨基酸的位置。在550位附近氨基酸疏水性较强。不含明显疏水区,亲水性氨基酸多于疏水性氨基酸。分析氨基酸残基的得分可知,该蛋白质不含明显的疏水区,亲水性的氨基酸基本均匀分布在整个肽链中,且多于疏水性氨基酸。
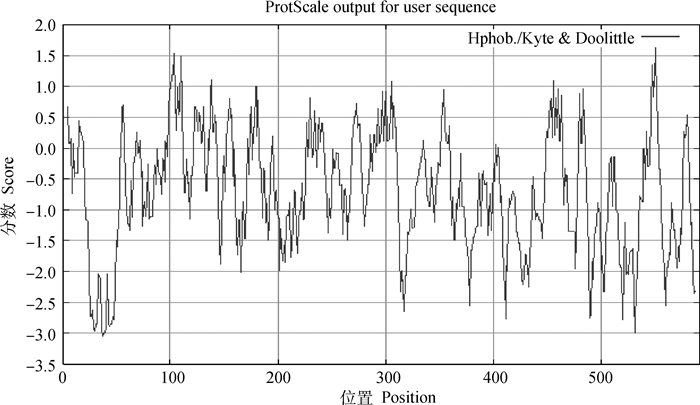
|
纵坐标代表的是蛋白质疏水性分值,横坐标代表氨基酸位置 Longitudinal coordinates represents the hydrophobicity score of proteins, abscissa represents the position of amino acids 图 4 猪Nrf2蛋白疏水性图谱 Fig. 4 The protein hydrophobicity value map of Nrf2 of pig |
使用SOPMA程序对蛋白二级结构进行预测分析,发现大部分为α-螺旋(43.49%),其间有部分的β-折叠、β-转角和无规卷曲。
2.2.5 Nrf2蛋白跨膜区和亚细胞定位预测使用Ex PASy中的TMHMM和TMPRED软件比较分析Nrf2蛋白,并未发现跨膜区。但通过SMART发现在481~545位存在碱性亮氨酸拉链(bZIP),E-value为2.75×10-12。使用targetp1.1预测Nrf2蛋白的亚细胞定位,Nrf2蛋白亚细胞定位于非叶绿体转运肽(cTP)和线粒体导肽(mTP),也不是分泌通道信号肽(SP),可能在其他位置。使用PSORTⅡ软件对Nrf2蛋白的预测发现,73.9%位于细胞核,21.7%位于细胞质,4.3%位于线粒体。
2.2.6 Nrf2蛋白信号肽与二硫键预测使用SignalP4.1软件预测Nrf2蛋白潜在的信号肽。其中C-score为剪切位点分值;S-score为信号肽分值;Y-score为综合剪切位点分值。Max. C-score为0.127,S-score小于0.5。故猪Nrf2蛋白不存在信号肽,且不是分泌性蛋白。
2.2.7 Nrf2蛋白糖基化及磷酸化位点预测通过NetNGlyc 1.0及NetOGlyc 3.1软件分别对Nrf2蛋白进行N-连接和O-连接糖基化位点预测,发现在216、225、325、334位点存在N-连接糖基化位点,且有66个O-连接糖基化位点。用Netphos 2.0预测Nrf2的磷酸化位点,发现有多个丝氨酸磷酸化位点、苏氨酸磷酸化位点、酪氨酸磷酸化位点。
2.2.8 物种间Nrf2核苷酸和氨基酸序列同源性比较将猪Nrf2基因CDS区核苷酸分别与人(NM_001145412.3)、大鼠(XM_006234396.3)、小鼠(NM_010902.4)的Nrf2基因进行比较,通过DNAMAN计算同源性,结果分别为:90.8%、82.5%、82.7%。4个物种间的同源性为89.49%。将上述4个物种Nrf2的氨基酸序列进行比对(图 5),计算同源性结果分别为:90.8%、82.5%、82.7%。物种间的同源性为88.73%。因此,通过序列比对可以得知,猪Nrf2核苷酸序列和氨基酸序列均与人的同源性最高。

|
图 5 物种间Nrf2氨基酸序列同源性比较 Fig. 5 Alignment of Nrf2 amino acid sequence among different species |
采用染色体步移技术从大蒲莲猪基因组中克隆得到Nrf2基因5′侧翼2 091 bp的DNA序列,扩增产物经1%琼脂糖凝胶电泳检测,结果如图 6A所示。切胶回收扩增产物,连接转化,菌液PCR筛选阳性菌液(图 6B)。使用在线分析软件对所得的序列进行分析预测,该序列存在一个转录起始位点(TSS),且检测到TATA-box,经预测分析不存在CpG岛(图 7)。
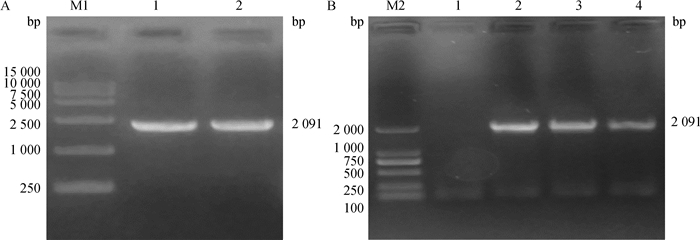
|
A. Nrf2基因启动子PCR结果;B. Nrf2基因菌液PCR鉴定。M1和M2为DNA相对分子质量标准;图A中泳道1~2和图B中泳道1~4均代表Nrf2基因5′侧翼 A. PCR result of promoter of Nrf2 gene; B. Microbial PCR identification of Nrf2 gene. M1 and M2 are 15 000 and 2 000 bp DNA marker, respectively; Both lanes 1-2 in figureA and 1-4 in figureB represent 5′ flanks of Nrf2 gene 图 6 猪Nrf2基因启动子和菌液PCR琼脂糖凝胶电泳图 Fig. 6 The agarose gel electrophoresis of PCR of Nrf2 gene promoter and microbial PCR identification |
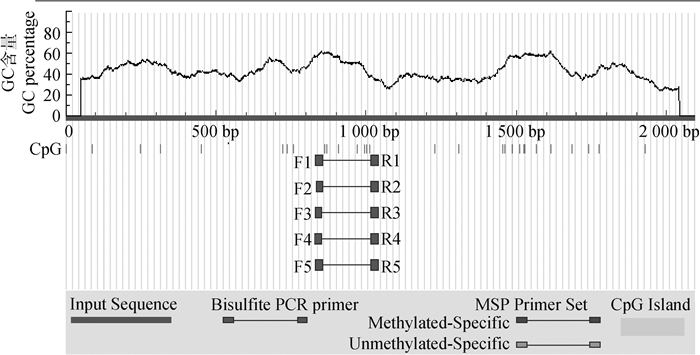
|
图 7 猪Nrf2基因5′侧翼CpG岛预测 Fig. 7 Prediction of 5′ flanking CpG island of pig Nrf2 gene |
以克隆得到的5′侧翼序列为模板,使用设计的特异性引物扩增得到不同长度的片段,产物经1%琼脂糖凝胶电泳检测,PCR产物大小与预测相符,成功得到了Nrf2基因不同长度启动子片段(图 8)。
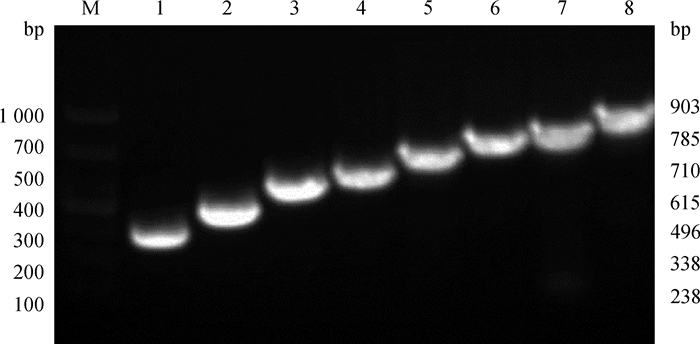
|
M. DNA相对分子质量标准;1~8.分别为Nrf2基因不同长度的5′侧翼序列PCR产物 M. The DNA marker; 1-8. The PCR products of Nrf2 gene 5′ flanking sequence 图 8 猪Nrf2基因5′侧翼不同长度片段 Fig. 8 Fragments with various lengths of 5′flanking sequence of pig Nrf2 gene |
双荧光素酶检测结果如图 9所示,其中pGL3-B相对活性最高,且显著高于pGL3-C、pGL3-D、pGL3-E、pGL3-F、pGL3-G(P<0.05),PGL3-A与pGL3-B差异不显著,pGL3-C相对活性是pGL-B的70%,pGL3-D活性是pGL3-A的50%,缺少了-785~-710 bp的区域活性下降,但pGL3-A活性却低于pGL3-B。因此推测,在-903~-710 bp区域内可能存在正调控区或增强子。
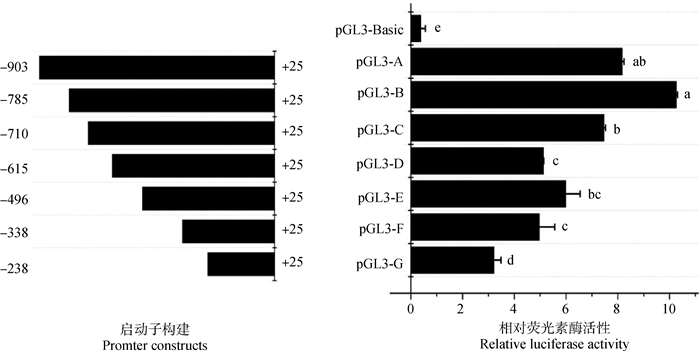
|
pGL3-A~pGL3-G.重组质粒的相对活性;pGL3-Basic为阴性对照的活性;不同小写字母表示差异显著(P < 0.05) pGL3-A-pGL3-G. The relative activities of recombinant vectors; pGL3-Basic. The relative activity of blank recombinant vector; The different lowercase letters indicate significant difference(P < 0.05) 图 9 猪Nrf2基因5′侧翼序列的启动子活性检测 Fig. 9 Detection of promoter activity of Nrf2 gene 5′ flanking sequence of pig |
氧化应激产生的ROS不及时清除会对细胞造成氧化损伤,进而可能导致衰老、癌变和畸形。Keap1-Nrf2-ARE通路是目前发现的最重要的抗氧化途径[6, 13]。Nrf2基因是与抗氧化应激有关的基因,对去除活性氧自由基和抑制肿瘤具有重要作用。除此之外,Nrf2能调节胰岛素分泌,与脂质代谢有关;Nrf2一定程度上抑制因化学药物诱发的癌症[14];在小鼠中,Nrf2可以抑制肺癌的发生[15]。因此,Nrf2基因作为重要的抗氧化应激基因,不仅与糖尿病等氧化应激相关的疾病有关,也对癌症发生有较大影响。
糖基化修饰涉及蛋白质结构和功能的改变。Nrf2中既存在O端糖基化,也有N端糖基化,表明Nrf2受到糖基化调节。N端糖基化一般发生在内质网和高尔基体,而O端糖基化仅发生在高尔基体内[16]。对于Nrfs家族中其他转录因子如Nrf1糖基化修饰研究较多,而对于Nrf2糖基化修饰研究较少,关于Nrf2糖基化修饰假设需要进一步的验证。
氨基酸序列分析发现,Nrf2在481~545位存在碱性亮氨酸拉链(bZip)序列,表明大蒲莲猪Nrf2是CNC家族的成员[17]。信号肽和一定数量的跨膜区是分泌蛋白的重要特征[18-19],核因子Nrf2既不是分泌蛋白,也不是跨膜蛋白,对Nrf2亚细胞定位预测结果也表明它主要在细胞核中发挥功能。蛋白质不稳定系数可以用来评定蛋白质的稳定性,若蛋白质不稳定系数达到40以上,表明蛋白质结构不稳定[20]。对Nrf2蛋白进行理化分析,得到其不稳定系数为60.91,表明核因子Nrf2在生物体内不能稳定存在。有研究表明,人发生氧化应激时,磷酸化可使无活性的Nrf2肽链上的丝氨酸残基结合位点构象发生改变,激活Nrf2,从而Nrf2不易被蛋白酶识别,从而不易被蛋白酶分解,增加蛋白的稳定性[21-22]。当Nrf2活化后进入细胞核内与Maf蛋白结合后才能识别ARE序列,从而起始基因转录[23],而这与上述分析结果相吻合。
有研究将不同长度启动子片段插入到报告基因,构建表达载体,通过转染细胞来检测启动子对报告基因表达水平的影响,分析对目的基因启动子区活性起重要作用的序列[24-25]。pGL3-Basic是一类常用的不含启动子但含有萤火虫荧光素酶报告基因序列的载体,其萤火虫荧光素酶活性可反映启动子区的活性[26];而质粒pGL 4.74含有海肾荧光素酶报告基因序列,可以作为内参使用[27-28]。人、小鼠等物种Nrf2均存在CpG岛,且发生甲基化,并且CpG岛甲基化修饰会抑制Nrf2蛋白的表达[29],而猪Nrf2经预测启动子区不含CpG岛,表明猪Nrf2基因启动子序列与人、小鼠存在差异,这可能也会引起功能差异,但是需要后续试验进一步验证;Nrf2基因表达于多个器官,是一种氧化应激基本表达的关键转录因子,其介导抗氧化相关Ⅱ相解毒酶的表达。本研究使用染色体步移技术扩增得到Nrf2基因5′侧翼2 091 bp(-2 065~+25 bp)长度的片段,Nrf2 5′侧翼区起始密码子ATG上游序列含有典型的TATA-box,符合真核生物基因典型启动子结构。双荧光素酶检测分析发现,-903~-710 bp(PGL3-A)存在正调控区或增强子,并且通过生物学信息学分析发现,这段区域存在SP1、AP-1、TBP、CRE-BP1等转录因子结合位点,也表明该区域存在正调控区或增强子。
4 结论本研究克隆获得了大蒲莲猪Nrf2基因的完整cDNA序列,全长2 358 bp,其中包含5′UTR 87 bp,3′UTR 495 bp以及CDS区1 776 bp,编码591个氨基酸。Nrf2基因的克隆及功能分析为进一步研究基因功能奠定了基础。本研究还克隆得到了Nrf2基因5′侧翼区2 091 bp(-2 065~+25 bp),并且发现,上游-903~-710 bp区域内可能存在正调控区或增强子,Nrf2基因启动子区活性研究为探究Nrf2基因表达调控机制提供了理论依据,为进一步探索猪抗氧化应激性能的遗传改良奠定了基础。
| [1] | MA Q. Role of nrf2 in oxidative stress and toxicity[J]. Annu Rev Pharmacol Toxicol, 2013, 53: 401–426. DOI: 10.1146/annurev-pharmtox-011112-140320 |
| [2] | HERNÁNDEZ P, ZOMEÑO L, ARIÑO B, et al. Antioxidant, lipolytic and proteolytic enzyme activities in pork meat from different genotypes[J]. Meat Sci, 2004, 66(3): 525–529. DOI: 10.1016/S0309-1740(03)00155-4 |
| [3] | LEPETSOS P, PAPAVASSILIOU A G. ROS/oxidative stress signaling in osteoarthritis[J]. Biochim Biophys Acta, 2016, 1862(4): 576–591. DOI: 10.1016/j.bbadis.2016.01.003 |
| [4] | LI S J, SONG Z Y, LIU T T, et al. Polysaccharide from Ostrea rivularis attenuates reproductive oxidative stress damage via activating Keap1-Nrf2/ARE pathway[J]. Carbohydr Polym, 2018, 186: 321–331. DOI: 10.1016/j.carbpol.2018.01.075 |
| [5] |
林谦, 邱磊, 云龙, 等. 核因子E2相关因子2调控机体抗氧化途径特性及其与畜禽的健康和肉品质的关系[J]. 动物营养学报, 2014, 26(6): 1421–1429.
LIN Q, QIU L, YUN L, et al. Nuclear factor erythroid-2-related factor 2 mediated antioxidant pathway character and its relation on health and meat quality of livestock and poultry[J]. Chinese Journal of Animal Nutrition, 2014, 26(6): 1421–1429. DOI: 10.3969/j.issn.1006-267x.2014.06.001 (in Chinese) |
| [6] | KANSANEN E, JYRKKÄNEN H K, LEVONEN A L. Activation of stress signaling pathways by electrophilic oxidized and nitrated lipids[J]. Free Radical Biol Med, 2012, 52(6): 973–982. DOI: 10.1016/j.freeradbiomed.2011.11.038 |
| [7] | JIN X L, WANG K, LIU L, et al. Nuclear factor-like factor 2-antioxidant response element signaling activation by tert-butylhydroquinone attenuates acute heat stress in bovine mammary epithelial cells[J]. J Dairy Sci, 2016, 99(11): 9094–9103. DOI: 10.3168/jds.2016-11031 |
| [8] | LEISER S F, MILLER R A. Nrf2 Signaling, a mechanism for cellular stress resistance in long-lived mice[J]. Mol Cell Biol, 2010, 30(3): 871–884. DOI: 10.1128/MCB.01145-09 |
| [9] | DENICOLA G M, KARRETH F A, HUMPTON T J, et al. Oncogene-induced Nrf2 transcription promotes ROS detoxification and tumorigenesis[J]. Nature, 2011, 475(7354): 106–109. DOI: 10.1038/nature10189 |
| [10] | ZHANG Y, XU H Y, MU J H, et al. Inactivation of ADAMTS18 by aberrant promoter hypermethylation contribute to lung cancer progression[J]. J Cell Physiol, 2019, 234(5): 6965–6975. DOI: 10.1002/jcp.v234.5 |
| [11] | TOJI N, KOSHI K, FURUSAWA T, et al. A cell-based interferon-tau assay with an interferon-stimulated gene 15 promoter[J]. Biomed Res (Tokyo, Japan), 2018, 39(1): 13–20. DOI: 10.2220/biomedres.39.13 |
| [12] | GASTEIGER E, GATTIKER A, HOOGLAND C, et al. ExPASy:the proteomics server for in-depth protein knowledge and analysis[J]. Nucleic Acids Res, 2003, 31(13): 3784–3788. DOI: 10.1093/nar/gkg563 |
| [13] | LU M C, JI J A, JIANG Z Y, et al. The Keap1-Nrf2-ARE pathway as a potential preventive and therapeutic target:an update[J]. Med Res Rev, 2016, 36(5): 924–963. DOI: 10.1002/med.21396 |
| [14] | QIN S, HOU D X. Multiple regulations of Keap1/Nrf2 system by dietary phytochemicals[J]. Mol Nutr Food Res, 2016, 60(8): 1731–1755. DOI: 10.1002/mnfr.v60.8 |
| [15] | XU C J, HUANG M T, SHEN G X, et al. Inhibition of 7, 12-dimethylbenz(a)anthracene-induced skin tumorigenesis in C57BL/6 mice by sulforaphane is mediated by nuclear factor E2-related factor 2[J]. Cancer Res, 2006, 66(16): 8293–8296. DOI: 10.1158/0008-5472.CAN-06-0300 |
| [16] | SHIBATA T, OHTA T, TONG K I, et al. Cancer related mutations in NRF2 impair its recognition by Keap1-Cul3 E3 ligase and promote malignancy[J]. Proc Natl Acad Sci U S A, 2008, 105(36): 13568–13573. DOI: 10.1073/pnas.0806268105 |
| [17] | HÄGGLUND P, BUNKENBORG J, ELORTZA F, et al. A new strategy for identification of N-glycosylated proteins and unambiguous assignment of their glycosylation sites using HILIC enrichment and partial deglycosylation[J]. J Proteome Res, 2004, 3(3): 556–566. DOI: 10.1021/pr034112b |
| [18] |
李航, 段惠军. Nrf2/ARE信号通路及其调控的抗氧化蛋白[J]. 中国药理学通报, 2011, 27(3): 300–303.
LI H, DUAN H J. Nrf2/ARE pathway and downstream antioxidant genes[J]. Chinese Pharmacological Bulletin, 2011, 27(3): 300–303. (in Chinese) |
| [19] | ZHANG C L, WANG G Z, JI Z B, et al. Molecular cloning, characterisation and mRNA expression analysis of the sheep myosin light chain 1 gene[J]. Gene, 2015, 569(1): 51–59. DOI: 10.1016/j.gene.2015.04.048 |
| [20] |
胡古月, 赵刚, 陈颖钰, 等. 支原体分泌蛋白研究进展[J]. 畜牧兽医学报, 2017, 48(9): 1572–1578.
HU G Y, ZHAO G, CHEN Y Y, et al. Progress on secretory proteins of mycoplasma[J]. Acta Veterinaria et Zootechnica Sinica, 2017, 48(9): 1572–1578. (in Chinese) |
| [21] | HOFFMAN A, TALESKI G, QIAN H, et al. Methylenetetrahydrofolate reductase deficiency deregulates regional brain amyloid-β protein precursor expression and phosphorylation levels[J]. J Alzheimers Dis, 2018, 64(1): 223–237. |
| [22] | HUANG H C, NGUYEN T, PICKETT C B. Phosphorylation of Nrf2 at ser-40 by protein kinase C regulates antioxidant response element-mediated transcription[J]. J Biol Chem, 2002, 277(45): 42769–42774. DOI: 10.1074/jbc.M206911200 |
| [23] |
李玉华, 付杰丽, 李培飞, 等. 金黄色葡萄球菌对体外保存猪精子活力及蛋白磷酸化修饰的影响[J]. 畜牧兽医学报, 2017, 48(9): 1665–1673.
LI Y H, FU J L, LI P F, et al. Effects of Staphylococcus aureus on motility and protein phosphorylation modification of boar sperm during storage in vitro[J]. Acta Veterinaria et Zootechnica Sinica, 2017, 48(9): 1665–1673. (in Chinese) |
| [24] | GRANKVIST K, MARKLUND S, SEHLIN J, et al. Superoxide dismutase, catalase and scavengers of hydroxyl radical protect against the toxic action of alloxan on pancreatic islet cells in vitro[J]. Biochem J, 1979, 182(1): 17–25. DOI: 10.1042/bj1820017 |
| [25] |
韩凤桐, 林秀坤, 刘娣, 等. 牛Sry启动子调控序列的鉴定[J]. 中国农业科学, 2010, 43(14): 2996–3004.
HAN F T, LIN X K, LIU D, et al. Identification of the regulation sequences of bovine Sry promoter[J]. Scientia Agricultura Sinica, 2010, 43(14): 2996–3004. DOI: 10.3864/j.issn.0578-1752.2010.14.019 (in Chinese) |
| [26] |
张冬杰, 汪亮, 刘娣, 等. 猪黑素皮质激素受体-4基因(MC4R)启动子克隆及其分析[J]. 农业生物技术学报, 2011, 19(4): 734–739.
ZHANG D J, WANG L, LIU D, et al. Cloning and activity analysis of promoter of pig melanocortortin-4 receptor gene (MC4R)[J]. Journal of Agricultural Biotechnology, 2011, 19(4): 734–739. DOI: 10.3969/j.issn.1674-7968.2011.04.020 (in Chinese) |
| [27] |
石元, 陈伟, 曾勇庆, 等. 猪CuZnSOD基因启动子的克隆鉴定及分析[J]. 生物工程学报, 2014, 30(2): 213–222.
SHI Y, CHEN W, ZENG Y Q, et al. Cloning and analysis of promoter of pig copper zinc superoxide dismutase gene (CuZnSOD)[J]. Chinese Journal of Biotechnology, 2014, 30(2): 213–222. (in Chinese) |
| [28] |
陈涛, 马立霞, 崔景香, 等.猪miR-331-3p过表达载体的构建及其对细胞增殖的影响[J/OL].生物工程学报, (2019-02-26).https://doi.org/10.13345/j.cjb.180434.
CHEN T, MA L X, CUI J X, et al.Construction of miR-331-3p overexpression vector and its effect on the cell proliferation[J/OL]. Chinese Journal of Biotechnology.(2019-02-26).https://doi.org/10.13345/j.cjb.180434. (in Chinese) |
| [29] |
付聪丽, 卞涛. 核转录因子E2相关因子2启动子CpG岛甲基化对其蛋白表达及COPD的影响[J]. 国际呼吸杂志, 2017, 37(19): 1498–1502.
FU C L, BIAN T. Relation of promoter CpG island methylation of Nrf2 gene with Nrf2 expression and COPD[J]. International Journal of Respiration, 2017, 37(19): 1498–1502. DOI: 10.3760/cma.j.issn.1673-436X.2017.19.014 (in Chinese) |



