2. 中国农业科学院北京畜牧兽医研究所, 北京 100193
2. Institute of Animal Science, Chinese Academy of Agricultural Sciences, Beijing 100193, China
白细胞分化抗原13(cluster of differentiation 13, CD13)也称为氨肽酶N(aminopeptidase N, APN),是一种金属离子依赖的蛋白酶。它也是一类单次跨膜的Ⅱ型膜结合金属糖蛋白,存在于细胞表面,占据膜蛋白总量的8%[1]。CD13相对分子量约150 ku, 大约由960~980个氨基酸残基构成。猪CD13由963个氨基酸残基组成,可被胰蛋白酶分解成2个亚基,分别为95 ku的氮端(氨基端)和50 ku的碳端(羧基端)。CD13蛋白由3部分构成:一个短的细胞质区、一个跨膜域和一个细胞外的外域[1]。CD13电子域由4个部分组成,C-末端所在的结构域Ⅳ最大,而猪的冠状病毒识别位置就位于结构域Ⅳ[2]。
CD13广泛分布于哺乳动物的多种组织及其细胞表面, 在中枢神经系统的突触膜、粒细胞、单核细胞、肾近曲小管上皮和肺细胞表面都有表达,尤其在小肠微绒毛表面高表达[1]。CD13基因也在各种癌症细胞中异常高表达[3-4],如非小细胞肺癌[5]、胰腺癌[6]、肝癌[7]等。肠道中,CD13主要功能是水解肽、酰胺等结构中的酰胺键, 催化蛋白质和肽的N-末端氨基酸切割从而释放出不同的N-中性氨基酸,特别是丙氨酸[8]。CD13介导的肽水解和活化对于调节几种生理过程尤为重要,例如控制血压平衡(血管紧张素)和细胞趋化性,调节心血管生成等[9]。研究表明,CD13可以降解细胞外基质蛋白参与肿瘤细胞的侵袭、生长;可以降解白细胞介素8,从而降低机体免疫功能;可以降解组织相容性复合物Ⅱ的黏附抗原决定簇,从而降低T细胞对肿瘤细胞表面抗原的识别能力,导致机体对肿瘤的免疫抑制或免疫耐受[10]。CD13也与肿瘤转移运动和肿瘤血管生成有关[11]。最近相关文献报道,CD13在调节精子功能和早期胚胎发育中起着重要作用[12-13]。CD13主要是Ⅰ型冠状病毒细胞结合受体,病毒通过与CD13结合后进入宿主细胞,引起宿主发生病毒感染[14]。早先报道,人冠状病毒229E[15]和猪传染性胃肠炎病毒(transmissible gastroenteritis coronavirus, TGEV)[16]细胞受体都是CD13。刘博奇等[17]将CD13表达出7段不同的重组蛋白,检测出TGEV的主要结合位置在36~153、349~591、592~963位氨基酸。李宝贤等[18]构建了表达CD13的真核系统,建立了稳定表达CD13的犬肾细胞系,并进一步明确了CD13是猪流性腹泻病毒(porcine epidemic diarrhea virus, PEDV)的结合受体,又通过免疫荧光与中和试验发现,CD13还具有介导PEDV复制的能力。
本研究利用CRISPR/Cas9技术制备CD13基因敲除的猪IPEC-J2细胞(猪空肠上皮细胞系)系,从而为在细胞水平研究CD13功能提供试验材料。
1 材料与方法 1.1 试验材料猪IPEC-J2细胞为华中农业大学何启盖老师惠赠,质粒载体为保存的pX330-GFP和pX330-RFP载体,感受态细胞为DH5α。
1.2 主要试剂Basic Primary Fibroblasts Nucleofector Kit转染试剂盒购自Lonza公司;胎牛血清(fetal bovine serum, FBS)、D-PBS、DMEM/F12、人重组表皮生长因子(recombinant human epidermal growth factor, EGF)、胰岛素转铁蛋白硒(insulin-transferrin-selenium, ITS)均购自Gibco公司;载体连接试剂盒、血液/细胞/组织基因组DNA提取试剂盒、无内毒素质粒大提试剂盒均购自天根生化科技(北京)有限公司;ANPEP Polyclonal Antibody购自ABClonal公司。
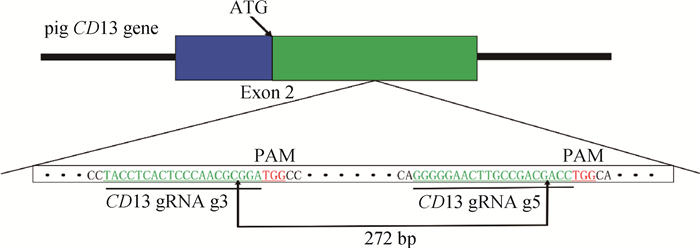
1.3 猪CD13基因双gRNA的设计选定猪CD13基因(GenBank登录号:NC_010449.5)第2外显子起始密码子ATG之后的区域作为打靶区域,利用麻省理工学院张峰实验室开发的网站(http://crispr.mit.edu)对打靶区域进行评分,从候选序列中选择2个合适的向导RNA(guide RNA, gRNA),分别命名为g3、g5。
1.4 猪CD13基因编辑载体构建将g3连接到pX330-GFP载体上命名为pX330-GFP-g3;将g5连接到pX330-RFP载体上命名为pX330-RFP-g5。根据g3、g5序列合成互补配对的寡聚核苷酸合成寡核苷酸:CD13-g3-gRNA-F:AAACACCGTACCTCACTCCCAACGCGGA,CD13-g3-gRNA-R:CTCTAAAACTCCGCGTTGGGAGTGAGGTA;CD13- g5-gRNA-F:AAACACCGGGGGGAACTTGCCGAC-GACC,CD13-g5-gRNA-R:CTCTAAAACGGTCGT-CGGCAAGTTCCCCC。
载体构建步骤:1)将每对寡核苷酸稀释至10 μmol·L-1后,各取10 μL分别98 ℃反应10 min,在自然冷却至室温的条件下进行退火;2)将实验室制备的线性化载体pX330-GFP和pX330-RFP分别与1对退火的寡核苷酸16 ℃连接1 h,随后转化DH5α感受态细胞,涂布于含氨苄的LB平板进行生长,挑取单菌落扩大培养并测序,测序引物为U6-FWD;3)将序列正确的菌落进行扩大培养,用去内毒素大提质粒试剂盒提取pX330-GFP-g3和pX330-RFP-g5质粒,所提质粒纯化后用于细胞转染。
1.5 猪IPEC-J2细胞的培养与转染转染前复苏IPEC-J2细胞于10 cm细胞培养皿中,用含有10% FBS、5 ng·mL-1 EGF、1%的ITS、1%双抗的DMEM/F12培养基培养,当细胞达到80%左右汇合度时即可进行转染。步骤按照Basic Primary Fibroblasts Nucleofector Kit(Lonza)试剂盒说明书进行操作,转染后48 h于倒置荧光显微镜下观察细胞发光情况。
1.6 流式细胞术分选双荧光标记的阳性细胞将转染48 h后的细胞用D-PBS漂洗,0.25%胰酶-EDTA溶液消化2 min,并用含20% FBS的DMEM/F12培养基中和。随后,以未转染的IPEC-J2细胞作为空白对照,使用流式细胞术对转染细胞进行双荧光筛选。取同时被激发红绿双荧光且强度最高的细胞打入含有培养基的96孔细胞培养板中,每孔1个细胞。
分别提取未转染细胞、转染后48 h的细胞和流式分选后混池细胞的基因组DNA进行PCR扩增,观察片段敲除效果。具体使用的引物和PCR扩增条件:根据g3、g5在基因组上的位置,在它们两侧分别设计引物,上下游引物分别为CD13-F: 5′-CTCCCTTCTCACCCTCACC-3′, CD13-R: 5′-AAGGTCCCGAATCCAAGC-3′。PCR反应体系:EX Taq 10 μL,CD13-F 0.5 μL,CD13-R 0.5 μL,DNA 2 μL,ddH2O 7 μL,共20 μL。扩增条件:95 ℃预变性5 min;95 ℃变性30 s,65 ℃退火30 s,72 ℃延伸1 min,共34个循环;72 ℃ 10 min。扩增产物用2%琼脂糖凝胶电泳分离检测,凝胶成像系统拍照。
1.7 单克隆细胞的培养及基因型鉴定将分选后细胞置于37 ℃、5% CO2的细胞培养箱中培养,每2 d更换1次培养基。然后将细胞转入48孔培养板中进行培养,并从每孔取少量细胞进行基因组DNA提取和PCR扩增(引物、条件同“1.6”),同时将PCR产物送北京天一辉远生物科技有限公司测序。
1.8 Western blot检测细胞中CD13蛋白的表达水平取野生型细胞和敲除型细胞分别培养,待细胞长满后,置于蛋白裂解液中,裂解细胞,冰上孵育30 min。将蛋白样品与蛋白变性buffer混匀,99 ℃变性10 min。分别取对照组5 μL、试验组10 μL样品用10% Western blot电泳胶进行电泳分离,蛋白质经电泳分离后用200 mA 2 h转移到硝酸纤维素膜上。取出硝酸纤维素膜,放置于TBST配制的5%脱脂奶粉溶液中,室温封闭2 h。用TBST洗去膜上面的多余奶粉后放入由TBST配制的1:1 000稀释一抗中,放入摇床上4 ℃过夜,TBST洗膜3次,每次10 min。用1:2 000稀释的二抗与膜在室温下孵育40 min,TBST洗膜3次,每次洗膜10 min,化学发光仪显色拍照,β-actin为内参蛋白。
2 结果 2.1 猪CD13基因的gRNA设计在猪CD13基因第2外显子设计2个gRNAs,分别命名为g3、g5(图 1)。2个gRNAs选用的前间区序列邻近基序(protospacer adjacent motif, PAM)均为TGG。2个gRNAs序列如表 1所示。
|
方框区域为第2外显子序列,蓝色方框区域为第2外显子的5′UTR序列,绿色方框为第2外显子上蛋白编码序列 The box region is the 2nd exon sequence, the blue box region is the 5′UTR sequence of the 2nd exon, and the green box is the protein coding sequence on the 2nd exon 图 1 猪CD13基因双gRNA基因编辑示意图 Fig. 1 Schematic diagram of double gRNA-directed editing porcine CD13 gene |
|
|
表 1 gRNAs靶位点核苷酸序列 Table 1 The target sequences of gRNAs |
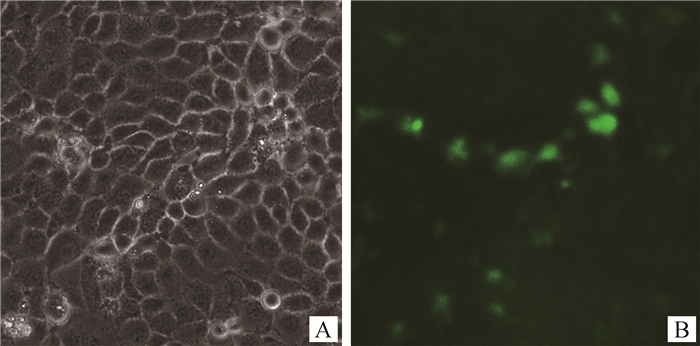
为了便于对阳性细胞基因型进行检测,将构建好的pX330-GFP-g3和pX330-RFP-g5质粒共转染IPEC-J2细胞,48 h后荧光显微镜下观察细胞发光情况,结果显示部分细胞能够表达GFP蛋白(图 2)。
|
A.明场下的细胞;B. GFP表达情况 A. Cells under bright field; B. Expression of GFP 图 2 细胞转染效率检测(100×) Fig. 2 Detection of cells transfection efficiency(100×) |
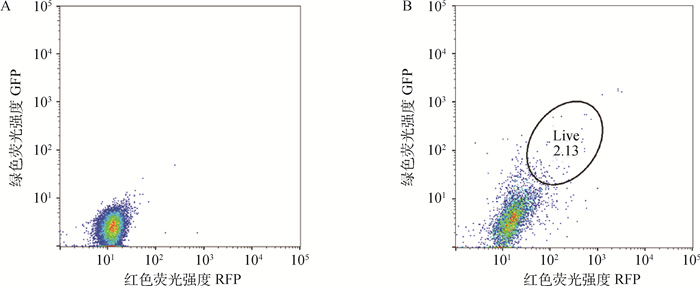
同时转染pX330-GFP-g3和pX330-RFP-g5质粒的IPEC-J2细胞,经流式细胞术分选后,取总细胞量2.13%的阳性细胞进行单细胞分离培养和混池细胞PCR检测(图 3)。
|
A.未转染细胞;B.转染48 h后细胞:圆圈中细胞表示分选得到的阳性细胞 A. Untransfected cells; B. Cells after 48 h of transfection: The cells in the circle are the positive cells sorted 图 3 流式细胞术分选阳性细胞 Fig. 3 Positive cells sorted by flow cytometry |
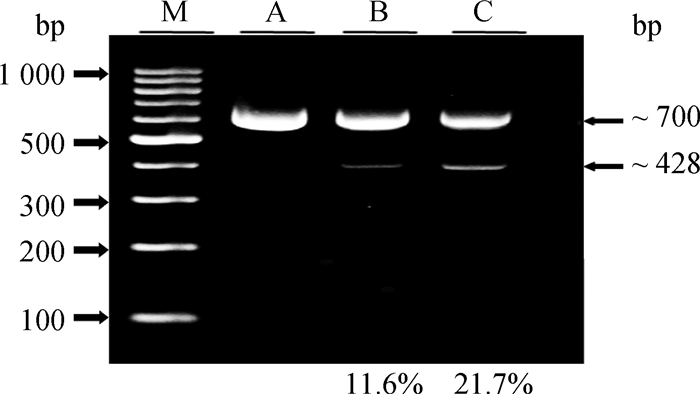
对未转染细胞、转后48 h的细胞和流式分选后混池细胞进行PCR扩增,观察PCR扩增结果。未转染细胞只在700 bp处有一条带;转后48 h的细胞和流式分选后混池细胞在700和428 bp处各有1条条带,说明同时转染双gRNA后,CD13发生片段敲除。经灰度值统计显示,富集前和富集后的片段敲除效率分别为11.6%和21.7%(图 4)。
|
M. DNA相对分子质量标准;A.未转染细胞;B.转染后48 h未分选的细胞;C.转染后48 h流式分选的细胞 M. 100 bp DNA Ladder marker; A. Untransfected cells; B. The Unsorted cells at 48 h after transfection; C. The sorted cells at 48 h after transfection 图 4 流式富集前后CD13基因敲除效率检测 Fig. 4 Detection of knockout efficiency of CD13 gene before and after flow cytometry |
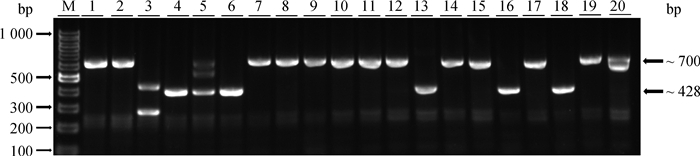
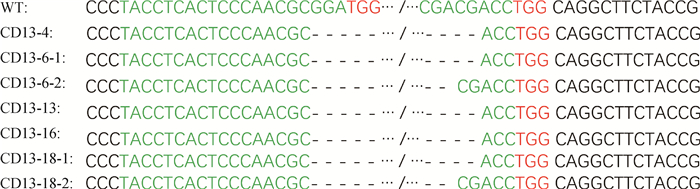
本试验共获得20株单克隆细胞。提取20株细胞的基因组DNA并进行PCR扩增,PCR产物用2%琼脂糖凝胶电泳检测。电泳结果显示,13株细胞没有发生片段缺失,条带大小为700 bp;7株细胞发生片段缺失,其中1株为单等位基因片段缺失,6株细胞为CD13双等位基因片段缺失(图 5)。为了获得野生型和纯合片段敲除细胞系,如图 5所示,选择15株细胞(5株为双等位基因片段缺失细胞:泳道4、6、13、16、18;10株为未发生片段缺失细胞:泳道1、2、8、10、11、12、15、17、19、20)的PCR产物进行测序,测序结果表明,10株未发生片段缺失的细胞都为野生型,5株双等位基因片段缺失(图 6)的细胞中有3株(泳道4、13、16)为纯合缺失272 bp,2株(泳道6、18)为杂合片段缺失(其中1条链缺失272 bp,另1条链缺失270 bp)。综上,20株细胞中1株是单等位基因片段缺失细胞,3株是双等位基因杂合片段缺失细胞,3株是双等位基因纯合片段缺失细胞。
|
M. DNA相对分子质量标准;1、2、7、8、9、10、11、12、14、15、17、19、20.未发生片段缺失;3.双等位基因杂合片段缺失;5.单等位基因片段缺失;4、6、13、16、18.双等位基因片段缺失 M. 100 bp DNA Ladder marker; 1, 2, 7, 8, 9, 10, 11, 12, 14, 15, 17, 19, 20. No fragment deletion; 3. Biallelic heterozygous fragment deletion; 5. Single allele fragment deletion; 4, 6, 13, 16, 18, Biallelic fragment deletion 图 5 单克隆细胞PCR产物电泳图 Fig. 5 The PCR products electropherogram of monoclonal cells |
|
红色标记为PAM序列,绿色标记为gRNA序列 Red represent the PAM sequences, green represent the gRNA sequences 图 6 5个双等位基因片段敲除克隆的基因型 Fig. 6 The genotypes of 5 biallelic fragment deletion clones |
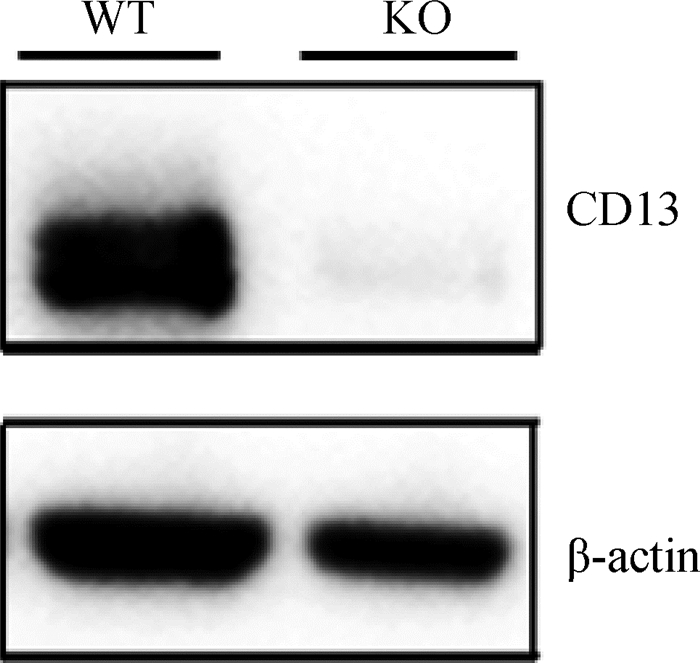
选择野生型细胞系和纯合敲除型细胞系在蛋白水平上检测CD13表达情况。Western blot结果显示,野生型细胞中CD13蛋白表达量较高,而双等位基因纯合敲除型细胞中几乎检测不到CD13的表达,表明对CD13基因进行了成功敲除(图 7)。
|
WT.野生型细胞;KO.纯合敲除细胞 WT. Wild type cells; KO. Homozygous knockout cells 图 7 CD13蛋白水平检测 Fig. 7 Detection of CD13 protein expression level |
CRISPR是原核生物抵御病毒或外源质粒入侵的获得性免疫系统,CRISPR/Cas9由gRNA通过碱基互补配对识别靶DNA序列,引导Cas9蛋白特异性切割,实现基因组特定DNA片段的敲除、敲入或替换。CRISPR/Cas9技术与锌指核酸酶技术(zinc finger nuclease, ZFN)和转录激活因子样效用物核酸酶技术(transcription activator-like effector nuclease, TALEN)相比,具有操作简单、成本低、效率高和适用范围广等优点,现已广泛应用于小鼠[19-21]、大鼠[22]、鸡[23]、猪[24-26]、牛[27]、羊[28]、猴子[29]、斑马鱼[30]、果蝇[31]、线虫[32]等动物的基因组定点修饰。
猪在我国养殖范围广泛,其不仅是重要的农业经济动物,也是生物学、医学和分子遗传学基础研究中的重要模式生物。因此,对猪基因组进行精准的编辑修饰,生产基因编辑猪,对畜牧生产、医学、生物医药等领域都具有重要价值。在畜牧生产领域已经利用CRISPR/Cas9技术获得无外源标记的高瘦肉率的MSTN基因编辑猪[33]和低背膘厚的IGF2基因编辑猪[34]。密苏里大学Prather团队制备了CD163基因敲除猪,细胞水平和个体水平攻毒试验发现其具有同时抵抗1型和2型PRRSV的能力[35]。本实验室也制备了无外源标记的CD163双等位基因编辑猪[36]。
Zhu等[37]利用CRISPR/Cas9技术设计了2个gRNAs靶向作用于IPI-2I细胞(猪回肠上皮细胞系)中CD13基因的第1外显子区域,并构建到pMID18-T质粒载体,利用脂质体转染到IPI-2I细胞并进行药物筛选获得IPI-2I细胞的CD13基因敲除单克隆细胞,并进行测序和Western blot验证,系统地建立了CD13基因敲除的IPI-2I细胞系。使用TGEV和猪德尔塔病毒(porcine deltacoronavirus, PDCoV)对CD13敲除细胞进行攻毒试验,结果表明,CD13基因敲除后的IPI-2I细胞对PDCoV的感染具有抑制作用(差异不显著);研究表明,CD13影响PDCoV病毒进入的早期,不影响IPI-2I细胞中的病毒组装和释放;对于TGEV病毒的感染则可完全阻止[37]。Whitworth等[38]利用CRISPR/Cas9技术在CD13基因第2外显子设计了6个gRNAs,并分别构建到pX330载体,电转染到猪成纤维细胞后挑取单克隆,以检测基因编辑效率。选用效率高的2号gRNA和没有编辑效果的3号gRNA,以及Cas9 mRNA共同注射到受精卵细胞质中,培养5 d后转移到代孕猪中进行妊娠,获得12头猪,其中9头猪发生基因编辑。使用PEDV和TGEV对F1代猪进行攻毒,结果表明,CD13基因敲除能完全阻止TGEV的感染,而不能阻止PEDV的感染。Li等[39]采用CRISPR/Cas9技术敲除人肝癌细胞的CD13基因和猪睾丸细胞的CD13基因,敲除后的细胞仍会感染PEDV。Nam和Lee[40]及Shirato等[41]的研究表明,在猪肾和睾丸细胞中过表达猪CD13基因略微增加了PEDV的感染力,表明猪CD13具有促进PEDV复制的作用。Ji等[42]利用siRNA技术敲降IPEC-J2细胞中CD13的表达,PEDV的感染量没有改变,TGEV的感染量下降。Kamau等[43]报道,PEDV可以识别猪的APN,其作用位点位于APN蛋白的第7结构域。以上结果表明,CD13是TGEV的受体,但作为PDCoV、PEDV的受体存在争议。
本研究在猪CD13基因第2外显子设计2个gRNAs并分别构建载体,电转染IPEC-J2细胞48 h后,通过流式细胞术分选具有双荧光标记的细胞,富集后片段敲除效率为富集前的1.87倍,表明通过富集的方法可以提高筛选效率。通过对单克隆细胞进行PCR扩增、测序,CD13基因发生片段缺失细胞株数占总单克隆细胞株数的35%,CD13双等位基因敲除细胞株数占总单克隆细胞株数的30%,双等位纯合片段敲除细胞株数占总单克隆细胞株数的15%;在发生片段缺失的7株细胞中,6株为双等位基因敲除细胞株(占比为85.7%),其中3株为双等位基因纯合缺失(占比为42.9%)。这些数据说明,本试验设计的gRNA对CD13基因实现了高效的编辑。通过对双等位基因纯合片段敲除型细胞进行Western blot检测,显示双等位基因纯合片段敲除型细胞几乎检测不到CD13蛋白,说明CD13基因被成功敲除,本研究成功获得了CD13基因敲除的IPEC-J2细胞。IPEC-J2细胞系是非转化、非致瘤性小肠细胞系,能分泌粘蛋白,产生细胞因子和趋化因子,具有与原始肠道组织相似的Toll样受体,保留了较好的上皮特性和迁移能力,是一种优良的研究肠道病原微生物的模型[44-45]。本试验将CRISPR/Cas9基因编辑技术和流式细胞术相结合,高效地获得了CD13基因纯合敲除的IPEC-J2细胞系。下一步计划使用PEDV、TGEV、PDCoV等对本试验所获得的CD13基因敲除的细胞进行攻毒试验,研究CD13与这些病原的相互作用机制。
4 结论本研究利用CRISPR/Cas9基因编辑技术成功制备出猪CD13双等位基因敲除的IPEC-J2细胞系,为研究CD13基因与猪肠道致病微生物的相互作用储备了试验材料。
| [1] | MINA-OSORIO P. The moonlighting enzyme CD13:old and new functions to target[J]. Trends Mol Med, 2008, 14(8): 361–371. DOI: 10.1016/j.molmed.2008.06.003 |
| [2] | REGUERA J, SANTIAGO C, MUDGAL G, et al. Structural bases of coronavirus attachment to host aminopeptidase N and its inhibition by neutralizing antibodies[J]. PLoS Pathog, 2012, 8(8): e1002859. DOI: 10.1371/journal.ppat.1002859 |
| [3] | RANOGAJEC I, JAKIĆ-RAZUMOVIĆ J, PUZOVIĆ V, et al. Prognostic value of matrix metalloproteinase-2 (MMP-2), matrix metalloproteinase-9 (MMP-9) and aminopeptidase N/CD13 in breast cancer patients[J]. Med Oncol, 2012, 29(2): 561–569. DOI: 10.1007/s12032-011-9984-y |
| [4] | WICKSTRÖM M, LARSSON R, NYGREN P, et al. Aminopeptidase N (CD13) as a target for cancer chemotherapy[J]. Cancer Sci, 2011, 102(3): 501–508. DOI: 10.1111/cas.2011.102.issue-3 |
| [5] | ZHANG Q, WANG J H, ZHANG H Q, et al. Expression and clinical significance of aminopeptidase N/CD13 in non-small cell lung cancer[J]. J Cancer Res Ther, 2015, 11(1): 223–228. DOI: 10.4103/0973-1482.138007 |
| [6] | PANG L, ZHANG N, XIA Y, et al. Serum APN/CD13 as a novel diagnostic and prognostic biomarker of pancreatic cancer[J]. Oncotarget, 2016, 7(47): 77854–77864. |
| [7] | DAI X M, HUANG T, YANG S L, et al. Peritumoral EpCAM is an independent prognostic marker after curative resection of HBV-related hepatocellular carcinoma[J]. Dis Markers, 2017, 2017: 8495326. |
| [8] | LIPSCOMB W, STRÄTER N. Recent advances in zinc enzymology[J]. Chem Rev, 1996, 96(7): 2375–2434. DOI: 10.1021/cr950042j |
| [9] | CORTI A, CURNIS F, ARAP W, et al. The neovasculature homing motif NGR:more than meets the eye[J]. Blood, 2008, 112(7): 2628–2635. DOI: 10.1182/blood-2008-04-150862 |
| [10] | SELI E, SENTURK L M, BAHTIYAR O M, et al. Expression of aminopeptidase N in human endometrium and regulation of its activity by estrogen[J]. Fertil Steril, 2001, 75(6): 1172–1176. DOI: 10.1016/S0015-0282(01)01779-4 |
| [11] | BHAGWAT S V, LAHDENRANTA J, GIORDANO R, et al. CD13/APN is activated by angiogenic signals and is essential for capillary tube formation[J]. Blood, 2001, 97(3): 652–659. DOI: 10.1182/blood.V97.3.652 |
| [12] | KHATUN A, RAHMAN M S, RYU D Y, et al. Elevated aminopeptidase N affects sperm motility and early embryo development[J]. PLoS One, 2017, 12(8): e0184294. DOI: 10.1371/journal.pone.0184294 |
| [13] | KHATUN A, KANG K H, RYU D Y, et al. Effect of aminopeptidase N on functions and fertility of mouse spermatozoa in vitro[J]. Theriogenology, 2018, 118: 182–189. DOI: 10.1016/j.theriogenology.2018.06.005 |
| [14] | MASTERS P S. The molecular biology of coronaviruses[J]. Adv Virus Res, 2006, 66: 193–292. DOI: 10.1016/S0065-3527(06)66005-3 |
| [15] | YEAGER C L, ASHMUN R A, WILLIAMS R K, et al. Human aminopeptidase N is a receptor for human coronavirus 229E[J]. Nature, 1992, 357(6377): 420–422. DOI: 10.1038/357420a0 |
| [16] | DELMAS B, GELFI J, L'HARIDON R, et al. Aminopeptidase N is a major receptor for the enteropathogenic coronavirus TGEV[J]. Nature, 1992, 357(6377): 417–420. DOI: 10.1038/357417a0 |
| [17] |
刘博奇, 李广兴, 王衡, 等. 猪传染性胃肠炎病毒特异性受体APN功能区的初步鉴定[J]. 黑龙江畜牧兽医, 2009(6): 21–23.
LIU B Q, LI G X, WANG H, et al. Initial identification of the domain of TGEV specific receptor APN[J]. Heilongjiang Animal Science and Veterinary Medicine, 2009(6): 21–23. DOI: 10.3969/j.issn.1004-7034.2009.06.009 (in Chinese) |
| [18] |
李宝贤, 马广鹏, 葛俊伟, 等. 猪流行性腹泻病毒功能性受体的鉴定[J]. 病毒学报, 2009, 25(3): 220–225.
LI B X, MA G P, GE J W, et al. Aminopeptidase N is a functional receptor for the PEDV coronavirus[J]. Chinese Journal of Virology, 2009, 25(3): 220–225. DOI: 10.3321/j.issn:1000-8721.2009.03.011 (in Chinese) |
| [19] | MIZUNO S, DINH T T H, KATO K, et al. Simple generation of albino C57BL/6J mice with G291T mutation in the tyrosinase gene by the CRISPR/Cas9 system[J]. Mamm Genome, 2014, 25(7-8): 327–334. DOI: 10.1007/s00335-014-9524-0 |
| [20] | WANG H Y, YANG H, SHIVALILA C S. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering[J]. Cell, 2013, 153(4): 910–918. DOI: 10.1016/j.cell.2013.04.025 |
| [21] | FUJIHARA Y, IKAWA M. CRISPR/Cas9-based genome editing in mice by single plasmid injection[J]. Methods Enzymol, 2014, 546: 319–336. DOI: 10.1016/B978-0-12-801185-0.00015-5 |
| [22] | GUAN Y T, SHAO Y J, LI D L, et al. Generation of site-specific mutations in the rat genome via CRISPR/Cas9[J]. Methods Enzymol, 2014, 546: 297–317. DOI: 10.1016/B978-0-12-801185-0.00014-3 |
| [23] |
张琦, 黄娇娇, 杨彩侠, 等. CRISPR/Cas9介导RB1基因敲除及其在鸡前脂肪细胞分化、增殖中的功能研究[J]. 畜牧兽医学报, 2016, 47(9): 1775–1784.
ZHANG Q, HUANG J J, YANG C X, et al. CRISPR/Cas9 mediated RB1 knockout and its impact on chicken preadipocytes differentiation and proliferation[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(9): 1775–1784. (in Chinese) |
| [24] | HAI T, TENG F, GUO R F, et al. One-step generation of knockout pigs by zygote injection of CRISPR/Cas system[J]. Cell Res, 2014, 24(3): 372–375. DOI: 10.1038/cr.2014.11 |
| [25] | RUAN J X, LI H G, XU K, et al. Highly efficient CRISPR/Cas9-mediated transgene knockin at the H11 locus in pigs[J]. Sci Rep, 2015, 5: 14253. DOI: 10.1038/srep14253 |
| [26] | HUANG L, HUA Z D, XIAO H W, et al. CRISPR/cas9-mediated ApoE-/- and LDLR-/- double gene knockout in pigs elevates serum LDL-C and TC levels[J]. Oncotarget, 2017, 8(23): 37751–37760. |
| [27] | GAO Y P, WU H B, WANG Y S, et al. Single Cas9 nickase induced generation of NRAMP1 knockin cattle with reduced off-target effects[J]. Genome Biol, 2017, 18: 13. DOI: 10.1186/s13059-016-1144-4 |
| [28] | CRISPO M, MULET A P, TESSON L, et al. Efficient generation of myostatin knock-out sheep using CRISPR/Cas9 technology and microinjection into zygotes[J]. PLoS One, 2015, 10(8): e0136690. DOI: 10.1371/journal.pone.0136690 |
| [29] | KANG Y, ZHENG B, SHEN B, et al. CRISPR/Cas9-mediated Dax1 knockout in the monkey recapitulates human AHC-HH[J]. Hum Mol Genet, 2015, 24(25): 7255–7264. DOI: 10.1093/hmg/ddv425 |
| [30] | VARSHNEY G K, PEI W H, LAFAVE M C, et al. High-throughput gene targeting and phenotyping in zebrafish using CRISPR/Cas9[J]. Genome Res, 2015, 25(7): 1030–1042. DOI: 10.1101/gr.186379.114 |
| [31] | GRATZ S J, UKKEN F P, RUBINSTEIN C D, et al. Highly specific and efficient CRISPR/Cas9-catalyzed homology-directed repair in Drosophila[J]. Genetics, 2014, 196(4): 961–971. DOI: 10.1534/genetics.113.160713 |
| [32] | DICKINSON D J, GOLDSTEIN B. CRISPR-based methods for Caenorhabditis elegans genome engineering[J]. Genetics, 2016, 202(3): 885–901. DOI: 10.1534/genetics.115.182162 |
| [33] |
张冬杰, 刘娣, 张旭, 等. 利用CRISPR-Cas9系统定点突变猪MSTN基因的研究[J]. 畜牧兽医学报, 2016, 47(1): 207–212.
ZHANG D J, LIU D, ZHANG X, et al. Study of pig MSTN gene point mutation based on the CRISPR-Cas9 system[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(1): 207–212. (in Chinese) |
| [34] |
吴金青, 梅瑰, 刘志国, 等. 应用SSA报告载体提高ZFN和CRISPR/Cas9对猪IGF2基因的打靶效率[J]. 遗传, 2015, 37(1): 55–62.
WU J Q, MEI G, LIU Z G, et al. Improving gene targeting efficiency on pig IGF2 mediated by ZFNs and CRISPR/Cas9 by using SSA reporter system[J]. Hereditas, 2015, 37(1): 55–62. (in Chinese) |
| [35] | WELLS K D, BARDOT R, WHITWORTH K M, et al. Replacement of porcine CD163 scavenger receptor cysteine-rich domain 5 with a CD163-like homolog confers resistance of pigs to genotype 1 but not genotype 2 porcine reproductive and respiratory syndrome virus[J]. J Virol, 2017, 91(2): e01521–16. |
| [36] |
魏迎辉, 刘志国, 徐奎, 等. CD163双等位基因编辑猪的制备及传代[J]. 中国农业科学, 2018, 51(4): 770–777.
WEI Y H, LIU Z G, XU K, et al. Generation and propagation of cluster of differentiation 163 biallelic gene editing pigs[J]. Scientia Agricultura Sinica, 2018, 51(4): 770–777. (in Chinese) |
| [37] | ZHU X Y, LIU S D, WANG X L, et al. Contribution of porcine aminopeptidase N to porcine deltacoronavirus infection[J]. Emerg Microbes Infect, 2018, 7: 65. |
| [38] | WHITWORTH K M, ROWLAND R R R, PETROVAN V, et al. Resistance to coronavirus infection in amino peptidase N-deficient pigs[J]. Transgenic Res, 2019, 28(1): 21–32. DOI: 10.1007/s11248-018-0100-3 |
| [39] | LI W T, LUO R, HE Q G, et al. Aminopeptidase N is not required for porcine epidemic diarrhea virus cell entry[J]. Virus Res, 2017, 235: 6–13. DOI: 10.1016/j.virusres.2017.03.018 |
| [40] | NAM E, LEE C. Contribution of the porcine aminopeptidase N (CD13) receptor density to porcine epidemic diarrhea virus infection[J]. Vet Microbiol, 2010, 144(1-2): 41–50. DOI: 10.1016/j.vetmic.2009.12.024 |
| [41] | SHIRATO K, MAEJIMA M, ISLAM M T, et al. Porcine aminopeptidase N is not a cellular receptor of porcine epidemic diarrhea virus, but promotes its infectivity via aminopeptidase activity[J]. J Gen Virol, 2016, 97(10): 2528–2539. DOI: 10.1099/jgv.0.000563 |
| [42] | JI C M, WANG B, ZHOU J Y, et al. Aminopeptidase-N-independent entry of porcine epidemic diarrhea virus into Vero or porcine small intestine epithelial cells[J]. Virology, 2018, 517: 16–23. DOI: 10.1016/j.virol.2018.02.019 |
| [43] | KAMAU A N, PARK J E, PARK E S, et al. Porcine amino peptidase N domain Ⅶ has critical role in binding and entry of porcine epidemic diarrhea virus[J]. Virus Res, 2017, 227: 150–157. DOI: 10.1016/j.virusres.2016.10.004 |
| [44] | HE C M, DENG J, HU X, et al. Vitamin A inhibits the action of LPS on the intestinal epithelial barrier function and tight junction proteins[J]. Food Funct, 2019, 10(2): 1235–1242. DOI: 10.1039/C8FO01123K |
| [45] | KARIMI S, JONSSON H, LUNDH T, et al. Lactobacillus reuteri strains protect epithelial barrier integrity of IPEC-J2 monolayers from the detrimental effect of enterotoxigenic Escherichia coli[J]. Physiol Rep, 2018, 6(2): e13514. DOI: 10.14814/phy2.13514 |



