2. 中国农业科学院北京畜牧兽医研究所, 北京 100193
2. Institute of Animal Science, Chinese Academy of Agricultural Sciences, Beijing 100193, China
藏羊(Tibetan sheep)又称藏系羊,是中国三大粗毛绵羊品种之一,形成历史悠久。据薄吾成[1]考究,藏羊是由古羌人驯化的羌羊流传而来,原产于陕西西部和甘肃大部,后随着羌人的迁徙,顺着青藏高原东端横断山脉的河谷南下,经世代繁衍逐渐扩散于我国的西藏、青海、甘肃、云南、四川等高海拔的青藏高原地区,其中青海省是藏羊的主要产区。藏羊对青藏高原恶劣的生态环境具有很强的适应能力,抗病力强,可饲范围广,是青藏高原地区农牧民的主要生产和生活资料[2]。根据《中国畜禽遗传资源志-羊志》[3]记载,把我国藏区的羊种资源按其生态环境,并结合其生产、经济特点,主要分为高原型(草地型)和山谷型两大类,其中以高原型为主。高原型藏羊四肢结实,体格较大,体型似长方形;而山谷型藏羊体格较小,结构紧凑似圆桶状。藏羊头部多带有黑色、褐色或杂色斑块,这一保护机制可使机体免受强烈紫外线辐射并防止产生雪盲[4]。由于各地湿热条件差别明显、地势高低悬殊以及人工选育等,不同区域的藏系绵羊在体型、外貌、生产性能[5]和生态适应性[6-7]等方面存在明显的差异,形成了各具特色的地域类型和亚型。目前,有关藏系绵羊的表型资料不全,更缺乏来自遗传学和分子生物学方面的信息,对藏系绵羊品系分化方面的研究仍处于较低的水平。
随着测序和基因分型技术的迅猛发展,从过去低通量、耗时的限制性片段多态(RFLP)标记[8-10]到如今高通量、高密度的单核苷酸多态性(SNP)标记[11-14],基因检测效率大幅提高,为追踪不同群体经历的选择提供了可能。本研究使用Affymetrix Ovine SNP 600K芯片对我国5个省区的20个藏系绵羊群体(共384个个体)进行全基因组检测,以期从分子层面上对藏系绵羊品种进行种质资源分析,为研究藏系绵羊各群体间的亲缘关系及遗传分化提供参考。
1 材料与方法 1.1 试验动物和样品采集选取西藏、青海、甘肃、云南和四川地区的20个藏系绵羊群体共计384只成年绵羊作为研究对象,群体内的个体来自多个农户养殖场的不同家系,各群体内的个体公母各半,采集耳组织样或血样带回实验室,-80 ℃保存备用,具体采样信息见表 1。
|
|
表 1 样品采集信息 Table 1 Sample collection information |
利用Promega基因组DNA提取试剂盒提取血样和耳组织样的DNA,要求单个基因组DNA样品的浓度大于50 ng·μL-1,且单个样品的总量大于3 μg。用Affymetrix Ovine 600K芯片进行基因型测定,对获得的样本和SNP用Plink[15]软件进行质量控制,质控标准为个体检出率(call rate)大于0.9、位点检出率大于0.9、最小等位基因频率(MAF)大于0.05、位点要符合哈迪温-伯格平衡(HWE)。质控参数:--mind 0.1 --geno 0.1 --maf 0.05 --hwe 1e-5。
1.3 藏系绵羊群体遗传多样性分析群体遗传多样性是指一个群体内所有个体的遗传变异总和,自然环境的选择以及通过基因交流向群体中引入新的等位基因的人工选育均能影响群体遗传多样性。本研究通过Plink软件(参数:--het)计算藏系绵羊群体的杂合度和近交系数,了解藏系绵羊群体的多样性。
1.3.1 杂合度杂合度是指1个位点具有2个或2个以上等位基因的状态,用于衡量群体中的遗传变异程度[16]。杂合度越高,表明群体内遗传多样性系数越高。杂合度又分为观测杂合度(Ho)和期望杂合度(He),观测杂合度是随机抽取的2个样本的等位基因不相同的概率,期望杂合度是根据种群内当前优势等位基因的分布频率推算出的理论值,计算公式:
| $He = \sum\limits_{i = 1}^m {\left( {1 - \sum\limits_{j = 1}^m {P_{ij}^2} } \right)} /n$ |
其中,m为基因座位数;n为每个基因座位的等位基因数;Pij为第i基因座位第j等位基因的频率。
1.3.2 近交系数近交系数是指形成合子的2个配子来自同一共同祖先的概率。传统的计算方法是通过通径系数原理,在假定群体中祖先没有亲缘关系的前提下计算系谱中的径路系数。即根据近亲交配的世代数,计算某个体同一位点上的两个等位基因来自共同祖先的概率:
| $F = \sum {\left[ {{{\left( {\frac{1}{2}} \right)}^{{n_1} + {n_2} + 1}}\left( {1 + {F_A}} \right)} \right]} $ |
其中,n1表示父本到共同祖先的代数;n2表示母本到共同祖先的代数;FA表示共同祖先自身的近交系数。
1.4 藏系绵羊群体结构分析为避免互相连锁的SNPs对群体结构分析的影响,本研究选取经Plink修剪后相互之间连锁不平衡系数(r2)小于0.2的SNP进行主成分分析(PCA)、进化树分析(NJ-tree)和群体结构分析(STRUCTURE)。参数:indep-pairwise 50 5 0.2。
1.4.1 主成分分析(PCA)主成分分析(principal components analysis)是指将一组可能存在多元相关的变量转化为线性不相关的变量,从多元因素中选出主要影响因素进行分析。群体进化中,PCA结果能够揭示群体结构,常用于校正群体分层[17-18]。本研究通过Plink软件(参数:--pca)进行主成分分析,输出结果用R软件画图展现。
1.4.2 系统发育树构建(NJ-tree)进化树是以类似树状分支的图形分析各种(类)生物之间亲缘关系,每一个结点表示一个分类学单元(属、种群、个体等),结点间的距离能够反映彼此间的分离程度。本研究基于贝叶斯模型构建的遗传距离矩阵进行进化树分析(参数:--distance-matrix),由MEGA[19]软件编辑和输出进化树图形,研究不同藏系绵羊群体间的进化关系。
1.4.3 群体结构(STRUCTURE)群体结构分析主要是研究遗传变异导致各群体产生分化的一种动态变化规律,能够根据不同品种的分化走向确定品种所属类型[20]。通过假定不同的祖先来源数目K,推断群体中所有个体的祖先成分,以此来判断各个个体的归属情况。本研究使用ADMIXTURE[21]软件基于贝叶斯模型进行STRUCTURE分析,利用各位点的等位基因及基因型数据来构建聚类模型,从群体层面上研究所有群体的分层情况。
2 结果 2.1 藏系绵羊样品DNA分型及质控384个样品DNA的分型结果显示,大部分样本的位点检出率达到94%以上,检测到604 554个SNPs位点;质控后,10个个体被去除(均属浪卡子绵羊群体),剩余374只绵羊和423 178个SNPs位点用于后续分析。
2.2 藏系绵羊群体遗传多样性分析对西藏、青海、甘肃、云南和四川地区的20个藏系绵羊群体384个个体进行杂合度和近交系数分析,结果如表 2所示。这20个藏系绵羊群体期望杂合度相差不大,均在0.326 0左右。其中,西藏浪卡子绵羊(LKZ)的观测杂合度最低,为0.235 0,近交系数最高,为0.279 6;青海山谷型藏羊(SG)的观测杂合度最高,为0.323 4,近交系数最低,为0.009 0。整体而言,西藏和云南地区藏系绵羊的遗传多样性相对较低,近交系数相对较高,而青海、甘肃、四川地区藏系绵羊的遗传多样性相对较高,近交系数相对较低。这可能是山谷型藏系绵羊分布地域狭窄,加之被毛毛质较差和体格较小,近年来其种群资源量明显下降,因此表现出较低的遗传多样性[22]。
|
|
表 2 藏系绵羊群体的杂合度和近交系数分析 Table 2 Analysis of heterozygosity and inbreeding coefficient of Tibetan sheep populations |
质控后的数据文件经Plink修剪后剩余124 464个相互之间连锁不平衡系数(r2 < 0.2)的SNPs用于藏系绵羊的群体关系分析。
2.3.1 藏系绵羊群体的主成分分析对西藏、青海、甘肃、云南和四川地区的20个藏系绵羊群体进行主成分分析,结果如图 1A所示,PC1能够解释12.64%的遗传变异,很明显地将云南地区的藏系绵羊(宁蒗绵羊NL、剑川绵羊JC)以及四川地区的贡嘎绵羊GG从所有群体中区分开;PC2能够解释9.92%的遗传变异,在PC2正值方向,西藏地区的左贡绵羊ZG单独聚在一起,展现出较高的分化程度,PC2负值方向,青海地区的黑藏羊HZ个体较分散,但能与其他藏羊群体区别开,山谷型藏羊SG亦表现出一定程度的分化趋势。而剩余的14个藏系绵羊群体相对聚集在一起,分化程度较低。提取这14个相对分化程度较低的群体的基因组信息进行主成分分析,结果发现(图 1B),PC1能解释10.60%的遗传变异,将西藏地区的藏系绵羊群体(除了阿旺绵羊AW)与其他地区的藏系绵羊区分开,而青海、甘肃、四川地区的8个藏系绵羊群体聚集在一起。进一步提取青海、甘肃、四川相对聚集的8个藏系绵羊群体以及西藏阿旺绵羊的基因组信息进行单独主成分分析,结果如图 1C所示,阿旺绵羊相对单独聚集,而其他8个藏羊群体混杂在一起,较难识别群体结构,可能存在较近的亲缘关系[23]。
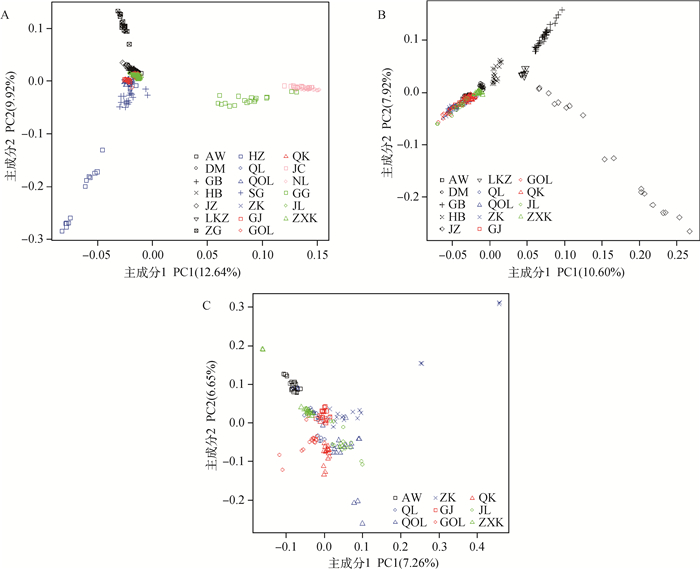
|
A. 20个群体的PCA;B. 14个群体的PCA;C. 9个群体的PCA。黑色、蓝色、红色、粉色、绿色标记分别代表西藏、青海、甘肃、云南、四川地区的藏系绵羊 A. PCA of 20 populations; B. PCA of 14 populations; C. PCA of 9 populations. Black, blue, red, pink and green markers represent Tibetan sheep in Tibet, Qinghai, Gansu, Yunnan and Sichuan regions, respectively 图 1 藏系绵羊群体的主成分分析 Fig. 1 Principal component analysis of Tibetan sheep populations |
系统发育树结果显示(图 2),云南地区的藏系绵羊(宁蒗绵羊、剑川绵羊)和四川地区的贡嘎绵羊聚集在一支,与其他藏系绵羊群体遗传距离较远,青海地区的黑藏羊、山谷型藏羊以及西藏地区的左贡绵羊也显示出相对较高的分化程度,这一结果与PCA结果一致。西藏地区的藏系绵羊群体基本能与其他地区的藏系绵羊分开,而青海地区的欧拉羊QOL、泽库羊ZK,甘肃地区的甘加羊GJ、欧拉羊GOL、乔科羊QK以及四川地区的贾洛绵羊JL混杂在一起,分化程度较低,存在基因交流的可能。四川甘孜州的扎西卡绵羊ZXK部分与西藏群体较近,部分与青海祁连绵羊QL聚集在一起。
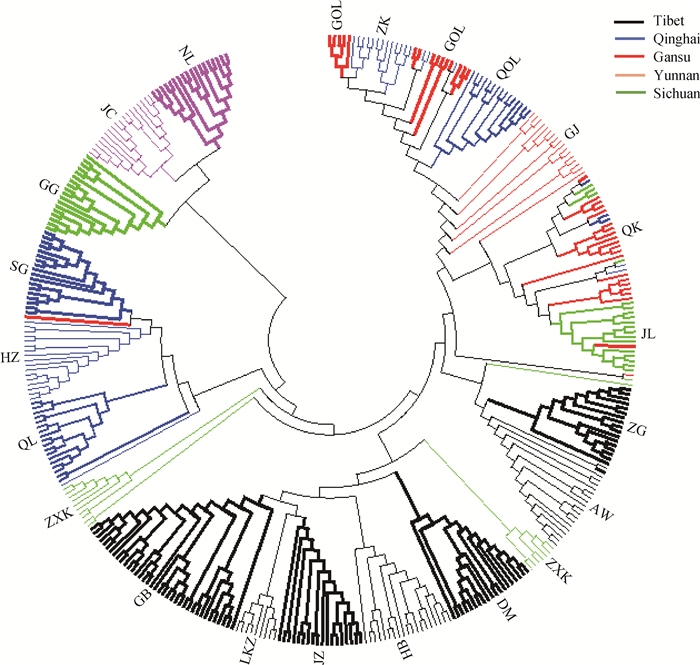
|
图 2 藏系绵羊群体的邻接树分析 Fig. 2 NJ-tree analysis of Tibetan sheep populations |
ADMIXTURE软件的交叉验证程序能够帮助选择最佳K值,与其他K值相比,良好的K值将表现出较低的交叉验证误差。在默认设置下(--cv)进行交叉验证,通过比较不同K值下交叉验证错误估计的标准误差可以发现(图 3),CV error(K=6)是最佳的建模选择。
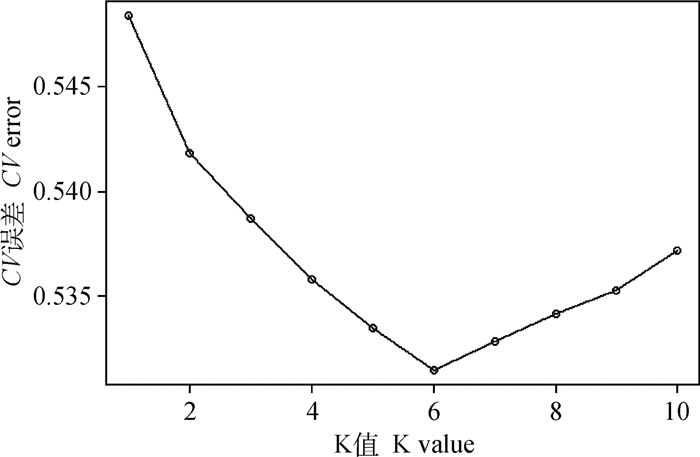
|
图 3 藏系绵羊群体的交叉验证 Fig. 3 Cross-validation of Tibetan sheep populations |
STRUCTURE结果显示(图 4),K=2时,云南地区的藏系绵羊(宁蒗绵羊、剑川绵羊)和四川地区的贡嘎绵羊首先从群体中分出来;K=3~5时,青海黑藏羊、西藏左贡绵羊、青海山谷型藏羊依次从群体中分开,分化程度相对较高。这一结果与PCA、NJ-tree保持一致。K=6时,西藏地区的藏系绵羊(岗巴绵羊GB、江孜绵羊JZ、浪卡子LKZ)表现出一定的分化趋势。但不论K为何值时,青海、甘肃、四川地区的草地型藏羊都没有分离,说明这几个群体的起源应该相同,没有明显的分化。
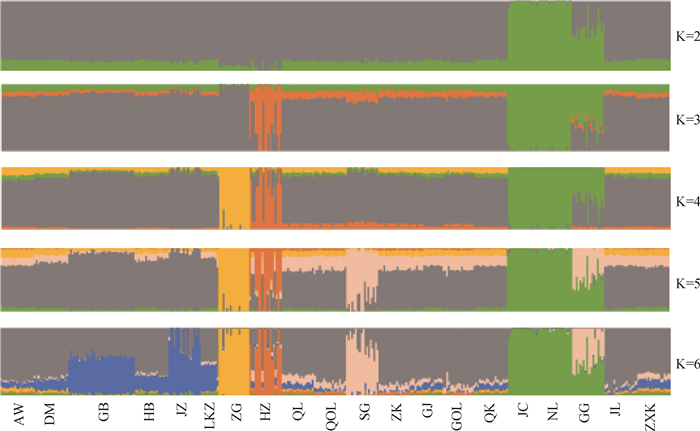
|
图 4 藏系绵羊群体的STRUCTURE分析 Fig. 4 STRUCTURE analysis of Tibetan sheep populations |
藏系绵羊主要分布在青藏高原及其毗邻的川、滇、甘等高寒地区,地域环境的特殊性和复杂性使得藏系绵羊形成了各具特色的地域类型和亚型,存在地域水平的多样性。青海省的藏羊主要分为高原型、山谷型、欧拉型和贵德黑裘皮羊(黑藏羊),后根据青海省不同区域的生态条件特点,青海藏羊又被分为欧拉型藏羊、白藏羊(祁连藏羊)、黑藏羊[24]。甘肃省的藏羊主要分为高原型、欧拉型和岷县黑裘皮羊,其中高原型藏羊主要分布于甘肃省甘南藏族自治州的各县,按其地域和特性又分为甘加型、欧拉型和乔科型3个亚型[25]。欧拉羊主产于甘肃省的玛曲县及毗邻地区以及青海省河南蒙古族自治县、久治县等地,总数达到70多万只,其中青海省约有54万只,甘肃省约有18万只。云南省的藏羊主要为山谷型,主要分布于云南省邵通市、曲靖市、丽江市及保山市腾冲县等地。四川省藏羊主要分为高原型和山谷型,其中高原型藏羊中心产区分布在若尔盖县辖曼乡唐克镇、辖曼牧场等,红原、阿坝、松潘、壤塘等县均有分布,目前存栏量超过70万只;山谷型藏羊中心产区分布在阿坝州小金、金川、茂县、松潘等县,黑水、理县、马尔康、壤塘等有少量分布,存栏量在6万只左右。山谷型藏羊在藏区各省都有分布,覆盖范围很广泛,根据地域命名法可分为阿旺绵羊、多玛绵羊、岗巴绵羊、霍尔巴绵羊、青海黑藏羊、宁蒗绵羊、贾洛绵羊、贡嘎绵羊等多个地方群体,主要分布在西藏自治区和青海的班玛、昂次,云南的昭通、曲靖、丽江,四川省的阿坝南部等地[26]。对于藏系绵羊的品种的划分和认知大都停留在地域和形态学水平,缺乏来自遗传学和分子生物学方面的信息支撑,难以对藏系绵羊的品系分化和形成进行统一。
本研究以西藏、青海、甘肃、云南和四川地区的20个藏系绵羊品种为研究对象,基于600K芯片数据分析藏系绵羊间的遗传关系和遗传多样性。遗传多样性结果显示,西藏、云南地区藏系绵羊的近交系数明显普遍高于青海、甘肃和四川地区的藏系绵羊。这可能与近年来对草地型藏羊的本品种选育有关,通过品种改良或引入优良性状品种进行杂交,用以提高草地型藏羊的生产性能[27-28],因此导致主产草地型绵羊的青海、甘肃和四川地区的藏系绵羊有着较高的遗传多样性。而西藏、云南地区相对保守,并且受到地理隔离等影响,导致资源形成一定程度的地理隔离。而群体关系分析也反映了这一特点,通过PCA、NJ-tree和STRUCTURE结果可以看出,西藏和云南地区的藏系绵羊基本能保持地域特异性,各个藏羊资源独立形成聚类,可以单独作为新的藏羊资源品种。另外,青海的山谷型藏羊和黑藏羊也单独形成群体聚类,也可以单独作为新的藏羊资源品种。
相反,从PCA、NJ-tree结果可以看出,青海、甘肃和四川地区的草地型藏羊(包括青海欧拉羊、泽库羊、甘加羊、甘肃欧拉羊、乔科羊、贾洛绵羊)在遗传距离、群体结构中差异不大,显示混乱聚集;四川的扎西卡绵羊部分与西藏群体聚集,部分与青海祁连绵羊聚集,表现出一定的群体内分化,这一结果与前期研究相符。杨晓军和赵有璋[29]利用RAPD技术对青海和甘肃的8个藏系绵羊群体进行遗传多样性分析时也指出,通过比较群体间的遗传距离发现,青海和甘肃地区的草地型藏羊亲缘关系较近,存在一定的基因交流,并且从UPGMA法构建的系统发育树结果看出,欧拉型和甘加型藏羊是由较早分化出来的乔科型藏羊或类似群体中分化而来,分化时间不长。徐琳娜[30]利用微卫星标记技术也证实,聚类分析中青海草地型藏羊和甘肃甘加羊、欧拉羊、乔科羊聚为一类,与青海黑藏羊分开。
STRUCTURE结果也显示,青海、甘肃和四川地区的草地型藏羊难以分离,可能存在相似的进化历程,没有明显的分化。因此可以基本断定,青海、甘肃和四川地区的草地型藏羊能够归为一种类型。近期,欧拉羊新资源认定,也将青海和甘肃的欧拉羊合并,也说明本研究的结果与基于表型和性能测定的资源认定结果相符合,为新资源的鉴定提供了有效的技术支撑。
4 结论西藏和云南地区的藏系绵羊群体基本能保持地域特异性,相互独立聚集,遗传多样性较低。而青海、甘肃和四川地区的草地型绵羊聚类混乱,在遗传距离和群体结构中差异不大。
| [1] |
薄吾成. 试论藏羊渊源[J]. 西北农林科技大学学报:自然科学版, 1986(2): 79–82.
BO W C. On the origin of Tibetan sheep[J]. Journal of Northwest A&F University:Natural Science Edition, 1986(2): 79–82. (in Chinese) |
| [2] |
向泽宇, 王长庭. 青藏高原藏羊遗传资源的现状, 存在问题及对策[J]. 中国畜牧兽医文摘, 2011, 27(2): 1–4.
XIANG Z Y, WANG C T. Current status of genetic resources of Tibetan sheep in Qinghai-Tibet plateau, existing problems and countermeasures[J]. Animal Husbandry and Veterinary Digesl of China, 2011, 27(2): 1–4. (in Chinese) |
| [3] |
国家畜禽遗传资源委员会.
中国畜禽遗传资源志-羊志[M]. 北京: 中国农业出版社, 2011.
China National Commisson of Animal Genetic Resources. Animal genetic resources in China sheep and goats[M]. Beijing: China Agriculture Press, 2011. (in Chinese) |
| [4] |
徐琳娜, 李国林. 藏系绵羊遗传多样性的研究进展[J]. 畜牧兽医杂志, 2012, 31(4): 56–58, 60.
XU L N, LI G L. Research progress on genetic diversity of Tibetan sheep[J]. Journal of Animal Science and Veterinary Medicine, 2012, 31(4): 56–58, 60. (in Chinese) |
| [5] |
孙晓萍, 刘建斌, 袁超, 等. 藏绵羊高海拔适应性及生产性能分析[J]. 今日畜牧兽医, 2018, 34(4): 50–52.
SUN X P, LIU J B, YUAN C, et al. Analysis of high altitude adaptability and production performance of Tibetan sheep[J]. Today Animal Husbandry and Veterinary Medicine, 2018, 34(4): 50–52. DOI: 10.3969/j.issn.1673-4092.2018.04.050 (in Chinese) |
| [6] |
贾荣莉. 高原藏羊肺组织学结构及特点[J]. 中国兽医科技, 1997, 27(2): 43–44.
JIA R L. Histological structure and characteristics of lung of Tibetan sheep on plateau[J]. Chinese Veterinary Science, 1997, 27(2): 43–44. (in Chinese) |
| [7] |
王欣荣, 吴建平. 藏羊脑动脉系统结构特征与高原适应性研究[J]. 家畜生态学报, 2013, 34(8): 36–40.
WANG X R, WU J P. Study on structural features of the cerebral arterial system associatedwith plateau adaptability in Tibetan sheep[J]. Acta Ecologae Animalis Domastici, 2013, 34(8): 36–40. DOI: 10.3969/j.issn.1673-1182.2013.08.007 (in Chinese) |
| [8] |
涂正超, 张亚平. 藏绵羊线粒体DNA遗传多样性研究[J]. 畜牧兽医学报, 1998, 29(2): 132–135.
TU Z C, ZHANG Y P. Mitochondrial DNA polymorphism in Zang sheep based on restriction endonuclease cleavage patterns[J]. Acta Veterinaria et Zootechnica Sinica, 1998, 29(2): 132–135. DOI: 10.3321/j.issn:0366-6964.1998.02.008 (in Chinese) |
| [9] |
熊勇.藏系绵羊ZFX、ZFY基因片段的克隆测序及进化研究[D].成都: 西南民族大学, 2004.
XIONG Y.Cloning, sequencing and evolution of ZFX and ZFY gene fragments of Tibetan sheep[D].Chengdu: Southwest Minzu University, 2004.(in Chinese) |
| [10] |
李祥龙, 张增利, 巩元芳, 等. 我国主要地方绵羊品种mtDNA D-loop区PCR-RFLP研究[J]. 遗传, 2006, 28(2): 165–170.
LI X L, ZHANG Z L, GONG Y F, et al. Study on mtDNA D-loop of Chinese main indigenous sheep breeds using PCR-RFLP[J]. Hereditas, 2006, 28(2): 165–170. (in Chinese) |
| [11] | KIJAS J W, LENSTRA J A, HAYES B, et al. Genome-wide analysis of the world's sheep breeds reveals high levels of historic mixture and strong recent selection[J]. PLoS Biol, 2012, 10(2): e1001258. DOI: 10.1371/journal.pbio.1001258 |
| [12] | WEI C H, WANG H H, LIU G, et al. Genome-wide analysis reveals population structure and selection in Chinese indigenous sheep breeds[J]. BMC Genomics, 2015, 16: 194. DOI: 10.1186/s12864-015-1384-9 |
| [13] | LV F H, AGHA S, KANTANEN J, et al. Adaptations to climate-mediated selective pressures in sheep[J]. Mol Biol Evol, 2014, 31(12): 3324–3343. DOI: 10.1093/molbev/msu264 |
| [14] | YANG J, LI W R, LV F H, et al. Whole-genome sequencing of native sheep provides insights into rapid adaptations to extreme environments[J]. Mol Biol Evol, 2016, 33(10): 2576–2592. DOI: 10.1093/molbev/msw129 |
| [15] | PURCELL S, NEALE B, TODD-BROWN K, et al. PLINK:a tool set for whole-genome association and population-based linkage analyses[J]. Am J Hum Genet, 2007, 81(3): 559–575. |
| [16] | NEI M. Molecular population genetics and evolution[J]. Front Biol, 1975, 40: 198–199. |
| [17] | PATTERSON N, PRICE A L, REICH D. Populationstructure and eigenanalysis[J]. PLoS Genet, 2006, 2(12): e190. DOI: 10.1371/journal.pgen.0020190 |
| [18] | PRICE A L, PATTERSON N J, PLENGE R M, et al. Principal components analysis corrects for stratification in genome-wide association studies[J]. Nat Genet, 2006, 38(8): 904–909. DOI: 10.1038/ng1847 |
| [19] | TAMURA K, PETERSON D, PETERSON N, et al. MEGA5:molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Mol Biol Evol, 2011, 28(10): 2731–2739. DOI: 10.1093/molbev/msr121 |
| [20] | KIM J J, VERDU P, PAKSTIS A J, et al. Use of Autosomal loci for clustering individuals and populations of East Asian origin[J]. Hum Genet, 2005, 117(6): 511–519. DOI: 10.1007/s00439-005-1334-8 |
| [21] | ALEXANDER D H, NOVEMBRE J, LANGE K. Fastmodel-based estimation of ancestry in unrelated individuals[J]. Genome Res, 2009, 19(9): 1655–1664. DOI: 10.1101/gr.094052.109 |
| [22] |
晁燕, 祁世荣, 张延生. 青海藏系绵羊高原型与山谷型类群的遗传多样性和分化研究[J]. 黑龙江畜牧兽医, 2016(11): 102–104.
CHAO Y, QI S R, ZHANG Y S. Studyon genetic diversity and differentiation of Tibetan sheep plateau and valley types in Qinghai[J]. Heilongjiang Animal Science and Veterinary Medicine, 2016(11): 102–104. (in Chinese) |
| [23] | INTARAPANICH A, SHAW P J, ASSAWAMAKIN A, et al. Iterative Pruning PCA improves resolution of highly structured populations[J]. BMC Bioinform, 2009, 10: 382. DOI: 10.1186/1471-2105-10-382 |
| [24] |
王荣鑫, 马森, 张才骏, 等. 青海藏系绵羊九项生理参数测定[J]. 青海畜牧兽医学院学报, 1987(1): 24–28, 39.
WANG R X, MA S, ZHANG C J, et al. Determination of nine physiological parameters of Tibetan sheep in Qinghai[J]. Journal of Qinghai College of Animal Science and Veterinary Medicine, 1987(1): 24–28, 39. (in Chinese) |
| [25] |
甘肃省畜牧厅.
甘肃省畜禽品种志[M]. 兰州: 甘肃人民出版社, 1986.
Chief Editor of Gansu Animal Husbandry Department. Records of domestic animal and fowl breeds of Gansu Province[M]. Lanzhou: Gansu People's Publishing House, 1986. (in Chinese) |
| [26] |
李沐森, 郭文场. 藏羊的类型分布及生活习性[J]. 特种经济动植物, 2016(3): 5–8.
LI M S, GUO W C. Types, distribution and living habits of Tibetan sheep[J]. Special Economic Animal and Plant, 2016(3): 5–8. DOI: 10.3969/j.issn.1001-4713.2016.03.004 (in Chinese) |
| [27] |
吉金凤, 祁红霞, 何文, 等. 乔科羊产肉性能测定分析[J]. 畜牧兽医杂志, 2007, 26(1): 82–83.
JI J F, QI H X, HE W, et al. Determination and analysis of meat performance of Qiaoke sheep[J]. Journal of Animal Science and Veterinary Medicine, 2007, 26(1): 82–83. DOI: 10.3969/j.issn.1004-6704.2007.01.037 (in Chinese) |
| [28] |
李昕, 常明华, 全晓毅, 等. 欧拉羊杂交改良高原型藏羊效果分析[J]. 中国畜牧兽医文摘, 2015, 31(1): 55, 73.
LI X, CHANG M H, QUAN X Y, et al. Effect analysis of plateau Tibetan sheep improved by Oula sheep hybridization[J]. Chinese Journal of Animal Husbandry and Veterinary Medicine, 2015, 31(1): 55, 73. (in Chinese) |
| [29] |
杨晓军, 赵有璋. 藏系绵羊遗传多样性及其品种(系)分化的研究[J]. 中国草食动物, 2006, 26(4): 19–22.
YANG X J, ZHAO Y Z. Study on the genetic diversity and the breeds differentiation of Tibet sheep[J]. China Herbivore Science, 2006, 26(4): 19–22. DOI: 10.3969/j.issn.2095-3887.2006.04.006 (in Chinese) |
| [30] |
徐琳娜.甘肃青海藏系绵羊群体遗传多样性的微卫星分析[D].兰州: 甘肃农业大学, 2010.
XU L N.Analysis on genetic diversity in Tibet sheep populations in Gansu and Qinghai Provinces by microsatellite markers[D]. Lanzhou: Gansu Agricultural University, 2010.(in Chinese) http://cdmd.cnki.com.cn/article/cdmd-10733-1011026133.htm |



