ADAR是一个多基因家族,其基因数目在物种间存在差异。目前,哺乳动物中主要有3种ADAR基因,即ADAR1、ADAR2和ADAR3。三者编码的蛋白在哺乳动物中高度保守,都具有数量不等的双链RNA结合结构域和一个高度保守的C端脱氨酶结构域[1],但仅有ADAR1和ADAR2具有催化RNA编辑发生的活性。
ADAR1最早发现于牛肝提取物中,是首个被鉴定出的A-to-I RNA编辑酶[2-4]。ADAR1在生物体内广泛表达,其中在神经系统中表达量相对较高[5]。在人和小鼠中,ADAR1存在多个转录本[6-7]。缺乏ADAR1脱氨酶活性的小鼠会因红细胞生成障碍和细胞非正常凋亡而死于胚胎早期[8-9]。通过RNA编辑,ADAR1可以改变密码子进而改变蛋白结构和功能,影响可变剪接,影响miRNA的结合和成熟等[10]。目前,已报道ADAR1在胚胎发育[11]、神经系统功能调节[12]、病毒感染[13]、自身免疫抑制[14]以及多种人肿瘤的发生发展[10, 15]中发挥重要作用。猪因其解剖、生理生化特点与人极为相似,常被用来制作人疾病模型。因此,对猪ADAR1基因的研究有利于揭示人疾病分子机理。目前,该基因在猪中仅有一段预测的序列,在一定程度上阻碍了其功能研究的开展。
本研究以大白猪脾组织mRNA为模板,利用RT-PCR、5′ RACE和3′ RACE克隆得到ADAR1 cDNA全长序列,并对该序列及其编码的蛋白进行生物信息学分析,同时应用荧光定量PCR技术检测ADAR1基因在大白猪各组织及背部脂肪沉积过程中的表达情况,为进一步研究猪ADAR1基因的功能奠定基础。
1 材料与方法 1.1 试验材料试验所用大白猪均来自中国农业科学院北京畜牧兽医研究所昌平种猪场。4头35日龄大白猪,屠宰后用无菌剪刀采集心、肝、脾、肺、肾、脑、肌肉、小肠和背部脂肪9种组织,并迅速置于液氮中保存备用。选择3窝个体(每窝4头,共12头),屠宰间隔时间为60 d左右,每窝分别于7、60、120和180日龄时各屠宰1头,采集背部皮下脂肪组织,并迅速置于液氮罐中。ADAR1具有影响病毒感染的作用,而仔猪断奶后经过1周的适应,体况相对稳定,并具备一定的免疫能力。因此,选择35日龄大白猪的脾组织用于cDNA全长扩增。
1.2 主要试剂动物组织总RNA提取试剂盒、2×Taq PCR MasterMix均购自天根生化科技有限公司(北京);RACE试剂盒(SMARTer RACE 5′/3′ Kit)、反转录试剂盒(PrimeScriptTM RT Reagent Kit)和荧光定量染料(SYBR® Premix Ex TaqTM Ⅱ)均购于TaKaRa公司(大连)。
1.3 组织RNA提取及cDNA合成使用动物组织总RNA提取试剂盒提取大白猪9种组织样品的总RNA,并用NanoDrop2000超微量分光光度计测定浓度和纯度,用1%琼脂糖凝胶电泳检测完整性。取1 μg RNA,按照反转录试剂盒说明书合成cDNA,并置于-20 ℃保存备用。
1.4 引物设计参考ADAR1基因mRNA序列(NCBI登录号:XM_021089756.1),使用NCBI数据库中的Primer-BLAST设计基因克隆和荧光定量PCR引物(表 1)。用于RACE克隆的巢式PCR引物的5′末端添加一段长度为15 bp的序列“GATTACGCCAAGCTT”,便于PCR产物的连接测序。所有引物均由英潍捷基(上海)贸易有限公司合成。
|
|
表 1 猪ADAR1基因cDNA克隆和荧光定量PCR引物 Table 1 Primers for cDNA clone and real-time PCR of porcine ADAR1 |
参考猪ADAR1基因的mRNA预测序列,设计其保守区段的扩增引物(A1~A6),以35日龄大白猪脾cDNA为模板,经PCR扩增获得其中间序列。PCR反应体系为25 μL:cDNA模板1 μL,上、下游引物(10 μmol·L-1)各1 μL,2×Taq PCR MasterMix 12.5 μL以及ddH2O 9.5 μL。PCR反应条件:94 ℃预变性3 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min,35个循环;72 ℃延伸10 min;4 ℃保存。反应产物用1.5%琼脂糖凝胶电泳检测,条带明亮且长度符合预期的产物交由北京诺赛基因组研究中心有限公司进行Sanger测序。
根据所获得的猪ADAR1基因中间序列,设计5′和3′RACE特异性引物。以35日龄大白猪脾组织RNA为模板,按照SMARTer RACE cDNA第一链合成说明书合成5′和3′RACE所用cDNA后,使用UPM和外侧引物(5A-outer和3A-outer)进行第一轮PCR扩增。为确保RACE PCR的特异性,扩增产物经30倍稀释后作为模板,使用UPS和内侧引物(5A-inner和3A-inner)进行第二轮巢式PCR。PCR产物经琼脂糖凝胶电泳分离后使用琼脂糖凝胶回收试剂盒纯化。纯化后的产物连接到Linearized pRACE载体,并转化至感受态细胞Stellar Competent Cells,均匀涂板后,37 ℃过夜培养。挑选阳性菌落,交由北京诺赛基因组研究中心有限公司进行Sanger测序。测序获得的所有片段使用DNAMAN软件拼接组装,得到ADAR1基因的cDNA全长序列。
1.6 猪ADAR1基因生物信息学分析利用NCBI数据库中的ORF finder(https://www.ncbi.nlm.nih.gov/orffinder/)查找开放阅读框,并获得预测的蛋白序列。利用DNAMAN软件分析猪ADAR1与人、黑猩猩、猕猴、长臂猿、黄牛、绵羊、山羊、小鼠、鸡和斑马鱼的核酸序列和氨基酸序列一致性。使用Ensembl数据库中的BLAST/BLAT程序(http://asia.ensembl.org/Multi/Tools/Blast?db=core),并依据GT-AG法则确定外显子和内含子边界。使用ExPaSy中的ProtParam(https://web.expasy.org/protparam/)和ProtScale (https://web.expasy.org/protscale/)工具分析蛋白的基本理化性质和亲-疏水性。使用TMHMM Server v. 2.0(http://www.cbs.dtu.dk/services/TMHMM/)预测跨膜结构。利用在线软件SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)和SignalP 4.1 Server(http://www.cbs.dtu.dk/services/SignalP/)预测蛋白质二级结构和信号肽位点。利用SMART(http://smart.embl-heidelberg.de/)预测蛋白的保守结构域。应用SWISS-MODEL(https://www.swissmodel.expasy.org/)构建蛋白质三级结构。应用MEGA-X软件绘制ADAR1蛋白序列系统发育树。
1.7 荧光定量PCR检测猪ADAR1基因的组织表达利用ABI QuantStudioTM 7 Flex实时荧光定量PCR仪检测ADAR1在大白猪不同组织(心、肝、脾、肺、肾、脂肪、肌肉、大脑和小肠)及不同日龄大白猪背膘(7、60、120和180日龄)中的表达量。反应总体积为20 μL:10 μL 2×SYBR®Premix Ex Taq Ⅱ,上、下游引物(10 μmol·L-1)各0.4 μL,0.4 μL 50×ROX Reference Dye Ⅱ,2 μL cDNA和6.8 μL ddH2O。PCR程序:95 ℃预变性30 s;95 ℃变性5 s,60 ℃ 34 s,40个循环;反应结束后分析熔解曲线。以GAPDH为内参,每个样品3个技术性重复。采用2-△△Ct法计算ADAR1基因在大白猪各组织及不同日龄大白猪背膘中的表达量。
1.8 统计分析使用Graphpad prism 7作图,使用SAS 9.2的GLM过程进行单因素方差分析,P < 0.05表示差异显著。
2 结果 2.1 猪ADAR1基因克隆及序列分析对ADAR1基因的保守部分进行扩增、测序并拼接,获得了一条长为3 053 bp的中间序列。5′和3′ RACE分别获得了长度为1 132和2 965 bp的5′和3′末端序列。将3段序列拼接后,获得了长度为6 259 bp的猪ADAR1基因cDNA全长序列。RACE扩增得到的产物经琼脂糖凝胶电泳检测结果如图 1所示。
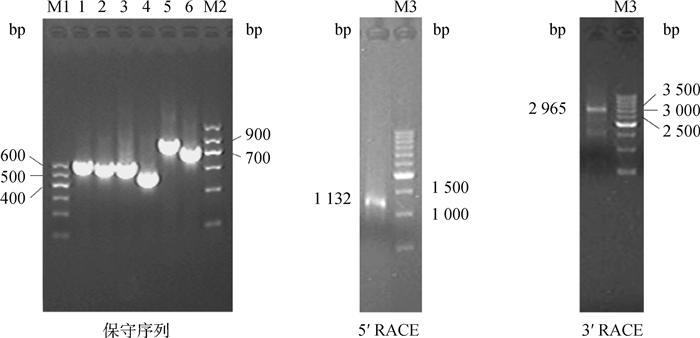
|
M1、M2、M3. DNA相对分子质量标准;1~6. A1~A6 PCR产物 M1. Marker I; M2. Marker Ⅱ; M3. 500 bp DNA Ladder; 1-6. PCR products of A1-A6 图 1 猪ADAR1基因RACE产物的琼脂糖凝胶电泳图 Fig. 1 Gel electrophoresis results of RACE products of porcine ADAR1 |
序列分析表明,ADAR1基因cDNA序列包含一个长153 bp的5′ UTR,一个长3 438 bp的CDS区,一个长2 641 bp的3′ UTR和一个poly(A)尾巴。该基因起始密码子为ATG,终止密码子为TAA,共编码1 145个氨基酸。利用Ensembl网站中的BLAST/BLAT工具进行序列分析,发现ADAR1基因共包含15个外显子,长度在94~2 879 bp之间,且剪接供体和受体均符合GT-AG法则(图 2、表 2)。
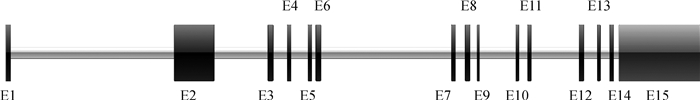
|
外显子用盒子表示,内含子用线表示 Exons are shown as boxes, introns are shown as lines 图 2 猪ADAR1基因结构图 Fig. 2 Structure of porcine ADAR1 gene |
|
|
表 2 猪ADAR1基因外显子-内含子边界 Table 2 Exon-intron boundaries of the porcine ADAR1 gene |
猪ADAR1基因的CDS核酸序列与牛和羊的一致性最高,分别为89.2%和89.0%,其次是人等灵长类动物,约为86%,与斑马鱼的一致性最低,为52.7%(表 3)。猪ADAR1基因的氨基酸序列与牛的一致性最高(87.3%),其次是羊(86.9%),与斑马鱼的一致性最低(65.1%)(表 3)。应用MEGA-X软件,根据ADAR1的氨基酸序列构建猪、人、黑猩猩、猕猴、长臂猿、黄牛、绵羊、山羊、小鼠、鸡和斑马鱼的进化树,分析猪ADAR1与各物种在进化上的关系。结果表明,猪与牛、羊ADAR1的亲缘关系最近,其次是灵长类(图 3)。
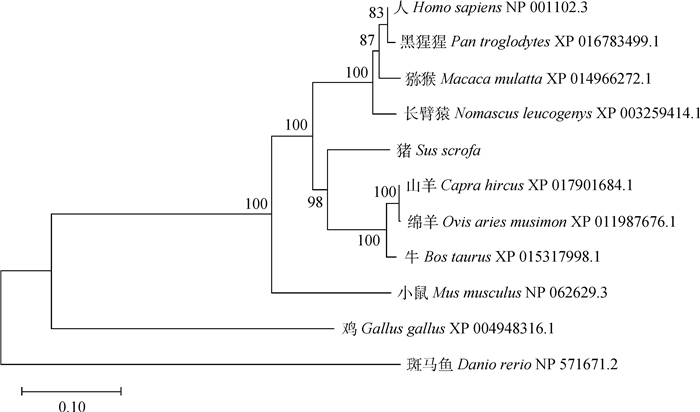
|
图 3 不同物种基于ADAR1的系统进化树 Fig. 3 Phylogenetic tree of ADAR1 in different species |
|
|
表 3 猪ADAR1基因CDS区与其它物种间的一致性分析 Table 3 Identity analysis of ADAR1 CDS between the pig and other species |
基本理化性质分析表明,预测ADAR1蛋白的分子式为C5622H8856N1594O1696S42,包含17 810个原子,理论等电点为8.61,分子量是127.26 ku,脂肪系数70.72,半衰期30 h,不稳定系数高达48.49,属于不稳定蛋白。该蛋白共含有20种氨基酸,其中亮氨酸含量最高(9.1%),色氨酸含量最低(1.1%)。疏水性预测分析显示,ADAR1蛋白疏水性得分在第724位氨基酸最低(-2.96),在第804位氨基酸最高(1.99),总平均疏水性为-0.59,推测该蛋白属于亲水性蛋白(图 4A)。二级结构预测显示,该蛋白含有α-螺旋、延伸片段、β-折叠和无规则卷曲共4种二级结构,分别占总长度的29.7%、13.0%、6.6%和50.7%。因此,该蛋白可归类为混合型蛋白。用SMART对该蛋白的结构域进行预测发现,ADAR1含有2个Zα结构域,3个双链RNA结合结构域和一个脱氨酶结构域(图 4B)。2个Zα结构域分别位于第129~195和236~304位氨基酸;3个双链RNA结合结构域分别位于第449~515、560~626和672~738位氨基酸;1个脱氨酶结构域位于第758~1 141位氨基酸。TMHMM和SignalP预测显示,该蛋白没有跨膜结构域和信号肽。使用SWISS-MODEL构建了猪ADAR1三级结构,由第757~1 143位氨基酸构成(图 4C)。
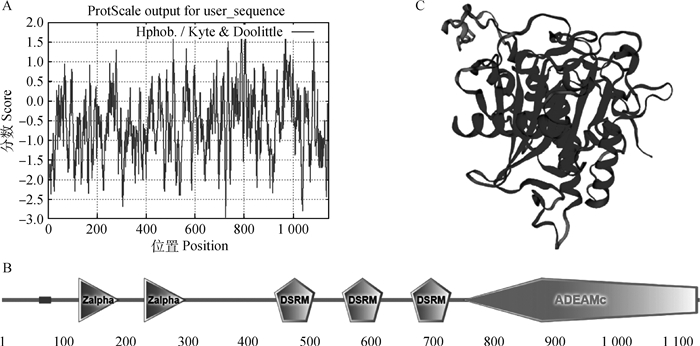
|
A.亲-疏水性分析;B.结构域预测;C.三级结构预测 A. Hydrophilic-hydrophobic property prediction; B. Structure domain prediction; C. Tertiary structure prediction 图 4 ADAR1蛋白结构与功能分析 Fig. 4 Structure and function prediction of the porcine ADAR1 protein |
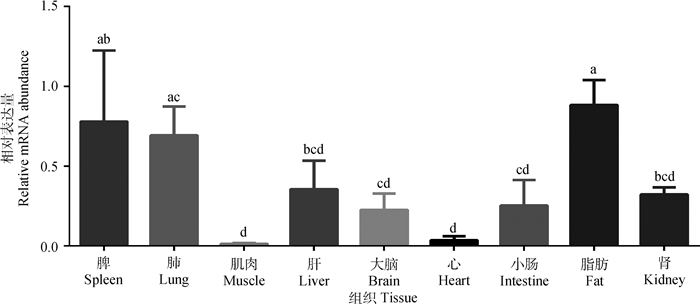
运用荧光定量PCR检测ADAR1基因在大白猪多种组织中的表达情况,结果如图 5所示。在所检测的9种组织中,ADAR1均有表达,其中,在脂肪、脾和肺中高表达,在心和肌肉中表达较低,并且其在脂肪中表达量显著高于除脾和肺外的其它组织(P < 0.05),在肌肉、肝、大脑、心、小肠和肾中的表达量无显著性差异(P>0.05)。
|
不同字母表示差异显著(P < 0.05);相同字母表示差异不显著(P>0.05);误差线表示标准差,下同 The different letters show statistically significant differences (P < 0.05); The same letters show that the difference is not statistically significant (P > 0.05);Error bars represent standard deviations, the same as below 图 5 猪ADAR1在9种组织的相对表达量 Fig. 5 Relative expressions of the porcine ADAR1 in 9 tissues |
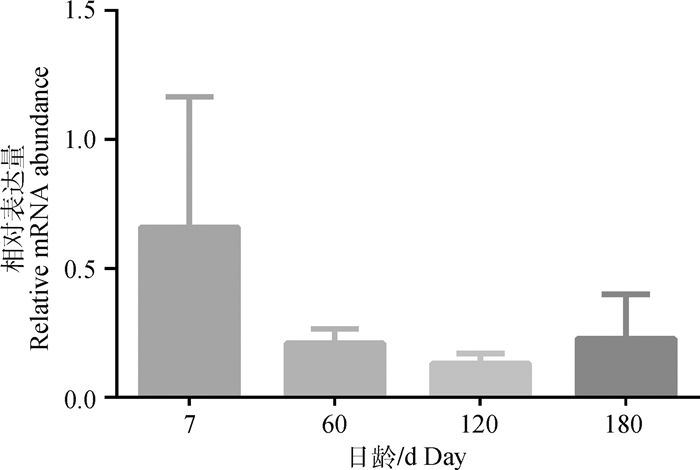
利用荧光定量PCR,以GAPDH为内参,检测ADAR1基因在不同日龄大白猪背膘中的表达量,以研究其在背部脂肪沉积过程中的表达规律。结果表明,ADAR1在不同日龄背膘中表达量无显著差异,但在7日龄时表达量最高,其次是180日龄,在120日龄时表达量最低,整体表现出随个体发育其表达量具有先降低后升高的趋势(图 6)。
|
图 6 ADAR1在大白猪不同日龄背膘中的相对表达量 Fig. 6 Relative expressions of porcine ADAR1 in backfat of Large White pigs on different ages |
本研究利用RACE技术,克隆获得了一条长为6 259 bp的猪ADAR1基因cDNA全长序列,其包含153 bp的5′ UTR,3 438 bp的CDS区,2 641 bp的3′ UTR和一个poly(A)尾巴。与NCBI中的猪ADAR1基因预测mRNA相比,本研究获得的ADAR1 mRNA在长度上增加了57 bp,预测的氨基酸序列一致性为99.3%。根据Ensembl blast的结果及GT-AG法则划分内含子与外显子边界,该基因共包含15个外显子,与NCBI中的预测结果一致,但比人ADAR1的外显子少2个[16]。该基因CDS序列与牛羊一致性最高,约为89%,与人、鼠已知序列的一致性在80%以上。猪ADAR1蛋白包含1 145个氨基酸,涵盖全部20种氨基酸。在人和小鼠中,主要存在2种ADAR1异构体,根据其分子量分别命名为p150和p110,前者由干扰素诱导表达,后者组成型表达[17-18]。研究表明,ADAR1 p150属于核质穿梭蛋白,p110主要位于细胞核[18]。本研究中,预测的ADAR1蛋白未含有任何有利于蛋白穿透细胞膜或者细胞器膜的信号肽和跨膜结构,但其包含了大量的与信号肽跨膜功能相关的α-螺旋[19]。同时,保守结构域预测显示,猪ADAR1蛋白具有2个Zα结合结构域,与人p150一致[20]。因此,推测本研究获得的猪ADAR1转录本可能编码ADAR1 p150蛋白。猪ADAR1蛋白与灵长动物、牛和羊具有高度一致性(≥85%),表明ADAR1在哺乳动物中高度保守。进化树分析显示,猪ADAR1蛋白能够与其它物种聚到一起,为ADAR1的保守性提供了进一步的证据。
ADAR1基因在机体各组织中广泛表达,在脑和脾中表达量较高[5, 21]。本研究在9种组织中检测了ADAR1的表达量,发现其在脾和肺组织中表达量较高,在肌肉中表达量最低。脾是机体内重要的免疫器官[22],而肺是机体内最易受到细菌病毒感染的器官,ADAR1在脾和肺中的高表达表明其与机体免疫存在紧密关系,这与报道的ADAR1具有影响病毒感染的作用相吻合[23-24]。前期研究发现,脂肪组织中存在大量RNA编辑事件[25]。因此,笔者对不同日龄大白猪背膘中ADAR1基因的表达情况进行了检测。结果发现,ADAR1在背部脂肪沉积过程中均有表达,并表现出了随日龄增加先下降后上升的趋势,但由于所用个体数量有限,还需使用更多样本来进一步验证。
RNA编辑是一种重要的转录后修饰过程。近年来,随着高通量测序技术的快速发展及相关软件的开发[26],RNA编辑位点的数目呈现出爆炸式增长。目前,已在人中鉴定出450多万个A-to-I RNA编辑位点[27]。A-to-I编辑已知由ADAR酶家族催化发生。作为ADAR家族的一员,ADAR1可催化腺嘌呤水解脱氨形成次黄嘌呤。猪ADAR1脱氨酶结构域与人、鼠(序列来源于UniProt,https://www.uniprot.org/)的一致性分别为96.4%和95.8%,表现出高度保守。ADAR1既可编辑细胞RNA,也可编辑病毒RNA,并可根据靶序列组成和结构进行位点特异性编辑[28]。通过RNA编辑,ADAR1表达量的改变会引起一系列生理变化。在多发性骨髓瘤中,ADAR1介导的GLI1编辑促进肿瘤的恶性自我更新[29]。ADAR1介导的RNA编辑能够通过调控miRNA的生成加速造血细胞周期,对维持正常的造血细胞增殖是必不可少的[30]。ADAR1在维持正常的神经系统功能中也发挥着重要作用,其可通过编辑谷氨酸受体B亚基上的Q/R位点减弱谷氨酸受体对钙离子的通透性,进而影响神经信号的传导[12]。ADAR1在病毒感染中可发挥抗病毒作用,其通过编辑病毒RNA引起结构变化,进而导致病毒RNA降解,达到抗病毒的作用[23]。此外,ADAR1还可以不依赖RNA编辑影响胚胎干细胞中的神经分化和病毒感染[24, 31]。因此,ADAR1具有较高的研究价值,其在猪经济性状中的作用还需深入挖掘
4 结论本研究成功克隆了猪ADAR1基因cDNA全长序列。该序列长6 259 bp,包含153 bp的5′UTR,3 438 bp的CDS区,2 641 bp的3′UTR和一个poly(A)尾巴,共有15个外显子。ADAR1在心、肝、脾、肺、肾、脑、肌肉、小肠和背部脂肪中均表达,同时在背膘中表现出随日龄增加表达量先下降后上升的趋势。本研究为进一步探索ADAR1的功能奠定了基础。
| [1] | BASS B L. RNA editing by adenosine deaminases that act on RNA[J]. Annu Rev Biochem, 2002, 71: 817–846. DOI: 10.1146/annurev.biochem.71.110601.135501 |
| [2] | KIM U, GARNER T L, SANFORD T, et al. Purification and characterization of double-stranded RNA adenosine deaminase from bovine nuclear extracts[J]. J Biol Chem, 1994, 269(18): 13480–13489. |
| [3] | KIM U, WANG Y, SANFORD T, et al. Molecular cloning of cDNA for double-stranded RNA adenosine deaminase, a candidate enzyme for nuclear RNA editing[J]. Proc Natl Acad Sci U S A, 1994, 91(24): 11457–11461. DOI: 10.1073/pnas.91.24.11457 |
| [4] | WANG Q D. RNA editing catalyzed by ADAR1 and its function in mammalian cells[J]. Biochemistry (Mosc), 2011, 76(8): 900–911. DOI: 10.1134/S0006297911080050 |
| [5] | HUNTLEY M A, LOU M, GOLDSTEIN L D, et al. Complex regulation of ADAR-mediated RNA-editing across tissues[J]. BMC Genomics, 2016, 17: 61. DOI: 10.1186/s12864-015-2291-9 |
| [6] | ORLANDI C, BARBON A, BARLATI S. Activity regulation of adenosine deaminases acting on RNA (ADARs)[J]. Mol Neurobiol, 2012, 45(1): 61–75. |
| [7] | HOOD J L, MORABITO M V, MARTINEZ Ⅲ C R, et al. Reovirus-mediated induction of ADAR1(p150) minimally alters RNA editing patterns in discrete brain regions[J]. Mol Cell Neurosci, 2014, 61: 97–109. DOI: 10.1016/j.mcn.2014.06.001 |
| [8] | HARTNER J C, SCHMITTWOLF C, KISPERT A, et al. Liver disintegration in the mouse embryo caused by deficiency in the RNA-editing enzyme ADAR1[J]. J Biol Chem, 2004, 279(6): 4894–4902. DOI: 10.1074/jbc.M311347200 |
| [9] | HARTNER J C, WALKLEY C R, LU J, et al. ADAR1 is essential for the maintenance of hematopoiesis and suppression of interferon signaling[J]. Nat Immunol, 2009, 10: 109–115. DOI: 10.1038/ni.1680 |
| [10] | XU L D, OHMAN M. ADAR1 editing and its role in cancer[J]. Genes (Basel), 2019, 10(1): 12. |
| [11] | WANG Q, KHILLAN J, GADUE P, et al. Requirement of the RNA editing deaminase ADAR1 gene for embryonic erythropoiesis[J]. Science, 2000, 290(5497): 1765–1768. DOI: 10.1126/science.290.5497.1765 |
| [12] | SEEBURG P H, HARTNER J. Regulation of ion channel/neurotransmitter receptor function by RNA editing[J]. Curr Opin Neurobiol, 2003, 13(3): 279–283. DOI: 10.1016/S0959-4388(03)00062-X |
| [13] | PUJANTELL M, FRANCO S, GALVÁN-FEMENÍA I, et al. ADAR1 affects HCV infection by modulating innate immune response[J]. Antiviral Res, 2018, 156: 116–127. DOI: 10.1016/j.antiviral.2018.05.012 |
| [14] | NAKAHAMA T, KATO Y, KIM J I, et al. ADAR1-mediated RNA editing is required for thymic self-tolerance and inhibition of autoimmunity[J]. EMBO Rep, 2018, 19(12): e46303. DOI: 10.15252/embr.201846303 |
| [15] | NEMLICH Y, BARUCH E N, BESSER M J, et al. ADAR1-mediated regulation of melanoma invasion[J]. Nat Commun, 2018, 9: 2154. DOI: 10.1038/s41467-018-04600-2 |
| [16] | WANG Y, ZENG Y, MURRAY J M, et al. Genomic organization and chromosomal location of the human dsRNA adenosine deaminase gene:the enzyme for glutamate-activated ion channel RNA editing[J]. J Mol Biol, 1995, 254(2): 184–195. DOI: 10.1006/jmbi.1995.0610 |
| [17] | GEORGE C X, SAMUEL C E. Human RNA-specific adenosine deaminase ADAR1 transcripts possess alternative exon 1 structures that initiate from different promoters, one constitutively active and the other interferon inducible[J]. Proc Natl Acad Sci U S A, 1999, 96(8): 4621–4626. DOI: 10.1073/pnas.96.8.4621 |
| [18] | PATTERSON J B, SAMUEL C E. Expression and regulation by interferon of a double-stranded-RNA-specific adenosine deaminase from human cells:evidence for two forms of the deaminase[J]. Mol Cell Biol, 1995, 15(10): 5376–5388. DOI: 10.1128/MCB.15.10.5376 |
| [19] | BLOM N, SICHERITZ-PONTÉN T, GUPTA R, et al. Prediction of post-translational glycosylation and phosphorylation of proteins from the amino acid sequence[J]. Proteomics, 2004, 4(6): 1633–1649. DOI: 10.1002/(ISSN)1615-9861 |
| [20] | NISHIKURA K. A-to-I editing of coding and non-coding RNAs by ADARs[J]. Nat Rev Mol Cell Biol, 2016, 17(2): 83–96. DOI: 10.1038/nrm.2015.4 |
| [21] | PICARDI E, MANZARI C, MASTROPASQUA F, et al. Profiling RNA editing in human tissues:towards the inosinome Atlas[J]. Sci Rep, 2015, 5: 14941. DOI: 10.1038/srep14941 |
| [22] |
蔡雯祎, 熊显荣, 陈通, 等. KDM2B基因克隆及其在牦牛组织和卵母细胞减数分裂过程中的表达研究[J]. 畜牧兽医学报, 2018, 49(3): 534–541.
CAI W Y, XIONG X R, CHEN T, et al. Molecular cloning of KDM2B gene and its expression pattern in tissues and the process of oocyte meiosis in yak[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(3): 534–541. (in Chinese) |
| [23] | GONZALES-VAN S R, SARNOW P. Making the mark:the role of adenosine modifications in the life cycle of RNA viruses[J]. Cell Host Microbe, 2017, 21(6): 661–669. DOI: 10.1016/j.chom.2017.05.008 |
| [24] | CACHAT A, ALAIS S, CHEVALIER S A, et al. ADAR1 enhances HTLV-1 and HTLV-2 replication through inhibition of PKR activity[J]. Retrovirology, 2014, 11: 93. DOI: 10.1186/s12977-014-0093-9 |
| [25] | ZHANG Y B, ZHANG L C, YUE J W, et al. Genome-wide identification of RNA editing in seven porcine tissues by matched DNA and RNA high-throughput sequencing[J]. J Anim Sci Biotechnol, 2019, 10: 24. DOI: 10.1186/s40104-019-0326-9 |
| [26] |
张跃博, 颜华, 王立刚, 等. 哺乳动物RNA编辑及其检测方法[J]. 畜牧兽医学报, 2018, 49(11): 2299–2309.
ZHANG Y B, YAN H, WANG L G, et al. RNA Editing and its detection methods in mammalian[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(11): 2299–2309. (in Chinese) |
| [27] | PICARDI E, D'ERCHIA A M, LO GIUDICE C, et al. REDIportal:a comprehensive database of A-to-I RNA editing events in humans[J]. Nucleic Acids Res, 2017, 45(D1): D750–D757. DOI: 10.1093/nar/gkw767 |
| [28] | SAMUEL C E. Adenosine deaminases acting on RNA (ADARs) are both antiviral and proviral[J]. Virology, 2011, 411(2): 180–193. DOI: 10.1016/j.virol.2010.12.004 |
| [29] | CREWS L, LAZZARI E, MONDALA P, et al. ADAR1-mediated GLI1 editing promotes malignant self-renewal in multiple myeloma[J]. Blood, 2017, 130(Suppl 1): 1204. |
| [30] | JIANG Q F, ZIPETO M A, SANTOS N D, et al. RNA editing enzyme ADAR1 accelerates normal hematopoiesis cell cycle by regulating microRNA biogenesis[J]. Blood, 2016, 128(22): 887. |
| [31] | CHEN T, XIANG J F, ZHU S S, et al. ADAR1 is required for differentiation and neural induction by regulating microRNA processing in a catalytically independent manner[J]. Cell Res, 2015, 25(4): 459–476. DOI: 10.1038/cr.2015.24 |



