2. 湖北省农业科学院畜牧兽医研究所农业部畜禽细菌病防治制剂创制重点实验室, 武汉 430070
2. Key Laboratory of Prevention and Control Agents for Animal Bacteriosis(Ministry of Agriculture), Institute of Animal Husbandry and Veterinary Science, Hubei Academy of Agricultural Sciences, Wuhan 430070, China
多杀性巴氏杆菌(Pasteurella multocida, Pm)是一种重要的革兰阴性机会致病菌,能够感染多种动物甚至是人,主要导致呼吸系统疾病和败血症[1-2]。Pm至少可以被分为5种荚膜基因型(A、B、D、E、F)及8种LPS基因型(L1~L8)[3-4]。目前已知的Pm主要毒力因子包括荚膜、脂多糖(LPS)、黏附因子、毒素(PMT)、铁摄取相关蛋白、唾液酸代谢相关蛋白、透明质酸酶以及外膜蛋白(OMP)[5]。Pm基因组中含有大量的基因编码这些毒力因子,这些毒力基因构成了Pm致病的分子基础[6-7]。
猪是Pm的宿主之一。Pm可以感染导致猪的多种临床表现,包括呼吸困难、萎缩性鼻炎、肺炎、败血症甚至死亡,其流行严重危害我国及世界养猪业的健康发展[1-2]。当前Pm感染依然是我国养猪业中最严重的危害之一,然而其所受到的重视程度远不及链球菌、副猪嗜血杆菌、猪繁殖与呼吸综合征病毒等病原。目前依然缺乏对我国猪群中流行的Pm较为系统的流行病学尤其是分子流行病学资料。因此,本研究针对2013年12月—2017年12月4年间我国猪群中流行的Pm进行了分离鉴定和分子流行病学调查,对于进一步了解猪致病性Pm的生物学特性及疫苗研发具有一定的参考价值。
1 材料与方法 1.1 试验材料样品为2013年12月—2017年12月4年间来源于全国各地区(湖北、湖南、广东、广西、安徽、福建、河北、河南、黑龙江、江苏、江西、内蒙古、山西、上海、四川、海南、贵州、重庆、甘肃、陕西、山东、辽宁、新疆等23个省/市/自治区)规模化猪场患有疑似呼吸系统疾病猪的肺、鼻拭子、气管、肝等样品,置-80 ℃保存以供检测和细菌分离。
Pm参考菌株HB03(荚膜A型;GenBank登录号:CP003328)[8]、HN04(荚膜B型;GenBank登录号:PPVE00000000)[9]、HN05(荚膜D型不产毒素;GenBank登录号:PPVF00000000)[9]、HN06(荚膜D型产毒素;GenBank登录号:CP003313)[10]及HN07(荚膜F型;GenBank登录号:CP007040)[11]由本实验室分离并保存,在本研究中用作参考菌株。
1.2 细菌的分离培养及鉴定选取组织中病变严重的部位用70%酒精处理表面后用经高压灭菌的剪刀剪碎,然后加入适量的0.9%无菌生理盐水进行匀浆处理。用无菌接种环蘸取组织匀浆划线接种于含NAD(10 μg·mL-1)及5%新生牛血清的TSA平板上,然后将平板置于37 ℃恒温条件下培养24~48 h。待平板上长出肉眼可见的菌落后,按照《临床微生物学手册》(第十版)中的标准方法对菌落进行纯化培养。然后根据菌落的形态挑取疑似Pm的单菌落进行革兰染色鉴定、生化鉴定以及利用Townsend等[3]建立的PCR方法扩增Pm的KMT1基因作最终确认。
1.3 荚膜分型、脂多糖分型、MLST基因分型Pm的荚膜基因型通过Townsend等[3]所建立PCR方法确定。PCR反应在20 μL反应体系中进行,包括:2 μL模板DNA、10 μL 2 × Taq Master Mix (Dye Plus, 南京Vazyme)、10 μmol·L-1上、下游引物(见参考文献[3])各1 μL、PCR反应用水6 μL。PCR反应程序:95 ℃预变性10 min,94 ℃变性45 s、56 ℃退火5 s,72 ℃延伸1 min,30个循环;72 ℃延伸10 min。PCR反应结束后产物利用1%琼脂糖进行凝胶电泳分析。PmHB03、HN04、HN06以及HN07在PCR反应中用作参考菌株。
Pm的脂多糖基因型通过Harper等[4]所建立PCR方法确定。PCR反应采用25 μL反应体系:2 μL模板DNA、12.5 μL 2 × Taq Master Mix (Dye Plus, 南京Vazyme)、10 μmol·L-1上、下游引物(见参考文献[4])各1 μL、PCR反应用水8.5 μL。PCR反应程序:95 ℃预变性10 min;94 ℃变性30 s、54 ℃退火30 s,72 ℃延伸2.5 min,30个循环;72 ℃延伸5 min。PCR反应结束后产物利用1%琼脂糖进行凝胶电泳分析。
多序列位点分型按照PmMLST官网(http://pubmLst.org/pmultocida)推荐的PCR方法(https://pubmlst.org/pmultocida/multihost/Pmultocida_MLST_Protocols.pdf)分别扩增7个管家基因:腺苷酸环化酶基因(adk)、5-烯醇丙酮酰莽草酸-3-磷酸合成酶基因(aroA)、嘌呤核苷酸磷酸化酶基因(deoD)、谷氨酸脱氢酶基因(gdhA)、葡萄糖-6磷酸脱氢酶基因(g6pd)、苹果酸脱氢酶基因(mdh)、磷酸葡萄糖异构酶基因(pgi)。PCR反应结束后产物利用1%琼脂糖进行凝胶电泳分析。扩增条带大小正确的产物送往BGI(深圳)利用对应的测序引物(https://pubmlst.org/pmultocida/multihost/Pmultocida_MLST_Protocols.pdf)进行测序,并将序列提交至PmMLST官网中的多宿主来源PmMLST数据库(Multiple host MLST database)进行MLST基因分型。
1.4 主要毒力基因检测Pm的毒力因子编码基因检测按照Khamesipour等[12]建立的PCR方法进行。一共检测23种毒力因子编码相关基因,具体如下:与菌毛和黏附素编码相关的基因ptfA、fimA、hsf-1、hsf-2、pfhA、tadD;毒素编码基因toxA;与铁元素摄取和代谢有关的基因exbB、exbD、tonB、hgbA、hgbB、Fur、tbpA;唾液酸代谢酶编码基因nanB和nanH;透明质酸酶编码基因pmHAS;外膜蛋白编码基因ompA、ompH、oma87、plpB;超氧化物歧化酶编码基因sodA和sodC。PCR反应在20 μL体系中进行,包括:2 μL模板DNA、10 μL 2 × Taq Master Mix (Dye Plus, 南京Vazyme)、10 μmol·L-1上、下游引物(见参考文献[12])各1 μL、PCR反应用水6 μL。PCR反应程序:95 ℃预变性10 min;94 ℃变性45 s,55 ℃退火45 s,72 ℃延伸1 min,30个循环;72 ℃延伸10 min。PCR反应结束后产物利用1%琼脂糖进行凝胶电泳分析。
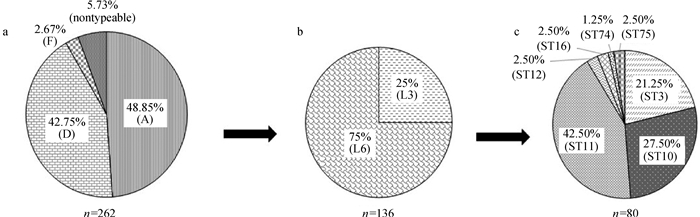
2 结果 2.1 我国猪群中流行的多杀性巴氏杆菌的荚膜基因型笔者从2013年12月—2017年12月4年间来源于全国各地区规模化猪场患有疑似呼吸系统疾病猪的肺、鼻拭子、气管、肝等样品中分离鉴定出262株Pm。对262株猪源Pm进行荚膜基因型鉴定,共鉴定出三种不同的荚膜基因型A、D、F以及“未定荚膜型”(荚膜基因型不属于A、B、D、E、F中的任何一种),分别含有128、112、7及15株,所占百分比分别为48.85%、42.75%、2.67%、5.73%(图 1a)。
|
图 1 262株猪Pm的荚膜基因型(a)、脂多糖基因型(b)以及多位点序列型(c)所占的百分比分布 Fig. 1 Percent distribution of the capsular types (a), LPS genotypes (b) and MLST genotyes (c) among the Pm isolates from pigs in this study |
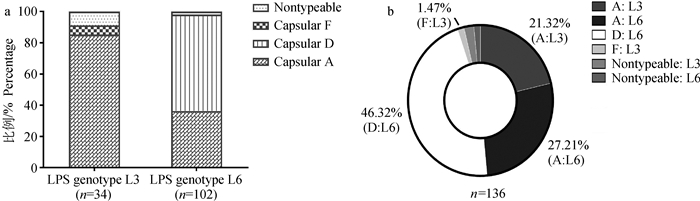
从所分离鉴定的262株Pm中随机选取136株进行LPS基因型鉴定,仅鉴定出两种LPS基因型L3和L6,分别含有34株和102株,所占比例分别为25.00%和75.00%(图 1b);编码L3型LPS的Pm分别为荚膜A、D和F型,各有29、0、2株;编码L6型LPS的Pm分别为荚膜A、D和F型,各有37、63、0株(图 2a)。136株猪Pm中共包含4种荚膜:LPS基因型组合A: L3、A: L6、D: L6、F: L3,各有29、37、63、2株(图 2b)。
|
图 2 136株猪多杀性巴氏杆菌的荚膜基因型和脂多糖基因型的百分比分布 Fig. 2 Percentage of the capsular types and LPS genotypes among the 136 isolates from pigs |
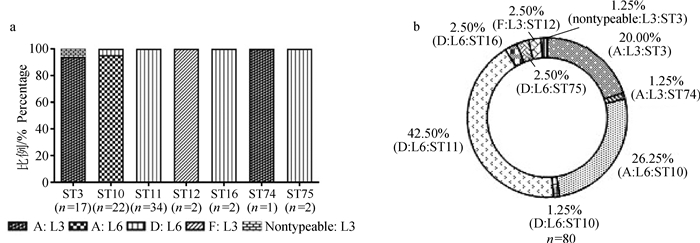
进一步从鉴定了脂多糖基因型的136株Pm中随机选取80株进行MLST基因型检测,共鉴定出7种MLST基因型ST3、ST10、ST11、ST12、ST16、ST74及ST75,分别含有17、22、34、2、2、1、2株,所占比例分别为21.25%、27.50%、42.50%、2.50%、2.50%、1.25%、2.50%(图 1c);就MLST基因型与荚膜基因型和LPS基因型的关系而言,ST3型菌株的荚膜:LPS基因型主要为A: L3(94.12%);ST10型菌株的荚膜:LPS基因型主要为A: L6(95.45%);ST11型菌株的荚膜:LPS基因型全部为D: L6型;ST12型菌株的荚膜:LPS基因型全部为F: L3;ST16型菌株的荚膜:LPS基因型全部为D: L6;ST74型菌株的荚膜:LPS基因型全部为A: L3;ST75型菌株的荚膜:LPS基因型全部为D: L6(图 3a)。80株猪多杀性巴氏杆菌中共包含8种荚膜:LPS:MLST基因型组合A: L3: ST3、A: L3: ST74、A: L6: ST10、D: L6: ST10、D: L6: ST11、D: L6: ST16、D: L6: ST75、F: L3: ST12,分别有16、1、21、1、34、2、2和2株(图 3b)。
|
图 3 80株猪多杀性巴氏杆菌的MLST基因型与荚膜基因型和脂多糖基因型的关系 Fig. 3 Percentage of the capsular types and LPS genotypes in each MLST genotype among the 80 Pm isolates from pigs |
针对NCBI数据库中已经公布全基因组序列的13株猪Pm进行荚膜基因型、LPS基因型以及MLST基因型分析,发现除了HN04的荚膜为B型外,其余的猪Pm的荚膜均为A、D或者F型;除了荚膜为B型的HN04编码产生L2型LPS外,其余菌株的LPS基因型均为L3或者L6;在13株猪Pm中一共鉴定出了5种MLST基因型——ST3、ST10、ST11、ST12、ST44;如果将13株猪Pm的荚膜基因型、LPS基因型以及MLST基因型组合起来看会发现,虽然荚膜A型Pm既编码L3型LPS又编码L6型LPS,但是A: L3型Pm全部为MLST基因型ST3,而A: L6型菌株全部为MLST基因型ST10;相对地,荚膜D型Pm的基因型则全部为D: L6: ST11型;而荚膜F型Pm的基因型全部为F: L3: ST12型(表 1)。
|
|
表 1 已知全基因组序列的多杀性巴氏杆菌的基因型特征 Table 1 Genetic characteristics of the 14-genome sequence-known P. multocida strains |
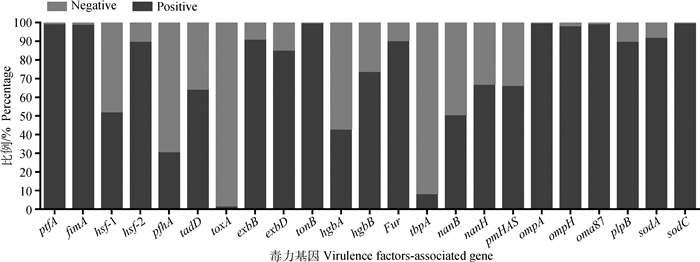
对全部262株Pm的23种毒力基因(ptfA、fimA、hsf-1、hsf-2、pfhA、tadD、toxA、exbB、exbD、tonB、hgbA、hgbB、Fur、tbpA、nanB、nanH、pmHAS、ompA、ompH、oma87、plpB、sodA、sodC)进行检测,结果显示部分毒力基因如ptfA、fimA、exbB、tonB、Fur、ompA、ompH、oma87、sodA、sodC等基因的检出率在90.00%以上,而toxA与tbpA的检出率低于10.00%,此外,部分毒力基因,如hsf-1、pfhA、tadD、hgbA、hgbB、nanB、nanH和pmHAS,其检出率在30.00%~80.00%(图 4)。
|
图 4 262株猪多杀性巴氏杆菌中23种毒力基因的分布百分比(n=262) Fig. 4 Percent distribution of the 23 virulence factors-associated genes among the 262 Pm originated from pigs |
进一步分析发现部分毒力基因的分布表现出一定的“基因型偏好性”,其中tadD和pmHAS表现出“荚膜基因型偏好性”强于“LPS基因型偏好性”;而pfhA、hgbA和hgbB表现出“LPS基因型偏好性”强于“荚膜基因型偏好性”;toxA、hsf-1和nanB则表现出特定的“荚膜:LPS基因型偏好性”,toxA仅在基因D: L6型Pm中被检测到,hsf-1在D: L6型Pm中的检出率显著高于其在其他荚膜:LPS基因型Pm中的检出率,nanB在基因型A: L6型Pm中的检出率显著高于其在其他荚膜:LPS基因型菌株中的检出率(图 5)。
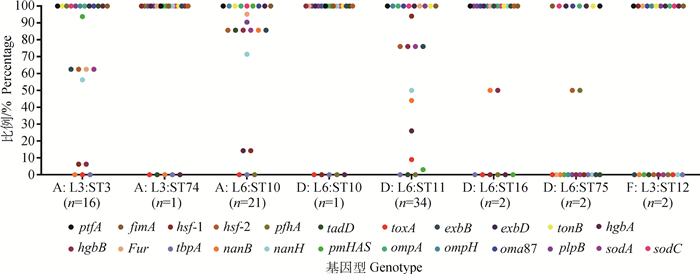
|
图 5 不同毒力基因在不同荚膜:脂多糖:MLST基因型的多杀性巴氏杆菌中所占的百分比 Fig. 5 Percent distribution of different virulence factors associated genes among different Pm capsular types: LPS genotypes: MLST genotypes |
本研究利用PCR方法对2013年12月—2017年12月4年间从我国病猪中分离鉴定的262株Pm进行了荚膜基因型、LPS基因型、MLST基因型以及毒力基因的鉴定。本研究的结果显示,当前在我国猪群中流行的Pm优势荚膜基因型依然为A和D型,与现有的关于我国猪多杀性巴氏杆菌的报道一致[13-17]。值得注意的是,我国猪群中流行的Pm优势荚膜基因型是随着时间的变化而不断变化的。据报道20世纪90年代以前在我国猪群中流行的多杀性巴氏杆菌的荚膜基因型以A和B型为主,其次是D型[18];大约30年后,即21世纪前十年,A和D型成了我国猪群中流行的多杀性巴氏杆菌的优势荚膜基因型,并且D型的检出率要显著高于A型(54.9% vs 39.5%),其次才是B型[19];然而在2010年以后,虽然A和D型依旧是我国猪群中流行的多杀性巴氏杆菌的优势荚膜基因型,但是A型所占的比例已经高于D型(49.3% vs 47.6%)[13]。而在本研究中,笔者发现荚膜A和D型所占的比例(48.85% vs 42.75%)在继续增加,这可能意味着在将来荚膜A型Pm有可能变成我国猪群中占主导地位的流行菌株。此外,一些在猪Pm中不常见的荚膜基因型(如F型)也被检出,已知荚膜F型Pm主要流行于禽类特别是火鸡,与禽霍乱的发生密切相关,此前极少有该型菌株分离自猪群的报道[2]。然而,最近几年我国常有F型Pm分离病猪的报道[15, 17, 20-22];笔者的后期研究表明荚膜F型Pm对猪具有较强的致病性[11],这意味着在我国猪群中流行的Pm可能发生了变化,并且F型也有可能成为未来我国猪群中流行的Pm重要荚膜基因型之一。
就LPS基因型而言,L3和L6型是当前在我国猪群流行的Pm主要LPS基因型,并且以L6为主;这一结果与王林柏等[23]的结果有较大差异,这可能是因为王林柏等所检测的样本数据量较小,在他们的研究中,只检测了4株猪Pm。相反地,笔者的检测样本量要大得多,因此笔者推测在我国猪群中流行的Pm脂多糖基因型应为L3和L6型。为了证实这一推测,笔者将GenBank数据库中已经公布全基因组序列的猪Pm进行了荚膜、LPS和MLST基因分型,结果发现这些Pm的LPS基因型多为L3和L6型(表 1),与笔者的检测结果相符。据报道,L3和L6型LPS的外核结构与脊椎动物许多细胞表面含有的P、Pk以及福斯曼抗原的低聚糖结构非常类似,这种相似的结构有助于表面被L3和L6型LPS包裹的细菌躲避宿主的免疫系统[24-25],这一机制也可以解释在猪群中流行的多杀性巴氏杆菌主要编码产生L3和L6型脂多糖的原因。如果将荚膜基因型和LPS基因型结合起来分析,发现荚膜:LPS基因型为D: L6的组合(46.32%)要显著高于其他组合(A: L3、A: L6以及A: L3)。值得一提的是,本实验室前期曾对保存的分离自猪萎缩性鼻炎临床病例的7株产毒Pm进行了荚膜和脂多糖分型,发现这些产毒Pm全部为D: L6型菌株[26]。这意味着荚膜基因型:LPS基因型为D: L6的组合可能是一个在临床上与由Pm导致的猪呼吸道疾病密切相关的基因型组合。
对于MLST基因型而言,ST11、ST10及ST3是我国猪群中流行的Pm常见MLST基因型,其中ST11型菌株占主导地位。国外有学者也曾报道ST11是在西班牙猪群中流行的Pm中最常见的MLST基因型[27];而本实验室保存的分离自猪萎缩性鼻炎临床病例的7株产毒多杀性巴氏杆菌的MLST基因型也都为ST11型[26],这说明ST11型在临床上可能与猪呼吸道疾病密切相关。此外,也有学者报道多杀性巴氏杆菌ST11型与兔的巴氏杆菌肺炎存在一定的关系[28],这些结果均说明ST11有可能为Pm一个重要的致病型。除了ST11外,ST3和ST10所占的比例也很高,并且本研究首次鉴定出了ST74和ST75型,这两种MLST基因型在多杀性巴氏杆菌中还未见报道。
本研究还发现Pm的荚膜基因型及LPS基因型与MLST基因型之间可能存在一定的关系,例如本研究中鉴定的所有A: L3型菌株的MLST基因型均为ST3,而所有A: L6型菌株的MLST基因型均为ST10,所有D: L6型菌株的MLST基因型均为ST11,所有F: L3型菌株的MLST基因型均为ST12,并且本研究在针对已经公布了全基因组序列的13株猪Pm进行荚膜基因型、LPS基因型及MLST基因型进行分析时也发现了相同的规律(表 1),这意味着在猪群中流行的Pm可能表现出特定的基因型特征。当然还需要更多的临床流行病学数据来对这一猜测进行确认。如果将所有80株猪Pm的荚膜基因型、LPS基因型和MLST基因型组合起来分析,笔者发现D: L6: ST11型菌株所占的比例(42.50%)要远远高于其他基因型组合,而已有的报道显示所有分离自猪萎缩性鼻炎临床病例的产毒Pm也全部为D: L6: ST11型菌株[26],这说明荚膜:脂多糖:MLST基因型组合D: L6: ST11是我国猪群中流行的Pm的主要基因型,并且该基因型可能与我国猪群中由Pm导致的呼吸道疾病密切相关。
就毒力基因而言,已有的研究显示在Pm中,参与细菌黏附定植(ptfA、fimA、hsf-2)、铁摄取(exbB、exbD、tonB、Fur)的基因以及超氧化物歧化酶基因(sodA、sodC)、外膜蛋白(ompA、ompH、oma87、plpB)基因在猪多杀性巴氏杆菌中广泛存在且具有较高的检出频率[19, 29-30],这些基因在本研究中的检出率都高于85.00%(图 4),并且这些基因在不同基因型的Pm中的检出率也都非常高,且无显著差异(图 5)。值得一提的是这些毒力基因在其他宿主来源的Pm中的检出率也非常高[12, 29, 31-32],这些毒力基因或许可以作为比较好的免疫原候选基因,其对应的蛋白或许可以适合作为疫苗添加成分提供针对不同基因型和/或不同宿主来源的Pm感染的保护力。
4 结论当前在我国猪群中流行的多杀性巴氏杆菌(Pm)基因型存在一定程度的多样性,但是荚膜:LPS:MLST基因型A: L3: ST3、A: L6: ST10、D: L6: ST11菌株在我国猪群中占主导地位,并且D: L6: ST11所占的比例最高,可能与我国猪群中由Pm导致的呼吸道疾病密切相关,这对于猪致病性Pm的疫苗研发具有一定的参考价值。
| [1] | WILSON B A, HO M. Pasteurella multocida: from zoonosis to cellular microbiology[J]. Clin Microbiol Rev, 2013, 26(3): 631–655. DOI: 10.1128/CMR.00024-13 |
| [2] | WILKIE I W, HARPER M, BOYCE J D, et al. Pasteurella multocida: diseases and pathogenesis[M]//AKTORIES K, ORTH J H C, ADLER B. Pasteurella multocida. Berlin, Heidelberg: Springer, 2012, 361: 1-22. |
| [3] | TOWNSEND K M, BOYCE J D, CHUNG J Y, et al. Genetic organization of Pasteurella multocida cap loci and development of a multiplex capsular PCR typing system[J]. J Clin Microbiol, 2001, 39(3): 924–929. DOI: 10.1128/JCM.39.3.924-929.2001 |
| [4] | HARPER M, JOHN M, TURNI C, et al. Development of a rapid multiplex PCR assay to genotype Pasteurella multocida strains by use of the lipopolysaccharide outer core biosynthesis locus[J]. J Clin Microbiol, 2015, 53(2): 477–485. DOI: 10.1128/JCM.02824-14 |
| [5] | HARPER M, BOYCE J D, ADLER B. Pasteurella multocida pathogenesis: 125 years after pasteur[J]. FEMS Microbiol Lett, 2006, 265(1): 1–10. DOI: 10.1111/fml.2006.265.issue-1 |
| [6] | PENG Z, LIANG W, LIU W J, et al. Genomic characterization of Pasteurella multocida HB01, a serotype A bovine isolate from China[J]. Gene, 2016, 581(1): 85–93. |
| [7] | PENG Z, LIANG W, LIU W J, et al. Genome characterization of Pasteurella multocida subspecies septica and comparison with Pasteurella multocida subspecies multocida and gallicida[J]. Arch Microbiol, 2017, 199(4): 635–640. DOI: 10.1007/s00203-017-1341-x |
| [8] | LIANG W L, PENG Z, LIU W J, et al. Complete genome sequence of P. multocida serotype A strain HB03, isolated from swine[C]//Proceedings of the 23rd International Pig Veterinary Society Congress. Cancun, Mexico: IPVS, 2014, 2: 30. |
| [9] | PENG Z, LIANG W, WANG F, et al. Genetic and phylogenetic characteristics of Pasteurella multocida isolates from different host species[J]. Front Microbiol, 2018, 9: 1408. DOI: 10.3389/fmicb.2018.01408 |
| [10] | LIU W J, YANG M J, XU Z F, et al. Complete genome sequence of Pasteurella multocida HN06, a toxigenic strain of serogroup D[J]. J Bacteriol, 2012, 194(12): 3292–3293. DOI: 10.1128/JB.00215-12 |
| [11] | PENG Z, LIANG W, WANG Y G, et al. Experimental pathogenicity and complete genome characterization of a pig origin Pasteurella multocida serogroup F isolate HN07[J]. Vet Microbiol, 2017, 198: 23–33. DOI: 10.1016/j.vetmic.2016.11.028 |
| [12] | KHAMESIPOUR F, MOMTAZ H, MAMOREH M A. Occurrence of virulence factors and antimicrobial resistance in Pasteurella multocida strains isolated from slaughter cattle in Iran[J]. Front Microbiol, 2014, 5: 536. |
| [13] | LIU H S, ZHAO Z Q, XI X J, et al. Occurrence of Pasteurella multocida among pigs with respiratory disease in China between 2011 and 2015[J]. Irish Vet J, 2017, 70: 2. DOI: 10.1186/s13620-016-0080-7 |
| [14] | PENG Z, WANG H N, LIANG W, et al. A capsule/lipopolysaccharide/MLST genotype D/L6/ST11 of Pasteurella multocida is likely to be strongly associated with swine respiratory disease in China[J]. Arch Microbiol, 2018, 200(1): 107–118. DOI: 10.1007/s00203-017-1421-y |
| [15] |
林星宇, 胡凌, 王印, 等. 猪源多杀性巴氏杆菌的分离鉴定及荚膜血清型分型[J]. 浙江农业学报, 2016, 28(4): 558–562.
LIN X Y, HU L, WANG Y, et al. Isolation, identification and capsule serotyping of Pasteurella multocida originated from swine[J]. Acta Agriculturae Zhejiangensis, 2016, 28(4): 558–562. DOI: 10.3969/j.issn.1004-1524.2016.04.03 (in Chinese) |
| [16] |
席晓剑, 赵战勤, 薛云, 等. 猪源多杀性巴氏杆菌的病原流行病学及其毒力特性[J]. 中国兽医学报, 2015, 35(8): 1205–1210.
XI X J, ZHAO Z Q, XUE Y, et al. Prevalence and virulence of Pasteurella multocida in pig farms in central China[J]. Chinese Journal of Veterinary Science, 2015, 35(8): 1205–1210. (in Chinese) |
| [17] |
彭忠, 王庚, 吴广敬, 等. 猪源多杀性巴氏杆菌的分离与鉴定[J]. 中国兽医科学, 2015, 45(9): 943–951.
PENG Z, WANG G, WU G J, et al. Isolation and identification of Pasteurella multocida from swine[J]. Chinese Veterinary Science, 2015, 45(9): 943–951. (in Chinese) |
| [18] |
郭大和, 郑明, 潘松年. 我国多杀性巴氏杆菌荚膜抗原的血清学鉴定[J]. 畜牧兽医学报, 1979, 10(2): 67–78.
GUO D H, ZHENG M, PAN S N. Serological identification of capsular antigens of Pasteurella multocida in China[J]. Acta Veterinaria et Zootechnica Sinica, 1979, 10(2): 67–78. (in Chinese) |
| [19] | TANG X B, ZHAO Z Q, HU J Y, et al. Isolation, antimicrobial resistance, and virulence genes of Pasteurella multocida strains from swine in China[J]. J Clin Microbiol, 2009, 47(4): 951–958. DOI: 10.1128/JCM.02029-08 |
| [20] |
方超, 李润成, 魏榕, 等. 猪源荚膜血清F型多杀性巴氏杆菌的分离鉴定、毒力基因检测与致病性研究[J]. 中国预防兽医学报, 2017, 39(3): 210–214.
FANG C, LI R C, WEI R, et al. Identification, virulent factors detection and pathogenesis investigation of capsule serotype F Pasteurella multocida isolated from pigs[J]. Chinese Journal of Preventive Veterinary Medicine, 2017, 39(3): 210–214. (in Chinese) |
| [21] |
林星宇, 王印, 杨泽晓, 等. 猪源荚膜血清F型多杀性巴氏杆菌的分离鉴定[J]. 中国兽医科学, 2015, 45(6): 567–571.
LIN X Y, WANG Y, YANG Z X, et al. Isolation and identification of a Pasteurella multocida strain belonged to capsular type F from swine[J]. Chinese Veterinary Science, 2015, 45(6): 567–571. (in Chinese) |
| [22] |
王羽, 董文龙, 王巍, 等. 猪源荚膜血清F型多杀性巴氏杆菌分离鉴定[J]. 中国兽医杂志, 2017, 53(6): 31–33, 36.
WANG Y, DONG W L, WANG W, et al. Isolation and identification of a Pasteurella multocida strain belonged to capsular type F from swine[J]. Chinese Journal of Veterinary Medicine, 2017, 53(6): 31–33, 36. (in Chinese) |
| [23] |
王林柏, 孙久鹤, 郭东春, 等. 国内部分地区多杀性巴氏杆菌荚膜血清型和基因型的研究[J]. 中国预防兽医学报, 2016, 38(2): 116–119.
WANG L B, SUN J H, GUO D C, et al. Identification of capsule serotype and genotype of Pasteurella multocida in some areas of China[J]. Chinese Journal of Preventive Veterinary Medicine, 2016, 38(2): 116–119. DOI: 10.3969/j.issn.1008-0589.2016.02.07 (in Chinese) |
| [24] | HARPER M, ST MICHAEL F, JOHN M, et al. Pasteurella multocida Heddleston serovar 3 and 4 strains share a common lipopolysaccharide biosynthesis locus but display both inter-and intrastrain lipopolysaccharide heterogeneity[J]. J Bacteriol, 2013, 195(21): 4854–4864. DOI: 10.1128/JB.00779-13 |
| [25] | HARPER M, ST MICHAEL F, JOHN M, et al. Structural analysis of lipopolysaccharide produced by Heddleston serovars 10, 11, 12 and 15 and the identification of a new Pasteurella multocida lipopolysaccharide outer core biosynthesis locus, L6[J]. Glycobiology, 2014, 24(7): 649–659. DOI: 10.1093/glycob/cwu030 |
| [26] |
王豪男.我国部分省区猪源多杀性巴氏杆菌的分子流行病学调查[D].武汉: 华中农业大学, 2017.
WANG H N. Molecular epidemiology of Pasteurella multocida from swine in some provinces of China[D]. Wuhan: Huazhong Agricultural University, 2017. (in Chinese) |
| [27] | GARCÍA-ALVAREZ A, VELA A I, SAN MARTÍN E, et al. Characterization of Pasteurella multocida associated with ovine pneumonia using multi-locus sequence typing (MLST) and virulence-associated gene profile analysis and comparison with porcine isolates[J]. Vet Microbiol, 2017, 204: 180–187. DOI: 10.1016/j.vetmic.2017.04.015 |
| [28] | GARCÍA-ALVAREZ A, CHAVES F, FERNÁND-EZ A, et al. An ST11 clone of Pasteurella multocida, widely spread among farmed rabbits in the Iberian Peninsula, demonstrates respiratory niche association[J]. Infect Genet Evol, 2015, 34: 81–87. DOI: 10.1016/j.meegid.2015.07.018 |
| [29] | EWERS C, LVBKE-BECKER A, BETHE A, et al. Virulence genotype of Pasteurella multocida strains isolated from different hosts with various disease status[J]. Vet Microbiol, 2006, 114(3-4): 304–317. DOI: 10.1016/j.vetmic.2005.12.012 |
| [30] | GARCÍA N, FERNÁNDEZ-GARAYZÁBAL J F, GOYACHE J, et al. Associations between biovar and virulence factor genes in Pasteurella multocida isolates from pigs in Spain[J]. Vet Rec, 2011, 169(14): 362. DOI: 10.1136/vr.d4869 |
| [31] | FURIAN T Q, BORGES K A, LAVINIKI V, et al. Virulence genes and antimicrobial resistance of Pasteurella multocida isolated from poultry and swine[J]. Braz J Microbiol, 2016, 47(1): 210–216. DOI: 10.1016/j.bjm.2015.11.014 |
| [32] | SARANGI L N, THOMAS P, GUPTA S K, et al. Virulence gene profiling and antibiotic resistance pattern of Indian isolates of Pasteurella multocida of small ruminant origin[J]. Comp Immunol Microbiol Infect Dis, 2015, 38: 33–39. DOI: 10.1016/j.cimid.2014.11.003 |



