2. 青藏高原动物遗传资源保护与利用教育部重点实验室, 成都 610041
2. Key Laboratory of Qinghai-Tibetan Plateau Animal Genetic Resource Reservati on and Utilization, Chengdu 610041, China
诺如病毒(norovirus,NoV)属于杯状病毒科,诺如病毒属,是单链的RNA病毒,基因组全长7.3~7.5 kb,是世界范围内引起人类与多种动物肠道胃肠炎的重要病原之一[1]。根据基因组特征,可将NoV分为6个基因型,其中GI、GII和GIV可以感染人类,GII可感染猪,GIV可感染猫、狗,GV可感染小鼠,GVI可感染狗[2];感染牛的诺如病毒(bovine norovirus, BNoV)为基因型GIII,对人类健康并不够成威胁[3];基于BNoV基因序列,GIII型又可分为GIII.1和GIII.2两个亚型, 通过部分RdRp序列以及VP1序列的遗传进化分析,都可将两亚型区分开。BNoV是一种引起犊牛腹泻的病原,感染GIII.1毒株的小牛在肠道损伤和相关临床症状上都比感染GIII.2更严重[4-5]。
目前19个国家有检出BNoV的报道,地域上涵盖了欧洲、美洲、亚洲和非洲,其中GIII.1在意大利、韩国等9个国家检出,检出率在0.9%~10.7%;GIII.2型株在19个国家检出,但检出率差异较大,如在匈牙利、韩国和突尼斯等9国,检出率在3.8%~16.6%,而在美国、荷兰及挪威3国的检出率达到20.8%~80%,已成为当地犊牛腹泻的重要病原之一,并且该病毒在小牛中的健康带毒现象也较普遍[6-9]。尽管BNoV已经有广泛的地域分布,但目前尚无合适分离培养体系,对该病毒的认识至今仍然很有限,且基因组序列较少限制了对该病毒分子特征和遗传进化的深入了解[6]。
目前,GenBank数据库中共有5个BNoV基因组。该病毒基因组具有三个开放阅读框(ORF),从5′端开始,ORF1编码多聚蛋白,GIII.1型1 681 aa,GIII.2型1 685 aa,二者长度上相差4 aa;该多聚蛋白被病毒编码的蛋白酶(3CLPro)切割后分为6个成熟的非结构蛋白,从5′端到3′端分别如下:N端非结构蛋白NS1-2(p48),NS3核苷酸三磷酸酶(NTPase)/ RNA解旋酶,NS4蛋白(p22),NS5蛋白(VPg),NS6蛋白酶(3CLPro)和NS7 RNA依赖的RNA聚合酶(RdRp), 其中RdRp使用核糖核苷三磷酸(rNTP)作为底物,在RNA病毒基因组的复制中发挥核心作用[10]。ORF2编码主要衣壳蛋白(VP1),GIII.1型为519 aa,GIII.2型为522 aa,二者长度上相差3 aa;VP1包含两个主要结构域:一个为保守的S结构域,以及一个更可变的P结构域,该蛋白负责病毒的衣壳组装、受体识别、宿主特异性、毒株多样性和免疫原性[11-12];ORF3编码次要结构蛋白(VP2),是一种在NoV病毒颗粒中起稳定性作用的蛋白[13],GIII.1型224 aa,GIII.2型283 aa,两型间长度相差较大,达到59 aa。
最近,笔者实验室证明了BNoV GIII.1在中国的存在[7],但并未对BNoV在国内的流行情况及分子特征做进一步研究。本研究的目的是对国内5省区奶牛的BNoV感染情况进行调查,并对流行毒株的分子特征进行研究,为犊牛腹泻的防控提供参考。
1 材料与方法 1.1 奶牛犊牛粪便样本的采集147份奶牛犊牛(3 d~4月)粪便样本,包括93份腹泻样本和54份健康样本,分别来自四川、辽宁、河南、山东、陕西等5个省,采样时间为2017年11月到2018年5月。样本详细信息见表 1。
|
|
表 1 各省粪便样本BNoV检出情况 Table 1 Detection of BNoV in stool samples from each province |
粪便按照1:5的比例与PBS溶液涡旋混匀,将悬浮液进行5 000 r·min-1离心5 min后取上清液;使用TrizolTM试剂(TaKaRa,中国)按照说明从400 μL悬浮液中提取总RNA。使用PrimescriptTM反转录试剂盒(TaKaRa,中国)合成cDNA并储存于-20 ℃。
1.3 RT-PCR方法检测BNoV按照文献[14]的RT-PCR方法检测BNoV,上游引物:5′-AGTTAYTTTTCCTTYTAYGGBGA-3′,下游引物:5′-AGTGTCTCTGTCAGTCATCTTCAT-3′,靶基因为RdRp,扩增片段长度为532 bp(4 555—5 086 bp),PCR产物进行双向测序后进行遗传进化分析。
1.4 混合感染调查对BNoV阳性样本进一步进行牛轮状病毒(BRV),牛冠状病毒(BCoV),牛病毒性腹泻病毒(BVDV)3种病原的检测,其中冠状病毒检测方法参照本实验室建立的方法[15],检测引物为F: 5′-GGTAGCGGCGTTATTTCC-3′,R: 5′-CGCCATCTGAG-TGATTACTC-3′, 轮状病毒检测方法参照本实验室建立的方法[16],引物为F: 5′-CGAGTTGAACACCCAGAT-3′,R: 5′-GAGACGGGCATCTACACT-3′,牛病毒性腹泻病毒检测方法参照Gong等[17]建立的方法,引物为F: 5′-GCCATGCCCTTAGTAGGACT-3′, R:5′-CACCCTATCAGGCTGTRTYC-3′。
1.5 VP1和VP2基因片段的扩增参考GenBank中BNoV基因组序列,设计了扩增VP1基因片段的引物,上游引物:5′-TGCTCTGCCTCCTTGGTG-3′, 下游引物:5′-CCTGRTGGAARAGGATGTT-3′,预期产物长度为700 bp,包含了部分RdRp序列和505 bp的VP1片段。PCR产物进行双向测序,对其中的VP1序列做进一步分析。
参考GenBank中BNoV基因组序列,设计扩增VP2基因片段的引物,上游引物: 5′-GTCGGGTCCTGTTCGAGG-3′,下游引物: 5′-AATGGCATCCGATCTGTATT-3′,预期片段长度为806 bp, 包含803 bp的VP2片段。PCR产物进行双向测序,其中的VP2序列用于进一步分析。
1.6 序列和遗传进化分析使用Seqman7.0(DNASTAR)拼接序列;使用MEGA7.0进行多序列比对并构建基于邻近法的系统发育树。使用DNASTAR7.0软件(DNASTAR公司)的MegAlign程序确定核苷酸和推导的氨基酸序列的相似性。
1.7 统计学分析采用卡方检验计算腹泻犊牛粪便和健康犊牛粪便中BNoV检出率间的差异显著性。
2 结果 2.1 BNoV的检出率93份腹泻样本中有24份检出BNoV阳性,检出率为25.81%,极显著高于健康样本中的检出率9.26%(P < 0.01),表明BNoV与犊牛腹泻具有相关性。
在采集于四川省的腹泻粪便样本中检出率为34.78%(8/23),采集于辽宁省的腹泻粪便样本中检出率为75.00%(12/16),采集于河南省的腹泻粪便样本中检出率为11.11%(3/27),采集于山东省的腹泻粪便样本中检出率为9.09%(1/11),采集于陕西省的腹泻粪便样本中没有检出(0/16),具体情况见表 1。
2.2 BNoV与其他肠道病原的混合感染情况24份BNoV阳性的腹泻粪便样本的混合感染情况结果显示,有3个样本为诺如病毒单独感染,其他21个BNoV阳性样本存在与其他3种肠道病原的混合感染,见表 2。
|
|
表 2 BNoV与其他肠道病原混合感染情况 Table 2 Mixed infection of BNoV with other intestinal pathogens |
对29份样本的RdRp序列分析表明,24份样本为GIII.2型(样本编号:L1、L4、L9、L11、L12、L13、L16、L18、CZ03、CZ06、CZ10、CZ13、CZ15、CZ18、CZ19、CZ20、C5、A3、J4、J8、Z4、Z9、Z13、B10),其中20份为腹泻样本,4份为健康样本;有5份样本为GIII.1型(样本编号:L5、L6、L7、L8、B18),其中4份为腹泻样本,1份为健康样本。
5个GIII.1型毒株间RdRp片段核苷酸序列相似性为85%~100%,推导的氨基酸序列相似性为85.2%~100%,与GenBank中其他GIII.1毒株的核苷酸序列相似性为82.9%~90.3%,推导的氨基酸序列相似性84.8%~90.2%。基于核苷酸序列的遗传进化分析表明,5个毒株单独聚为一小支,与GenBank中其他的GIII.1型毒株有明显的遗传距离(图 1)。与GenBank中所有的GIII.1型毒株核苷酸序列相比,本研究测定的5个GIII.1型毒株间RdRp片段有5个核苷酸位点发生了共同变异(C4674T、CA4769T、GG4844AC、GT4907A、T4955C),并导致了3个氨基酸位点的变异(SY1590FC、C1615YS、L1562P)。
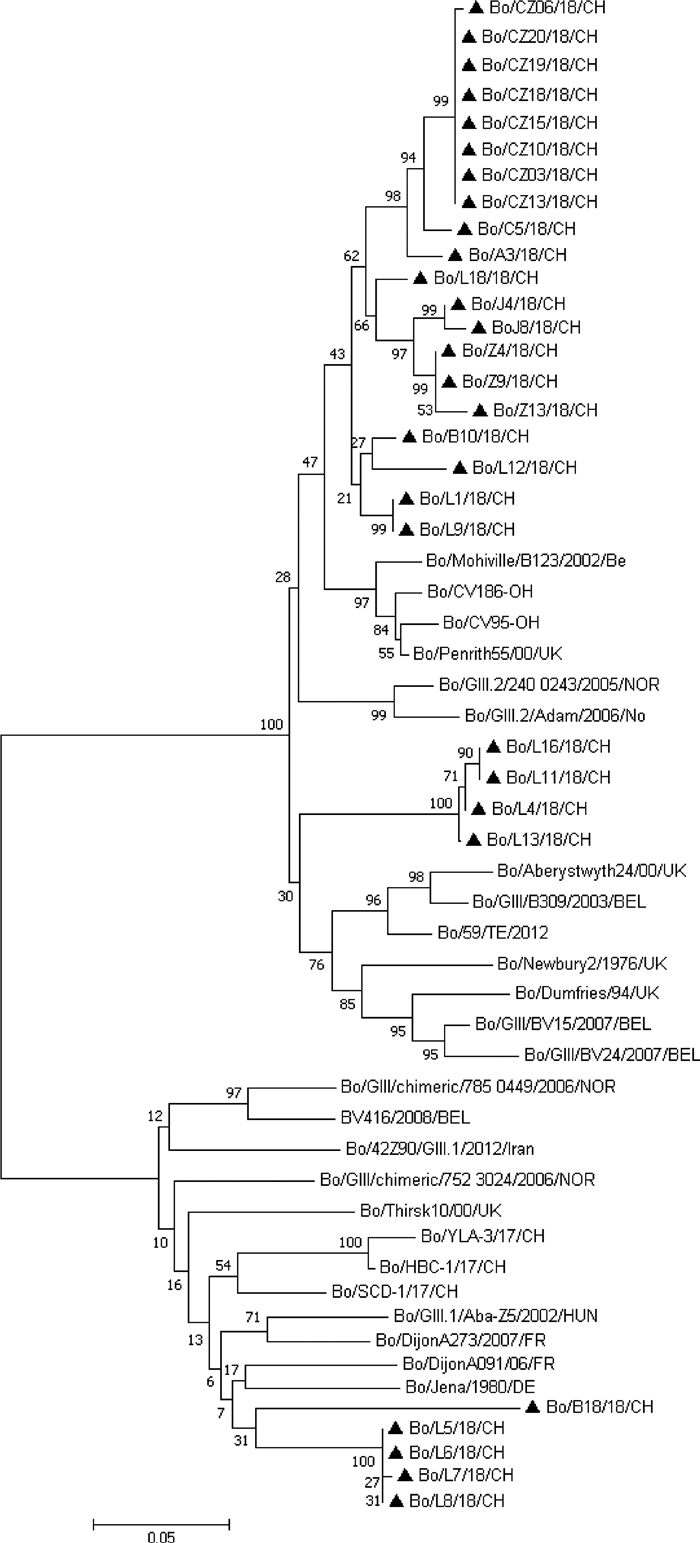
|
图 1 基于RdRp区域的遗传进化分析 Fig. 1 The phylogenetic tree of the partial nucleotide sequences of the RNA dependent RNA polymerase |
24个GIII.2型的毒株间核苷酸序列相似性为87.4%~100%,推导的氨基酸序列相似性为87.6%~100%,与GenBank中其他GIII.2型毒株核苷酸序列相似性为86.1%~94.8%,推导的氨基酸序列相似性为96.7~100%;基于核苷酸序列的遗传进化分析表明,本试验的24个毒株聚为2个独立的小支,其中一支包含了20个毒株,另外一支包含了4个毒株,见图 1。与GenBank中其他GIII.2型毒株相比,聚为独立1支的20个毒株的4836位点核苷酸均发生了变异(G4836CT);其余聚为独立1支的4个毒株共有17个核苷酸位点共同无义突变(CG4641A、G4656A、C4689T、C4685T、C4719T、A4756G、G4836T、G4845A、CT4851G、C4855TC、T4857GG、T4869CC、G4875TG、A4920T、T4977C、T4989C、GA5001T)。
本研究共获得18个VP1片段,其中17个为GIII.2序列,1个为GIII.1序列。17个GIII.2型毒株间的VP1序列核苷酸序列相似性为92.3%~100%(nt),推导的氨基酸序列相似性为96.5%~100%(aa),与GenBank中其他GIII.2型毒株的核苷酸序列相似性为85.2%~93.5%,推导的氨基酸序列相似性为94.9%~100%。根据其中的VP1区域片段进行遗传进化分析,17个样本聚为单独的一大支(图 2);将17个毒株的VP1核酸序列与GenBank中其他所有GIII.2型毒株的VP1核苷酸序列做对比,有2个位点发生了共同变异(G171A、GA327TC),这2处核苷酸变异没有引起氨基酸的改变。
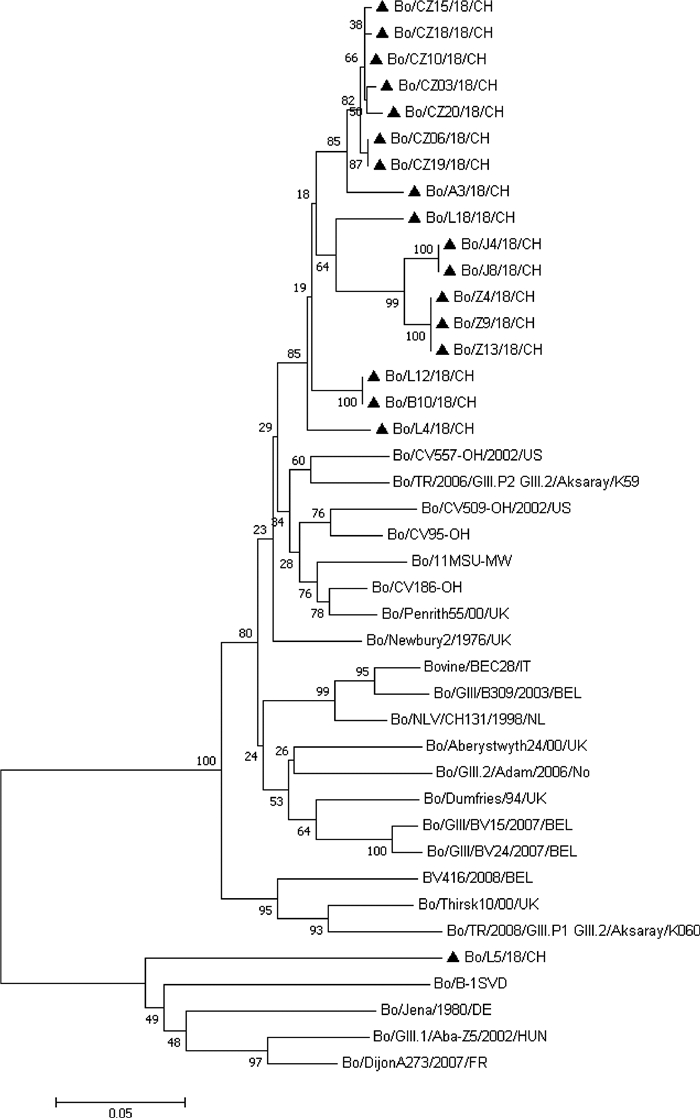
|
图 2 基于VP1区域(431 bp)的遗传进化分析 Fig. 2 The phylogenetic tree of the partial nucleotide sequences of the VP1 |
1个GIII.1型样本与GenBank中其他GIII.1型毒株的核苷酸序列相似性为81.7%~85.9%,推导的氨基酸序列相似性为91.6%~93.0%;遗传进化分析表明该毒株与其他已知的GIII.1型毒株聚为一大支(图 2),但单独聚为一小支;该毒株VP1核苷酸序列与GenBank中其他所有GIII.1型毒株的VP1核苷酸序列做对比,共有89个位点发生了改变,在氨基酸序列中共有6个位点为独特的变异,分别为E10D、P21S、N69Q、G82H、I103M、S131A。
获得了13个GIII.2型毒株的VP2基因片段(779 bp),13个毒株间核苷酸序列相似性为92.2%~100%,推导的氨基酸序列相似性为92.1%~100%,与GenBank里其他所有GIII.2毒株VP2基因核苷酸序列相似性为分别为81.8%~90.3%,推导的氨基酸序列相似性为84.3%~92.5%。基于VP2基因片段的遗传进化分析显示,13个毒株聚为单独的一大支,显示出独特的演化关系(图 3)。13个毒株VP2核苷酸序列与GenBank登录的所有GIII.2型毒株的VP2核苷酸序列对比,结果显示在12个位点上发生了共同变异(A379G、T371C、G416A、A431G、T435CC、A468G、T540C、G578A、T618C、T640AC、T717G、TC780A),并导致了6个位点氨基酸的共同变异(M124A、G139E、D144G、R193KQ、P197L、S214T)。
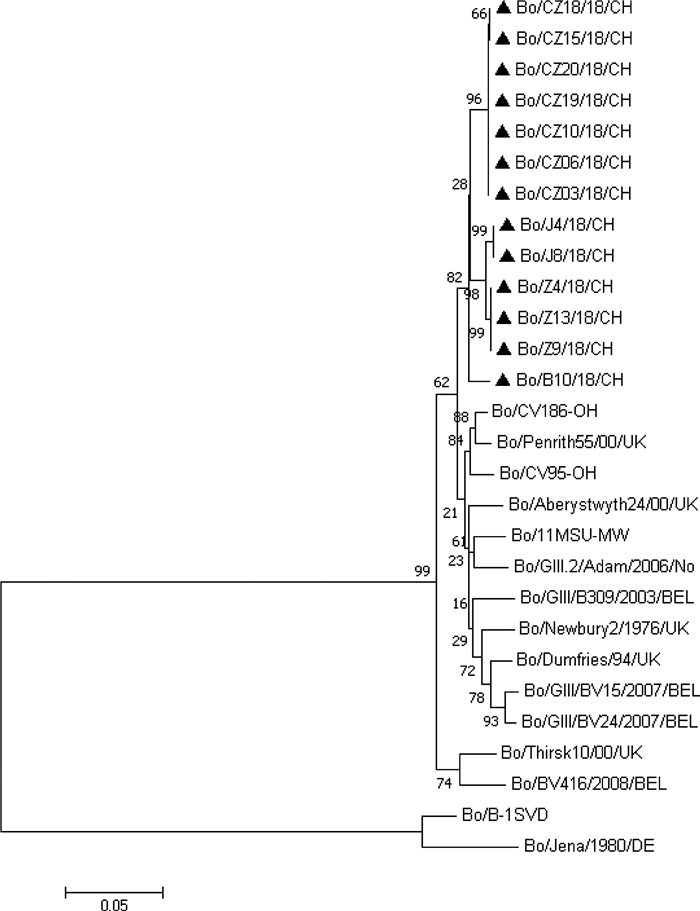
|
图 3 基于部分VP2序列的遗传进化分析 Fig. 3 The phylogenetic tree of the partial nucleotide sequences of the VP2 |
尽管目前尚无BNoV的分离培养体系,但在排除了其他病毒的含BNoV腹泻粪便无菌处理后感染新生无菌小牛后,引起急性持续性腹泻,并从粪便中获得较高含量的病毒[4];并且大量的病原流行病学资料也证明该病毒与犊牛腹泻具有相关性,因此,BNoV已是一种公认的犊牛腹泻的病原。本研究对国内5个省共计147份奶犊牛粪便样本进行了检测,结果表明BNoV在奶牛中普遍流行,腹泻样本中检出率达到25.81%,显著高于健康样本中的检出率,证明该病毒与犊牛腹泻相关,是一种导致国内犊牛腹泻的新发病原,并且有广泛的地域分布。在局部地区检出率高达75.0%,这与在美国部分地区BNoV的流行情况相似,有可能是一种重要的腹泻病原[18]。
混合感染调查结果显示,高达87.5%的BNoV与BRV、BCoV、BVDV三种病毒中的一种或几种存在共感染现象(表 1),与韩国的BNoV流行情况相似,其BNoV阳性样本中有超过60%的样本与其他肠道病毒存在混合感染[19]。BNoV感染不仅会引起犊牛持续性腹泻而且还会引起严重的绒毛萎缩以及绒毛上皮细胞的减少变薄[4],这将使犊牛对其他肠道病毒更易感并且临床表现更加严重。
犊牛腹泻是影响养牛业发展的一类重要疾病,给养殖生产带来了重大损失,且目前国内诊断犊牛腹泻时,往往只考虑到轮状病毒、冠状病毒和BVDV等几个主要导致牛腹泻的病原,对新发腹泻病毒关注很少。本研究的结果提示应将BNoV纳入国内犊牛腹泻病的常规诊断中,为奶牛腹泻病的科学防控提供依据。
3.2 RdRp的分子特征基于RdRp序列的遗传进化分析表明,本试验共获得5个GIII.1型与24个GIII.2型532 bp的 RdRp片段。有趣的是,本研究所得5个GIII.1型毒株与2017年本实验室首次在国内检测到的两个BNoV GIII.1毒株(Bo/YLA-3/17/CH、Bo/HBC-1/17/CH)遗传距离较远,这可能是样品来源地不同的原因,表明国内流行的BNoV GIII.1型毒株的RdRp存在遗传多样性。
NoV的RdRp是负责病毒基因组转录和复制的关键酶,准确启动RNA合成对于防止病毒遗传信息的损失至关重要[20], 值得注意的是,本试验获得的5个GIII.1型毒株独特的氨基酸的共同变异都位于编码RdRp拇指结构域的氨基酸序列上,对结合RNA和底物至关重要,因此所得毒株的这些氨基酸的共变异对RdRp的功能影响值得进一步关注。在人NoV的大流行毒株中,RdRp也显示出较低的复制保真度,其导致更高的突变率和病毒进化速率[21]。本研究检出的GIII.1型毒株在当地检出率超过20%,其RdRp的氨基酸变异对该毒株流行的影响值得进一步关注。
3.3 VP1区域分子特征本研究共获得1个GIII.1型与17个GIII.2型VP1片段,基于核苷酸的进化分析表明,毒株都聚为独立的一支,表现出了共进化的趋势;进一步分析发现,17个GIII.2型毒株,发生了较多的无义突变,在比对了GenBank中其他GIII.2毒株后也发现了类似的现象;通常来说,突变是RNA病毒的一种重要的进化方式,而目前的许多研究都表明了BNoV GIII.2呈现出相对缓慢的遗传进化,毒株间仅有较低的遗传差异[22-23]。目前,GIII.2 VP1无义突变在进化中的意义尚不清楚,有待进一步研究。
值得注意的是本研究测定的GIII.1型毒株序列,在高度保守的VP1 S区的部分氨基酸片段存在较多的共同变异,在杯状病毒的衣壳蛋白中, S结构域高保守,P1亚结构域仅有中等保守性,P2亚结构域为高度可变[24];S结构域对形成衣壳二十面体至关重要[25],且最近的一项研究表明在S结构域中有毒株特异性表位[26],本研究阐述的序列变异可能会对重要结构二十面体的形成和该特异性表位产生影响。
3.4 VP2分子特征VP2是BNoV中遗传进化差异最大的几个区域之一[27]。本研究测定的13个GIII.2型毒株的VP2基因片段在进化树上聚为单独的一大支,且有6个独特的氨基酸共同变异,体现了目前在国内流行的GIII.2型毒株独立的演化趋势和分子特征。VP2是一种微小的结构蛋白,目前尽管对BNoV VP2蛋白的功能及其在复制周期中的作用尚不清楚,但已经从感染NoV的人粪便中纯化的病毒颗粒中发现了VP2,VP2以一个或两个拷贝存在于每个病毒粒子,是病毒产生感染性所必需的,其很有可能涉及到衣壳组装和基因组衣壳化[14]。而国内流行的GIII.2型毒株与其他GIII.2毒株在VP2上的较大差异,可能会对该毒株的感染能力和组装有影响[14]。
4 结论研究证明BNoV已在国内的奶牛中广泛流行,是国内犊牛腹泻的新发病原,国内流行的BNoV毒株具有丰富的遗传多样性,包括GIII.1、GIII.2型,并且其RdRp、VP1和VP2基因具有独特的演化趋势,为国内犊牛腹泻的防控提供了重要参考。
| [1] | LIU B L, LAMBDEN P R, GÜNTHER H, et al. Molecular characterization of a bovine enteric calicivirus:relationship to the Norwalk-like viruses[J]. J Virol, 1999, 73(1): 819–825. |
| [2] | ZHENG D P, ANDO T, FANKHAUSER R L, et al. Norovirus classification and proposed strain nomenclature[J]. Virology, 2006, 346(2): 312–323. DOI: 10.1016/j.virol.2005.11.015 |
| [3] | OLIVER S L, DASTJERDI A M, WONG S, et al. Molecular characterization of bovine enteric caliciviruses:a distinct third genogroup of noroviruses (Norwalk-like viruses) unlikely to be of risk to humans[J]. J Virol, 2003, 77(4): 2789–2798. DOI: 10.1128/JVI.77.4.2789-2798.2003 |
| [4] | OTTO P H, CLARKE I N, LAMBDEN P R, et al. Infection of calves with bovine norovirus GⅢ. 1 strain Jena Virus:an experimental model to study the pathogenesis of norovirus infection[J]. J Virol, 2011, 85(22): 12013–12021. DOI: 10.1128/JVI.05342-11 |
| [5] | JUNG K, SCHEUER K A, ZHANG Z, et al. Pathogenesis of GⅢ. 2 bovine norovirus, CV186-OH/00/US strain in gnotobiotic calves[J]. Vet Microbiol, 2014, 168(1): 202–207. DOI: 10.1016/j.vetmic.2013.11.008 |
| [6] | DI FELICE E, MAUROY A, DAL POZZO F, et al. Bovine noroviruses:a missing component of calf diarrhoea diagnosis[J]. Vet J, 2016, 207: 53–62. DOI: 10.1016/j.tvjl.2015.10.026 |
| [7] | GUO Z J, HE Q F, YUE H, et al. First detection of nebovirus and norovirus from cattle in China[J]. Arch Virol, 2018, 163(2): 475–478. DOI: 10.1007/s00705-017-3616-6 |
| [8] | MOHAMED F F, MANSOUR S M G, EL-ARABY I E, et al. Molecular detection of enteric viruses from diarrheic calves in Egypt[J]. Arch Virol, 2017, 162(1): 129–137. DOI: 10.1007/s00705-016-3088-0 |
| [9] | POURASGARI F, KAPLON J, SANCHOOLI A, et al. Molecular prevalence of bovine noroviruses and neboviruses in newborn calves in Iran[J]. Arch Virol, 2018, 163(5): 1271–1277. DOI: 10.1007/s00705-018-3716-y |
| [10] | LEE J H, CHUNG M S, KIM K H. Structure and function of caliciviral RNA polymerases[J]. Viruses, 2017, 9(11): 329. DOI: 10.3390/v9110329 |
| [11] | CHEN R, NEILL J D, NOEL J S, et al. Inter-and intragenus structural variations in caliciviruses and their functional implications[J]. J Virol, 2004, 78(12): 6469–6479. DOI: 10.1128/JVI.78.12.6469-6479.2004 |
| [12] | PRASAD B V V, HARDY M E, DOKLAND T, et al. X-ray crystallographic structure of the Norwalk virus capsid[J]. Science, 1999, 286(5438): 287–290. DOI: 10.1126/science.286.5438.287 |
| [13] | YAO L, LI F L, WANG L Z, et al. Function of VP2 protein in the stability of the secondary structure of virus-like particles of genogroup Ⅱ norovirus at different pH levels:function of VP2 protein in the stability of NoV VLPs[J]. J Microbiol, 2014, 52(11): 970–975. DOI: 10.1007/s12275-014-4323-6 |
| [14] | SMILEY J R, HOET A E, TRÅVÉN M, et al. Reverse transcription-PCR assays for detection of bovine enteric caliciviruses (BEC) and analysis of the genetic relationships among BEC and human caliciviruses[J]. J Clin Microbiol, 2003, 41(7): 3089–3099. DOI: 10.1128/JCM.41.7.3089-3099.2003 |
| [15] |
何琪富, 郭紫晶, 李然, 等. 牛冠状病毒RT-PCR检测方法的建立及应用[J]. 畜牧兽医学报, 2018, 49(10): 2292–2298.
HE Q F, GUO Z J, LI R, et al. Establishment and application of a RT-PCR assay for detecting bovinecoronavirus[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(10): 2292–2298. (in Chinese) |
| [16] |
周芳, 岳华, 张斌, 等. 牦牛轮状病毒VP6基因序列分析及RT-PCR检测方法的建立与应用[J]. 畜牧兽医学报, 2016, 47(7): 1465–1473.
ZHOU F, YUE H, ZHANG B, et al. Establishment and application of an RT-PCR assay for yak rotavirus based on the sequence analysis of yak rotavirus VP6 gene[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(7): 1465–1473. (in Chinese) |
| [17] | GONG X W, LIU L H, ZHENG F Y, et al. Molecular investigation of bovine viral diarrhea virus infection in yaks (Bos gruniens) from Qinghai, China[J]. Virol J, 2014, 11: 29–29. DOI: 10.1186/1743-422X-11-29 |
| [18] | WISE A G, MONROE S S, HANSON L E, et al. Molecular characterization of noroviruses detected in diarrheic stools of Michigan and Wisconsin dairy calves:circulation of two distinct subgroups[J]. Virus Res, 2004, 100(2): 165–177. DOI: 10.1016/j.virusres.2003.11.014 |
| [19] | PARK S I, JEONG C, KIM H H, et al. Molecular epidemiology of bovine noroviruses in South Korea[J]. Vet Microbiol, 2007, 124(1-2): 125–133. DOI: 10.1016/j.vetmic.2007.03.010 |
| [20] | KAO C C, SINGH P, ECKER D J. De novo initiation of viral RNA-dependent RNA synthesis[J]. Virology, 2001, 287(2): 251–260. DOI: 10.1006/viro.2001.1039 |
| [21] | DEVAL J, JIN Z N, CHUANG Y C, et al. Structure(s), function(s), and inhibition of the RNA-dependent RNA polymerase of noroviruses[J]. Virus Res, 2017, 234: 21–33. DOI: 10.1016/j.virusres.2016.12.018 |
| [22] | DOMINGO E, HOLLAND J J. RNA virus mutations and fitness for survival[J]. Annu Rev Microbiol, 1997, 51: 151–178. DOI: 10.1146/annurev.micro.51.1.151 |
| [23] | MAUROY A, SCIPIONI A, MATHIJS E, et al. Complete genome sequence of a novel bovine norovirus:evidence for slow genetic evolution in genogroup Ⅲ genotype 2 noroviruses[J]. J Virol, 2012, 86(22): 12449–12450. DOI: 10.1128/JVI.02251-12 |
| [24] | HAN M G, SMILEY J R, THOMAS C, et al. Genetic recombination between two genotypes of genogroup Ⅲ bovine noroviruses (BoNVs) and capsid sequence diversity among BoNVs and Nebraska-like bovine enteric caliciviruses[J]. J Clin Microbiol, 2004, 42(11): 5214–5224. DOI: 10.1128/JCM.42.11.5214-5224.2004 |
| [25] | HARDY M E. Norovirus protein structure and function[J]. FEMS Microbiol Lett, 2010, 253(1): 1–8. |
| [26] | PARRA G I, SOSNOVTSEV S V, ABENTE E J, et al. Mapping and modeling of a strain-specific epitope in the Norwalk virus capsid inner shell[J]. Virology, 2016, 492: 232–241. DOI: 10.1016/j.virol.2016.02.019 |
| [27] | MAUROY A, SCIPIONI A, MATHIJS E, et al. Genetic and evolutionary perspectives on genogroup Ⅲ, genotype 2 bovine noroviruses[J]. Arch Virol, 2014, 159(1): 39–49. DOI: 10.1007/s00705-013-1791-7 |



