2. 河南省农业科学院动物免疫学 重点实验室, 农业部动物免疫学重点实验室, 河南省动物免疫学重点实验室, 郑州 450002;
3. 河南农业大学牧医工程学院, 郑州 450002;
4. 扬州大学江苏高校动物重要疫病与人兽共患防控协同创新中心, 扬州 225009
2. Key Laboratory of Animal Immunology of the Ministry of Agriculture, Henan Provincial Key Laboratory of Animal Immunology, Henan Academy of Agricultural Sciences, Zhengzhou 450002, China;
3. College of Animal Science and Veterinary Medicine, Henan Agricultural University, Zhengzhou 450002, China;
4. Jiangsu Co-Innovation Center for the Prevention and Control of Important Animal Infectious Disease and Zoonose, Yangzhou University, Yangzhou 225009, China
马立克病(Marek’s disease, MD)是由马立克病病毒(Marek’s disease virus, MDV)感染引起并导致家禽的免疫抑制和恶性组织淋巴性肿瘤病。MDV是双股DNA病毒,属于α疱疹病毒亚科[1],共有三种血清型:血清1型(MDV-1)、血清2型(MDV-2)以及火鸡疱疹病毒(HVT),其中仅有MDV-1具有致病/致瘤性。MDV-1在感染后4~8周内可在鸡体内引起急性T细胞淋巴瘤以及内脏、皮肤肿瘤[2]。MD是首个被证明病毒疫苗免疫接种可有效预防癌症发生的实例,因此MDV-1感染这种独特的天然病毒-宿主模型系统被认为是研究病毒诱导癌症发生机制的良好生物模型[3]。由于MDV基因组庞大,编码基因众多,其感染致病的机制、尤其是诱导肿瘤发生的分子机制至今仍是研究的焦点。
MicroRNA(miRNA)是一类长度介于22~24 nt的非编码小分子RNA,可通过种子序列5′末端的2~7位核苷酸与靶基因mRNA的3′-UTR碱基配对结合抑制其表达[4]。miRNA在转录后水平调节基因表达,影响多种生物学过程,包括细胞发育、分化、凋亡、疾病发生尤其是癌症生物学的各个方面[5-6]。动植物中已鉴定上万种miRNAs,病毒中也已发现了数百种[7]。MDV-1共编码14个miRNA前体,表达26个成熟体miRNA[8]。此前对这些病毒miRNA的体内动态表达谱分析发现,miR-M11-5p与MDV-1致病及致瘤的进程密切相关[9],而在对Mid基因簇各个miRNA进行基因缺失的致病性分析表明,与亲本毒株GX0101相比较,miR-M11-5p基因缺失毒株致瘤率明显增强,提示miR-M11-5p在MDV致瘤过程中可能作为肿瘤抑制因子发挥抑癌作用[10]。因此,miR-M11-5p调控MDV致瘤过程的分子机制值得进一步深入研究。
笔者利用hybrid-PCR方法[11]建立miR-M11-5p候选靶基因的cDNA文库,通过测序分析和双荧光素酶报告试验进行筛选,然后用miRNA过表达和RT-qPCR进行鉴定,以期发现miR-M11-5p的宿主靶基因,为揭示miR-M11-5p调控MDV致病及致瘤分子机制奠定基础。
1 材料与方法 1.1 毒株与细胞MDV超强毒株GX0101,由山东农业大学动物医学院崔治中教授馈赠。原代鸡胚成纤维细胞(chicken embryo fibroblast,CEF),由7~9日龄SPF鸡胚制备获得。人胚胎肾细胞HEK 239T细胞,由本实验室保存备用。
1.2 试剂与载体TRIzol® Reagent、lipofectamineTM 2000购自Invitrogen公司;3′-Full RACE Core Set with PrimeScript RTase、Ex Taq DNA聚合酶、pMD19-T(simple)、PrimeScriptTMRT reagent Kit with gDNA Eraser、SYBR® Premix Ex TaqTMⅡ(Tli RNaseH Plus)均购自宝生物工程(大连)有限公司;内切酶XhoⅠ、NotⅠ-HF购自NEB公司;Annealing Buffer for DNA Oligos (5×)购自碧云天生物技术公司;X-tremeGENE HP DNA Transfection Reagent购自Roche公司;DMEM、opti-MEM、Fetal Bovine Serum购自Gbico公司;0.25%胰蛋白消化酶购自新赛美公司;Dual-luciferase® Reporter Assay系统购自Promega公司;psiCHECK-2载体由河南省农业科学院动物免疫学重点实验室保存。
1.3 引物设计与合成候选靶基因3′-UTR扩增及RT-qPCR所需特异性引物对,均用Primer Premier 5.0设计(序列见表 1),由上海生物工程股份有限公司合成。Negative control、miR-M11-5p mimics和mut-miR-M11-5p mimics(序列见表 2),由上海吉玛制药技术有限公司合成。
|
|
表 1 候选靶基因3′-UTR扩增及RT-qPCR引物信息 Table 1 Primers used for amplifying the 3′-UTR and RT-qPCR analysis of the candidate target genes |
|
|
表 2 miRNA mimics合成信息 Table 2 miRNA mimics synthesis information |
利用7~9日龄SPF鸡胚制备CEF细胞,并用TRIzol试剂提取总RNA。测定RNA的OD260 nm/OD280 nm比值应在1.8~2.1范围内。将提取的RNA用3′-Full RACE Core Set with PrimeScript RTase试剂说明书反转录成cDNA,10 μL反应体系:RNA 1.0 μL、PrimeScript RTase (200 U·μL-1) 0.25 μL、RNase Inhibitor (40 U·μL-1) 0.25 μL、5×PrimeScript Buffer 2 μL、dNTP Mixture (10 mmol·L-1 each) 1 μL、3′RACE Adaptor (5 μmol·L-1) 1 μL、RNase Free dH2O 4.5 μL。以该cDNA为模板,按照试剂盒说明书进行两轮PCR反应。第一轮:用miR-M11-5p特异性锚定引物(5′-TCTCTRCRCGGTRRGGRRR-3′,“R”为“G”或“A”)为上游引物和试剂盒中所带3′-RACE Outer Primer (5′-TACCGTCGTTCCACTAGTAGTGATTT-3′)为下游引物,以反转录cDNA为模板进行。第二轮:用上述miR-M11-5p特异性锚定引物为上游引物、试剂盒中所带3′-RACE Inner Primer(5′-CGCGGATCCTCCACTAGTGATTTCACTATAGG-3′)为下游引物,以第一轮PCR产物为模板进行。将得到的第二轮PCR产物用1%的琼脂糖凝胶进行电泳分析,并用胶回收试剂盒回收纯化大小介于100 ~1 000 bp的3′RACE PCR终产物。将其连接到pMD19-T载体,转化至E. coli JM109中,涂布氨苄抗性LB琼脂平板,37 ℃培养箱倒置培养12 h,于超净工作台中挑取单个菌落于1 mL氨苄抗性LB液体培养基进行扩大培养。用miR-M11-5p特异性锚定引物和3′-RACE Inner Primer进行菌液PCR扩增,1%琼脂糖凝胶电泳分析,挑选阳性菌落送上海生工生物工程有限公司测序,测序结果用NCBI BLAST软件进行序列对比分析。
1.5 双荧光素酶报告载体构建 1.5.1 候选靶基因3′-UTR报告载体构建对miRNA结合靶点在3′-UTR的宿主候选靶基因优先进行扩增。从CEF基因组中提取RNA,反转录为cDNA为模板,用表 1所列的引物对(#1~37)进行PCR扩增。PCR反应体系:Ex Taq 10 μL,上下游引物各1 μL,cDNA 1 μL,ddH2O 7 μL。设置PCR程序为95 ℃ 5 min,98 ℃ 10 s,55 ℃ 30 s,72 ℃ 1 min,30个循环;72 ℃ 10 min。PCR产物用1%琼脂糖凝胶电泳分析。切胶回收目的片段,纯化后连接pMD19-T,16 ℃连接过夜,连接产物转化至E. coli JM109中,氨苄LB固体培养基涂板挑取单菌落,PCR鉴定后送测序。将目的片段用内切酶XhoⅠ/NotⅠ-HF从pMD19-T载体上切割并回收,克隆到经过同样酶切处理的psiCHECK-2载体上,16 ℃连接过夜,连接产物转化至E. coli JM109中,氨苄LB固体培养基涂板挑取单菌落。经PCR和双酶切鉴定,测序。用甘油将测序正确的菌种保存于-20 ℃冰箱中,并提取质粒用于后续试验。
1.5.2 候选靶基因3′-UTR突变载体构建设计合成5′和3′端分别是XhoⅠ或NotⅠ酶切位点的黏性末端、中间部分是与miR-M11-5p结合位点相对应的突变的候选靶基因3′-UTR片段的寡核苷酸(表 1,#38~40)。用ddH2O将寡核苷酸引物溶解至浓度为100 μmol·L-1,进行退火反应。ddH2O 12 μL、Annealing buffer 4 μL、正反寡核苷酸链各2 μL(100 μmol·L-1)。设置程序:95 ℃变性2 min,之后每90 s下降1 ℃,直至降到25 ℃。将退火后形成的双链结构直接与同样酶切处理过的psi-CHECK2载体上进行连接、转化、PCR、酶切鉴定和测序,方法同上。
1.6 双荧光素酶报告试验于转染前12~24 h将HEK 293T细胞接种到48孔板中。将吉玛公司合成的miR-M11-5p mimics、mut-miR-M11-5p mimics以及阴性对照negative control稀释为1 μmol·L-1。使用LipofectamineTM 2000,将300 ng psiCHECK-2-3′-UTR或psiCHECK-2-mut-3′-UTR分别与1 μL miR-M11-5p mimics、mut-miR-M11-5p mimics以及阴性对照negative control各自均匀混合于12 μL opti-MEM无血清培养基中,孵育5 min。按照LipofectamineTM 2000说明书将混合物与稀释液混合,室温下静置20 min。将转染复合物共转染至汇合度约为90%的48孔板HEK 293T细胞中,25 μL·孔-1,轻摇混匀。转染6 h后更换含有10% FBS的DMEM新鲜培养基。转染48 h后,收集细胞样品。用Promega公司Dual-Luciferase Reporter Assay System连续测定萤火虫和海肾荧光素酶活性。首先弃去培养基,用PBS清洗3次,向每孔中加入65 μL的细胞裂解液,振荡15 min,使细胞完全裂解。将细胞裂解物加入到不透光的白色酶标板里,加入100 μL LARⅡ混匀,用Ω酶标仪读取萤火虫荧光素酶活性值,立即再向每孔中加入100 μL Stop & Glo® Reagent混匀后,用luminescence conters检测海肾荧光素酶活性值。均做3个重复。最后统计分析海肾荧光素酶活性/萤火虫荧光素酶活性比值差异显著性。
1.7 RT-qPCR 1.7.1 总RNA的提取用Roche转染试剂X-treme GENE将miR-M11-5p、mut-miR-M11-5p及其阴性对照negative control转染至汇合度为80%~90%的6孔板CEF细胞中,于转染后24、48 h收集细胞样品。按照TRIzol RNA提取说明书提取总RNA,测定RNA的OD260 nm/OD280 nm在1.8~2.1范围内,于-20 ℃保存备用。
1.7.2 cDNA制备各取上述总RNA样品1 μg,按照Prime-ScriptTMRT reagent Kit with gDNA Eraser的操作说明书将RNA反转录成cDNA,反应体系为:5 × Primer Script RT Master mix 4 μL、RNA 1 μg、RNase-free H2O补足至20 μL,置于37 ℃水浴锅中保温15 min,进行cDNA合成反应,然后85 ℃水浴锅中热激5 s,测定cDNA浓度后,-20 ℃保存备用。
1.7.3 qPCR以CEF细胞基因组DNA为阴性对照,以鸡管家基因甘油醛-3-磷酸脱氢酶(glyceraldehyde phosphate dehydrogenase,GAPDH)基因作为内参基因,进行qPCR分析,每个样品均设置3个重复。qPCR反应体系:cDNA模板2 μL、qPCR上、下游引物各0.8 μL(表 1,#41~44)、SYBR Premix Ex Taq 10 μL、加ddH2O至20 μL。qPCR反应程序:95 ℃ 5 min预变性;95 ℃ 30 s,60 ℃ 34 s,40个循环。熔解曲线生成条件为7500 Fast荧光定量PCR仪,默认参数95 ℃ 15 s;60 ℃ 15 s,1.75 ℃·min-1升温至95 ℃维持15 s。
1.7.4 数据分析所有qPCR数据用2-△△Ct法进行计算。
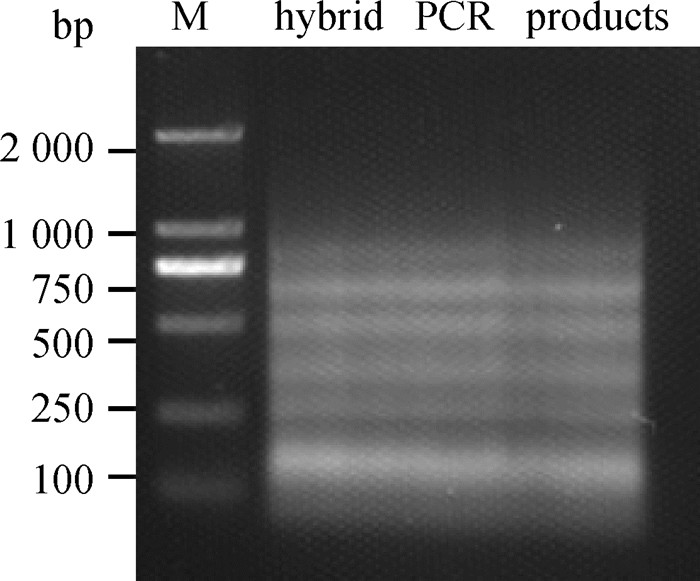
2 结果 2.1 miR-M11-5p候选靶基因cDNA文库的构建及鉴定利用3′-Full RACE和与miR-M11-5p(5′-UUUUCCUUACCGUGAGCUUAGA-3′)反向互补的特异性杂交引物3′-RRRGGRRTGGCRCRTCGRRTCT-5′(“R”为“G”或“A”),对CEF细胞cDNA进行hybrid-PCR扩增,经凝胶电泳分析显示扩增获得了不同长度的PCR产物(图 1)。回收纯化大小介于100~1 000 bp的PCR产物,连接至pMD19-T载体,转化E. coli JM109后用氨苄抗性LB固体培养基培养,挑取单菌落摇菌进行PCR及电泳鉴定,共获得373个可扩增出目的片段的阳性克隆,将其逐一进行测序得到有效基因序列280条。用BLAST软件在NCBI基因数据库中进行序列比对分析,共获得77个单一的基因序列(表 3),其中miR-M11-5p种子序列的结合位点位于3′-UTR、5′-UTR或CDS的候选靶基因各有37、5和35个。
|
M. DL2000 DNA相对分子质量标准 M. DL2000 DNA marker 图 1 hybrid-PCR产物凝胶电泳分析 Fig. 1 Electrophoresis analysis of hybrid-PCR products |
|
|
表 3 miR-M11-5p的候选宿主靶基因 Table 3 Candidate host mRNA targets for miR-M11-5p |
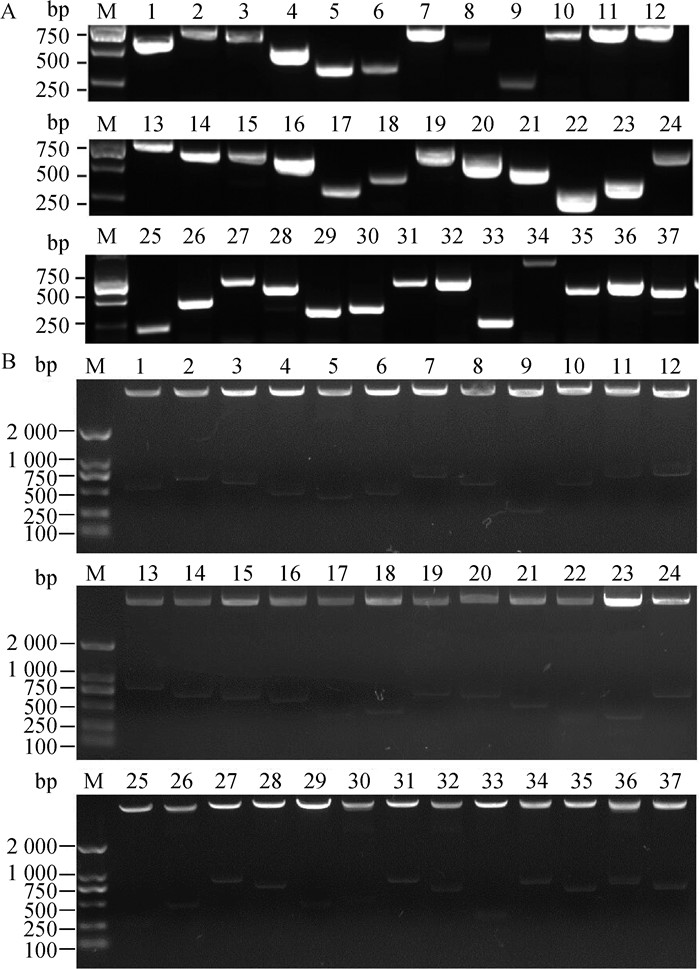
优先选择miRNA结合位点位于3′-UTR的候选靶基因进行双荧光素酶报告试验。利用表 1所列的特异性引物对(# 1~37)对miR-M11-5p的候选靶基因3′-UTR目的片段进行PCR扩增及凝胶电泳分析(图 2A),回收PCR产物连接至pMD19-T载体,经测序对比分析证实扩增的目的基因序列与预期完全一致。摇菌提取相应的pMD19-T-3′-UTR质粒,双酶切胶回收3′-UTR片段,电泳胶回收纯化后克隆至psiCHECK-2载体,经过酶切鉴定(图 2B)及测序分析,成功构建37个psi-CHECK2-3′-UTR质粒。
|
M. DL2000 DNA相对分子质量标准;1~37.候选靶基因3′-UTR编号(见表 1) DL2000 DNA marker; 1-37. 3′-UTRs of candidate target genes (see table 1) 图 2 候选靶基因3′-UTR的PCR扩增(A)及psiCHECK-2-3′-UTR质粒双酶切鉴定(B) Fig. 2 PCR amplification of the 3′-UTRs of candidate target genes (A) and restriction enzyme digestion of psiCHECK-2-3′-UTR plasmids(B) |
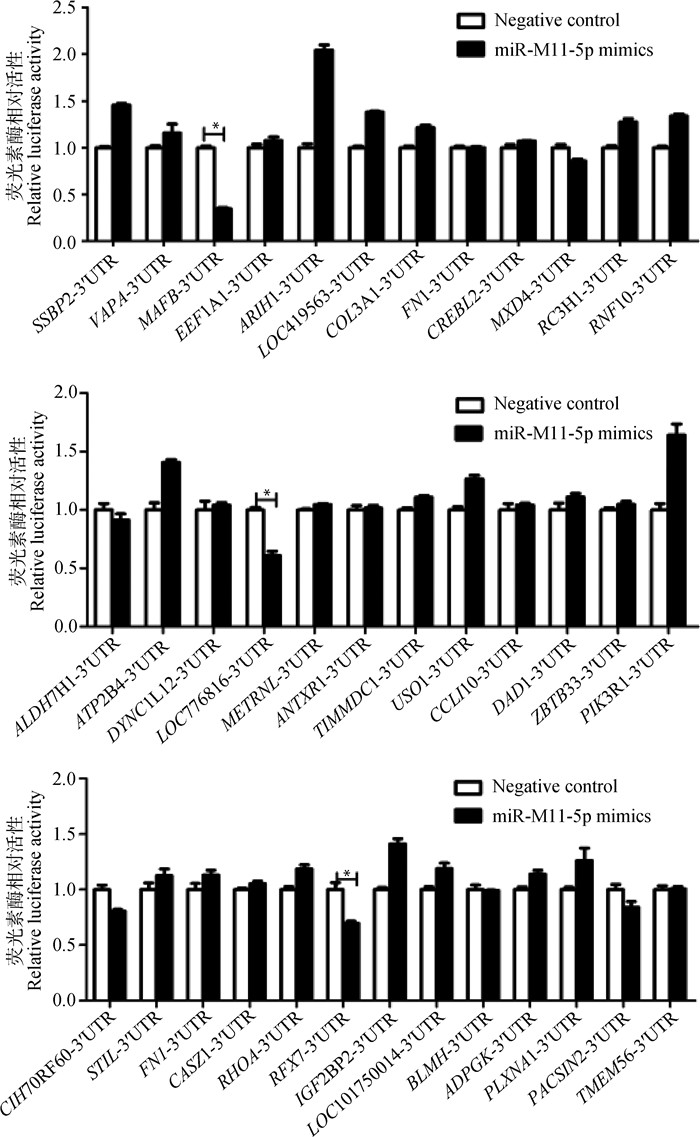
将37个不同候选靶基因的psi-CHECK2-3′-UTR质粒分别与miR-M11-5p mimics或阴性对照negative control共转染HEK 239T细胞,48 h后用Promega双荧光素酶报告系统分别测定转染细胞样品中萤火虫荧光素酶活性及海肾荧光素酶活性。通过计算海肾荧光素酶活性/萤火虫荧光素酶活性的比值来统计分析miR-M11-5p与候选靶基因3′-UTR的相互作用。与对照组相比,37个候选靶基因中三个候选靶基因MAFB(v-maf avian musculoaponeurotic fibrosarcoma oncogene homolog B,肌肉腱膜纤维肉瘤癌基因同源物B)、RFX7(regulatory factor X7,调节因子X7)和LOC776816的psiCHECK-2-3′-UTR质粒转染细胞中海肾荧光素酶活性/萤火虫荧光素酶活性比值均受到miR-M11-5p mimics的显著抑制,其余候选靶基因psiCHECK-2-3′-UTR质粒转染没有发生显著抑制(图 3)。
|
误差线代表三次独立的重复试验。*.差异性显著(P < 0.05) Error bars are derived from three independent replicates.*. significant difference (P < 0.05) 图 3 双荧光素酶报告试验初步分析miR-M11-5p与候选靶基因3′-UTR的相互作用 Fig. 3 Dual luciferase reporter assay for preliminary screening of candidate target genes for miR-M11-5p |
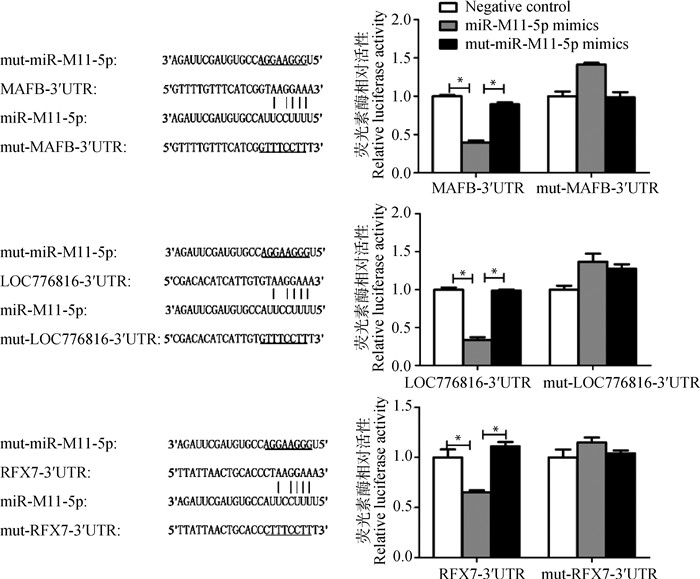
为了进一步证实miR-M11-5p与初筛候选靶基因MAFB、LOC776816和RFX7的特异性相互作用,将miR-M11-5p的2~7位种子序列进行突变,同时构建了相应的3′-UTR突变体质粒psiCHECK-2-mut-MAFB-3′-UTR、psiCHECK-2-mut-LOC776816-3′-UTR和psiCHECK-2-mut-RFX7-3′-UTR,然后共转染HEK 293T进行双荧光素酶报告试验。结果显示,突变后的miR-M11-5p失去抑制MAFB、LOC776816和RFX7这三个3′-UTR报告基因的活性,而3′- UTR突变后这些报告基因的活性也均不再被miR-M11-5p所抑制(图 4)。
|
误差线代表三次独立的重复试验;*.差异性显著(P < 0.05) Error bars are derived from three independent replicates; *. Significant difference (P < 0.05) 图 4 双荧光素酶报告试验对候选靶基因的突变验证 Fig. 4 Confirmation of the interactions between miR-M11-5p and candidate targets by dual luciferase reporter assay |
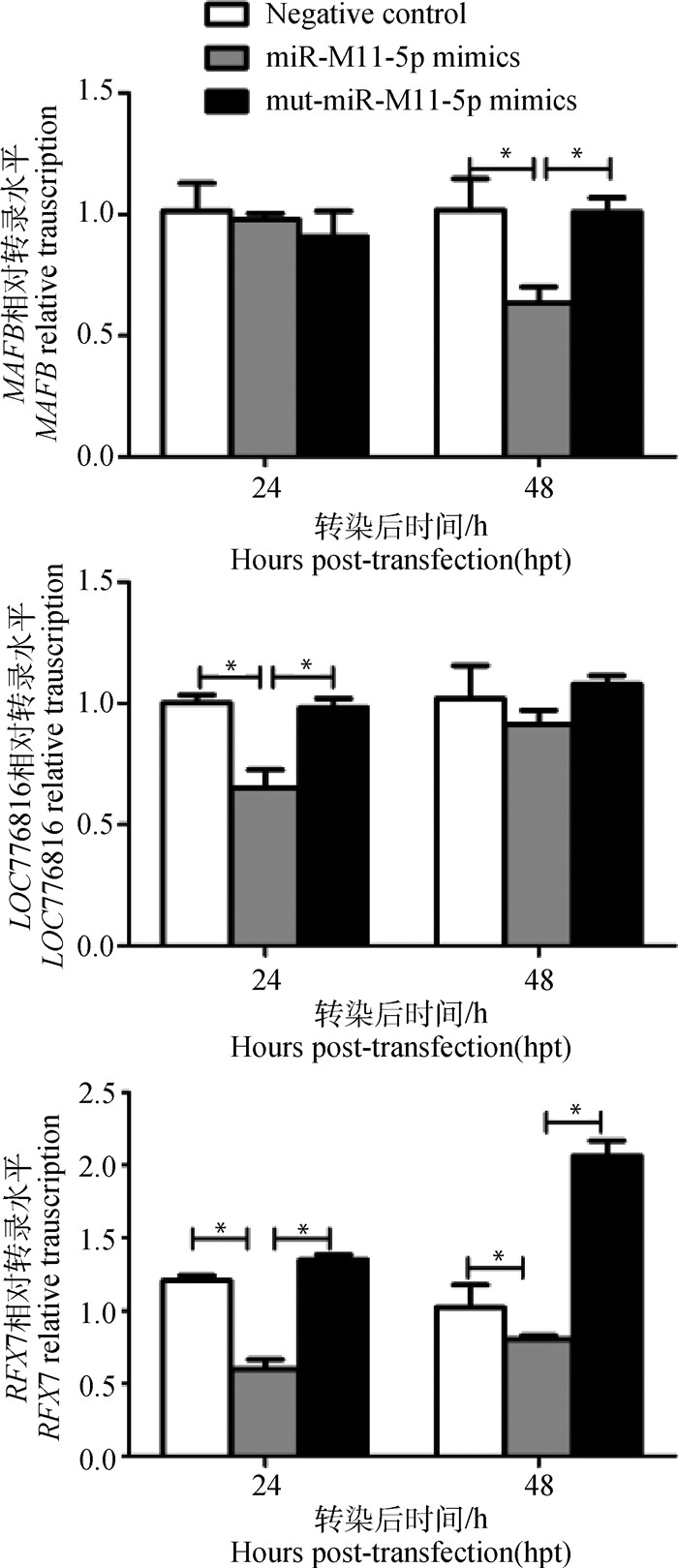
为了最终证实miR-M11-5p与MAFB-3′-UTR、LOC776816-3′-UTR以及RFX7-3′-UTR的体内相互作用,分别将miR-M11-5p mimics、mut-miR-M11-5p mimics或阴性对照negative control(表 2)转染至CEF细胞中,通过RT-qPCR检测MAFB、LOC776816和RFX7在转染后24和48 h的mRNA水平。结果(图 5)显示,在过表达24或48 h后miR-M11-5p均显著下调这3个候选靶基因的mRNA水平,证实MAFB、LOC776816和RFX7是miR-M11-5p的宿主靶基因。
|
误差线代表三次独立的重复试验;*.差异性显著(P < 0.05) Error bars are derived from three independent replicates.*. Significant difference (P < 0.05) 图 5 RT-qPCR分析mdv1-miR-M11-5p过表达CEF细胞中的靶基因mRNA水平 Fig. 5 Expression levels of target mRNA genes in miR-M11-5p over-expressed CEFs analyzed by RT-qPCR |
miRNA介导的基因表达调控依赖于种子序列与靶标的完全或近似完全的碱基配对,尤其是其5′端2~7位核苷酸(“种子序列”)是决定靶标特异性的重要因素[12-13]。病毒miRNA与靶mRNA的结合通常是不完全匹配的,所以宿主靶基因的发现鉴定非常困难。之前筛选鉴定miRNA的宿主靶基因多采用生物信息学软件预测(如TargetScan、RNAhybrid)和试验筛选(如HITS-CHIP、微阵列技术)的方法,这些方法主要依赖于miRNA/mRNA双链的匹配性和热力学稳定性以及miRNA在不同物种中的保守性。虽然这些方法在miRNA筛选靶基因中是可行的,但容易出现较高的假阳性结果,整个筛选过程工作量巨大。因此,筛选和鉴定miRNA的靶基因仍急需要一种快速、简便、高效、精准的方法。直接从mRNA逆转录的cDNA文库中捕获已知miRNA的靶标,是筛选鉴定靶基因简单且有效的方法[11]。本研究中笔者使用杂交PCR构建候选靶基因cDNA文库的方法,快速筛选获得了一批接近自然生物过程的miR-M11-5p候选宿主靶基因,进一步证实杂交PCR是筛选病毒miRNA靶标的有效方法,具有操作简单,成本低廉和高效准确的优点。
采用hybrid-PCR的方法,笔者建立了miR-M11-5p的宿主靶基因cDNA文库。在获得的77个宿主候选靶基因序列中,有37个miRNA结合位点位于mRNA基因的3′-UTR中。由于miRNA发挥调控作用主要是与mRNA的3′-UTR区域进行结合,因此,我们优先挑选这37个候选靶基因进行下一步验证试验。首先构建37个候选靶基因3′-UTR双荧光素酶报告质粒,通过双荧光素酶报告试验对miRNA与候选靶基因3′-UTR的体外相互作用进行分析验证,然后在CEF细胞中过表达miR-M11-5p mimics,利用RT-qPCR对候选靶基因的体内基因表达水平进行分析,最终鉴定MAFB、LOC776816和RFX7是miR-M11-5p的宿主靶基因。之前分析验证miRNA与靶mRNA的相互作用,主要采用miRNA过表达质粒转染细胞进行双荧光素酶报告试验及RT-qPCR分析。转染质粒虽然是一种相对简单的基因表达方式,但仍需构建质粒,并且质粒转染入核效率较低。在本研究中,笔者使用人工合成miRNA mimics替代miRNA过表达质粒进行转染用于后续相关试验分析,相关研究结果表明miRNA mimics易制备、转染效率高,可作为验证分析病毒miRNA靶基因的有效工具。
在已鉴定的3个宿主靶基因中,LOC776816编码蛋白目前尚无基因注释和相关功能报道。MAFB为MAF bZIP转录因子B,该基因编码的蛋白质是碱性亮氨酸拉链(bZIP)转录因子[14]。有研究表明,MAFB在各种细胞、组织和器官的发育和分化中起重要作用,包括破骨细胞生成[15],巨噬细胞分化[16],胰腺发育[17]和后脑中MAFB节段表达[18]。此外,也有研究表明,MAFB基因在携带染色体重排的人类骨髓瘤细胞中异常表达[19];在急性髓性白血病母细胞中观察到MAFB表达升高[20]。研究还发现miR-223通过靶向MAFB mRNA抑制鼻咽癌细胞的增殖和迁移[21]。这些数据表明MAFB在肿瘤发生发展过程中可能具有重要促癌作用,miR-M11-5p靶向并抑制MAFB的表达,也间接表明其可能在MD肿瘤发生过程中发挥重要抑癌作用。RFX7是调节因子X(RFX)转录因子家族的成员。有研究表明,转录因子RFX7可限制自然杀伤(NK)细胞的代谢并促进其维持和免疫,RFX7的缺失可导致NK细胞维持和体内免疫力的降低[22]。miR-M11-5p靶向并抑制RFX7基因表达,可能在MD肿瘤发生过程中细胞吞噬、免疫力发挥重要的负调控作用。综上所述,本研究的数据进一步佐证了笔者之前对miR-M11-5p是MD肿瘤抑制因子的推测,但其抑癌的分子机制仍有待进一步的深入研究。
4 结论通过hybrid-PCR构建了miR-M11-5p的候选靶基因cDNA文库。通过基因克隆和测序分析,从文库中初步获得77个候选宿主靶基因,其中37个miRNA结合位点位于mRNA基因3′-UTR中。通过双荧光素酶报告试验、miRNA过表达及RT-qPCR分析,最终鉴定MAFB、RFX7和LOC776816为miR-M11-5p的宿主靶基因。本研究为后续揭示miR-M11-5p抑癌的分子调控机制奠定了重要基础。
| [1] | DAVISON A J, EBERLE R, EHLERS B, et al. The order Herpesvirales[J]. Arch Virol, 2009, 154(1): 171–177. |
| [2] | WITTER R L, SCHAT K A. Diseases of poultry[M]. Ames: Iowa State University Press, 2003: 407-464. |
| [3] | OSTERRIEDER N, KAMIL J P, SCHUMACHER D, et al. Marek's disease virus:from miasma to model[J]. Nat Rev Microbiol, 2006, 4(4): 283–294. |
| [4] | BARTEL D P. MicroRNAs: genomics, biogenesis, mechanism, and function[J]. Cell, 2004, 116(2): 281–297. |
| [5] | FILIPOWICZ W, BHATTACHARYYA S N, SONENBERG N. Mechanisms of post-transcriptional regulation by microRNAs: are the answers in sight?[J]. Nat Rev Genet, 2008, 9(2): 102–114. DOI: 10.1038/nrg2290 |
| [6] | LEE Y S, DUTTA A. MicroRNAs in cancer[J]. Annu Rev Pathol, 2009, 4: 199–227. DOI: 10.1146/annurev.pathol.4.110807.092222 |
| [7] | GRIFFITHS-JONES S. The microRNA registry[J]. Nucleic Acids Res, 2004, 32(Database issue): D109–D111. |
| [8] |
罗俊, 滕蔓, 樊剑鸣, 等. 马立克氏病病毒编码的microRNA:从基因组学到功能研究[J]. 中国科学:生命科学, 2010, 40(6): 476–483.
LUO J, TENG M, FAN J M, et al. Marek's disease virus-encoded microRNAs:genomics, expression and function[J]. Scientia Sinica (Vitae), 2010, 40(6): 476–483. (in Chinese) |
| [9] |
赵朴.马立克氏病病毒miRNA的体内动态表达谱及对病毒基因的自我调控[D].杨凌: 西北农林科技大学, 2016.
ZHAO P. In vivo dynamic expression profiles of MDV miRNAs and their self-regulatory founctions on the viral protein-coding genes[D]. Yangling: Northwest A&F University, 2016. (in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10712-1016163801.htm |
| [10] |
滕蔓. Mid基因簇miRNAs在马立克病病毒致病中的潜在功能研究[D].兰州: 甘肃农业大学, 2017.
TENG M. Potential roles of the Mid-clustered microRNAs in the pathogenesis of Marek's disease virus[D]. Lanzhou: Gansu Agricultural University, 2017. (in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10733-1017824815.htm |
| [11] | HUANG Y J, QI Y, RUAN Q, et al. A rapid method to screen putative mRNA targets of any known microRNA[J]. Virol J, 2011, 8: 8. DOI: 10.1186/1743-422X-8-8 |
| [12] | BARTEL D P. MicroRNAs:target recognition and regulatory functions[J]. Cell, 2009, 136(2): 215–233. DOI: 10.1016/j.cell.2009.01.002 |
| [13] | STARK A, BRENNECKE J, RUSSELL R B, et al. Identification of drosophila microRNA targets[J]. PLoS Biol, 2003, 1(3): e60. DOI: 10.1371/journal.pbio.0000060 |
| [14] | TSUCHIYA M, MISAKA R, NITTA K, et al. Transcriptional factors, Mafs and their biological roles[J]. World J Diabetes, 2015, 6(1): 175–183. |
| [15] | SMINK J J, BÉGAY V, SCHOENMAKER T, et al. Transcription factor C/EBPβ isoform ratio regulates osteoclastogenesis through MafB[J]. EMBO J, 2009, 28(12): 1769–1781. DOI: 10.1038/emboj.2009.127 |
| [16] | KELLY L M, ENGLMEIER U, LAFON I, et al. MafB is an inducer of monocytic differentiation[J]. EMBO J, 2000, 19(9): 1987–1997. DOI: 10.1093/emboj/19.9.1987 |
| [17] | HANG Y, STEIN R. MafA and MafB activity in pancreatic β cells[J]. Trends Endocrinol Metab, 2011, 22(9): 364–373. DOI: 10.1016/j.tem.2011.05.003 |
| [18] | GRAPIN-BOTTON A, BONNIN M A, SIEWEKE M, et al. Defined concentrations of a posteriorizing signal are critical for MafB/Kreisler segmental expression in the hindbrain[J]. Development, 1998, 125(7): 1173–1181. |
| [19] | HANAMURA I, ⅡDA S, AKANO Y, et al. Ectopic expression of MAFB gene in human myeloma cells carrying (14;20)(q32;q11) chromosomal translocations[J]. Jpn J Cancer Res, 2001, 92(6): 638–644. DOI: 10.1111/cas.2001.92.issue-6 |
| [20] | YANG L, LIU Y N, ZHU L, et al. DNMT3A R882 mutation is associated with elevated expression of MAFB and M4/M5 immunophenotype of acute myeloid leukemia blasts[J]. Leuk Lymphoma, 2015, 56: 2914–2922. DOI: 10.3109/10428194.2015.1015123 |
| [21] | YANG W Y, LAN X, LI D M, et al. MiR-223 targeting MAFB suppresses proliferation and migration of nasopharyngeal carcinoma cells[J]. BMC Cancer, 2015, 15: 461. DOI: 10.1186/s12885-015-1464-x |
| [22] | CASTRO W, CHELBI S T, NIOGRET C, et al. The transcription factor Rfx7 limits metabolism of NK cells and promotes their maintenance and immunity[J]. Nat Immunol, 2018, 19(8): 809–820. |



