近年来,中国集约化牛场高产奶牛卵巢静止性乏情率较高,严重影响奶牛繁殖性能,降低了奶牛生产性能和经济价值。奶牛产后发生卵巢静止是由于产后发情周期出现异常,而导致奶牛卵巢上无卵泡发育,或出现卵泡却不能发育到优势卵泡,以至于奶牛在产后不能及时怀孕而延长产犊间隔,最终导致奶牛淘汰。
影响奶牛卵巢静止发生的主要因素为奶牛机体出现能量负平衡状态,营养可以直接通过日粮营养素或代谢中间体影响下丘脑-垂体-卵巢轴,进而影响卵泡发育[1]。直接影响卵泡的代谢物和营养物质有很多。研究较多的是卵泡内介质对卵泡生长和营养的影响,包括瘦素、葡萄糖-胰岛素、生长激素(growth hormone,GH)和IGF。但蛋白质组学对产后奶牛卵巢静止研究尚不明确。
蛋白质组学的出现,为当代生物科学提供了新的研究方向。组学和生物信息学的结合,构成了现代系统生物学的基础,并广泛应用于模式生物(包括啮齿类、酵母、人和家畜等)的研究领域。首先认识到基因组学和蛋白质组学研究能够成为系统生物学基础[2]。iTRAQ试剂拓展了研究蛋白质差异表达的动态能力,它能提供多种样本中蛋白的定量信息,包括时间过程分析及疾病标记的发现和诊断。蛋白质组学技术在奶牛业应用广泛,Min等[3]应用iTRAQ技术研究奶牛热应激,Dai等[4]研究基础饲料加喂高饲料日粮调节乳汁合成的机制,Zhang等[5]利用iTRAQ技术筛选患子宫内膜炎的奶牛子宫内膜与血浆的差异蛋白。因此,针对高产奶牛产后卵巢静止所致的乏情,本试验应用iTRAQ技术对卵巢静止奶牛进行了蛋白质组学、生物信息学研究,为今后深入探究奶牛产后乏情机制奠定基础。
1 材料与方法 1.1 试验动物与分组本试验选取黑龙江某集约化牛场,奶牛饲喂全混合日粮(TMR)组成:精料8~9 kg、青贮17~20 kg、干草3.5~4.0 kg、脂肪300~400 g。营养水平:DM 55.60%、粗蛋白16%、产奶净能7.322 MJ·kg-1、脂肪5.60%、NDF 39.10%、ADF 20.30%、钙180 g、磷116 g。
跟踪两组奶牛至产后60~90 d,在产后60 d开始观察外部表现,实施直肠检查或B超检查监测卵泡发育情况,并根据文献[6]奶牛产后卵巢静止判定标准,最终选择正常发情奶牛(CON)和卵巢静止奶牛(IO)各50头,两组试验奶牛基本信息见表 1。
|
|
表 1 两组试验奶牛产后基本信息 Table 1 The basic information of test cows in 2 groups |
早上7:00奶牛饲喂前经尾静脉采血。每头奶牛收集全血10 mL,3 000 r·min-1离心10 min,收集血清置于1.5 mL离心管中,每管600 μL,12 000 r·min-1离心10 min,收集上清液置于1.5 mL离心管中。
1.2.2 样品制备在血清样本中加入裂解缓冲液。置样品于超声波60 s,频率为0.2 s,振幅为22%。在室温下提取30 min,15 000 r·min-1,4 ℃离心20 min,收集上清液,即为蛋白质。采用Bradford法测定提取的蛋白质浓度[7]。本试验应用人血清/血浆高丰度蛋白试剂盒(美国MERCK公司)进行样本去高丰度蛋白试验,为降低奶牛个体差异的影响,试验选择混合样本的方法进行iTRAQ标记的蛋白质组学试验。每组随机选择15头奶牛进行蛋白质组学试验,其余35头用于验证试验,组内每5头奶牛进行混合成为1个混合样本,每组有3个混合样本(IO组:IO1、IO2、IO3;CON组:CON1、CON2、CON3)。CON组和IO组混合样品的蛋白平均浓度接近,分别为(3.21±0.16)g·L-1和(3.15±0.15)g·L-1,差异不显著(P>0.05)。取出200 μg蛋白溶液,按照iTRAQ试剂盒说明书操作进行蛋白酶解。
1.2.3 酶解肽段离线预分离及LC-MS/MS质谱分析(1) 高pH条件下的反相色谱分离。(2)纳升级反相色谱-Q Exactive进行蛋白质分析。采用Uniprot数据库(http://www.uniprot.org)。iTRAQ的质谱分析是由Thermo Q-Exactive型质谱完成,产生的质谱原始文件采用配套商用软件Proteome Discoverer1.4(Thermo公司)处理和筛选差异表达蛋白质。使用平均差异倍数计算并筛选差异表达蛋白质。
1.2.4 基于生物信息学技术的差异表达蛋白质功能分析根据DAVID在线数据软件对试验数据进行GO分析(gene ontology),使用在线软件STRING进行蛋白质网络互作分析,应用DAVID在线数据软件进行Pathway分析。
1.2.5 差异表达蛋白质的免疫印迹分析按照常规方法[8]选择4种差异蛋白进行Western blot验证,差异蛋白包括果糖2磷酸醛缩酶(fructose-bisphosphate aldolase,ALDOB)、胰岛素样生长因子结合蛋白2(insulin like growth factor binding peotein,IGFBP-2)、α-胰蛋白酶抑制剂重链H3(inter-alpha-trypsin inhibitor heavy chain H3,ITIH3)以及乳酸脱氢酶(L-lactate dehydrogenase,LDHB)。
1.2.6 差异表达蛋白质的ELISA分析应用ELISA试剂盒(南京金益柏公司)对样本中15个未处理血清样本进行差异蛋白的定量检测。所检测的差异蛋白为α-2-HS糖蛋白(AHSG)、载脂蛋白A4(APOA4)、果糖二磷酸醛缩酶(ALDOB)、谷胱甘肽过氧化物酶(GPX3)、透明质酸酶(SPAM1)、丙酮酸激酶(PKM2)和视黄醇结合蛋白4(RBP4)。
1.2.7 数据分析根据凝胶的分布及其灰度情况计算灰度值。应用SPSS软件(IBM,V20.0)中独立样本t检验的统计学方法,对Western blot的灰度值进行差异性分析及组间差异性比较。P < 0.05表示差异显著;P < 0.01表示差异极显著。
2 结果 2.1 样品去除高丰度蛋白随机选取两组中去除高丰度蛋白后的样品进行聚丙烯酰胺凝胶电泳检测(SDS-PAGE),以观察去除高丰度蛋白的效果(图 1)。图 1结果表明,白蛋白和IgG高丰度蛋白条带的去除效果明显。
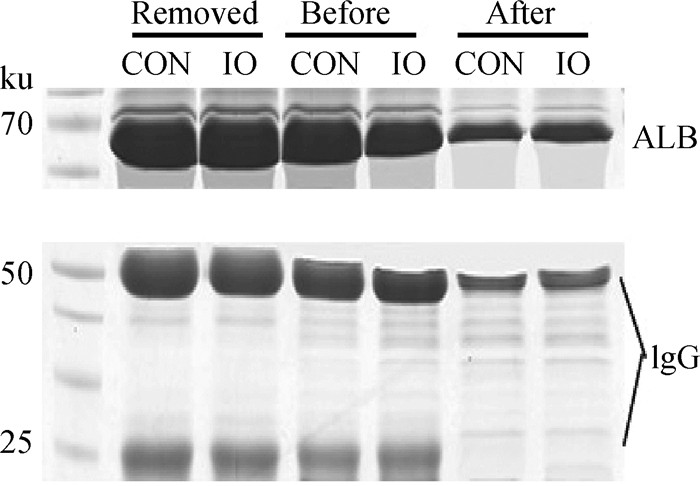
|
Removed.样品中去除掉的高丰度蛋白; Before.未处理样品; After.处理后样品; ALB.血清白蛋白 Removed. The high abundance proteins removed from the sample; Before.Untreated sample; After.Treated sample; ALB. Serum albumin 图 1 血液样本去除高丰度蛋白前、后以及去除掉的高丰度蛋白 Fig. 1 The high abundance proteins were removed before, after and the removed high abundance |
在本试验中,卵巢静止组表达上调的蛋白平均倍数变化>1,表达下调的蛋白平均倍数变化 < 1。选取平均倍数变化≥1.2,平均倍数变化≤0.8,且P < 0.05进行差异蛋白的筛选,共获得61种差异表达蛋白质(表 2)。
|
|
表 2 在两组奶牛间的差异表达蛋白质 Table 2 The differentially expressed proteins between dairy cows in two groups |
差异表达蛋白质GO富集分析表明,生物学过程包括34个注释(图 2),分子功能包括15个注释(图 3),细胞组成包括13个注释(图 4)。
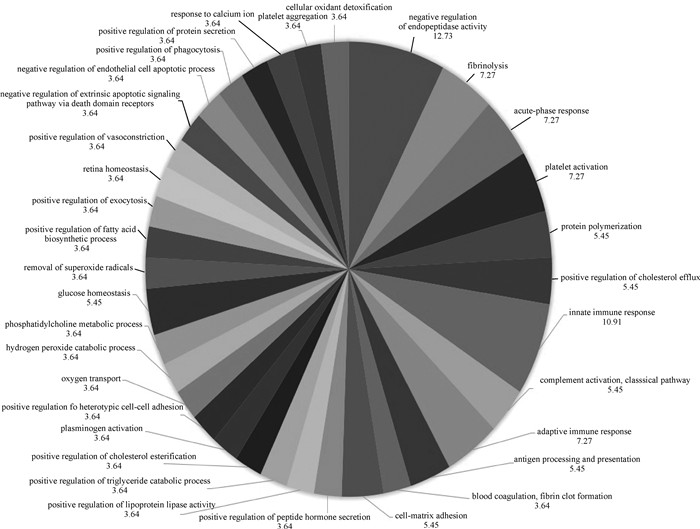
|
图 2 生物学过程注释比重 Fig. 2 The proportion of annotation from biological process analysis |

|
图 3 分子功能注释比重 Fig. 3 The proportion of annotation from molecular functional analysis |
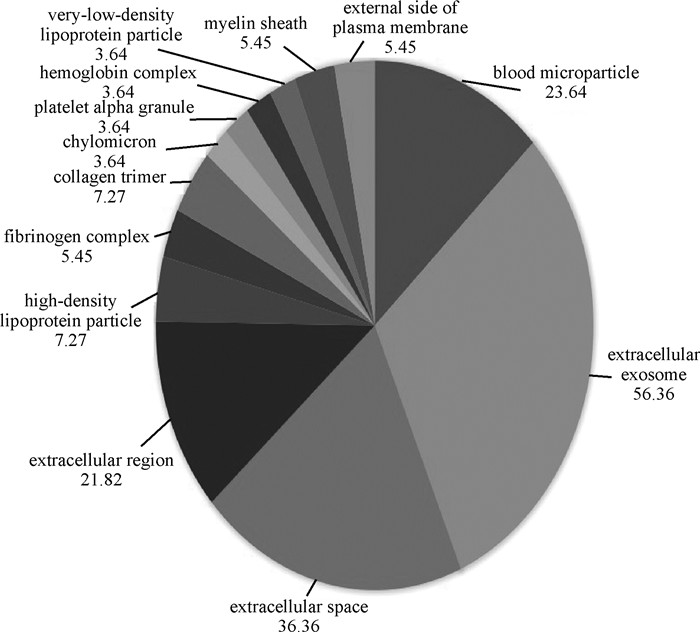
|
图 4 细胞组成注释比重 Fig. 4 The proportion of accession from cellular component analysis |
61种差异表达蛋白质进行蛋白质网络互作的分析结果如图 5所示,其中3种蛋白质未在数据库中获得匹配,分别是PEI、SPM和LW012。结果共得到57个节点,68条蛋白互作注释,蛋白质互作富集P < 10-16。在相互关联的中心是HP与PRDX2间的互作。
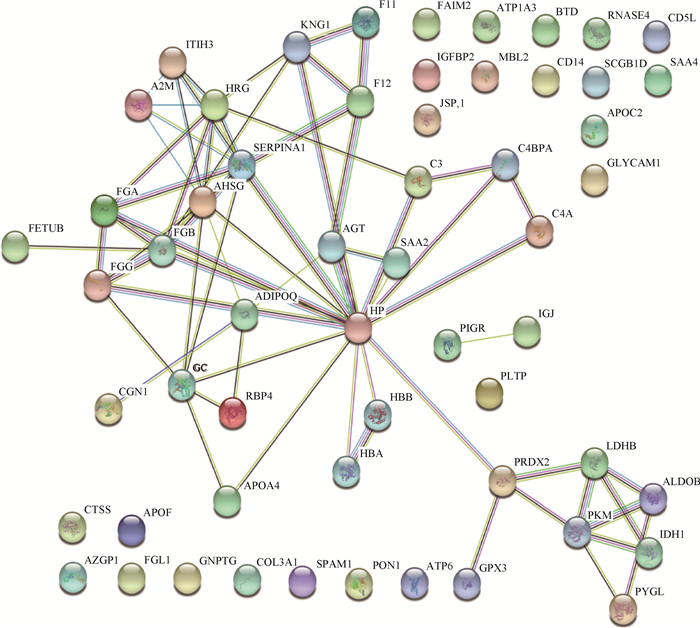
|
图 5 差异表达蛋白质的网络互作分析图 Fig. 5 The network interaction diagram of differentially expressed proteins |
信号通路分析结果见表 3,差异表达蛋白共涉及11个信号代谢通路。经过筛选发现糖酵解/糖异生、氨基酸的生物合成、胰高血糖素信号通路、维生素的消化与吸收可能与卵巢静止的发生存在相关性。
|
|
表 3 差异表达蛋白的信号通路分析 Table 3 Signal pathway analysis of differentially expressed proteins |
对61种差异蛋白生物信息学分析(包括GO、蛋白互作和Pathway),以及对差异表达蛋白相关文献和资料的查询,共筛选出14种差异表达蛋白质与卵巢静止发生存在关系,结果见图 6。

|
图 6 与奶牛卵巢静止发生存在关系的14种差异表达蛋白质的网络互作分析图 Fig. 6 The network interaction diagram of 14 differentially expressed proteins associated with inactive ovaries of cows |
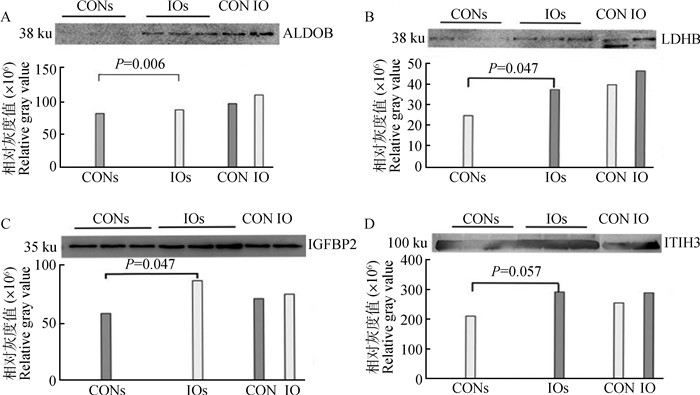
选择ALDOB、IGFBP2、ITIH3和LDHB 4种差异蛋白进行Western blot和灰度值分析,结果见图 7。图 7结果表明,4种蛋白在混合样本和单一样本中的相对表达量均与蛋白组学结果一致。
|
A. ALDOB;B.LDHB;C.IGFBP2;D.ITIH3。CONs.健康对照组混合样品;IOs.卵巢静止组混合样品;CON.健康对照组未混合单一样品; IO.卵巢静止组未混合单一样品 A.ALDOB; B.LDHB; C.IGFBP2;D.ITIH3.CONs.Mixed sample of the healthy control group; IOs.Mixed sample of the ovarian resting group; CON.Single sample in the healthy control group; IO.Single sample in the ovarian resting group 图 7 Western blot检测及灰度值分析 Fig. 7 Western blot detection and gray value analysis |
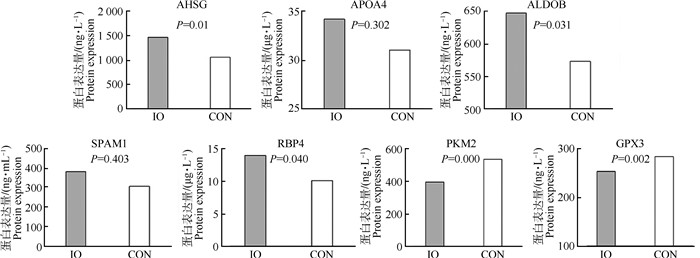
为了验证蛋白组学的结果,选择7种差异表达蛋白质应用ELISA方法进行验证,结果见图 8。有5种蛋白在两组间存在统计学差异,其中PKM2和GPX3在卵巢静止组中表达极显著性下调(P < 0.01);AHSG在卵巢静止组中表达极显著性上调(P≤0.01)、ALDOB和RBP4在卵巢静止组中表达显著性上调(P < 0.05)。另外两种蛋白APOA4和SPAM1在两组之间差异不显著(P>0.05),但在卵巢静止组中表达均上调。
|
图 8 ELISA方法检测蛋白表达量 Fig. 8 Proteins expression were detected with ELISA |
本试验应用组学技术获得了奶牛卵巢静止血浆差异蛋白,通过生物信息学分析挖掘它们与卵巢静止的关系。根据Western blot条带灰度值分析可知,4种蛋白在混合样本和单一样本中的相对表达量均与蛋白组学结果一致。其中ALDOB的灰度值在卵巢静止组中的混合样本和单一样本中表达极显著上调(P < 0.01),LDHB和IGFBP2的灰度值在卵巢静止组中的混合样本和单一样本中表达显著上调(P < 0.05),ITIH3的灰度值在两组混合样本之间虽然未表现统计学显著差异(P>0.05),但结果仍符合蛋白质组学结果表达趋势,在卵巢静止组中表达上调。这些表明蛋白质组学筛选差异表达蛋白与Western blot验证结果符合性高。
脂联素(ADIPOQ)主要是由白色脂肪组织产生的蛋白质,研究表明ADIPOQ可以调节卵巢功能,并在妊娠早期对胚胎产生影响[9]。在奶牛中,ADIPOQ及其受体存在于不同的细胞中,包括卵母细胞、卵泡膜、颗粒细胞、卵丘细胞和黄体细胞[10]。并且,奶牛卵巢生理状态与来自卵巢中卵泡细胞和黄体细胞中ADIPOQ及其受体的表达模式相关[11]。事实上,在卵泡期和黄体期,脂连蛋白、ADIPOQ受体1和ADIPOQ受体2的表达在优势卵泡中的颗粒细胞、卵丘细胞是高于闭锁卵泡的[12]。ADIPOQ还可能通过ERK1/2MAPK途径来减少胰岛素对类固醇生成的诱导,但它仅在牛卵泡膜细胞中对类固醇生成起抑制作用[13]。在本试验中应用蛋白组学技术发现卵巢静止奶牛中ADIPOQ的表达上调。这提示它可能通过调节胰岛素和IGF-1对卵泡发育发挥间接的影响,但有待于进一步研究证实。
谷胱甘肽过氧化物酶属于一个系统发生相关的酶家族,哺乳动物GPX1-4的催化中心是硒代半胱氨酸。谷胱甘肽是GPX3的底物,GPX3是保护细胞免受脂质过氧化氢和H2O2的主要抗氧化物酶[14],并催化游离氢过氧化物和其他氢过氧化物的还原[15]。过度的氧化应激可诱发卵泡细胞凋亡,并诱发闭锁[16]。而GPX3可以防止细胞凋亡氧化应激,并促进卵泡生长。在本试验中GPX3在卵巢静止奶牛血液中表达下调,提示卵巢静止发生时机体内氧化应激可能使卵泡细胞扩增失败,会导致未能形成促进卵母细胞生成的卵丘结构。
胰岛素样生长因子(IGF)系统由几个成员组成,包括IGF-1和IGF-2、两种受体,以及结合蛋白(IGFBP2、IGFBP2、IGFBP3、IGFBP4、IGFBP5和IGFBP6等)。IGFBPs存在于生物体液中,并通过抑制或增强靶细胞中两种IGF(IGF1和IGF2)的作用而发挥功能[17]。IGFBPs对IGF-1的生物利用度的调节,对于牛卵母细胞和卵泡的体外培养是必需的[18]。牛卵泡颗粒细胞和卵泡膜细胞中IGF1受体的表达在卵泡发育的最后阶段增加,在闭锁发生时减少[19]。尽管IGF1和胰岛素在卵泡发育后期发挥关键作用,然而本试验中奶牛卵巢静止发生时IGFBP2表达上调,且卵泡在早期就已经停止发育。
视黄醇结合蛋白4(RBP4)也称维生素A结合蛋白4。类维生素A的运输和代谢是由特异性结合蛋白介导和调节的[20],维生素A有局部调节卵泡发育的潜在作用,包括卵母细胞成熟。视黄酸是视黄醇的活化形式,也是其氧化形式[21]。视黄酸能够降低在颗粒细胞中FSH诱导表达的FSH受体和LH受体。由于LH受体和FSH受体的获得是卵泡发育、卵母细胞成熟、排卵和黄体化的关键。因此,维生素A及其衍生物的增加会影响卵泡的发育。本试验中卵巢静止奶牛血液中RBP4升高,但其在卵巢静止发生中的作用机制有待进一步探究。
4 结论本研究首次应用基于iTRAQ与LC-MS/MS联用技术,结合生物信息学、免疫印迹学、ELISA的方法,获得了卵巢静止奶牛61种血清差异表达蛋白质,其中与卵巢静止密切相关的14种蛋白质,其中GPX3、SCGB1D和PKM2表达下调,ADIPOQ、AHSG、APOA4、FETUB、ALDOB、SPAM1、LDHB、RBP4、IGFBP2、ITIH3及GLYCAM1表达上调。在主要的4种差异表达蛋白中,ADIPOQ、IGFBP2和RBP4可能是通过影响生殖激素生物过程影响卵泡发育,而GPX3会通过氧化应激而影响卵泡发育。
| [1] | SCARAMUZZI R J, CAMPBELL B K, DOWNING J A, et al. A review of the effects of supplementary nutrition in the ewe on the concentrations of reproductive and metabolic hormones and the mechanisms that regulate folliculogenesis and ovulation rate[J]. Reprod Nutr Dev, 2006, 46(4): 339. DOI: 10.1051/rnd:2006016 |
| [2] | IEKER T, GALITSKI T, HOOD L. A new approach to decoding life: systems biology[J]. Annu Rev Genomics Hum Genet, 2001, 2(1): 343. DOI: 10.1146/annurev.genom.2.1.343 |
| [3] | MIN L, CHENG J, ZHAO S, et al. Plasma-based proteomics reveals immune response, complement and coagulation cascades pathway shifts in heat-stressed lactating dairy cows[J]. J Proteomics, 2016: 14699–14708. |
| [4] | DAI W, CHEN Q, WANG Q, et al. Complementary transcriptomic and proteomic analyses reveal regulatory mechanisms of milk protein production in dairy cows consuming different forages[J]. Sci Rep, 2017, 14(7): 44234. |
| [5] | ZHANG S D, DONG S W, WANG D S, et al. Differential proteomic profiling of endometrium and plasma indicate the importance of hydrolysis in bovine endometritis[J]. J Dairy Sci, 2017, 100(1): 9324–9337. |
| [6] | XU C C, XIA C, SUN Y H, et al. Metabolic profiles using 1H-nuclear magnetic resonance spectroscopy in postpartum dairy cows with ovarian inactivity[J]. Theriogenology, 2016, 86(6): 1475–1481. DOI: 10.1016/j.theriogenology.2016.05.005 |
| [7] | BRADFORD M M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding[J]. Anal Biochem, 1976, 72(1-2): 248–254. DOI: 10.1016/0003-2697(76)90527-3 |
| [8] | KENNEDY R, ANDERSON G.Microscale Western blot: WO, US9182371[P]. 2015. |
| [9] | PALIN M F, BORDIGNON V V, MURPHY B D. Adiponectin and the control of female reproductive functions[J]. Vitam Horm, 2012, 90(2): 239–287. |
| [10] | MAILLARD V, UZBEKOVA S, GUIGNOT F, et al. Effect of adiponectin on bovine granulosa cell steroidogenesis, oocyte maturation and embryo development[J]. Reprod Biol Endocrin, 2010, 8(1): 23. |
| [11] | TABANDEH M R, HOSSEINI A, SAEB M, et al. Changes in the gene expression of adiponectin and adiponectin receptors (AdipoR1 and AdipoR2) in ovarian follicular cells of dairy cow at different stages of development[J]. Theriogenology, 2010, 73(5): 659–669. DOI: 10.1016/j.theriogenology.2009.11.006 |
| [12] | TABANDEH M R, GOLESTANI N, KAFI M, et al. Gene expression pattern of adiponectin and adiponectin receptors in dominant and atretic follicles and oocytes screened based on brilliant cresyl blue staining[J]. Anim Reprod Sci, 2012, 131(1-2): 30–40. DOI: 10.1016/j.anireprosci.2012.02.006 |
| [13] | LAGALY D V, AAD P Y, GRADO-AHUIR J A, et al. Role of adiponectin in regulating ovarian theca and granulosa cell function[J]. Mol Cell Endocrinol, 2008, 284(2): 38–45. |
| [14] | CHAISWING L, OBERLEY T D. Extracellular/microenvironmental redox state[J]. Antioxid Redox Signal, 2010, 13(4): 449–465. DOI: 10.1089/ars.2009.3020 |
| [15] | YU B P. Cellular defenses against damage from reactive oxygen species[J]. Physiol Rev, 1994, 74(1): 139–162. DOI: 10.1152/physrev.1994.74.1.139 |
| [16] | TILLY J L, TILLY K I. Inhibitors of oxidative stress mimic the ability of follicle-stimulating hormone to suppress apoptosis in cultured rat ovarian follicles[J]. Endocrinology, 1995, 136(1): 242–252. DOI: 10.1210/endo.136.1.7828537 |
| [17] | MONGET P, FABRE S, MULSANT P, et al. Regulation of ovarian folliculogenesis by IGF and BMP system in domestic animals[J]. Domest Anim Endocrinol, 2002, 23(1-2): 139–154. DOI: 10.1016/S0739-7240(02)00152-2 |
| [18] | THOMAS F H, CAMPBELL l B K, ARMSTRONG D G, et al. Effects of IGF-Ⅰ bioavailability on bovine preantral follicular development in vitro[J]. Reproduction, 2007, 133(6): 1121–1128. DOI: 10.1530/REP-06-0382 |
| [19] | ARMSTRONG D G, GUTIERREZC G, BAXTER G, et al. Expression of mRNA encoding IGF-Ⅰ, IGF-Ⅱ and type 1 IGF receptor in bovine ovarian follicles[J]. J Endocrinol, 2000, 165(1): 101–113. |
| [20] | NAPOLI J L. Retinoic acid biosynthesis and metabolism[J]. FASEB J, 1996, 10(9): 993–1001. DOI: 10.1096/fasebj.10.9.8801182 |
| [21] | RHINN M, DOLLÉ P. Retinoic acid signaling during development[J]. Development, 2012, 139(5): 843–858. DOI: 10.1242/dev.065938 |



