2. 北京奶牛中心, 北京 100192
2. Beijing Dairy Cattle Center, Beijing 100192, China
牛奶是一种至少含有10 000种物质的混合物,含有丰富的乳糖、乳蛋白、脂类和矿物质,有益于人体健康和疾病预防[1-2]。传统的乳制品检测手段如凯氏定氮法、分光光度计法、酸水解法、盖勃氏法、碱水解法等测定方法耗费时间长,分析速度慢,准确度不高。气象色谱法、高效液相色谱法、气象色谱-质谱联法等测定方法,准确可靠,但对仪器设备要求高,不能满足室外快速准确的测定。越来越多的研究表明,使用中红外光谱分析技术(mid-infrared spectrometry, MIRS)能够对牛奶中各种常规营养成分如蛋白质[3]、酪蛋白[4]、脂肪[5]、乳铁蛋白[6]、尿素含量[7]等进行测定,还能够预测和分析一些具有经济价值的新成分,如各类脂肪酸[8-10]、乳蛋白组成及结构[11]、凝乳特性[12-14]、滴定酸度[13-14]、矿物质[14-15]、酮体[16]、能量负平衡[17-19]、发病风险[20]和甲烷排放[21]。此外,探究MIRS在乳业中应用于表型预测或遗传目的[22-25]有效性的研究逐渐增多。
红外光位于微波光区和可见光区之间,处于红色光的外侧,故命名为“红外线”。根据其波长可以分为3个区:近红外光区(0.75~2.50 μm)、中红外光区(2.50~25.00 μm)、远红外光区(25.00~1 000.00 μm)[26],不同波长的光发挥不同的作用,中红外光主要应用于有机化合物结构的识别和鉴定。同时,它也被称为基频区,波长在2 500~25 000 nm(波数为4 000~400 cm-1)的电磁波是绝大多数有机物和无机离子的基频吸收带,也是红外光中最适合进行物质定性和定量分析的区域[27]。奶牛生产性能测定(dairy herd improvement, DHI)是通过MIRS对与性状相关的中红外(mid-infrared, MIR)光谱特定区段建立标准模型,再将该区域光谱值代入标准模型中估计出该性状值,很大程度上受测定机器、光谱区段、预测方程、分析方法、地区、品种等因素的影响。当前只有产奶量(milk yield, MY)、乳脂率(fat percentage, FP)和乳蛋白率(protein percentage, PP)包含于奶牛选择指数内,理论上原始MIR光谱数据能更好地反映各个测定指标的特性,且绝大部分牛奶成分的遗传变异性未知,所以对原始MIR光谱数据的研究是有必要的。
本研究旨在通过对DHI测定所得原始MIR光谱数据进行遗传分析,更好地探究牛奶成分的遗传规律,以期从遗传角度提高奶牛质量,为种牛选择提供参考。
1 材料与方法 1.1 数据来源本研究中原始MIR光谱数据和DHI数据来自北京奶牛中心,试验群体为北京三元绿荷奶牛养殖中心某牛场1 822头处于泌乳期的健康荷斯坦牛。该牛场每个月采集奶样,在北京奶牛生产性能测定中心进行DHI测定。参照北京地区系谱数据库(公牛13 092头,母牛437 877头)对本研究所涉及的荷斯坦牛进行系谱追溯,获得有效系谱记录共8 563条,包括公牛4 100头、母牛4 463头。
1.2 数据整理 1.2.1 DHI数据质控本研究收集了试验牛场2016年12月,2017年2、4、5、6、7、8、9、10月共9个月数据。按照如下标准对数据进行质控:(1)保留同时具有DHI数据和原始MIR光谱变量数据的奶样记录;(2)胎次保留1~5胎;(3)泌乳天数(days in milk, DIM)保留5~600 d的数据记录;(4)将DHI测定月份划为9个水平。质控后共得到9 807条可用奶样数据。
1.2.2 原始MIR光谱数据红外光谱中通常以波数来表示吸收谱带的位置,横轴单位为cm-1,它表示的是1 cm长度内波的数目,纵轴单位为吸光度(absorbance, A),或是透过率(transmittance, T),T=10-A,而T=Imilk/Iwater,其中I为透射度,由于牛奶是混合物,所以一般T < 1。本研究保留波数为4 000~649 cm-1区域的原始MIR光谱数据,共得到899个波数数据(理论上,每个奶样的每个波数有一个值,但实际生产中原始MIR光谱中波数不是连续的,也不是固定间隔的)。
1.3 统计分析使用EXCEL(v2013)对原始MIR光谱数据进行基本描述性统计,使用R(v3.51)对原始MIR光谱数据进行变异系数(coefficients of variation, CV)计算及相关性分析。
主成分分析(principal components analysis, PCA)是对多变量高维数据进行降维,在损失少量信息的前提下以达到简化的目的,即把多指标转化为几个指标的多元统计方法[28]。使用SAS(v9.2)对已剔除水(波数范围:830~650 cm-1、1 700~1 600 cm-1、3 470~3 040 cm-1)的MIR光谱数据进行主成分分析,并选取累计贡献率达85%以上的主成分。
使用DMU(v6.0)DMUAI模块采用AI-REML结合EM算法估计主成分及899个波数的方差组分和遗传力。
| $ \begin{array}{l} {Y_{ijklmn}} = \mu + Testmont{h_i} + Parit{y_j} + \alpha DI{M_k} + \\ \;\;\;\;\;\;\;\;\;\;\;\;\;\;{\rm{ \mathsf{ β} }}{e^{ - 0.05DIM}}_l + {A_m} + {e_{ijklmn}}1 \end{array} $ | (模型1) |
| $ \begin{array}{l} {Y_{ijklmn}} = \mu + Testmont{h_i} + Parit{y_j} + \alpha DI{M_k} + \\ \;\;\;\;\;\;\;\;\;\;\;\;\;\;\;{\rm{ \mathsf{ β} }}{e^{ - 0.05DIM}}_l + {A_m} + P{e_m} + {e_{ijklmn}} \end{array} $ | (模型2) |
模型1中Yijklmn表示前4个主成分,模型2中Yijklmn表示899个波数;μ为群体均值;Testmonthi为固定效应,表示测定月份(i=1~9);Parityj为固定效应,表示胎次(j=1~5);DIMk为协变量;el-0.05DIM为协变量表示e的指数函数;α、β为协变量的回归系数;Am为加性遗传效应;Pem为永久环境效应;eijklmn为随机残差。模型参考Zaalberg等[29]的研究。
遗传力通过模型3计算而来[30]。
| $ {h^2} = \sigma _A^2/\sigma _P^2 = \sigma _A^2/\left( {\sigma _G^2 + \sigma _E^2} \right) $ | (模型3) |
式中,h2为遗传力;σA2为加性遗传方差;σG2为遗传方差;σP2为表型方差;σE2为残差方差。
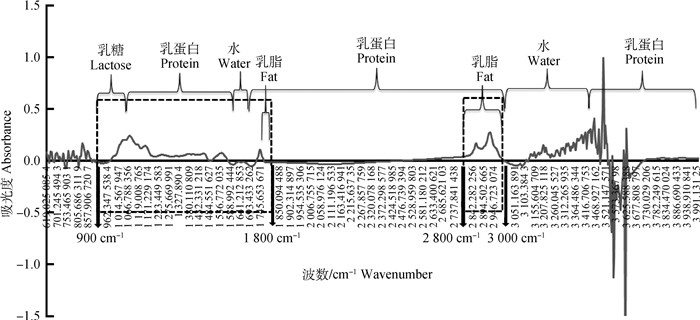
2 结果 2.1 牛奶MIRS相关光谱数据基本描述质控后获得9 807个奶样,每个奶样的MIRS光谱数据包含899个波数,以吸光度A为单位的光谱值个数有8 816 493个,其总和为229 752.585,均值为0.026,最大值为2.746,最小值为-2.343,极差为5.089。图 1为牛奶原始MIR光谱数据吸光度变化趋势,并标注了牛奶中主要成分的大致分布范围,可以看到与乳蛋白、水相关的光谱范围较大,而乳脂涉及区域较小。谱线在波数649 cm-1开始出现剧烈上下波动,而后轻微波动,之后呈现一段很长的平稳区,在3 066 cm-1开始波动式上升,后骤然下降,最后趋于平稳。水的存在造成830~650 cm-1、3 500~3 130 cm-1两个区域噪音干扰严重,谱线呈现杂乱无规律变化,干扰其它成分的红外吸收,1 700~1 600 cm-1是由于水O-H的弯曲振动造成,影响较小。但为避免引入噪音,许多研究都会把与水有关光谱区域剔除。对于1 800~900 cm-1区域,或说1 616~926 cm-1[31]、1 153~900 cm-1[32],它是一块指纹区,如C-O等结构大部分位于该区域内,这些键是牛奶中各组分(蛋白质、脂肪、乳糖等)的基础结构,可以说这些光谱值直接反映了牛奶状况。牛奶中红外光谱中每个变量对应的值都有意义,它可能是某个化学键的吸收峰值,也可能是不同物质交联键位的吸收峰值,但红外光吸收主要集中在虚线区为3 000~2 800 cm-1、1 800~900 cm-1。
|
图 1 牛奶中红外光谱的吸光度变化趋势 Fig. 1 Change trend of absorbance of mid-infrared spectrum in milk |
此外,表 1展示了部分有机物MIRS吸收峰[33],有研究表明,1 700~1 500 cm-1区域与乳蛋白键位有关,其中酰胺类(AmideⅠ、Ⅱ、Ⅲ)化学键分别对应的范围为1 705~1 558 cm-1、1 613~1 480 cm-1、1 280~1 200 cm-1[34];在另一个主要区域3 000~2 800 cm-1,与脂肪酸相关的酯键、碳氢键被认定于此,并被2 846 cm-1、2 915 cm-1两个波数把控[35];尿素在1 626 cm-1、1 597 cm-1、1 468 cm-1有吸收峰;波数为1 581~964 cm-1、1 812~1 697 cm-1、2 969~2 699 cm-1与丙酮有关;1 100~900 cm-1与多糖分子(乳糖)相关等。以上信息对建立相关标准模型具有参考价值。
|
|
表 1 部分有机化合物红外吸收峰 Table 1 Infrared absorption peak of the partial organic compounds |
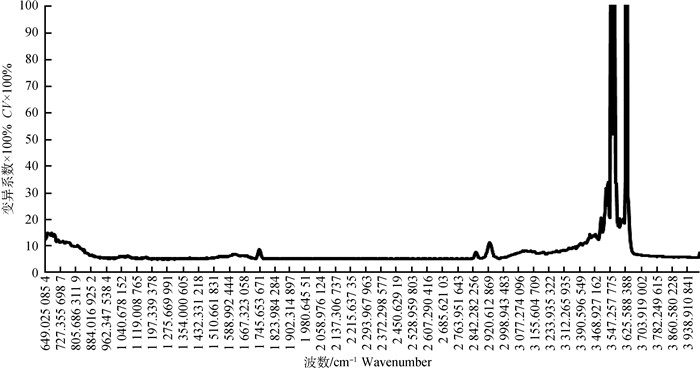
图 2表示的是MIR光谱每个变量之间的CV,表示数据相对变异程度大小。可以看到,两个CV大于30%的区域在3 574~3 521 cm-1、3 630~3 618 cm-1之间,包括16个变量,其余大部分处于20%以下,变化平稳。
|
图 2 899个波数的变异系数 Fig. 2 Variation coefficients of 899 wavenumbers |
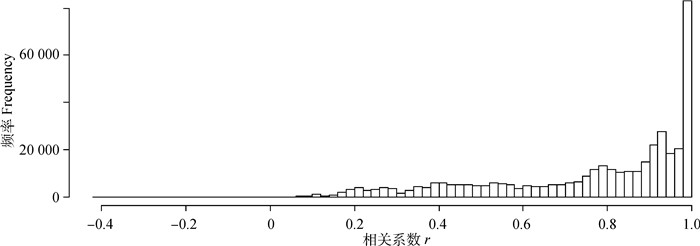
899个波数对应的光谱值之间相关系数(r)如图 3所示。r的“均值±标准差”为0.769 1±0.000 4,rMAX为0.999,rMIN为-0.405,极差值为1.405。其中大于0.900的相关系数的比例达到42.64%,相关系数范围在0.800~0.900、0.700~0.800之间的比例分别为14.66%、11.45%。从图 3中可以看出,大部分相关系数集中于0.500~1.000,占比为81.09%,说明不同变量之间具有较高的相关关系,关联紧密。
|
图 3 牛奶MIR光谱每个变量之间相关关系直方图 Fig. 3 Histogram of correlation between each variable of mid-infrared spectrum in milk |
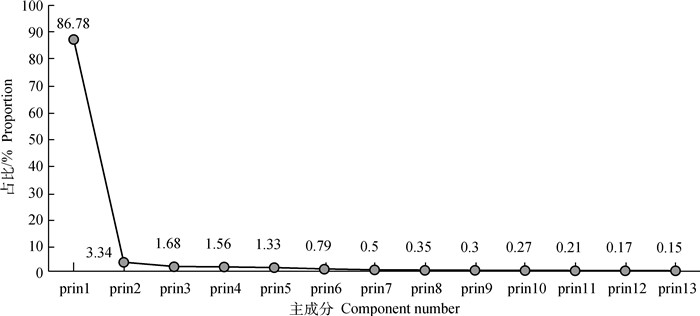
通过SAS进行PCA,得到剔去水的原始MIR光谱第一、二、三、四主成分特征值(eigenvalue)分别为615.277、23.689、11.942、11.074,占比分别为86.78%、3.34%、1.68%、1.56%,第一主成分解释方差在85%以上,即包含原始数据的信息总量达到了85%。并且可以从图 4看出,折线从第二主成分开始基本保持平稳状态。
|
图 4 剔除水的MIRS数据主成分碎石图 Fig. 4 Principal components accumulation of MIRS with the wave of water removed |
以吸光度(B)为单位, 利用模型1对前6个主成分进行遗传估计,表 2为剔除水区域的MIR光谱数据,结果显示,第一、第二、第三主成分h2分别为0.016±0.006、0.152±0.011、0.087±0.010,最高的遗传力为0.152。由于主成分保留了原始变量绝大多数信息,表明原始MIR光谱数据具有遗传特性。
|
|
表 2 剔除水后的MIR光谱数据主成分遗传力 Table 2 Principal component heritability of mid-infrared spectrum data of milk without water |
利用模型2对MIR光谱的899个波数进行遗传估计,如图 5所示。不同点代表 4 000~649 cm-1区域内899个波数的遗传力。从总体变化趋势来看,887~649 cm-1、3 625~3 498 cm-1属于极低遗传力区域,遗传力接近于0。1 708~887 cm-1遗传力开始变化,总体上提高,均值0.033。2 801~1 708 cm-1区域的遗传力没有太大变化,均值为0.013。从2 801 cm-1开始,遗传力又开始上升,直到3 495 cm-1,均值为0.050。4 000~3 629 cm-1,遗传力趋于稳定0.024。计算遗传力,以吸光度(B)为单位,有55个变量遗传力在0.100以上,hMAX2为0.604,hMIN2为0.209;以透射率(T)为单位,有20个变量的遗传力在0.100以上,hMAX2为0.518,hMIN2为0.103。
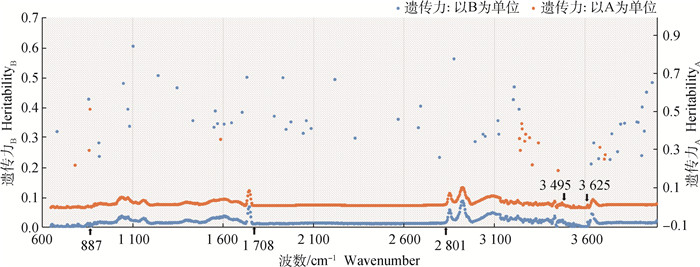
|
图 5 不同单位下遗传力随波数变化规律 Fig. 5 The variation of heritability with wave number under different units |
表 3表示的是899个波数的吸光度、透射率、变异系数随遗传力的变化规律。在以吸光度(B)为单位计算的遗传力中,有7个变量不收敛,没有遗传力,大约20%变量遗传力极低,73%变量遗传力在0.010~0.100之间,只有7%的变量具有中高遗传力。高低遗传力相比,高遗传力区域吸光度增大,透射率减少,变异系数随之变小。在以透射率(T)为单位计算的遗传力中,大约11%的变量遗传力极低,86%变量遗传力在0.010~0.100,中高遗传力的极少,占比3%。高低遗传力相比,随遗传力提高,吸光度增大,透射率减少,变异系数减少。
|
|
表 3 吸光度、透射率、变异系数随遗传力变化规律 Table 3 The change law of absorbance, transmittance and variation coefficient with heritability |
MIRS广泛用于多个领域,它不需要对样品进行复杂的预处理,或可以直接进行分析,借助定制化、简化的仪器可以对不同领域的待检样品进行在线大批量连续测定,自动地快速获得无损光谱。光谱包含了非常复杂的信息,对每个样本的描述是独一无二,反映了其物理和分子特性。此外,可直接对光谱进行评估,并且光谱数据易于储存。
本研究计算原始MIR光谱的吸光度A均值为0.026,在Bittante和Cecchinato[36]的研究中其均值为0.027,几乎相等。Soyeurt等[31]的研究显示,1 658~1 628 cm-1、3 444~3 105 cm-1两个MIR光谱区域的CV大于30%,本研究结果与之相比,没有重合,可能是由于本研究中两个区域的CV均小于20%,变异系数是衡量离散程度的无量纲量,变异系数高可能由噪音引起,所以在之后的研究中可以考虑对相关区域进行处理。不同波数之间具有较高的相关关系,关联紧密,因此可以使用主成分分析从另一个角度对光谱数据进行分析。
探究原始MIR光谱数据是否具有遗传特性,把水有关区段剔除后的MIR光谱数据进行主成分分析,得出的第一、第二、第三主成分h2分别为0.016±0.006、0.152±0.011、0.087±0.010,最高的遗传力为0.152。由于主成分保留了原始变量绝大多数信息,表明原始MIR光谱数据总体上具有遗传特性。Dagnachew等[37]对山羊奶的傅里叶红外光谱(fourier transform infrared spectra, FTIR)进行遗传评价,对原始数据进行主成分分析,前8个主成分遗传力为0.011~0.285。Soyeurt等[31]对荷斯坦奶牛的MIR光谱进行主成分分析,前46个主成分遗传力为0.000~0.350。外界环境、个体本身都会对遗传力估计造成影响。相比之下,本研究计算的主成分遗传力偏低,这与数据预处理、数据量、系谱、统计方法有关,而且并不是所有的MIR光谱区域都有遗传意义。此外,通过计算主成分遗传力,可以看出代表信息最多的第一主成分的遗传力并不是最高的,遗传力高的往往在后面几个指标出现,表明了光谱信息大部分并不是由基因导致的,更多的是一种加性的结果。
Bittante和Cecchinato[36]的研究显示,1 056个变量中约有16%不存在遗传力或遗传力小于0.050。有55%的变量遗传估计为0.050~0.100,遗传力在0.150~0.200、0.200~0.250内的变量占比分别为6%、4%,只有1%的变量遗传力在0.250以上,与之相比,本研究所用MIR光谱的变量遗传力大多集中于0.010~0.030之间,遗传力偏低,这大概与数据量有关,前者结果也表明,遗传力低的变量,平均透射率和变异系数高,并随着遗传力提升而减小。Zaalberg等[29]也表示,现研究中单个波数的遗传力处于低水平,这与遗传估计方法有关。本研究中,887~649 cm-1、3 625~3 498 cm-1属于极低遗传力区域,同时也是水分影响区域,分析可能是由于水导致残差效应方差增加比加性遗传效应方差快。
本研究通过简单的分析,对MIR光谱的基本特征进行探究。本研究中使用主成分分析方法和对每个变量进行遗传评估的方法对于寻找牛奶中各个乳成分相关的光谱区域是非常有帮助的,这有可能减少了未来应用光谱数据的大小。此外,还需更多研究对MIR光谱中遗传变异做出解释,找到最佳方法来选择光谱中相关区域并确定该物质。对牛奶中具有经济价值且测量成本高的新性状相关光谱区域进行建模,并利用化学计量法得到准确值,并对该模型不断校正,应用于生产中,从而不断提高我国牛奶的营养和质量。
4 结论牛奶的MIR光谱是一条潜藏许多牛奶组分信息的曲线,超过80%波数之间相关系数在0.500~1.000之间,同时,在3 574~3 521 cm-1、3 630~3 618 cm-1区域变量的变异程度很高。利用PCA对899个MIR光谱的波数进行降维,效果很好。对每个波数进行遗传估计,MIR光谱的遗传力大多集中于0.010~0.030之间,遗传力偏低,处于中高遗传力的变量占比约为7%,随遗传力提高,吸收度增大,透射率和变异系数降低。
致谢: 感谢北京奶牛中心在数据上的支持。
| [1] | PATTON S. Milk|milk in human health and nutrition[M]//FUQUAY J W.Encyclopedia of Dairy Sciences.2nd ed.Amsterdam: Elsevier, 2011: 607-612. |
| [2] | PEREIRA P C. Milk nutritional composition and its role in human health[J]. Nutrition, 2014, 30(6): 619–627. DOI: 10.1016/j.nut.2013.10.011 |
| [3] | ETZION Y, LINKER R, COGAN U, et al. Determination of protein concentration in raw milk by mid-infrared fourier transform infrared/attenuated total reflectance spectroscopy[J]. J Dairy Sci, 2004, 87(9): 2779–2788. DOI: 10.3168/jds.S0022-0302(04)73405-0 |
| [4] | BONFATTI V, DI MARTINO G, CARNIER P. Effectiveness of mid-infrared spectroscopy for the prediction of detailed protein composition and contents of protein genetic variants of individual milk of Simmental cows[J]. J Dairy Sci, 2011, 94(12): 5776–5785. DOI: 10.3168/jds.2011-4401 |
| [5] | SOYEURT H, DARDENNE P, GILLON A, et al. Variation in fatty acid contents of milk and milk fat within and across breeds[J]. J Dairy Sci, 2006, 89(12): 4858–4865. DOI: 10.3168/jds.S0022-0302(06)72534-6 |
| [6] | SOYEURT H, COLINET F G, ARNOULD V M R, et al. Genetic variability of lactoferrin content estimated by mid-infrared spectrometry in bovine milk[J]. J Dairy Sci, 2007, 90(9): 4443–4450. DOI: 10.3168/jds.2006-827 |
| [7] | SANTOS P M, PEREIRA-FILHO E R, RODRIGUEZ-SAONA L E. Rapid detection and quantification of milk adulteration using infrared microspectroscopy and chemometrics analysis[J]. Food Chem, 2013, 138(1): 19–24. DOI: 10.1016/j.foodchem.2012.10.024 |
| [8] | SOYEURT H, DARDENNE P, DEHARENG F, et al. Estimating fatty acid content in cow milk using mid-infrared spectrometry[J]. J Dairy Sci, 2006, 89(9): 3690–3695. DOI: 10.3168/jds.S0022-0302(06)72409-2 |
| [9] | SOYEURT H, DEHARENG F, GENGLER N, et al. Mid-infrared prediction of bovine milk fatty acids across multiple breeds, production systems, and countries[J]. J Dairy Sci, 2011, 94(4): 1657–1667. DOI: 10.3168/jds.2010-3408 |
| [10] | FERRAND-CALMELS M, PALHIÈRE I, BROCHARD M, et al. Prediction of fatty acid profiles in cow, ewe, and goat milk by mid-infrared spectrometry[J]. J Dairy Sci, 2014, 97(1): 17–35. DOI: 10.3168/jds.2013-6648 |
| [11] | DE MARCHI M, BONFATTI V, CECCHINATO A, et al. Prediction of protein composition of individual cow milk using mid-infrared spectroscopy[J]. Ital J Anim Sci, 2010, 8(S2): 399–401. |
| [12] | DE MARCHI M, TOFFANIN V, CASSANDRO M, et al. Prediction of coagulating and noncoagulating milk samples using mid-infrared spectroscopy[J]. J Dairy Sci, 2013, 96(7): 4707–4715. DOI: 10.3168/jds.2012-6506 |
| [13] | DE MARCHI M, FAGAN C C, O'DONNELL C P, et al. Prediction of coagulation properties, titratable acidity, and pH of bovine milk using mid-infrared spectroscopy[J]. J Dairy Sci, 2009, 92(1): 423–432. DOI: 10.3168/jds.2008-1163 |
| [14] | TOFFANIN V, DE MARCHI M, LOPEZ-VILLALOBOS N, et al. Effectiveness of mid-infrared spectroscopy for prediction of the contents of calcium and phosphorus, and titratable acidity of milk and their relationship with milk quality and coagulation properties[J]. Int Dairy J, 2015, 41: 68–73. DOI: 10.1016/j.idairyj.2014.10.002 |
| [15] | SOYEURT H, BRUWIER D, ROMNEE J M, et al. Potential estimation of major mineral contents in cow milk using mid-infrared spectrometry[J]. J Dairy Sci, 2009, 92(6): 2444–2454. DOI: 10.3168/jds.2008-1734 |
| [16] | GRELET C, BASTIN C, GELÉ M, et al. Development of Fourier transform mid-infrared calibrations to predict acetone, β-hydroxybutyrate, and citrate contents in bovine milk through a European dairy network[J]. J Dairy Sci, 2016, 99(6): 4816–4825. DOI: 10.3168/jds.2015-10477 |
| [17] | MCPARLAND S, BANOS G, WALL E, et al. The use of mid-infrared spectrometry to predict body energy status of Holstein cows[J]. J Dairy Sci, 2011, 94(7): 3651–3661. DOI: 10.3168/jds.2010-3965 |
| [18] | MCPARLAND S, BANOS G, MCCARTHY B, et al. Validation of mid-infrared spectrometry in milk for predicting body energy status in Holstein-Friesian cows[J]. J Dairy Sci, 2012, 95(12): 7225–7235. DOI: 10.3168/jds.2012-5406 |
| [19] | MCPARLAND S, LEWIS E, KENNEDY E, et al. Mid-infrared spectrometry of milk as a predictor of energy intake and efficiency in lactating dairy cows[J]. J Dairy Sci, 2014, 97(9): 5863–5871. DOI: 10.3168/jds.2014-8214 |
| [20] | LAINÉ A, BASTIN C, THERON L, et al.Potential of fine milk composition for cow udder health management[C]//Proceedings of the 39th ICAR Conference, Berlin.Germany: ICAR, 2014. |
| [21] | DEHARENG F, DELFOSSE C, FROIDMONT E, et al. Potential use of milk mid-infrared spectra to predict individual methane emission of dairy cows[J]. Animal, 2012, 6(10): 1694–1701. DOI: 10.1017/S1751731112000456 |
| [22] | FLEMING A, SCHENKEL F S, KOECK A, et al. Heritabilities of measured and mid-infrared predicted milk fat globule size, milk fat and protein percentages, and their genetic correlations[J]. J Dairy Sci, 2017, 100(5): 3735–3741. DOI: 10.3168/jds.2016-12243 |
| [23] | NARAYANA S G, SCHENKEL F S, FLEMING A, et al. Genetic analysis of groups of mid-infrared predicted fatty acids in milk[J]. J Dairy Sci, 2017, 100(6): 4731–4744. DOI: 10.3168/jds.2016-12244 |
| [24] | FLEMING A, SCHENKEL F S, MALCHIODI F, et al. Genetic correlations of mid-infrared-predicted milk fatty acid groups with milk production traits[J]. J Dairy Sci, 2018, 101(5): 4295–4306. DOI: 10.3168/jds.2017-14089 |
| [25] | HEIN L, SØRENSEN L P, KARGO M, et al. Genetic analysis of predicted fatty acid profiles of milk from Danish Holstein and Danish Jersey cattle populations[J]. J Dairy Sci, 2018, 101(3): 2148–2157. DOI: 10.3168/jds.2017-13225 |
| [26] |
钟海庆.
红外光谱法入门[M]. 北京: 化学工业出版社, 1984: 1-4.
ZHONG H Q. Introduction to infrared spectroscopy[M]. Beijing: Chemical Industry Press, 1984: 1-4. (in Chinese) |
| [27] |
董利锋, YANT H, 屠焰, 等. 中红外光谱技术在牛奶营养物质预测及奶牛相关特性分析上的应用[J]. 动物营养学报, 2016, 28(2): 326–334.
DONG L F, YAN T H, TU Y, et al. Applicationof mid-infrared spectrometry in milk nutrient components prediction and related traits analysis of dairy cows[J]. Chinese Journal of Animal Nutrition, 2016, 28(2): 326–334. DOI: 10.3969/j.issn.1006-267x.2016.02.004 (in Chinese) |
| [28] |
何晓群.
多元统计分析[M]. 2版. 北京: 中国人民大学出版, 2008: 152-167.
HE X Q. Multivariate statistical analysis[M]. 2nd ed. Beijing: China Renmin University Press, 2008: 152-167. (in Chinese) |
| [29] | ZAALBERG R M, SHETTY N, JANSS L, et al. Genetic analysis of Fourier transform infrared milk spectra in Danish Holstein and Danish Jersey[J]. J Dairy Sci, 2019, 102(1): 503–510. DOI: 10.3168/jds.2018-14464 |
| [30] |
刘澳星, 郭刚, 王雅春, 等. 中国荷斯坦牛初产日龄遗传评估及全基因组关联分析[J]. 畜牧兽医学报, 2015, 46(3): 373–381.
LIU A X, GUO G, WANG Y C, et al. Genetic analysis and genome wide association studies for age at first calving in Chinese Holsteins[J]. Acta Veterinaria et Zootechnica Sinica, 2015, 46(3): 373–381. (in Chinese) |
| [31] | SOYEURT H, MISZTAL I, GENGLER N. Genetic variability of milk components based on mid-infrared spectral data[J]. J Dairy Sci, 2010, 93(4): 1722–1728. DOI: 10.3168/jds.2009-2614 |
| [32] | KAROUI R, MOUAZEN A M, DUFOUR É, et al. A comparison and joint use of NIR and MIR spectroscopic methods for the determination of some parameters in European Emmental cheese[J]. Eur Food Res Technol, 2006, 223(1): 44–50. DOI: 10.1007/s00217-005-0110-2 |
| [33] | COATES J.Interpretation of infrared spectra, a practical approach[M]//MEYERS R A.Encyclopedia of Analytical Chemistry.Chichester: Wiley, 2006: 4-15. |
| [34] | SIVAKESAVA S, IRUDAYARAJ J. Rapid determination of tetracycline in milk by FT-MIR and FT-NIR spectroscopy[J]. J Dairy Sci, 2002, 85(3): 487–493. DOI: 10.3168/jds.S0022-0302(02)74099-X |
| [35] | DUFOUR E, MAZEROLLES G, DEVAUX M F, et al. Phase transition of triglycerides during semi-hard cheese ripening[J]. Int Dairy J, 2000, 10(1-2): 81–93. DOI: 10.1016/S0958-6946(00)00025-X |
| [36] | BITTANTE G, CECCHINATO A. Genetic analysis of the Fourier-transform infrared spectra of bovine milk with emphasis on individual wavelengths related to specific chemical bonds[J]. J Dairy Sci, 2013, 96(9): 5991–6006. DOI: 10.3168/jds.2013-6583 |
| [37] | DAGNACHEW B S, KOHLER A, ÅDNØY T. Genetic and environmental information in goat milk Fourier transform infrared spectra[J]. J Dairy Sci, 2013, 96(6): 3973–3985. DOI: 10.3168/jds.2012-5972 |



