2. 西北农林科技大学动物医学院, 杨凌 712100;
3. 中国兽医药品监察所, 北京 100081;
4. 农业部兽用药物与诊断技术北京科学观测实验站, 北京 100193
2. College of Veterinary Medicine, Northwest A & F University, Yangling 712100, China;
3. China Institute of Veterinary Drug Control, Beijing 100081, China;
4. Scientific Observation and Experiment Station of Veterinary Drugs and Diagnostic Technology of Beijing of Ministry of Agriculture, Beijing 100193, China
牛病毒性腹泻病毒(bovine viral diarrhea virus, BVDV)属于黄病毒科(Flaviridae)瘟病毒属(Pestivirus)[1-2],其基因组为单股正链RNA,由5′非编码区(5′UTR)、编码多聚蛋白的开放阅读框、3′非编码区(3′UTR)组成。BVDV基因组共编码12种蛋白,在基因组中分布顺序依次为5′-Npro-Capsid-Erns-E1-E2-P7-NS2/3-NS4A- NS4B-NS5A-NS5B-3′[3-5]。BVDV感染宿主细胞后即开始其感染循环,病毒基因组RNA作为模板翻译产生BVDV复制所需的各种病毒蛋白质[6]。在病毒蛋白的作用下,转录合成子代病毒RNA合成所需的负链RNA[6]。负链RNA可作为BVDV在细胞内复制的标志物。
荧光定量RT-PCR方法(reverse transcription real-time quantitative PCR, RT-qPCR)具有高灵敏度和重复性,被广泛用于病毒定量及病原检测中[7]。然而,标准的RT-qPCR方法不能区分RNA病毒复制过程中的正链和负链RNA,因此也不能对病毒进行绝对的定量[8]。而链特异性荧光定量RT-PCR方法(strand specific reverse transcription real-time quantitative PCR, ssRT-qPCR),不仅具有RT-qPCR方法的高灵敏度和重复性,也具有区分病毒复制过程中的正链和负链的能力[9-11]。ssRT-qPCR原理是利用5′端添加非病毒序列特异性的反转录引物分别合成病毒正、负链的cDNA,通过引入的非病毒序列标签区别病毒的正、负链,再通过非病毒序列标签和与之配对的病毒基因组序列上的引物分别对病毒的正、负链进行定量检测[8]。ssRT-qPCR一般包括正链特异性荧光定量RT-PCR方法[ss(+)RT-qPCR]和负链特异性荧光定量RT-PCR方法[ss(-)RT-qPCR]。
ssRT-qPCR在RNA病毒基因组复制的分子机制和调控机制研究、感染性病毒检测、宿主蛋白在病毒复制中的研究等方面具有广泛的应用。Richardson等[12]建立登革热病毒2型的ssRT-qPCR,并利用该方法对埃及伊蚊肠道组织和腿部病毒的复制进行定量分析,首次描述了登革热病毒2型在埃及伊蚊肠道组织中的变化规律。Haist等[13]建立了沙粒病毒的ssRT-qPCR方法,用于研究沙粒病毒基因组RNA复制的调节方式。Komurian-Pradel等[14]建立丙型肝炎病毒的ssRT-qPCR方法用于丙型肝炎病毒复制动力学的研究。Lagaye等[15]利用ssRT-qPCR方法评价了针对细胞自噬机制的新型抗丙型肝炎病毒药物的抗病毒效力。
为了对BVDV复制过程中正、负链RNA进行定量检测,针对BVDV的正、负链分别设计链特异性反转录引物和链特异性PCR引物,建立了BVDV ssRT-qPCR。
1 材料与方法 1.1 病毒与细胞牛病毒性腹泻病毒BVDV BJ-2013株、牛副流感病毒3型BPIV3-BJ株、牛传染性鼻气管炎病毒BRSV-BJ株、牛呼吸道合胞体病毒均为实验室保存。MDBK细胞由中国农业科学院北京畜牧兽医研究所宠物疫病防控创新团队保存。
1.2 主要试剂及仪器病毒DNA/RNA小量提取试剂盒为北京艾德莱生物技术有限公司产品;M-MLV反转录酶为TaKaRa公司产品;ChamQTM Universal SYBR qPCR Master Mix为南京诺唯赞生物科技有限公司产品;荧光定量PCR仪(ABI Q7 Flex 96孔)为美国ABI公司产品。
1.3 引物设计根据BVDV基因组序列,分别设计特异性反转录引物(BV+RT/BV-RT)及荧光定量PCR引物。参考文献[8],分别在特异性反转录引物的5′端添加非病毒序列。同时,设计用于构建标准品的引物,引物BV+F与BV+RT用于正链标准品模板的RT-PCR扩增;引物BV-R与BV-RT用于负链标准品模板的RT-PCR扩增。引物序列见表 1,非病毒序列为下划线标记部分。
|
|
表 1 本研究所用引物 Table 1 Primers used in this study |
参照试剂盒说明书提取BVDV的RNA,并利用M-MLV反转录酶合成cDNA。以合成的cDNA为模板,利用引物分别对BVDV正、负链标准品模板进行PCR扩增。回收预期目的条带后,连接至pMD18-T载体,鉴定阳性的重组质粒送北京铂尚生物技术有限公司进行测序验证。用紫外分光光度计测定重组质粒的质量浓度,并参照公式:重组质粒拷贝数(copies·μL-1)=6.02×1023(copies·mol-1)×浓度(g·mL-1) ×10-3/分子质量(g·mol-1),计算重组质粒拷贝数。将重组质粒分别进行10倍倍比稀释,保存备用。
1.5 荧光定量PCR反应条件根据ChamQTM Universal SYBR qPCR Master Mix试剂盒说明书确定荧光定量PCR体系为20 μL。以“1.4”中制备的pMD18T-BV+和pMD18T-BV-重组质粒为模板,分别对退火温度(56、58、60、62 ℃)及引物(10 μmol·L-1)用量(0.4、0.6、0.8、1.0 μL)进行优化。
1.6 标准曲线的建立选用浓度为102~107copies·μL-1的pMD18T-BV+和pMD18T-BV-重组质粒作为模板分别进行检测。以log(拷贝数)为横坐标,以Ct值为纵坐标绘制标准曲线,并进行线性回归分析。
1.7 敏感性试验采用浓度为101~107copies·μL-1的pMD18T-BV+和pMD18T-BV-重组质粒为模板,应用建立的方法进行检测,评估所建立方法的敏感性。
1.8 重复性试验采用浓度为105~107copies·μL-1的pMD18T-BV+和pMD18T-BV-重组质粒作为模板分别进行3批批内和批间重复试验。每批次批间和批内重复试验设置3个重复,以组内和组间的变异系数(coefficient of variation, CV)评估所建立方法的重复性。
1.9 特异性试验以牛副流感病毒3型BPIV3-BJ株、牛传染性鼻气管炎病毒BRSV-BJ株、牛呼吸道合胞体病毒BRSV的cDNA或DNA、ddH2O及重组质粒pMD18T-BV+和pMD18T-BV-为模板分别应用ss(+)RT-qPCR和ss(-)RT-qPCR方法进行检测,以评估所建立荧光定量PCR方法的特异性。
1.10 BVDV链特异性检测为了验证所建立ssRT-qPCR方法的可靠性,利用该方法对复制过程中BVDV正、负链RNA进行定量分析。以10、1、0.1 MOI的接毒剂量分别接种于培养在6孔细胞培养板中的MDBK细胞。在接毒后0、2、6、12、24、36、48 h分别收取细胞样品,用TRIzol Reagent提取细胞总RNA,利用测定细胞总RNA浓度后,取1 μg细胞总RNA利用特异性反转录引物BV+RT和BV-RT进行cDNA的合成。反转录过程:(1)总RNA 1 μg,特异性反转录引物(10 μmol·L-1)0.5 μL,RNase free H2O补至10 μL;反应条件为70 ℃ 10 min,冰浴3 min。(2)前述反应结束后,分别加入M-MLV反转录酶0.5 μL,5×Reverse Transcriptase M-MLV Buffer 4.0 μL,dNTPs Mixture(10 mmol·L-1) 2.0 μL,RRI 1.0 μL,RNase free H2O 2.5 μL。反转录条件:30 ℃,10 min;42 ℃,1 h;85 ℃,15 s;4 ℃保存。合成cDNA后,用所建立的ssRT-qPCR方法对BVDV正、负链进行定量检测。根据所建立的标准曲线计算每微克细胞总RNA中BVDV正、负链的量,以时间为横坐标,以每微克细胞总RNA中BVDV正链或负链的量的对数值为纵坐标绘制曲线,评估BVDV复制过程中BVDV正链或负链的动态变化。
2 结果 2.1 标准品DNA模板的制备为了构建ssRT-qPCR方法建立所需要的标准品质粒,以RT-PCR方法成功扩增得到BVDV正、负链标准品DNA模板(图 1),并将其连接至pMD18-T载体。将验证正确的重组质粒分别命名为pMD18T-BV+和pMD18T-BV-,分别作为BVDV ss(+)RT-qPCR方法和ss(-)RT-qPCR方法的标准品。
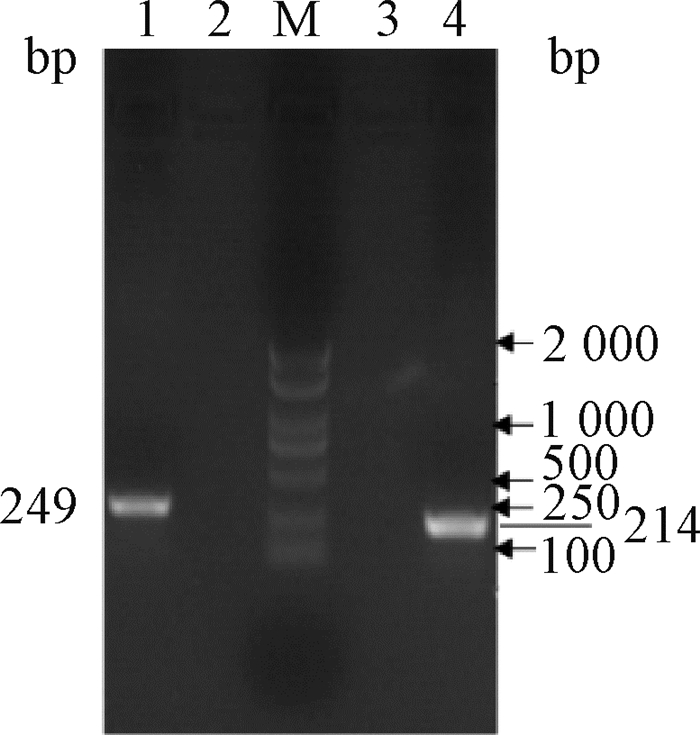
|
1.pMD18T-BV-;2.pMD18T-BV-阴性对照;M.DL2000相对分子质量标准;3.pMD18T-BV+阴性对照;4.pMD18T-BV+ 1.pMD18T-BV-; 2.Negative control for the amplification of pMD18T-BV-; M, DL2000 DNA marker; 3.Negative control for the amplification of pMD18T-BV+; 4.pMD18T-BV+ 图 1 标准品DNA模板的RT-PCR扩增 Fig. 1 The amplification of the template for ssRT-qPCR |
通过对ss(+)RT-qPCR和ss(-)RT-qPCR方法退火温度及引物用量的优化,最终确定其反应体系为20 μL:ChamQTM Universal SYBR qPCR Master Mix 10 μL、引物(10 μmol·L-1)各0.8 μL、模板1 μL、dd H2O 7.4 μL。BVDV ssRT-qPCR方法采用两步法,反应条件:95 ℃ 30 s;95 ℃ 5 s,60 ℃ 30 s(收集荧光),40个循环;熔解曲线:95 ℃ 15 s,60 ℃ 1 min,95 ℃ 15 s。
2.3 标准曲线的建立选用浓度为102~107copies·μL-1的pMD18T-BV+和pMD18T-BV-重组质粒作为模板,分别利用ss(+)RT-qPCR和ss(-)RT-qPCR方法进行检测并绘制标准曲线。结果显示,ss(+)RT-qPCR的标准曲线为y=-3.552x+41.13,线性相关系数R2为0.998 1(图 2a);ss(-)RT-qPCR标准曲线为y=-3.501x+43.24,线性相关系数R2为0.995 3(图 2b)。上述结果表明,在102~107拷贝范围内,所建立的SS(+)qRT-PCR和SS(-)qRT-PCR线性关系良好。
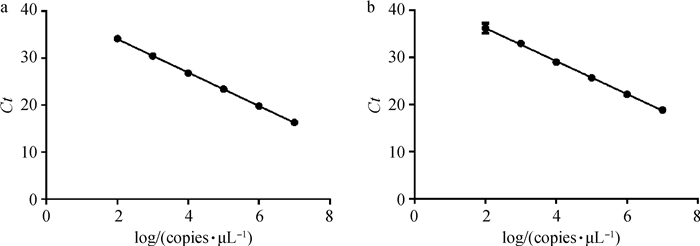
|
a. ss(+)RT-qPCR标准曲线;b. ss(-)RT-qPCR标准曲线 a. Standard curve of ss(+)RT-qPCR; b. Standard curve of ss(-)RT-qPCR 图 2 BVDV ssRT-qPCR标准曲线 Fig. 2 Standard curve of ssRT-qPCR |
选用浓度为101~107copies·μL-1的pMD18T-BV+和pMD18T-BV-重组质粒为模板,对建立的ss(+)RT-qPCR和ss(-)RT-qPCR方法敏感性进行评估,结果显示,在不同模板浓度条件下,两种方法的扩增曲线均呈现典型的S型曲线(图 3)。熔解曲线分析显示,所有的熔解曲线均为单一峰,说明不存在非特异性扩增。敏感性试验结果显示,ss(+)RT-qPCR方法最低检测限为10拷贝,而ss(-)RT-qPCR方法最低检测限为100拷贝。
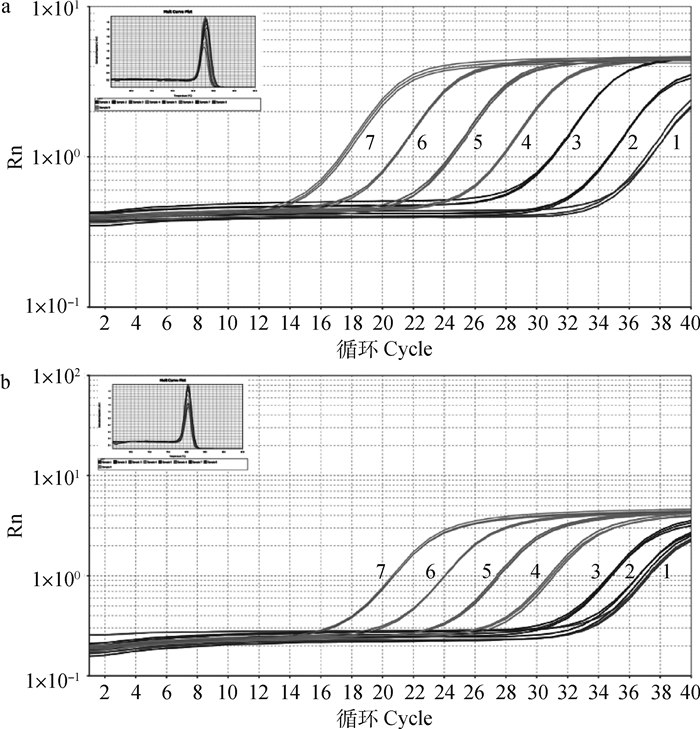
|
a. ss(+)RT-qPCR方法敏感性试验(1~7.浓度为101~107 copies·μL-1的pMD18T-BV+重组质粒模板);b. ss(-)RT-qPCR方法敏感性试验(1~7.浓度为101~107 copies·μL-1的pMD18T-BV-重组质粒模板) a. The sensitivity test for ss(+)RT-qPCR (1-7. Template of pMD18T-BV+ with 101-107 copies·μL-1concentration); b. The sensitivity test for ss(-)RT-qPCR (1-7. Template of pMD18T-BV-with 101-107 copies·μL-1concentration) 图 3 BVDV ssRT-qPCR方法敏感性试验 Fig. 3 The sensitivity test for BVDV ssRT-qPCR |
选用浓度为105~107copies·μL-1的pMD18T-BV+和pMD18T-BV-重组质粒作为模板分别进行3批批内和批间重复试验以评估ss(+)RT-qPCR和ss(-)RT-qPCR方法的可重复性。试验结果显示,所建立的ss(+)RT-qPCR和ss(-)RT-qPCR方法的批内和批间重复试验的变异系数均小于1%,说明所建立BVDV ssRT-qPCR方法具有良好的重复性(表 2、3)。
|
|
表 2 SS(+)qRT-PCR重复试验 Table 2 Inter-assay and intra-assay reproducibility test of the SS(+)qRT-PCR |
|
|
表 3 SS(-)qRT-PCR重复试验 Table 3 Inter-assay and intra-assay reproducibility test of the SS(-)qRT-PCR |
为了评估ss(+)RT-qPCR和ss(-)RT-qPCR方法的特异性,以牛副流感病毒3型BPIV3-BJ株、牛传染性鼻气管炎病毒BRSV-BJ株、牛呼吸道合胞体病毒的cDNA或DNA、ddH2O以及标准品为模板分别进行检测,结果显示仅含有pMD18T-BV+和pMD18T-BV-重组质粒的样品检测结果为阳性,其他检测结果均为阴性。该结果表明,所建立的ssRT-qPCR特异性较好(图 4)。
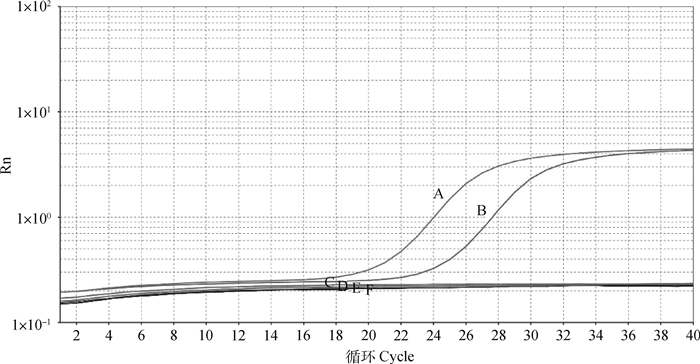
|
A. pMD18T-BV+;B. pMD18T-BV-;C.牛副流感病毒3型(BPIV3-BJ);D.牛传染性鼻气管炎病毒(BRSV-BJ);E.牛呼吸道合胞体病毒;F.ddH2O A. pMD18T-BV+; B. pMD18T-BV-; C. Bovine parainfluenza virus type 3(BPIV3-BJ); D. Infectious bovine rhinotracheitis virus(BRSV-BJ); E. Bovine respiratory syncytial virus; F. ddH2O 图 4 ssRT-qPCR特异性试验 Fig. 4 The specificity test of ssRT-qPCR |
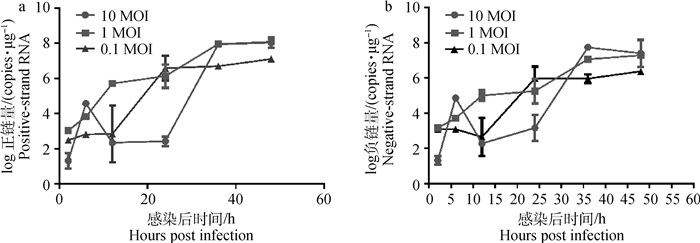
为了进一步验证所建立ssRT-qPCR方法的可靠性,利用该方法对复制过程中BVDV正、负链RNA分别进行了定量分析。在不同接毒剂量下,MDBK细胞内BVDV正、负链RNA的定量检测结果如图 5。由图 5可知,以0.1和10 MOI剂量感染MDBK后,细胞内BVDV正链和负链的动态变化趋势相似,BVDV感染细胞后2~6 h,BVDV正、负链RNA均呈现上升趋势,但在6~12 h间呈现下降趋势,12 h后呈现逐渐上升趋势,36~48 h间基本处于稳定期。以1 MOI剂量感染MDBK细胞,BVDV正、负链RNA的动态变化趋势略有差异。感染2 ~12 h间BVDV正、负链RNA整体呈上升趋势,在感染36 h后,BVDV正、负链RNA的量基本处于稳定期。10、1 MOI接毒剂量接种细胞后,BVDV正、负链RNA在感染后36 h基本维持稳定水平,以0.1 MOI接种细胞后BVDV正、负链RNA在感染后24 h即达到稳定水平。
|
a. BVDV正链RNA的动态变化;b. BVDV负链RNA的动态变化 a. Dynamic changes of BVDV positive-strand RNA; b. Dynamic changes of BVDV negative-strand RNA 图 5 不同MOI感染MDBK细胞后BVDV RNA的动态变化 Fig. 5 The dynamic changes of BVDV RNA in MDBK cells infected with different MOI |
荧光定量PCR方法因有高敏感性、高重复性、操作简单等优点而被广泛地应用于生命科学研究及动物疫病病原检测。荧光定量PCR方法包括探针法和染料法,其中染料法因成本较低,其应用更为广泛[7]。染料法虽不具有序列特异性,而且引物二聚体和其他非特异性扩增子也可能产生低水平的背景荧光,但是可借助熔解曲线分析和琼脂糖凝胶电泳分析评估其扩增的特异性[10]。链特异性荧光定量RT-PCR方法可区分RNA病毒的正、负链RNA,可排除正、负链RNA间的互相干扰,因此在感染性病毒检测、RNA病毒复制及调控机制以及宿主蛋白对病毒复制调控作用研究中已有应用[9]。
本研究中,建立的BVDV ss(+)RT-qPCR方法和ss (-)RT-qPCR方法,均具有良好的重复性,且与牛副流感病毒3型、牛传染性鼻气管炎病毒以及牛呼吸道合胞体病毒不存在交叉反应,其中ss(+)RT-qPCR方法最低可检测到10拷贝标准品质粒,而ss(-)RT-qPCR方法最低可检测到100拷贝标准品质粒。为了验证所建立的BVDV ss(+)RT-qPCR方法和ss(-)RT-qPCR方法的可靠性,利用该方法对BVDV在细胞复制过程中正、负链RNA进行定量分析。分析结果显示,以10、0.1 MOI剂量将BVDV接种于MDBK细胞后,BVDV正、负链RNA变化呈现先升后降再逐渐上升的趋势,在BVDV感染后36~48 h基本维持稳定;而以1 MOI剂量将BVDV接种于MDBK细胞后,BVDV正、负链RNA变化与以10、0.1 MOI剂量接种细胞后的变化存在差异,BVDV正、负链RNA变化虽有起伏但整体上呈上升趋势。BVDV基因组为单股正链RNA,其在细胞中复制时,需转录出负链RNA并与BVDV基因组RNA组成复制型双链RNA,之后在复制复合体的作用下启动子代病毒基因组的复制。在细胞中,BVDV负链RNA的出现表示BVDV子代病毒基因组RNA复制的开始,负链RNA的量与子代病毒RNA的复制的速度相关。负链RNA的增加表示在细胞中的BVDV子代RNA的复制速率逐渐提高,其含量达到稳定水平可能表示在细胞中BVDV复制与病毒释放之间达到了动态平衡状态。在之前的研究中,笔者利用荧光定量RT-PCR绘制BVDV一步生长曲线,发现在BVDV感染12 h后,细胞内产生的子代病毒才逐渐开始从细胞中释放,且在BVDV感染后12~48 h为其对数增长期[16]。众所周知,病原多种成分可以作为病原体相关分子模式被宿主模式识别受体所识别,如BVDV复制过程中出现的双链RNA(double strand RNA, dsRNA)、单链RNA(single strand RNA, ssRNA)以及核衣壳蛋白等[17-19]。病原体相关分子模式被宿主模式识别受体所识别后,通过一系列的信号转导激活细胞内固有免疫防御系统,抵御病原体的入侵和抑制病原体的复制[20-21]。固有免疫防御系统的抗病毒作用主要通过众多的抗病毒蛋白行使,如PKR和OAS蛋白[22-23]。PKR蛋白在固有免疫信号通路中发挥重要作用,可被dsRNA激活,而活化的PKR可促进IFN的产生, 可以调节和协同参与固有免疫防御系统中其他抗病毒机制[24-26]。不仅如此,PKR也可以通过使eIF-2α磷酸化来阻碍起始复合物的形成,从而直接行使抗病毒作用[27]。OAS蛋白可被dsRNA激活,通过OAS/RNAseL系统降解病毒RNA[28-30]。综合BVDV一步生长曲线及本研究中测定的BVDV感染细胞后BVDV正、负链RNA的定量分析结果,BVDV感染6~12 h呈下降趋势,可能是由复制过程中形成的dsRNA激活宿主细胞中抗病毒蛋白降解和病毒复制抑制作用造成的,但是在该阶段病毒RNA变化的原因需要进一步的验证。
4 结论建立了BVDV链特异性荧光定量RT-PCR方法——ss(+)RT-qPCR和ss(-)RT-qPCR,并利用该方法对BVDV在细胞内复制过程中正、负链RNA的动态变化过程进行描述,为研究宿主蛋白抗病毒机制、BVDV基因组RNA复制及调控机制等研究提供了研究手段。
| [1] | BROCK K V. The persistence of bovine viral diarrhea virus[J]. Biologicals, 2003, 31(2): 133–135. DOI: 10.1016/S1045-1056(03)00029-0 |
| [2] | RIDPATH J F, NEILL J D, VILCEK S, et al. Multiple outbreaks of severe acute BVDV in North America occurring between 1993 and 1995 linked to the same BVDV2 strain[J]. Vet Microbiol, 2006, 114(3-4): 196–204. DOI: 10.1016/j.vetmic.2005.11.059 |
| [3] | LACKNER T, MÜLLER A, PANKRAZ A, et al. Temporal modulation of an autoprotease is crucial for replication and pathogenicity of an RNA virus[J]. J Virol, 2004, 78(19): 10765–10775. DOI: 10.1128/JVI.78.19.10765-10775.2004 |
| [4] | FIRTH A E, ATKINS J F. A conserved predicted pseudoknot in the NS2A-encoding sequence of West Nile and Japanese encephalitis flaviviruses suggests NS1' may derive from ribosomal frameshifting[J]. Virol J, 2009, 6(1): 14. DOI: 10.1186/1743-422X-6-14 |
| [5] | SCHWEIZER M, PETERHANS E. Pestiviruses[J]. Annu Rev Anim Biosci, 2014, 2: 141–163. DOI: 10.1146/annurev-animal-022513-114209 |
| [6] | GOYAL S M, RIDPATH J F. Bovine viral diarrhea virus:diagnosis, management, and control[M]. Ames: Wiley Blackwell, 2005. |
| [7] | GINZINGER D G. Gene quantification using real-time quantitative PCR:an emerging technology hits the mainstream[J]. Exp Hematol, 2002, 30(6): 503–512. DOI: 10.1016/S0301-472X(02)00806-8 |
| [8] |
王洪梅, 贾春涛, 胡桂学, 等. 口蹄疫病毒链特异性荧光定量RT-PCR检测方法的建立[J]. 黑龙江畜牧兽医, 2015(11): 159–163.
WANG H M, JIA C T, HU G X, et al. Establishment of strand-specific reverse transcription quantitative PCR (ssRT-qPCR) assay for the detection of Foot-and-mouth disease virus[J]. Heilongjiang Animal Science and Veterinary Medicine, 2015(11): 159–163. (in Chinese) |
| [9] | VASHIST S, URENA L, GOODFELLOW I. Development of a strand specific real-time RT-qPCR assay for the detection and quantitation of murine norovirus RNA[J]. J Virol Methods, 2012, 184(1-2): 69–76. DOI: 10.1016/j.jviromet.2012.05.012 |
| [10] |
顾潮江, 李勇, 张玮莹, 等.口蹄疫病毒链特异性实时荧光定量RT-PCR技术的建立及应用[C]//第二届中国青年学者微生物遗传学学术研讨会论文集.武汉: 中国遗传学会微生物遗传学专业委员会, 2006: 1.
GU C J, LI Y, ZHANG W Y, et al. Establishment and application of strand-specific reverse transcription quantitative PCR assay for Foot-and-mouth disease virus[C]//Professional Committee of Microbial Genetics, Chinese Society of Genetics, the Second Chinese Young Scholars Symposium on Microbial Genetics. Wuhan: Professional Committee of Microbial Genetics, 2006: 1.(in Chinese) http://cpfd.cnki.com.cn/Article/CPFDTOTAL-ZGYL200610002110.htm |
| [11] | FENG L C, LINTULA S I, HO T H T, et al. Technique for strand-specific gene-expression analysis and monitoring of primer-independent cDNA synthesis in reverse transcription[J]. BioTechniques, 2012, 52(4): 263–270. |
| [12] | RICHARDSON J, MOLINA-CRUZ A, SALAZAR M I, et al. Quantitative analysis of dengue-2 virus RNA during the extrinsic incubation period in individual Aedes[J]. Amer J Trop Med Hyg, 2006, 74(1): 132–141. DOI: 10.4269/ajtmh.2006.74.132 |
| [13] | HAIST K, ZIEGLER C, BOTTEN J. Strand-specific quantitative reverse transcription-polymerase Chain reaction assay for measurement of arenavirus genomic and antigenomic RNAs[J]. PLoS One, 2015, 10(5): e0120043. DOI: 10.1371/journal.pone.0120043 |
| [14] | KOMURIAN-PRADEL F, PERRET M, DEIMAN B, et al. Strand specific quantitative real-time PCR to study replication of hepatitis C virus genome[J]. J Virol Methods, 2004, 116(1): 103–106. DOI: 10.1016/j.jviromet.2003.10.004 |
| [15] | LAGAYE S, BRUN S, GASTON J, et al. Anti-hepatitis C virus potency of a new autophagy inhibitor using human liver slices model[J]. World J Hepatol, 2016, 8(21): 902–914. DOI: 10.4254/wjh.v8.i21.902 |
| [16] |
刘存, 崔尚金. 牛病毒性腹泻病毒SYBR Green荧光定量PCR方法的建立及其应用[J]. 现代畜牧兽医, 2018(12): 9–13.
LIU C, CUI S J. Establishment and application of a SYBR Green real-time PCR assay for detection of bovine viral diarrhea virus[J]. Modern Journal of Animal Husbandry and Veterinary Medicine, 2018(12): 9–13. (in Chinese) |
| [17] | LESTER S N, LI K. Toll-like receptors in antiviral innate immunity[J]. J Mol Biol, 2014, 426(6): 1246–1264. DOI: 10.1016/j.jmb.2013.11.024 |
| [18] | KUMAR H, KAWAI T, AKIRA S. Pathogen recognition by the innate immune system[J]. Int Rev Immunol, 2011, 30(1): 16–34. |
| [19] | YONEYAMA M, ONOMOTO K, JOGI M, et al. Viral RNA detection by RIG-I-like receptors[J]. Curr Opin Immunol, 2015, 32: 48–53. DOI: 10.1016/j.coi.2014.12.012 |
| [20] | HOLL E K, ALLEN I C, MARTINEZ J. Holding the inflammatory system in check:TLRs and NLRs[J]. Mediators Inflamm, 2016, 2016: 8156816. |
| [21] | NAN Y C, NAN G X, ZHANG Y J. Interferon induction by RNA viruses and antagonism by viral pathogens[J]. Viruses, 2014, 6(12): 4999–5027. DOI: 10.3390/v6124999 |
| [22] |
杨超, 陈舜, 汪铭书, 等. OAS蛋白结构与抗病毒机制关系的研究进展[J]. 中国预防兽医学报, 2017, 39(8): 682–685.
YANG C, CHEN S, WANG M S, et al. Advances in the study of the relationship between the structure of OAS Protein and its Antiviral Mechanisms[J]. Chinese Journal of Preventive Veterinary Medicine, 2017, 39(8): 682–685. (in Chinese) |
| [23] | SEN G C, PETERS G A. Viral stress-inducible genes[J]. Adv Virus Res, 2007, 70: 233–263. DOI: 10.1016/S0065-3527(07)70006-4 |
| [24] | BERGMANN M, GARCIA-SASTRE A, CARNERO E, et al. Influenza Virus NS1 protein counteracts PKR-mediated inhibition of replication[J]. J Virol, 2000, 74(13): 6203–6206. DOI: 10.1128/JVI.74.13.6203-6206.2000 |
| [25] | DEB A, ZAMANIAN-DARYOUSH M, XU Z, et al. Protein kinase PKR is required for platelet-derived growth factor signaling of c-fos gene expression via Erks and Stat3[J]. EMBO J, 2001, 20(10): 2487–2496. DOI: 10.1093/emboj/20.10.2487 |
| [26] | SEN A, PRUIJSSERS A J, DERMODY T S, et al. The early interferon response to rotavirus is regulated by PKR and depends on MAVS/IPS-1, RIG-I, MDA-5, and IRF3[J]. J Virol, 2011, 85(8): 3717–3732. DOI: 10.1128/JVI.02634-10 |
| [27] | RAFIQUE S, IDREES M, ILYAS M, et al. Positional effect of phosphorylation sites 266 and 267 in the cytoplasmic domain of the E2 protein of hepatitis C virus 3a genotype:interferon Resistance analysis via Sequence Alignment[J]. Virol J, 2011, 8(1): 204. DOI: 10.1186/1743-422X-8-204 |
| [28] | SAWAI H, HIRANO A, MORI H, et al. Synthesis, characterization, and biological properties of 8-azido-and 8-amino-substituted 2', 5'-oligoadenylates[J]. J Med Chem, 2003, 46(23): 4926–4932. DOI: 10.1021/jm030035k |
| [29] | MORIN B, RABAH N, BORETTO-SOLER J, et al. High yield synthesis, purification and characterisation of the RNase L activators 5'-triphosphate 2'-5'-oligoadenylates[J]. Antiviral Res, 2010, 87(3): 345–352. DOI: 10.1016/j.antiviral.2010.06.003 |
| [30] | SHIBATA S, KAKUTA S, HAMADA K, et al. Cloning of a novel 2', 5'-oligoadenylate synthetase-like molecule, Oasl5 in mice[J]. Gene, 2001, 271(2): 261–271. DOI: 10.1016/S0378-1119(01)00508-X |



