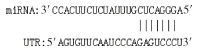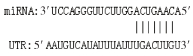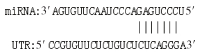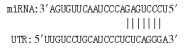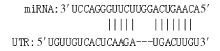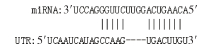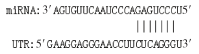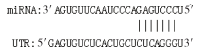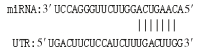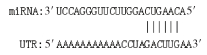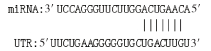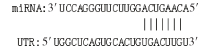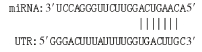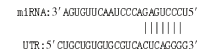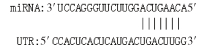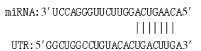睾丸是雄性动物繁殖最重要的生殖器官之一,形状为椭圆形,位于阴囊之内,左右各一,其生长发育受到睾丸发育相关基因的共同调控。睾丸主要分为曲细精管和睾丸间质两部分,曲细精管包含各级生精细胞和支持细胞等;睾丸间质由间质细胞、结缔组织等组成,是精子生成的重要场所[1]。自鼠类出生后睾丸发育高度依赖于间质细胞、支持细胞和生殖细胞的协调生长与分化[2]。因此,睾丸组织形态结构的完整性是雄性动物行使正常生殖功能的重要条件,探索睾丸的发育机制对繁殖性能具有积极意义。
microRNAs(miRNAs)是一类内源性的具有调控功能的非编码单链小RNA(长约20~25个核苷酸序列)。成熟的miRNAs与Ago (argonaute)等蛋白结合形成沉默复合体,以碱基互补配对的方式靶向作用于mRNA的3′UTR区域,从而将mRNA降解或抑制mRNA的翻译[3]。miRNA的作用涉及了所有的生命活动过程,包括生长、发育、细胞增殖和凋亡、物质代谢等,调控机体约60%以上的基因表达。其中一些miRNA在男性生殖细胞中表达丰富[4]。表明特定的miRNA具有调节生殖的功能[5]。例如,miR-19、miR-221/222、miR-122和miR-888已被证明在睾丸发育和精子形成中发挥重要作用[6]。也有研究表明,miR-125家族在调节卵母细胞和胚胎中合子基因组活化方面发挥着关键作用[7]。此外,miR-125b在牦牛、普通牛和犏牛睾丸组织中表达量较高,其调控靶基因涉及细胞凋亡机制可能与犏牛精子发生阻滞相关[8]。本课题组在前期研究miR-125b调控脂肪代谢机制的试验中发现,当miR-125b-2缺失时小鼠的繁殖性能出现障碍,产仔数显著下降,公鼠精子畸形率和不育率增加[9]。上述结果提示,miR-125b-2可能参与雄性生殖能力的调节。
转录组测序(RNA-Seq)可通过分析基因表达水平揭示转录性抑制因子,从而预测可能的分子机制。RNA-Seq数据库可以用于基因模型预测、新转录本识别和转录本表达的测定,具有更为灵敏和有效的特点[10]。迄今为止,RNA-Seq已被应用于大鼠附睾[11]、小鼠睾丸和附睾[12]转录组数据库的构建,并找到了很多与精子发生相关的基因和睾丸中特异性表达的基因。然而,目前尚未见miR-125b-2敲除后小鼠睾丸转录组的数据库以及相关报道。
本研究基于前期基础[7],通过对miR-125b-2敲除小鼠和野生型小鼠睾丸组织的转录组测序,获得两组睾丸的转录本数据库,通过生物信息学分析筛选miR-125b-2敲除前后睾丸组织中的差异表达基因,获得miR-125b-2可能调控的靶向功能基因及信号通路,为进一步研究miR-125b-2对睾丸精子发生的分子机制提供依据。
1 材料与方法 1.1 转基因小鼠的制备利用CRISPR/Cas9技术制备miR-125b-2基因敲除小鼠(由广州赛业生物科技有限公司完成),miR-125b-2 sgRNA序列为TCCCTGAGACCCTAACTTGTGA。在37只制备的小鼠中,利用PCR筛选出3只阳性(1母2公)小鼠。PCR引物:F:5′-ACATTACTGTAAGTTCTGATCTATA-3′,R:5′-GT-ACCGATTCTGAAGATTGTAT-3′,预期PCR产物大小为431 bp,通过测序鉴定小鼠的基因型。
1.2 试验动物选择及组织样品采集选择身体健康、无畸形、发育良好、食欲旺盛、有繁殖能力的性成熟野生型(WT)和miR-125b-2敲除型(KO)FVB/N小鼠各3只。颈部脱臼处死,解剖取睾丸,两组小鼠的睾丸(分别命名为WT组和KO组)放入高压灭菌后的冻存管,置于液氮中保存,用于总RNA的提取。
1.3 mRNA文库的构建及测序 1.3.1 总RNA提取用Trizol提取6只公鼠睾丸的总RNA,再用DNase Ⅰ去除总RNA中污染的基因组DNA。1%琼脂糖凝胶电泳检测其完整性,Nanodrop-1000核酸蛋白测定仪测定总RNA的浓度和纯度。转录组测序由杭州联川科技有限公司完成。
1.3.2 测序文库的建立将每组3个重复的样品进行混合得到总RNA,总RNA样品经质检合格后,采用磁珠(dT)法对mRNA进行富集,用片段化缓冲剂(fragment buffer)将mRNA分割成短片段,6碱基随机引物扩增短片段的mRNA,合成第1条cDNA链,再加入缓冲液、RNaseH、dNTPs和DNA polymerase Ⅰ合成第2条cDNA链。双链产物用AMPure XP beads纯化,用T4 DNA聚合酶和Klenow DNA聚合酶将DNA末端修复为平末端,加poly(A)尾,加接头,AMPureXP beads进行片段选择,最后进行PCR扩增,获得测序文库。文库质检合格后采用Illumina HiSeqTM4000进行测序。
1.3.3 测序结果质控分析采用Illumina配对端RNA-Seq方法,分别对WT和KO组转录组进行测序。首先,根据以下标准将低质量reads去掉,获得高质量的有效数据(valid data):1)去除带接头的reads;2)去除含有无法确定碱基信息的比例大于5%的reads;3)去除低质量reads(质量值Q≤10的碱基数占整个read的20%以上)。最终WT和KO组各检测到56 391 004和48 969 660个reads,检验率均高达99%以上。
1.3.4 RNA-Seq比对获得有效数据后,采用StringTie对valid data进行拼接以获取后续分析的参考序列。用Hisat对valid data进行参考基因组比对(http://ftp.ensembl.org/pub/release-88/gtf/mus_musculus/)。
1.3.5 转录丰度估计和差异表达检测对于比对结果采用FPKM(fragments per kilobase of exon model per million mapped reads)来测量每千碱基的片段在每百万片段的外显子映射,度量基因表达的丰度值,进而分析基因的表达水平。差异表达基因的筛选首先用FC (fold-change,倍数变化)和统计检测P值初选,然后用R语言的FDR(false discovery ratio)[13]对P值进行校正。差异表达基因(DEGs)的选择阈值是|log2(fold-change)|≥1,P<0.05。利用Gene Ontology数据库对DEGs进行GO分类,主要包括3类:生物学过程、分子功能和细胞组成。通过KEGG(kyoto encyclopedia of genes and genomes)找出差异基因显著富集的通路(pathway)。GO和KEGG检验均采用Benjamini Hochberg方法进行多重比对。最后利用实时定量PCR对RNA-Seq数据进行验证(该部分资料已发表[7]),基因差异表达水平均采用2-ΔΔCT方法[14]计算。
1.4 Targetscan和miRada预测靶关系通过软件Targetscan[15](5.0版本)和miRada [16](3.3a版本)一起预测可能与miR-125b-2存在直接靶关系的基因,根据Targetscan的阈值大于50和miRada的阈值小于-10为标准,最终获得可能存在靶向关系的候选基因。
1.5 数据处理与分析采用SPSS 17.0的单因素方差分析,并采用Duncan法进行检验,数据表示为“均数±标准差(x±s)”,*表示差异显著(P<0.05),**表示差异极显著(P<0.01)。
2 结果 2.1 小鼠基因型的鉴定剪取WT和KO小鼠的尾巴,提取DNA并通过引物特异性扩增,最后测序和比对。结果显示,KO组小鼠miR-125b-2基因成熟序列区缺失7 bp,如图 1所示。

|
图 1 通过Clustal X软件比对WT和KO组小鼠miR-125b-2序列 Fig. 1 The miR-125b-2 sequences comparison between WT and KO mice using Clustal X software |
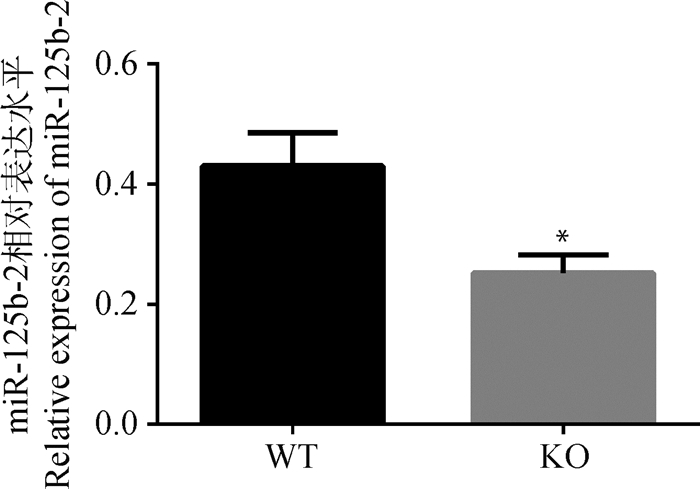
Stem-loop qPCR检测结果显示,与WT组相比,KO组睾丸组织中miR-125b-2的表达水平显著降低(P<0.05),如图 2所示。
|
图 2 WT和KO小鼠睾丸中miR-125b-2的表达水平(n=6) Fig. 2 The relative expression of miR-125b-2 in WT and KO mice testis (n=6) |
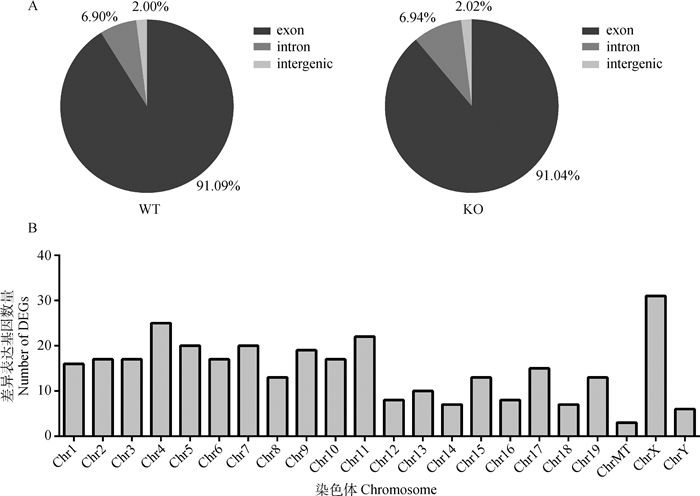
将RNA-seq测序结果与参考基因组的valid data进行比对,并根据参考基因组的区域信息分为exon(外显子)区、intron(内含子)区和intergenic(基因间隔区域)区。比对结果显示,WT和KO两组测序结果分别比对到exon、intron和intergenic区域的比例基本一致,且91%的reads比对到exon序列上(图 3A)。将筛选出的324个差异表达基因(DEGs)进行染色体定位分析,发现差异表达基因分布在Chr X染色体上最多,其次是Chr4和Chr11(图 3B)。
|
A.通过基因组比对获得的转录本分类;B.差异表达基因在染色体上的分布 A. Transcripts classification obtained by genomic alignment; B. Distribution of differentially expressed genes on chromosomes 图 3 差异表达基因在基因组上的分布 Fig. 3 Distribution of differentially expressed genes in genome |
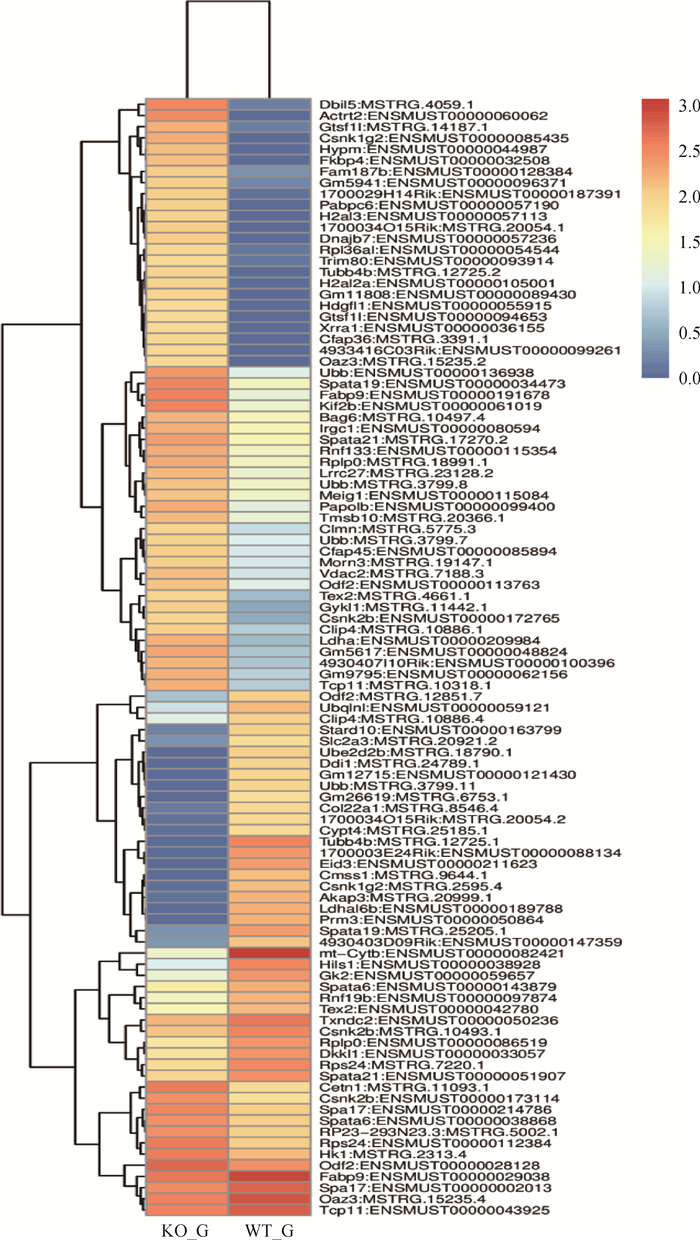
在|log2(fold-change)|≥1,P<0.05的选择标准下检验了32 667个基因,共获得324个DEGs(180个DEGs已注释),其中173个基因表达上调,151个基因表达下调,如图 4所示。
|
上图表示各个基因的变化趋势。红色表示上调表达水平,蓝色表示下调表达水平 The graph indicates the gene change tendency. Red color shows the up-regulation expression, while blue color shows the down-regulation expression 图 4 miR-125b-2敲除小鼠睾丸中差异表达基因的聚类热图 Fig. 4 Heatmap of differentially expressed genes in mouse testis with miR-125b-2 knockout |
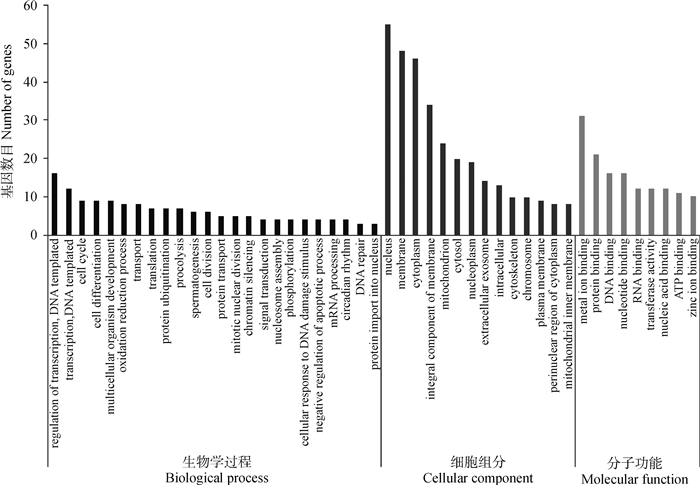
对180个已注释的DEGs进行GO功能分类,结果如图 5所示。110个DEGs富集在生物学过程(biological process),其中80个为显著富集(P<0.05),显著富集程度前5(P<1×10-5)的生物学过程分别为染色质沉默(chromatin silencing)、染色体浓缩(chromosome condensation)、精子染色质缩合(sperm chromatin condensation)、线粒体翻译的正调控(positive regulation of mitochondrial translation)和线粒体电子转运载体-细胞色素c到氧的电子传递(mitochondrial electron transport, cytochrome c to oxygen);144个DEGs富集在细胞组成(cellular component),其中有22个为显著富集(P<0.05),显著富集程度前4个(P<1×10-5)的细胞组成分别为核小体(nucleosome)、染色体(chromosome)、线粒体内膜预序列转位酶复合物(mitochondrial inner membrane presequence translocase complex)和F-actin蛋白复合物(F-actin capping protein complex);132个DEGs富集在分子功能(molecular function),其中有41个为显著富集(P<0.05),显著富集程度前3个(P<0.05)的分子功能分别为DNA聚合酶结合(DNA polymerase binding)、URM1激酶活性(URM1 activating enzyme activity)和核糖体的结构组成(structural constituent of ribosome)。
|
图 5 miR-125b-2敲除后小鼠睾丸中差异表达基因GO聚类分析 Fig. 5 Gene ontology classification of the DEGs in mice testis after miR-125b-2 knockout |
通过KEGG数据库中的Pathway富集分析来确定DEGs参与的主要生化代谢途径和信号转导途径。结果分析发现,KO组睾丸中差异表达的基因分布在74条通路中,其中差异表达基因富集的前10个KEGG通路中有7条信号通路达到显著水平(P<0.05),如表 1所示,它们依次是RNA转运、信使RNA监视通路、合硒化合物新陈代谢、真核生物的核糖体生成、酒精中毒、乙醛酸和二羧酸代谢和全身性红斑狼疮。
|
|
表 1 差异表达基因富集的前10个KEGG通路 Table 1 Top ten enriched KEGG pathways of DEGs |
针对睾丸转录组测序结果筛选KO组表达量显著上调的候选基因,通过Targetscan和miRada软件共同对所有测序结果进行miR-125b-2的靶基因预测,Targetscan软件在阈值大于50的条件下预测出6 257个靶基因,miRada在阈值小于-10的条件下预测出12 992个靶基因,两个软件预测的交集基因数一共有4 424个。Targetscan单独预测的打分值是TargetScan score,值越大越好;而miRada预测结果是Miranda energy,其绝对值越大越好。首先通过基因的显著性进行筛选,然后参考这两个值,绝对值越大,预测结果越可靠。在Targetscan和miRada设置的阈值分别为50和-10的原则下,最终筛选出19个符合条件的靶基因,如表 2所示。
|
|
表 2 miR-125b-2的靶基因预测 Table 2 Target genes prediction of miR-125b-2 |
睾丸作为精子发生的器官,其正常发育对动物的繁殖力以及种公畜的精液质量具有重要的意义。睾丸的发育受到生殖激素的影响,也受到各种分子机制的调控。精子发生是生殖细胞发育的一个复杂过程,睾丸中许多基因均参与了这一过程,任何基因表达的变化或调控的缺陷都会破坏精子的形成,导致男性不育[17]。而miRNAs通过将mRNA降解或抑制mRNA的翻译可实现对基因表达的调控,如miR-125b在牦牛、普通牛和犏牛睾丸组织中表达量均较高,可能参与犏牛精子发生阻滞的调节[9]。本课题组前期研究发现,miR-125b-2缺失引起小鼠繁殖性能下降,并降低小鼠的精子质量[7]。本研究通过制备miR-125b-2敲除小鼠,采用RNA-Seq方法检测了miR-125b-2敲除前后睾丸组织中基因表达水平的差异。结果显示,miR-125b-2敲除后基因表达水平受最大影响的前4个基因(Smcp、mt-Nd2、mt-Cytb和Hils1)都与线粒体功能和精子生成有关。线粒体是生理条件下发挥精子功能的关键细胞器[18],mt-Cytb的mtDNA拷贝数每增加一个单位,精子游动能力下降4%[19]。精子线粒体相关的富含半胱氨酸的蛋白质(Smcp)在生精细胞的减数分裂后期具有重要的调节作用[20]。NADH脱氢酶(ND2)的亚基2是由mt-ND2基因编码的,在控制线粒体活性氧的产生方面起着关键作用[21]。精子特异性连接组蛋白(HILS1)[22]在哺乳动物精子发生过程中,可能调控基因转录和染色质重塑。以上结果提示,miR-125b-2可能在线粒体相关基因的表达水平和精子发生调控中发挥着重要作用。
GO结果显示,被注释的180个DEGs的功能主要富集到5个生物学过程,分别为染色质沉默、染色体浓缩、精子染色质缩合、线粒体翻译的正调控和线粒体电子转运。染色质结构被用来作为雄性动物精子完整性和繁殖能力的检测指标[23]。据报道,精子染色质损伤与精子运动活力、存活率和精子畸形率有关,但不会影响其运动方式和顶体完整率[24]。精子染色质的完整性降低,精液的黏度会随之升高,精子活力也下降[25]。同时,KEGG分析结果显示,所有DEGs的生物学功能主要富集在3条信号通路上,分别为RNA转运、信使RNA监视通路和合硒化合物新陈代谢。研究表明,这几种信号通路对精子发生相关的泛素化酶作用和繁殖功能是至关重要的[26-27]。上述GO和KEGG分析结果均表明,miR-125b-2可能与精子生成的功能调节有关。
此外,在靶基因预测结果中发现,miR-125b-2直接靶向的3个基因(Papolb、Tssk和Slc22a14)与精子发生和不孕不育有很大的关系。睾丸特异性细胞质多聚(A)聚合酶(Papolb)通过其腺苷酸化来调节精子的形成[28]。睾丸特异性丝氨酸/苏氨酸激酶1(Tssk)和激酶2的缺失可使单倍体功能不全而导致男性不育[29],可能作为一个避孕的潜在靶点[30]。溶质载体22a14(Slc22a14)是精子发生相关跨膜蛋白,在雄性生殖细胞中高度表达,当Slc22a14缺失时公鼠精子运动能力下降,表现为高度不育[31],在弱精子症患者的精子中表现为低表达[32]。因此,结果提示miR-125b-2的敲除使精子发生相关基因Papolb、Tssk1和Slc22a14的表达水平显著上调,进而影响了小鼠的繁殖性能。
4 结论本研究利用转录组学筛选和分析了miR-125b-2敲除对小鼠睾丸发育相关基因及信号通路的影响,发现小鼠睾丸组织中324个DEGs可能与miR-125b-2的调控有关,这些DEGs多数都与精子发生和线粒体功能有关,并主要参与RNA转运、信使RNA监视和合硒化合物新陈代谢等通路,其中与精子发生相关基因Papolb、Tssk1和Slc22a14的表达量显著上调。这些结果为进一步研究miR-125b-2对小鼠繁殖性能的调节机制提供了良好的基础。
| [1] |
张逢, 陈指龙, 易思亮, 等. 细菌脂多糖急性暴露通过炎症反应诱导小鼠睾丸损伤[J]. 畜牧兽医学报, 2019, 50(5): 1099–1105.
ZHANG F, CHEN Z L, YI S L, et al. Acute exposure of bacterial lipopolysaccharide induces testicular injury in mice through inflammatory response[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(5): 1099–1105. (in Chinese) |
| [2] | PICUT C A, REMICK A K, DE RIJK E P C T, et al. Postnatal development of the testis in the rat:morphologic study and correlation of morphology to neuroendocrine parameters[J]. Toxicol Pathol, 2015, 43(3): 326–342. DOI: 10.1177/0192623314547279 |
| [3] | FABIAN M R, SONENBERG N, FILIPOWICZ W. Regulation of mRNA translation and stability by microRNAs[J]. Annu Rev Biochem, 2010, 79: 351–379. DOI: 10.1146/annurev-biochem-060308-103103 |
| [4] | HE Z P, KOKKINAKI M, PANT D, et al. Small RNA molecules in the regulation of spermatogenesis[J]. Reproduction, 2009, 137(6): 901–911. DOI: 10.1530/REP-08-0494 |
| [5] | ZHANG Q W, WANG Q, ZHANG Y, et al. Comprehensive analysis of microrna-messenger RNA from white yak testis reveals the differentially expressed molecules involved in development and reproduction[J]. Int J Mol Sci, 2018, 19(10): 3083. DOI: 10.3390/ijms19103083 |
| [6] | WU J W, BAO J Q, KIM M, et al. Two miRNA clusters, miR-34b/c and miR-449, are essential for normal brain development, motile ciliogenesis, and spermatogenesis[J]. Proc Natl Acad Sci U S A, 2014, 111(28): E2851–E2857. DOI: 10.1073/pnas.1407777111 |
| [7] | KIM K H, SEO Y M, KIM E Y, et al. The miR-125 family is an important regulator of the expression and maintenance of maternal effect genes during preimplantational embryo development[J]. Open Biol, 2016, 6(11): 160181. DOI: 10.1098/rsob.160181 |
| [8] |
廖珂.牦牛、普通牛和犏牛睾丸组织microRNA的比较研究[D].成都: 西南民族大学, 2016.
LIAO K.Comparative analysis of microRNA in yak, ordinary cattle and catttle-yak testis tissue[D]. Chengdu: Southwest Minzu University, 2016.(in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10656-1016776881.htm |
| [9] | LI L L, ZHU Y L, CHEN T, et al. miR-125b-2 knockout in testis is associated with targeting to the PAP gene, mitochondrial copy number, and impaired sperm quality[J]. Int J Mol Sci, 2019, 20(1): 148. DOI: 10.3390/ijms20010148 |
| [10] | MORTAZAVI A, WILLIAMS B A, MCCUE K, et al. Mapping and quantifying mammalian transcriptomes by RNA-Seq[J]. Nat Methods, 2008, 5(7): 621–628. DOI: 10.1038/nmeth.1226 |
| [11] | JELINSKY S A, TURNER T T, BANG H J, et al. The rat epididymal transcriptome:comparison of segmental gene expression in the rat and mouse epididymides[J]. Biol Reprod, 2007, 76(4): 561–570. DOI: 10.1095/biolreprod.106.057323 |
| [12] | JOHNSTON D S, JELINSKY S A, BANG H J, et al. The mouse epididymal transcriptome:transcriptional profiling of segmental gene expression in the epididymis[J]. Biol Reprod, 2005, 73(3): 404–413. DOI: 10.1095/biolreprod.105.039719 |
| [13] | BENJAMINI Y, HOCHBERG Y. Controlling the false discovery rate:a practical and powerful approach to multiple testing[J]. J Roy Stat Soc B, 1995, 57(1): 289–300. |
| [14] | LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the method[J]. Methods, 2001, 25(4): 402–408. DOI: 10.1006/meth.2001.1262 |
| [15] | NAM J W, RISSLAND O S, KOPPSTEIN D, et al. Global analyses of the effect of different cellular contexts on microRNA targeting[J]. Mol Cell, 2014, 53(6): 1031–1043. DOI: 10.1016/j.molcel.2014.02.013 |
| [16] | BETEL D, WILSON M, GABOW A, et al. The microRNA.org resource:targets and expression[J]. Nucleic Acids Res, 2008, 36(S1): D149–D153. |
| [17] | ABHARI A, ZARGHAMI N, SHAHNAZI V, et al. Significance of microRNA targeted estrogen receptor in male fertility[J]. Iran J Basic Med Sci, 2014, 17(2): 81–86. |
| [18] | PEÑA F J, RODRÍGUEZ MARTÍNEZ H, TAPIA J A, et al. Mitochondria in mammalian sperm physiology and pathology:a review[J]. Reprod Domest Anim, 2009, 44(2): 345–349. DOI: 10.1111/j.1439-0531.2008.01211.x |
| [19] | DARR C R, MORAES L E, CONNON R E, et al. The relationship between mitochondrial DNA copy number and stallion sperm function[J]. Theriogenology, 2017, 94: 94–99. DOI: 10.1016/j.theriogenology.2017.02.015 |
| [20] | CULLINANE D L, CHOWDHURY T A, KLEENE K C. Mechanisms of translational repression of the Smcp mRNA in round spermatids[J]. Reproduction, 2015, 149(1): 43–54. DOI: 10.1530/REP-14-0394 |
| [21] | ZHANG W W, HOU L L, WANG T, et al. The expression characteristics of mt-ND2 gene in chicken[J]. Mitochondrial DNA A DNA Mapp Seq Anal, 2016, 27(5): 3787–3792. |
| [22] | YAN W, MA L, BURNS K H, et al. HILS1 is a spermatid-specific linker histone H1-like protein implicated in chromatin remodeling during mammalian spermiogenesis[J]. Proc Natl Acad Sci U S A, 2003, 100(18): 10546–10551. DOI: 10.1073/pnas.1837812100 |
| [23] | LARSON K L, DEJONGE C J, BARNES A M, et al. Sperm chromatin structure assay parameters as predictors of failed pregnancy following assisted reproductive techniques[J]. Hum Reprod, 2000, 15(8): 1717–1722. DOI: 10.1093/humrep/15.8.1717 |
| [24] |
张宁, 张黎, 丁晓萍, 等. 精子染色质结构分析与精液参数的相关性研究[J]. 中华男科学, 2003, 9(3): 166–169.
ZHANG N, ZHANG L, DING X P, et al. Analysis of correlation between parameters of sperm chromatin structure assay and semen routine analysis[J]. National Journal of Andrology, 2003, 9(3): 166–169. DOI: 10.3969/j.issn.1009-3591.2003.03.002 (in Chinese) |
| [25] | GOPALKRISHNAN K, PADWAL V, BALAIAH D. Does seminal fluid viscosity influence sperm chromatin integrity?[J]. Arch Androl, 2000, 45(2): 99–103. DOI: 10.1080/014850100418783 |
| [26] |
郭曰帅.精子发生相关泛素化修饰的蛋白质组学研究[D].南京: 南京医科大学, 2015.
GUO Y S.Proteomics study on spermatogenesis related ubiquitination modification[D]. Nanjing: Nanjing Medical University, 2015.(in Chinese) |
| [27] |
林长光, 林金玉, 刘东霞, 等. 不同硒源对断奶仔猪生长性能、血清抗氧化能力和血浆硒含量的影响[J]. 畜牧兽医学报, 2013, 44(11): 1790–1796.
LIN C G, LIN J Y, LIU D X, et al. Effect of different selenium sources on growth performance, plasma selenium content and anti-oxidative capacity in weaned piglets[J]. Acta Veterinaria et Zootechnica Sinica, 2013, 44(11): 1790–1796. (in Chinese) |
| [28] | KASHIWABARA S I, TSURUTA S, OKADA K, et al. Adenylation by testis-specific cytoplasmic poly(A) polymerase, PAPOLB/TPAP, is essential for spermatogenesis[J]. J Reprod Dev, 2016, 62(6): 607–614. DOI: 10.1262/jrd.2016-116 |
| [29] | XU B F, HAO Z L, JHA K N, et al. Targeted deletion of Tssk1 and 2 causes male infertility due to haploinsufficiency[J]. Dev Biol, 2008, 319(2): 211–222. DOI: 10.1016/j.ydbio.2008.03.047 |
| [30] | XU B, HAO Z L, JHA K N, et al. Validation of a testis specific serine/threonine kinase[TSSK] family and the substrate of TSSK1 & 2, TSKS, as contraceptive targets[J]. Soc Reprod Fertil Suppl, 2007, 63: 87–101. |
| [31] | MARUYAMA S Y, ITO M, IKAMI Y, et al. A critical role of solute carrier 22a14 in sperm motility and male fertility in mice[J]. Sci Rep, 2016, 6: 36468. DOI: 10.1038/srep36468 |
| [32] |
侯方圆, 李玉山, 杨夕阳, 等. 溶质载体家族22成员14和精子相关抗原6在特发性弱精子症患者精子中的表达[J]. 中华男科学杂志, 2017, 23(8): 703–707.
HOU F Y, LI Y S, YANG X Y, et al. Expressions of SLC22A14 and SPAG6 proteins in the ejaculated sperm of idiopathic asthenozoospermia patients[J]. National Journal of Andrology, 2017, 23(8): 703–707. (in Chinese) |