蒙古马作为外蒙古国与我国内蒙古自治区的优秀品种,蕴含了宝贵的遗传资源。经过自然环境的长期适应,形成了耐粗饲、耐寒、强适应性和强抵抗力等优良特点,此外,还被用于肉食、产奶与骑乘[1]。由于蒙古马优秀的耐力,使赛马同摔跤、射箭并称为“蒙古族三竞技”[2]。但是至今,蒙古马与其它哺乳动物相比,优秀运动能力在基因水平的证据还很缺乏。通过基因组中正向选择基因的筛选分析,可以了解某物种在进化上特有的适应性。例如在蝙蝠中,参与修复DNA损伤的基因的正选择被视为物种对自由基活性氧损伤的响应,而飞行期间高代谢产生的自由基活性氧对DNA具有破坏作用[3]。在牦牛中,已经证实几种基因参与细胞对缺氧反应的适应性进化[4]。在纯血马中,一些主要负责脂肪酸氧化,增加胰岛素敏感性和肌肉力量的基因被认为与其运动表现相关[5]。
综上,作为珍贵遗传资源的蒙古马,本研究通过对其全基因组测序和生物信息学分析来揭示蒙古马蛋白编码基因的种类和数量,可以得到蒙古马运动相关的正向选择基因,并为竞技型家畜的育种提供理论基础。
1 材料与方法 1.1 材料本试验的血液样品采自于1匹内蒙古锡林郭勒盟正白旗的体型健硕、年龄为12岁的雌性蒙古马。用采血针采集其颈静脉血50 mL,置于预先加有抗凝剂的离心管中,立即放入液氮带回实验室,置于-80℃冰箱保存。
1.2 构建蒙古马DNA文库及其全基因组测序使用Puregene Tissue Core Kit A(Qiagen)对马全血基因组DNA进行抽提,根据全基因组鸟枪测序策略[6],使用Illumina文库构建试剂盒构建插入长度分别为170、500、800、2 000、5 000、10 000、20 000 bp的DNA测序文库。使用第二代高通量测序仪Illumina HiSeq 2000对各个文库进行双末端测序。
1.3 测序数据过滤和K-mer分析采用软件SOAPec_v2.01(http://sourceforge.net/projects/soapdenovo2/files/ErrorCorrection/SOAPec_v2.01.tar.gz/download)将测序得到的原始数据进行校正和过滤以减少测序错误对组装的影响。K-mer是指将reads分成包含k个碱基的字符串,采用SOAP de novo(http://soap.genomics.org.cn)中的K-merfreq软件包对K-mer进行分析,估计基因组的大小,选取的K值为17[7]。用全部小片段reads重新进行K-mer分析,17-mer的期望频数是44,考虑到深度低于10是低频的17-mer,以该值作为低频和高频K-mer的临界值进行纠错。对于有低频K-mer出现的reads,通过改变某些碱基可以使得整个reads上的K-mer都为高频,从而纠正测序导致的错误[7]。
1.4 蒙古马基因组组装和注释使用SOAP de novo(http://soap.genomics.org.cn)软件完成基因组组装,并对组装好的基因组数据进行GC含量统计。编码基因的结构预测是利用共线性关系进行的同源预测,根据蒙古马基因组与Ensembl-64 (http://asia.ensembl.org/index.html)中Homo sapiens和Equus caballus的共线性关系来进行同源预测,将得到的基因集定为核心基因集,运用GLEAN软件整合Homolog同源预测、De novo从头预测和cDNA/EST预测所得的基因构成Glean基因集,将Glean的基因集与核心基因集做cds相关的overlap比较,对于预测位置较一致的基因,用Glean基因来代替核心基因集中的基因以使该基因更加完整;同时向核心基因集中补充cDNA/EST预测结果所支持的其他基因,以得到最后的基因集(表 1),进而对得到的基因集的蛋白序列进行功能注释。
|
|
表 1 编码基因结构预测的基因集及预测方法 Table 1 Gene set of coding gene structure prediction and prediction method |
使用似然比检验(likelihood ratio tests,LRT)来筛选正选择基因(positive selected gene,PSG)[16]。非同义替换率(nonsynonymous substitution rate,Ka)与同义替换率(synonymous substitution rate,Ks)的比值(Ka/Ks) > 1时,认为此基因的进化过程受到了正向选择[17]。对特有基因和正向选择的蛋白编码基因使用GOseq[18]、UniProt(https://www.uniprot.org/)、KOBAS(2.0)[19]和Enrichr(http://amp.pharm.mssm.edu/Enrichr/)进行GO富集和KEGG通路富集分析,并使用Equus caballus基因组数据通过miRBase(http://www.mirbase.org/)对靶向结合mRNA中3′UTR区域的miRNA进行预测。
2 结果 2.1 蒙古马全基因组测序数据统计及K-mer分析通过测序获得原始数据281.4 Gb,经过滤后获得数据227.5 Gb,测序深度为75.83×,GC含量为41.3%,选取K=17,分析17-mer估算出该蒙古马基因组大小约为2.76 Gb。
2.2 蒙古马特有基因和正向选择基因的筛选通过基因功能注释得到该蒙古马的蛋白编码基因共19 475种。与负鼠、小鼠、人、纯血马、牛、狗和熊猫共7种哺乳动物全基因组数据比较,进一步获得了编码包括核酸酶敏感元件结合蛋白1、跨膜4结构域亚家族成员4A、Dachshund家族转录因子1、Dachshund家族转录因子2、成纤维细胞生长因子结合蛋白1、SLAM家族成员8、T淋巴细胞表面抗原Ly-9、类炭疽毒素受体和脂滴包被蛋白4的DACH2、SLAMF8、LY9、PLIN4等38种蒙古马特有基因(表 2),以及MAPK13、CCN2、ANO1、FSCN3、BTG1、TF、STK11等45种正向选择基因(图 1)。其中,马的5号和6号染色体被更多的正向选择基因所富集。
|
|
表 2 蒙古马基因组中的特有基因 Table 2 Specific genes in Mongolian horse genome |
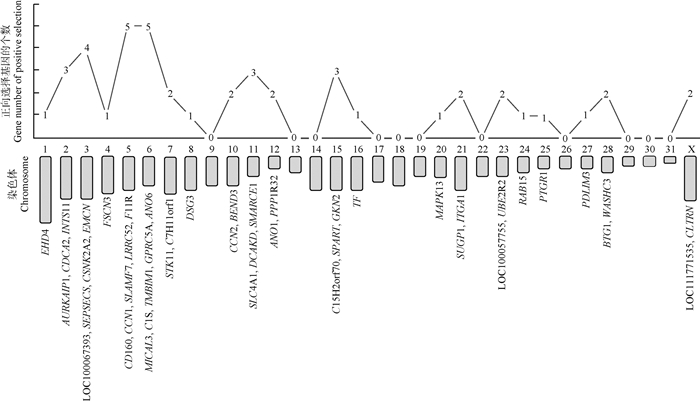
|
图 1 蒙古马正向选择基因的染色体分布 Fig. 1 The distribution of positive selective genes on chromosomes in Mongolian horse |
通过GO分析,包括编码9类蛋白的38种蒙古马特有基因被富集到了15个分子功能的条目,44个生物过程的条目,5个细胞组分的条目,其中,以GO条目的统计学显著性(P < 0.05)为划分基础,蒙古马潜在特异性地调控双链DNA结合(GO:0003690)、成纤维细胞生长因子结合(GO:0017134)、GTP酶结合(GO:0051020)、mRNA稳定化(GO:0048255)、蛋白质定位到细胞器(GO:0033365)等生物学功能和过程(图 2)。通过KEGG通路富集分析,38种特有基因被富集到了PPAR信号通路(PPAR signaling pathway)中促进脂肪细胞分化的脂滴包被蛋白Perilipin上。通过miRNA靶向结合蛋白编码基因mRNA的3′UTR种子区域的预测发现,特有基因mRNA中的DACH2、PLIN4、LOC100061331、LOC100061252等8种是eca-miR-450b-5p、eca-miR-15a、eca-miR-105、eca-miR-9000、eca-miR-9012等12种miRNAs的潜在靶基因(表 3)。
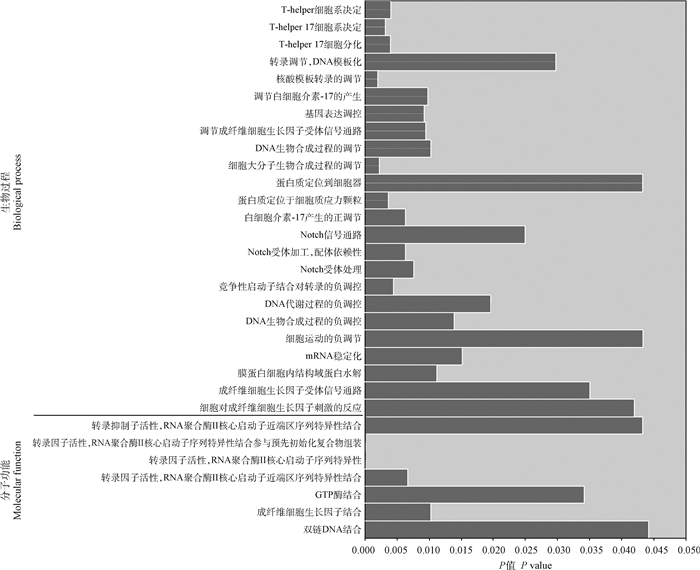
|
图 2 蒙古马特有基因的GO富集分析 Fig. 2 GO enrichment analysis of specific genes in Mongolian horse |
|
|
表 3 蒙古马特有基因mRNA对应的潜在miRNAs Table 3 The corresponding miRNAs targeting to mRNA of specific genes in Mongolian horse |
与负鼠、小鼠、人、纯血马、牛、狗和熊猫的全基因组数据比较,45种蒙古马正向选择蛋白编码基因经GO分析被富集到了92个分子功能的条目,340个生物过程的条目,44个细胞组分的条目,其中,以GO条目的统计学显著性(P < 0.05)为划分基础,蒙古马更倾向于调控锚蛋白结合(GO:0030506)、FAD绑定(GO:0071949)、亚铁结合(GO:0008198)、离子门控通道活性(GO:0022839)等分子功能(图 3),心血管系统发育(GO:0072358)、细胞离子稳态(GO:0006873)、成肌分化的正调节(GO:0045663)、调节血管重塑(GO:0060312)等生物过程(图 4)。通过KEGG通路富集分析,45种正向选择蛋白编码基因被富集到了FoxO信号通路、硒化合物代谢、紧密连接等68条通路上(图 5)。通过miRNA靶向结合蛋白编码基因mRNA的3′UTR种子区域的预测发现,正向选择蛋白编码基因mRNA中的MAPK13、UBE2R2、EHD4、AURKAIP1、SUGP1等19种是eca-miR-302c、eca-miR-8974、eca-miR-197、eca-miR-483、eca-miR-9077等26种miRNAs的潜在靶基因(表 4)。
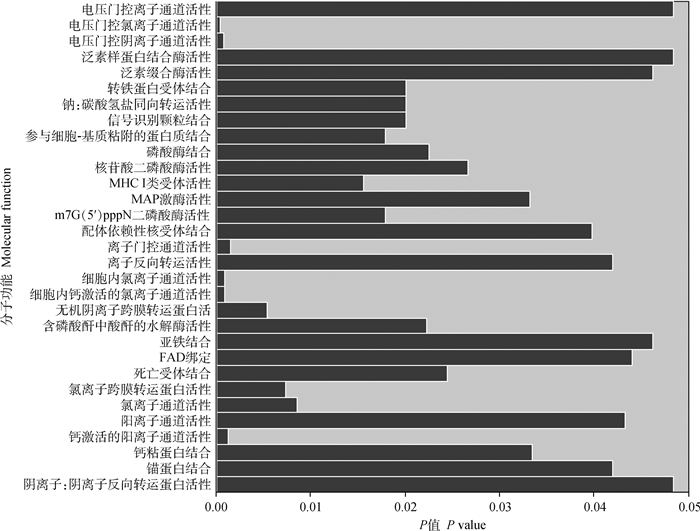
|
图 3 蒙古马正向选择的蛋白编码基因的GO分子功能富集分析 Fig. 3 GO molecular function enrichment analysis of protein-coding genes of positive selection in Mongolian horse |
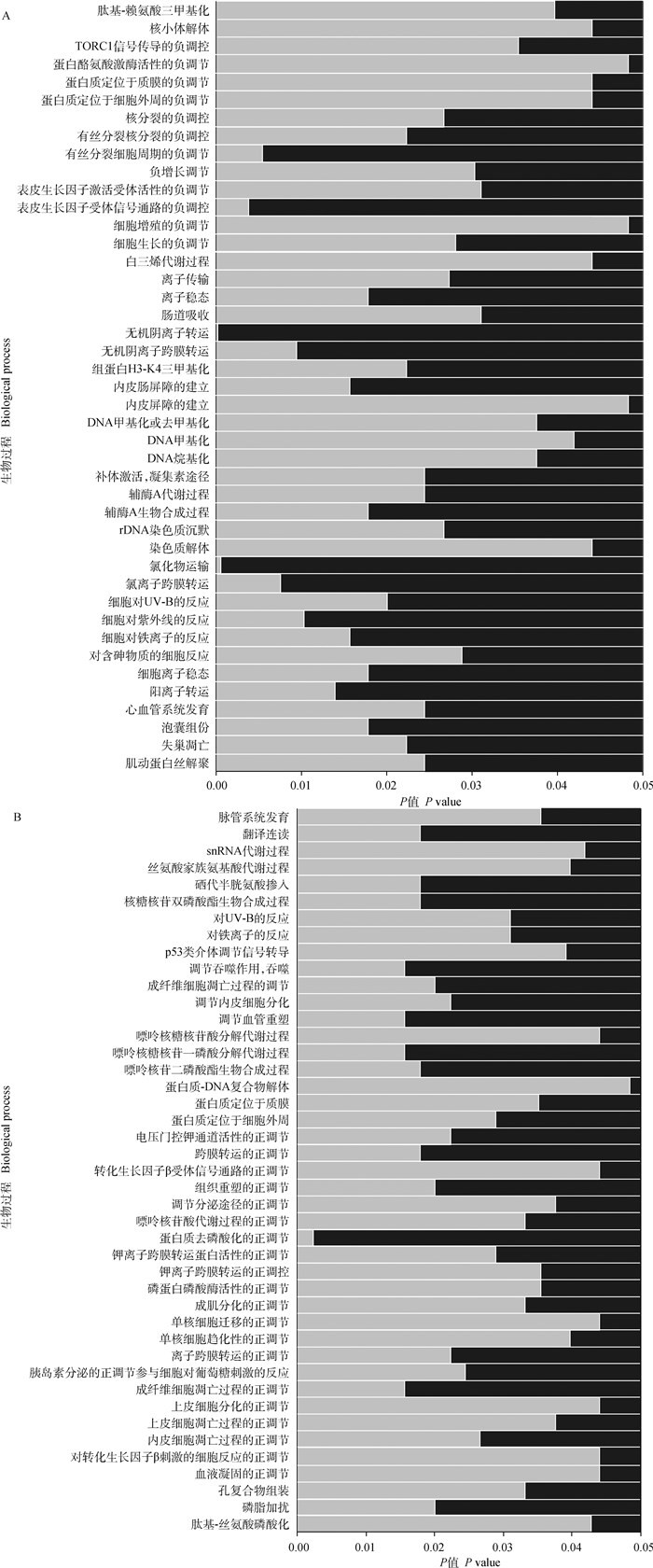
|
图 4 蒙古马正向选择的蛋白编码基因的GO生物过程富集分析 Fig. 4 GO biological process enrichment analysis of protein-coding genes of positive selection in Mongolian horse |
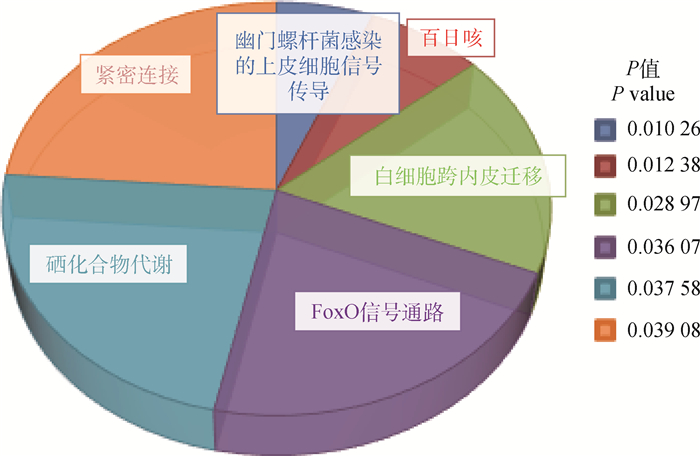
|
图 5 蒙古马正向选择的蛋白编码基因富集的KEGG通路 Fig. 5 Enriched KEGG pathways of protein-coding genes of positive selection in Mongolian horse |
|
|
表 4 蒙古马正向选择的蛋白编码基因mRNA对应的潜在miRNAs Table 4 The corresponding miRNAs targeting to mRNA of protein-coding genes of positive selection in Mongolian horse |
家畜的运动性状对于靶向竞技的遗传育种是十分重要的,但是蒙古马较其它哺乳动物而言,其与运动相关的基因种类、数量和功能还知之甚少。肌肉是在大多数动物中起到产生力和运动作用的软组织。它们主要负责维持和改变姿势、运动及内部器官功能等。通过对经高强度训练的耐力运动员测试发现,运动期间运动员较对照参与者有着更强的心肌收缩能力[20]。同时,慢性低强度运动可以减轻主动脉带状小型猪的心肌细胞收缩功能障碍[21]。较高的肌丝钙离子敏感性、增强的钙离子结合和pH调节能力则作为增加心肌收缩力的潜在机制[22]。此外,血管的生成可以促进肌肉骨骼组织修复[23],前十字韧带重建[24],运动介导的血管愈合和适应[25];改善和加速骨骼肌愈合[26],耐力运动的适应[27]。对大鼠的研究发现,肌肉经胰岛素刺激后葡萄糖的摄取增强,同时骨骼肌毛细血管化的增强与胰岛素敏感性的增强有关[28]。常规耐力运动下,细胞质内的线粒体可以提高骨骼肌氧化底物的能力,从而为肌肉工作产生ATP[29],并促进有氧代谢和运动能力[30]。在呼吸肌无力的呼吸衰竭中,呼吸肌功能受损会导致患者呼吸困难增加,运动能力下降[31]。以往的研究还表明,抗氧化基因的表达可以使大鼠更好的响应运动耐力,同时所有类型的运动都可以通过增加抗氧化能力和减少氧化应激来有效抑制骨骼肌萎缩[32-33]。在跟腱断裂患者肢体固定期间的辅助间歇性气动压缩可以通过上调Ⅰ型胶原合成来有效地增强早期愈合反应,而没有任何副作用[34]。
以上述研究为基础,正向选择的蛋白编码基因被进一步分类。MAPK13、CCN2、FSCN3、BTG1、TF、STK11、LOC100057755、EHD4、C1S、DSG3、MICAL3、PDLIM3、ITGA1、TMBIM1、SLC4A1、F11R、SPART作为蒙古马运动相关正向选择的蛋白编码基因,它们的功能被集中在肌动蛋白丝结合和肌动蛋白丝束组装(FSCN3)、成肌分化的正调节(BTG1)、肌动蛋白丝结构(TF、PDLIM3)、肌动蛋白结合(MICAL3)、肌肉的结构成分(PDLIM3)、肌动蛋白结构组织(F11R)、心脏发育(PDLIM3)、心肌收缩的正调节(CCN2)、肌肉收缩(ITGA1)、脉管系统发育(STK11)、钙离子结合(EHD4、C1S、DSG3)、调节细胞内pH(SLC4A1)、血管生成(CCN2)、血管生成的正调节(BTG1)、血管扩张(ITGA1)、正调节血管重塑(TMBIM1)、调节线粒体膜电位(SPART)、细胞对过氧化氢的反应(MAPK13)、细胞对DNA损伤刺激的反应(STK11)、氧化应激诱导的存活信号的关键组成部分(LOC100057755)、对缺氧的反应(CCN2)、组织稳态(STK11)、胶原生物合成过程的正调节(CCN2)。而eca-miR-302c、eca-miR-8974、eca-miR-9077、eca-miR-302b、eca-miR-326、eca-miR-190a、eca-miR-1468则通过与mRNA的3′UTR作用分别潜在靶向调控运动相关正向选择的蛋白编码基因MAPK13、LOC100057755、EHD4、MICAL3、TMBIM1和F11R等。
4 结论本试验通过全基因组测序和生物信息学分析,筛选出了与负鼠、小鼠、人、纯血马、牛、狗和熊猫共7种哺乳动物相比蒙古马特有基因38种,运动相关的正向选择的蛋白编码基因17种,这些运动相关的正向选择基因主要涉及了肌动蛋白丝结构、心肌收缩的正调节、钙离子结合、调节细胞内pH、血管生成、胶原生物合成过程的正调节等生物学功能。此外,还鉴别出了与12种特有基因、7种运动正向选择蛋白编码基因存在潜在调控关系的miRNAs。这些基因进一步为靶向竞技型家畜的分子育种提供了良好的理论基础。
| [1] |
芒来, 杨虹. 蒙古马遗传多样性研究进展[J]. 遗传, 2008, 30(3): 269–276.
DUGARJAVIIN M, YANG H. Progress in the study of genetic diversity of Mongolian horse[J]. Hereditas (Beijing), 2008, 30(3): 269–276. DOI: 10.3321/j.issn:0253-9772.2008.03.004 (in Chinese) |
| [2] |
栾桂芝, 郭聪. 蒙古族三竞技与现代体育三竞技的比较及其发展[J]. 大连民族大学学报, 2007, 9(6): 140–141.
LUAN G Z, GUO C. Mongolian three-game competition and modern three-game competition[J]. Journal of Dalian Nationalities University, 2007, 9(6): 140–141. DOI: 10.3969/j.issn.1009-315X.2007.06.044 (in Chinese) |
| [3] | ZHANG G J, COWLED C, SHI Z L, et al. Comparative analysis of bat genomes provides insight into the evolution of flight and immunity[J]. Science, 2013, 339(6118): 456–460. DOI: 10.1126/science.1230835 |
| [4] | QIU Q, ZHANG G J, MA T, et al. The yak genome and adaptation to life at high altitude[J]. Nat Genet, 2012, 44(8): 946–949. DOI: 10.1038/ng.2343 |
| [5] | GU J J, ORR N, PARK S D, et al. A genome scan for positive selection in thoroughbred horses[J]. PLoS One, 2009, 4(6). |
| [6] | LI R Q, FAN W, TIAN G, et al. The sequence and de novo assembly of the giant panda genome[J]. Nature, 2010, 463(7279): 311–317. DOI: 10.1038/nature08696 |
| [7] | LI R Q, ZHU H M, RUAN J, et al. De novo assembly of human genomes with massively parallel short read sequencing[J]. Genome Res, 2010, 20(2): 265–272. |
| [8] | HARRIS B.LASTZ: release1.02.00, built January 12, 2010[EB/OL].http://www.bx.psu.edu/miller_lab/dist/README.lastz-1.02.00/README.lastz-1.02.00a.html. |
| [9] | GERTZ E M, YU Y K, AGARWALA R, et al. Composition-based statistics and translated nucleotide searches: improving the TBLASTN module of BLAST[J]. BMC Biol, 2006, 4: 41. DOI: 10.1186/1741-7007-4-41 |
| [10] | SHE R, CHU J S C, WANG K, et al. GenBlastA:enabling BLAST to identify homologous gene sequences[J]. Genome Res, 2009, 19(1): 143–149. |
| [11] | BIRNEY E, CLAMP M, DURBIN R. GeneWise and genomewise[J]. Genome Res, 2004, 14(5): 988–995. DOI: 10.1101/gr.1865504 |
| [12] | STANKE M, KELLER O, GUNDUZ I, et al. AUGUSTUS:ab initio prediction of alternative transcripts[J]. Nucleic Acids Res, 2006, 34(Web Server issue): W435–W439. |
| [13] | SAFDAR M.GenScan: bioinformatics software for structure prediction and analysis of -gene[EB/OL]. (2010-07-21). https://www.biotecharticles.com/Bioinformatics-Article/GenScan-Bioinformatics-Software-For-Structure-Prediction-and-Analysis-of-Gene-284.html. |
| [14] | KENT W J. BLAT-the BLAST-like alignment tool[J]. Genome Res, 2002, 12(4): 656–664. DOI: 10.1101/gr.229202 |
| [15] | HAAS B J, SALZBERG S L, ZHU W, et al. Automated eukaryotic gene structure annotation using EVidenceModeler and the program to assemble spliced alignments[J]. Genome Biol, 2008, 9(1): R7. |
| [16] | LI H Q, STEPHAN W. Maximum-likelihood methods for detecting recent positive selection and localizing the selected site in the genome[J]. Genetics, 2005, 171(1): 377–384. |
| [17] | YANG Z H, BIELAWSKI J P. Statistical methods for detecting molecular adaptation[J]. Trends Ecol Evol, 2000, 15(12): 496–503. DOI: 10.1016/S0169-5347(00)01994-7 |
| [18] | YOUNG M D, WAKEFIELD M J, SMYTH G K, et al. Gene ontology analysis for RNA-seq:accounting for selection bias[J]. Genome Biol, 2010, 11(2): R14. DOI: 10.1186/gb-2010-11-2-r14 |
| [19] | MAO X Z, CAI T, OLYARCHUK J G, et al. Automated genome annotation and pathway identification using the KEGG Orthology (KO) as a controlled vocabulary[J]. Bioinformatics, 2005, 21(19): 3787–3793. DOI: 10.1093/bioinformatics/bti430 |
| [20] | CARLSSON L, LIND B, LAAKSONEN M S, et al. Enhanced systolic myocardial function in elite endurance athletes during combined arm-and-leg exercise[J]. Eur J Appl Physiol, 2011, 111(6): 905–913. DOI: 10.1007/s00421-010-1712-3 |
| [21] | HIEMSTRA J A, VETETO A B, LAMBERT M D, et al. Chronic low-intensity exercise attenuates cardiomyocyte contractile dysfunction and impaired adrenergic responsiveness in aortic-banded mini-swine[J]. J Appl Physiol (1985), 2018, 124(4): 1034–1044. DOI: 10.1152/japplphysiol.00840.2017 |
| [22] | WISLØFF U, LOENNECHEN J P, FALCK G, et al. Increased contractility and calcium sensitivity in cardiac myocytes isolated from endurance trained rats[J]. Cardiovasc Res, 2001, 50(3): 495–508. DOI: 10.1016/S0008-6363(01)00210-3 |
| [23] | HUARD J. Stem cells, blood vessels, and angiogenesis as major determinants for musculoskeletal tissue repair[J]. J Orthop Res, 2019, 37(6): 1212–1220. DOI: 10.1002/jor.24058 |
| [24] | LI H, LI J Y, JIANG J, et al. An osteogenesis/angiogenesis-stimulation artificial ligament for anterior cruciate ligament reconstruction[J]. Acta Biomater, 2017, 54: 399–410. DOI: 10.1016/j.actbio.2017.03.014 |
| [25] | WILHELM E N, GONZÁLEZ-ALONSO J, PARRIS C, et al. Exercise intensity modulates the appearance of circulating microvesicles with proangiogenic potential upon endothelial cells[J]. Am J Physiol Heart Circ Physiol, 2016, 311(5): H1297–H1310. DOI: 10.1152/ajpheart.00516.2016 |
| [26] | OTA S, UEHARA K, NOZAKI M, et al. Intramuscular transplantation of muscle-derived stem cells accelerates skeletal muscle healing after contusion injury via enhancement of angiogenesis[J]. Am J Sports Med, 2011, 39(9): 1912–1922. DOI: 10.1177/0363546511415239 |
| [27] | IVERSEN N, KRUSTRUP P, RASMUSSEN H N, et al. Mitochondrial biogenesis and angiogenesis in skeletal muscle of the elderly[J]. Exp Gerontol, 2011, 46(8): 670–678. |
| [28] | AKERSTROM T, LAUB L, VEDEL K, et al. Increased skeletal muscle capillarization enhances insulin sensitivity[J]. Am J Physiol Endocrinol Metab, 2014, 307(12): E1105–E1116. DOI: 10.1152/ajpendo.00020.2014 |
| [29] | BOOTH F W, RUEGSEGGER G N, TOEDEBUSCH R G, et al. Endurance exercise and the regulation of skeletal muscle metabolism[J]. Prog Mol Biol Transl Sci, 2015, 135: 129–151. DOI: 10.1016/bs.pmbts.2015.07.016 |
| [30] | PARK J Y, WANG P Y, MATSUMOTO T, et al. p53 improves aerobic exercise capacity and augments skeletal muscle mitochondrial DNA content[J]. Circ Res, 2009, 105(7): 705–712. DOI: 10.1161/CIRCRESAHA.109.205310 |
| [31] | SU C M, WANG I C, LIU S C, et al. Hypoxia induced mitogenic factor (HIMF) triggers angiogenesis by increasing interleukin-18 production in myoblasts[J]. Sci Rep, 2017, 7(1): 7393. DOI: 10.1038/s41598-017-07952-9 |
| [32] | KUMAR R, NEGI P S, SINGH B, et al. Cordyceps sinensis promotes exercise endurance capacity of rats by activating skeletal muscle metabolic regulators[J]. J Ethnopharmacol, 2011, 136(1): 260–266. DOI: 10.1016/j.jep.2011.04.040 |
| [33] | CAI M X, WANG Q A, LIU Z W, et al. Effects of different types of exercise on skeletal muscle atrophy, antioxidant capacity and growth factors expression following myocardial infarction[J]. Life Sci, 2018, 213: 40–49. DOI: 10.1016/j.lfs.2018.10.015 |
| [34] | ALIM M A, DOMEIJ-ARVERUD E, NILSSON G, et al. Achilles tendon rupture healing is enhanced by intermittent pneumatic compression upregulating collagen type Ⅰ synthesis[J]. Knee Surg Sports Traumatol Arthrosc, 2018, 26(7): 2021–2029. DOI: 10.1007/s00167-017-4621-8 |



