2. 甘肃农业大学动物医学院, 兰州 730070
2. College of Veterinary Medicine, Gansu Agricultural University, Lanzhou 730070, China
疯草(locoweed)是豆科棘豆属和黄芪属有毒植物的统称,广泛分布于亚洲、北美洲、南美洲和大洋洲等地区。由于疯草毒性灾害在世界范围内对草原畜牧业危害最为严重,现已被列入世界性“生态经济病”之一[1]。天然草地疯草的不断蔓延与扩散,引致疯草灾害多发和频发,大批牲畜中毒、死亡。目前,中国西部天然草地动物因疯草中毒死亡所造成的经济损失仍在持续剧增[2]。
现已确证引起疯草中毒的主要毒性物质是吲哚里西啶生物碱苦马豆素[3-5]。研究表明,疯草内普遍存在能够产生苦马豆素的内生真菌Alternaria Section Undifilum oxytropis[6-9],余永涛等[10]最新研究表明从直立黄芪中共分离到43株内生真菌,确定有13株菌丝中含有苦马豆素,进一步证实了该结果。国内外学者已从与疯草的毒性关系、在疯草中的传播机制和苦马豆素合成机制等多个方面有力地证实,Alternaria Section Undifilum oxytropis普遍存在于疯草植物组织内部,而且与疯草中苦马豆素的合成紧密相关[11-15]。有学者推测Alternaria Section Undifilum oxytropis中苦马豆素的合成代谢通路可能与豆类丝核菌、金龟子绿僵菌相似[16-17]。学者们对Alternaria Section Undifilum oxytropis进行了全基因测序,研究结果表明Alternaria Section Undifilum oxytropis全基因为70.05 Mb,通过分子生物学手段分析推测其中有164个基因位点所对应的氨基酸或酶可能参与苦马豆素的合成途径,并进一步假设存在有P6C途径和P2C途径[18-19],但上述结果仍需要进一步的研究进行验证确认。
但是目前关于Alternaria Section Undifilum oxytropis合成苦马豆素的代谢通路仍然没有阐释清楚。因此,明确Alternaria Section Undifilum oxytropis合成苦马豆素的代谢通路,结合基因敲除、基因沉默等技术手段,阻断或抑制苦马豆素合成相关基因的表达,有望为人工控制内生真菌合成苦马豆素,降低和消除疯草的毒性提供新的途径,从根本上解决动物疯草中毒病奠定基础,可以达到脱毒育种改良和抗肿瘤[20-21]、抗病毒[22]等开发药用价值利用的双赢。
利用化学诱变剂诱导菌株发生突变,筛选培养获得目标突变株,近年来亦有很多的研究报道,如Shirnalli等[23]利用N-methyl-N′-nitro-N-nitrosoguanidine诱变,获得了2株高生物量、高藻蓝蛋白和蛋白质含量的钝顶螺旋藻突变株。白晓南[24]对变异黄芪内生真菌Alternaria Section Undifilum oxytropis通过去除细胞壁,制备成原生质体后,用不同浓度的亚硝基胍诱导进行诱变选育,获得8株产SW差异显著菌株。Sandesh等[25]利用紫外线、EMS和MNNG对雨生红球藻进行诱变,并利用除草剂草铵膦进行突变株的筛选,获得的突变株总类胡萝卜素和虾青素含量提高了23%~59%,从转录水平上分析发现类胡萝卜素合成途径相关基因的表达均明显高于野生型。另研究表明,化学诱变具有成本低、使用方便、诱变作用专一性强等特点,其中亚硝基胍因其诱变效果稳定良好,被认为是化学诱变首选诱变剂。
近些年来随着高通量测序技术的出现以及生物信息技术的发展为疯草内生真菌基因组及SW合成通路的研究带来了新的思路。本研究通过化学诱变剂对原始菌株进行诱变,并利用高通量测序技术对SW产量变化较大的D4突变菌株及原始菌株进行基因组从头测序,通过差异基因及相关关键酶的注释,对比分析,结合KEGG通路,对疯草内生真菌合成SW通路进行了预测分析,为进一步阐明疯草内生真菌产苦马豆素的生物合成机制奠定了研究基础。
1 材料与方法 1.1 试验菌株Alternaria Section Undifilum oxytropis(编号:CICC2493)购自中国工业微生物菌株保藏中心,经复苏后-80 ℃保存。
1.2 主要试剂与仪器亚硝基胍购自上海叶源生物科技公司;真菌基因组DNA提取试剂盒、DL1000 marker、EDTA购自索莱宝公司;DynabeadsTMMyOneTMStreptavidin C1 AMPure XP A63880购自美国贝克曼库尔特有限公司;PGMTM Controls 200Kit、E-GelTM 50 bp DNA ladder、lon XpressTM Barcode Adapters 1-16 kit、Ion PGMTM Calibration Standard、Ion shearTMPlus Reagents Kit、Ion Plus Fragment Library Kit、Ion torrent PGMTM lnstall solutions测序所需的试剂盒均购自Thermo Scientific公司;centrifuge 5424R eppenderf高速离心机(德国Eppendorf公司);PCR扩增仪2700(美国AB公司);荧光定量PCR Life Tech(applied biosystems)购自赛默飞公司;超微量分光光度计Bio Spectrometer eppendorf(德国Eppendorf公司);Ion torrent PGMTM测序平台(美国Applied Biosystems);沃特浦实验室专用超纯水机(WP-UP-WF-30)购自四川沃特尔水处理设备有限公司;数控超声波清洗器(KD-300DE)购自昆山市超声仪器有限公司。
1.3 试验方法 1.3.1 内生真菌Alternaria Section Undifilum oxytropis亚硝基胍诱变取0.5 mg·mL-1亚硝基胍2、4、6、8、10、12 μL,加1 mL的磷酸盐缓冲液(pH=7.0),依次加入0.5 mL孢子悬液,28 ℃ 120 r·min-1恒温培养箱中反应5、10、15、20、25、30 min,反应完成后立即在4 ℃条件下10 000 r·min-1离心10 min,弃上清,加无菌水离心清洗,重复5次,最后一次离心完成后弃上清,加入0.5 mL磷酸盐缓冲液,振荡均匀后涂于PDA培养基上置于恒温箱中24 d,记录再生菌落数,每个试验组设5个平行, 计算致死率:
| $ \begin{array}{l} 致死率/\% = \\ \frac{{空白对照组菌落-诱变处理组菌落}}{{空白对照组菌落}} \times 100\% 。\end{array} $ |
选取经亚硝基胍诱变处理,生长良好的菌株分别接种于PDA培养基上,28 ℃,120 r·min-1,培养24 d,收集菌丝及发酵液,利用α-甘露糖苷酶活性分析法检测菌丝中SW含量,并将SW含量变化差异较大的菌株在改良察氏液体培养基中连续传代5代,每一代培养24 d,结束后检测发酵液中SW含量,检测、评价突变菌株产SW稳定性。
1.3.3 内生真菌基因组DNA提取分别使用玻璃珠和液氮破壁法,反复研磨真菌菌丝发白为止,加入玻璃珠,高速振荡30 min,55 ℃水浴蛋白酶K消化时间分别为30、45、60 min,水浴完成后,按索莱宝真菌DNA基因组提取试剂盒(Fungi Genomic DNA Extraction Kit)操作说明书分别对筛选获得的D4突变株和原始菌株进行DNA基因组提取。将提取获得的D4突变株和原始菌株的DNA,经0.8%的琼脂糖凝胶电泳,观察、选取条带亮且无弥散条带的DNA,采用超微量紫外分光光度计检测DNA的OD值。
1.3.4 DNA测序文库的构建 1.3.4.1DNA纯化、打断、再纯化:选用贝克曼公司DynabeadsTMMyOneTMStreptavidin C1 AMPure XP(REF-A63880)做纯化处理,本试验应用Ion ShearTMPlus Reagents Kit试剂盒操作说明先配制50 μL的反应体系(DNA 25 μL,10×Ligase Buffer 5 μL,去离子水10 μL,Ion ShearTM plus enzyme Mix Ⅱ 2 μL,Ion ShearTM plus stup buffer 5 μL)。
1.3.4.2Barcode标签添加:参考使用Ion XpressTM Barcode Adapters Kit试剂盒,将上述反应体系原始菌株、D4突变株分别按说明添加Barcode标签,原始菌株1号Barcode标签,D4突变株2号Barcode标签,按100 μL的反应体系(DNA 25 μL,10×Ligase Buffer 5 μL,Ion P1 Adapter 2 μL,Ion XpressTM Barcode X 2 μL,Dntp Mix 2 μL,Unlease-free water 49 μL,DNA Ligase 2 μL,Nick Repair polymerase 4 μL),在0.2 mL PCR管中进行连接,PCR的反应条件为25 ℃ 15 min、72 ℃ 5 min、4 ℃ 30 min。待反应完成后重复以上DNA纯化步骤。
1.3.4.3文库的扩增与凝胶电泳检测、DNA片段纯化:应用Ion Plus Fragment Library Kit试剂盒,取2个PCR管依次100 μL PlatiunmTM PCR Supermix Hight Fidelity、25 μL文库、5 μL引物(Library Amplification Primer Mix),扩增反应条件:先95 ℃变性5 min,然后95 ℃变性15 s,58 ℃退火15 s,70 ℃延伸60 s,共8个循环,待扩增反应完成,加6×DNA Loading Buffer在0.2%的琼脂凝胶糖、1×TEB电泳液、电压90 V条件下电泳40 min,电泳完成后在凝胶成像仪下观察电泳条带。
1.3.5 荧光定量PCR文库质量参考使用Ion Library TaqManTM定量试剂盒确定文库的浓度,检测文库的质量按照试剂盒说明(REF-4468802)进行操作,将文库稀释100倍配制反应体系。标准曲线的制作,参照Ion Library TaqManTM荧光定量试剂盒PCR手册检测文库质量。试验采用的荧光定量反应条件为99 ℃ 2 min、99 ℃ 15 s、60 ℃, 4 min 20个循环,保持10 min、荧光收集,并建立标准曲线计算文库的质量。
1.3.6 带模板的ISPS扩增与回收将2个DNA文库分别稀释为100 pmol·L-1,并等量混合。Ion PGMTM Hi-QTM OT2 Reagents,将反应所需的酶放置室温,Ion PGMTM Hi-QTM Reagent Mix涡旋30 s、1 000 r·min-1离心2 s,室温溶解后放置2~8 ℃保持备用,Ion PGMTM Hi-QTM Enzyme Mix 1 000 r·min-1离心2 s后置于冰上备用,Ion PGMTM Hi-QTM ISPS放置于室温备用。
1.3.7 富集带模板的ISPS准确配制1 mol·L-1 NaOH 40 μL加TweenTM solution 280 μL混匀过滤,将DynabeadsTM MyOneTM streptavidin C1 beads 2 s涡旋30 s,离心DynabeadsTM MyOneTM streptavidin C1 beads 2 s,用移液枪吹打至beads中黑色芯块散开后转移13 μL至新的EP管中,放置于磁力架上3 min弃上清,再向管中加入130 μL wash solution涡旋30 s,离心2 s。然后按照Ion oneTuchTMES说明进行操作,富集带模板ISPS。
1.3.8 基因组的测序首先对仪器进行清洗、氯洗、初始化,然后进行以下操作:将富集后的含有模板ISPS加入含有5 μL的control Ion SpherTM Particles的PCR管中,15 500 r·min-1离心2 min,弃上清,确定引物在冰上解冻后,离心5 s,向ISPS中加入12 μL测序引物,反复吹打混匀,在PCR仪上按92 ℃ 15 min、37 ℃ 2 min。将扩增好的ISPS加入3 μL Ion PGMTM Hi-QTM View sequencing polymerase,室温孵育5 min,加至Lon 318芯片,上机测序分析。
1.3.9 数据统计分析利用SPSS19.0统计学软件对亚硝基胍诱变筛选获得突变株与原始菌株的SW含量进行差异显著性分析(P < 0.05)。并利用BLAST、GO软件对突变株和原始菌株进行基因差异分析。
2 结果 2.1 亚硝基胍诱变随亚硝基胍诱变处理时间、作用剂量的增加,致死率也不断升高(图 1)。当浓度为0.5 mg·mL-1的亚硝基胍6 μL,与菌液作用5 min时,致死率达到58.3%,同样作用时间下,亚硝基胍12 μL时,致死率达到100%。

|
图 1 不同剂量和作用时间下亚硝基胍对诱变内生真菌的致死率 Figure 1 Mortality rate of endogenic fungi induced by nitrosoguanidine under different doses and time of action |
优选半数致死率附近的剂量及反应时间作为最佳诱变条件的原则,选取亚硝基胍作用剂量2 μL、作用时间15 min,亚硝基胍作用剂量6 μL、作用时间5 min,2组各3株诱变菌株选取菌落形态、长势良好的内生菌株6株(编号为D1、D2、D3、D4、D5、D6),并将上述6株内生真菌接种于改良察氏液体培养基中发酵培养,每代培养24 d,连续传代5代,经α-甘露糖苷酶活性分析法检测菌丝及发酵液中SW含量(表 1),D1和D4诱变菌株产SW含量较其他4株稳定(图 2)。
|
|
表 1 诱变菌株产SW含量变化检测(n=5) Table 1 The content changes of swainsonine in mutant strains(n=5) |
|
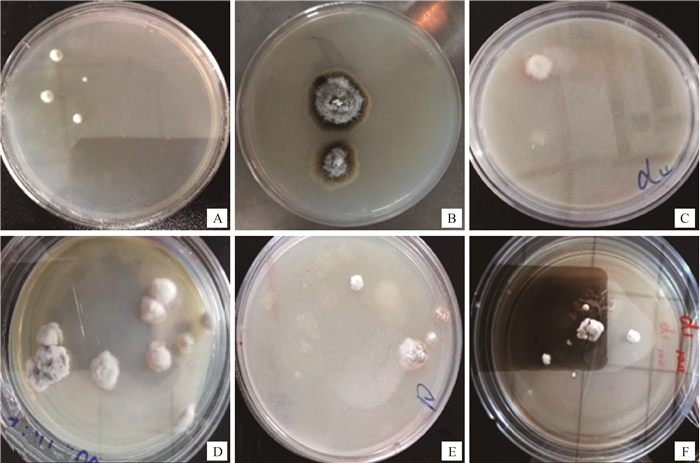
A、B为原始菌株生长菌落形态; C为诱变后D4突变株培养菌落形态;D为D4突变株传代培养后的菌落形态;E为诱变后D1突变株培养菌落形态;F为D1突变株传代培养后的菌落形态 A and B were the colony morphology of the original strain; C wasthe colony morphology of D4 mutant strain after mutagenesis; D was the colony morphology of D4 mutant after subculture; E was the colony morphology of D1 mutant strain; F was the colony morphology of D1 mutant after subculture 图 2 诱变前后突变菌株菌落的形态 Figure 2 Morphology of mutant colony before and after mutagenesis |
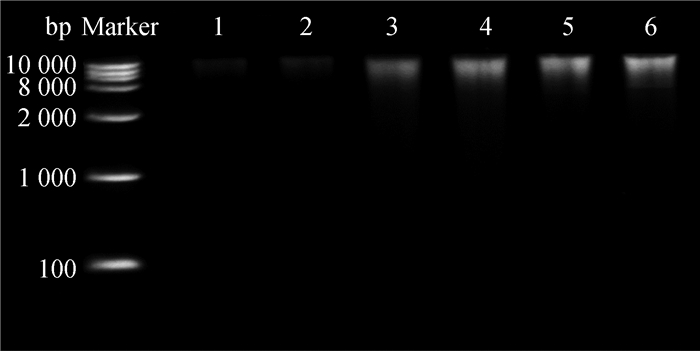
利用琼脂糖凝胶电泳检测提取真菌DNA(图 3)。结果表明,原始菌株和D4突变株经玻璃珠破壁1 h,蛋白酶消化45 min后电泳条带没有弥散现象,而其余的条带均有明显的条带弥散现象,可以用于下一步试验。
|
1、2分别为原始菌株和D4突变株玻璃珠破壁1 h、蛋白酶消化45 min后提取;3、4分别为原始菌株和D4突变株玻璃珠破壁30 min、蛋白酶消化30 min后提取;5、6分别为原始菌株和D4突变株液氮研磨后玻璃珠破壁30 min、蛋白酶消化45 min后提取 1 and 2 were extracted from the original strain and the D4 mutant of the glass beads 1 hour and the proteinase digested 45 min, respectively; 3 and 4 were extracted from the original and D4 mutant glass beads, 30 min, and protease digested 30 min, respectively; 5 and 6 were the original strain and the D4 mutant, after lapping the liquid nitrogen of the glass beads, and the digestion of the protease after the digestion of 45 min 图 3 原始菌株和D4突变株DNA提取结果 Figure 3 DNA extraction results |
利用贝克曼C1磁珠纯化后,得到的DNA再通过超微量分光光度计在260 nm下测定3次(表 2),结果表明,DNA质量良好可以满足基因测序的要求。
|
|
表 2 DNA OD值检测结果 Table 2 Result of DNA OD value |
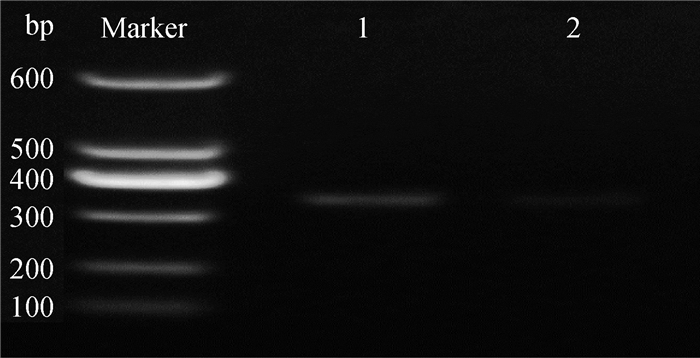
将基因文库的反应体系通过扩增后,用2%的琼脂糖凝胶电泳,结果如图 4,说明原始菌株与D4突变菌株DNA已被酶切打断为长度在300~400 bp之间的片段,且纯化后无其他杂带,可以进行下一步试验。
|
1.D4突变株DNA酶切结果;2.原始菌株DNA酶切结果 1. DNA enzyme digestion of D4 mutant strain; 2. DNA enzyme digestion of the original strain 图 4 D4突变株与原始菌株DNA酶切结果 Figure 4 Enzyme cutting results of D4 mutant and original strain DNA |
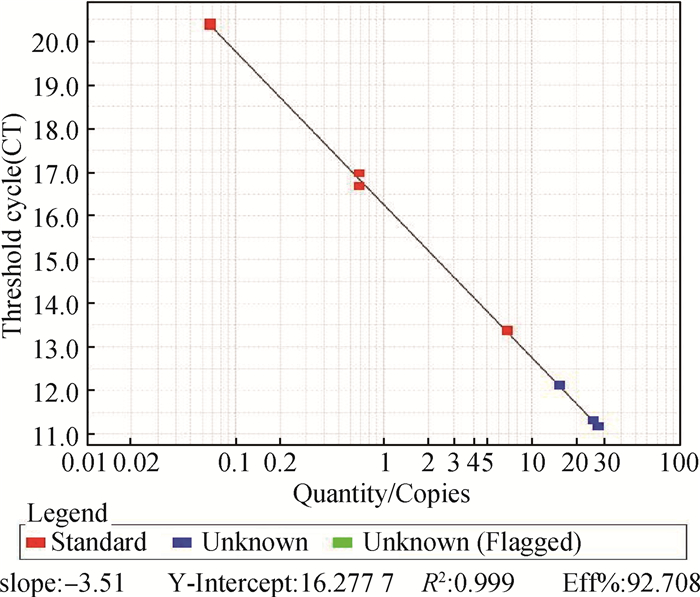
采用倍比稀释法建立标准曲线,荧光定量PCR检测结果显示R2=0.999,标准曲线线性关系良好(图 5)。扩增效率Eff%达到92.708,循环数标准差都控制在1个Cycle以内,故本标准曲线作为文库的定量计算是可行的。根据荧光定量PCR反应结果,标准对照品检测结果良好,重复检验结果稳定,根据文库ISPs1的浓度为15.291文库ISPs 2为27.017,表明基因文库质量良好(表 3)。
|
图 5 荧光定量PCR检测基因文库标准曲线 Figure 5 The standard curve of gene library by fluorescence quantitative PCR detection |
|
|
表 3 荧光定量PCR检测基因文库 Table 3 Detection of gene library by fluorescence quantitative PCR |
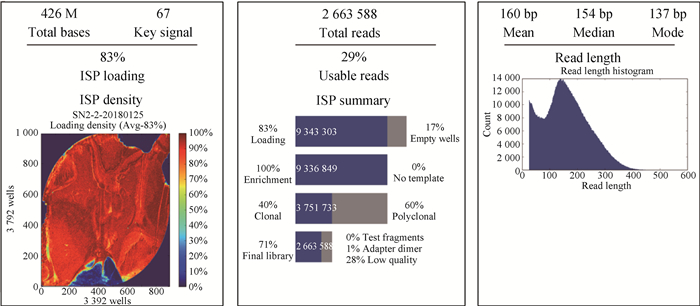
构建的测序文库中有效区域为83%(图 6)。原始数据为426 Mb, 获取2 663 588条Reads,所得片段大部分集中在137~160 bp,平均读取长度为154 bp。
|
图 6 基因文库测序结果 Figure 6 Sequencing results of gene library |
将基因测序原始数据首先用SPAdes Genome Assembler软件进行优化,去掉多余的低质量基因碎片,整合成一个fasta文件,并利用MIRA软件优化去掉重复序列,优化数据参数,整合、剪切、对连接位点识别连接。
2.9 测序长度评估利用MIRA软件程序对测序DNA进行拼接,结果如表 4所示,两株菌的Contig为12、13,原始菌株N50=23 120,覆盖深度73,D4突变株N50=15 451,覆盖深度74.63。
|
|
表 4 DNA拼接结果参数 Table 4 Parameters of DNA splicing result |
D4突变株得到序列共19条(通过MIRA拼接后的片段数),其中最短片段为2 281 bp,最长片段为37 408 bp, 原始菌株共计21条片段,最短片段为1 506 bp, 最长片段为37 408 bp。将其基因片段序列进行BLAST对比分析,结果表明D4突变株、原始菌株基因序列与Alternaria oxytropis SW生物合成基因簇全序列(ID: KY365741.1)相似度达99%,与slafractonia leguminicola swnk(Swnk)基因完整的CDS(ID:KY365746.1)相似度80%,说明D4突变株和原始菌株均包含有与SW生物合成基因簇和SwnK基因[26]。
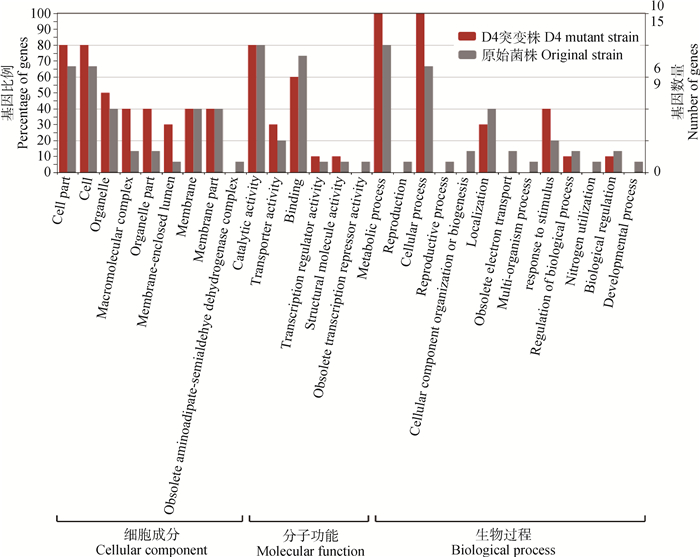
2.11 GO分析利用同源性分析在GO数据库中搜索得到的注释结果如下:分子功能、细胞成分和生物过程。在细胞组分中,“cell”和“cell part”是前两类,原始菌株和D4突变株分别匹配了812和659个基因。在生物过程中原始菌株和D4突变株分别匹配了“reproductive process” 101和0个基因、“cellular process” 725和1 015个基因,分子功能“binding”原始菌株和D4突变株分别匹配了761和609个基因,“metabolic process”代谢过程中,原始菌株和D4突变株分别匹配了812和1 015个基因(图 7)。
|
图 7 原始菌株与D4突变株GO功能注释基因分布及分类 Figure 7 Distribution and classification of functional annotation genes between original and D4 mutant strain by GO |
由于D4突变株、原始菌株均与Alternaria oxytropis具有高度基因相似性,利用BLAST对比分析,结合GO分析,结合Alternaria oxytropis中SW生物合成关键基因和关键酶,注释到SW生物合成的关键基因SwnK、SwnH2、SwnH1、SwnR和关键酶pyrroline-5-carboxylate reductase、formate/glycerate dehydrogenase catalytic、saccharopine reductase-like protein等。虽然原始菌株也注释到关键酶putative amino acid transporter,但突变菌株D4没有注释到putative amino acid transporter, 结合突变菌株D4产SW含量下降,推测putative amino acid transporter对内生真菌合成苦马豆素至关重要(见表 5、图 8、图 9)。
|
|
表 5 原始菌株与D4突变菌株注释到的SW合成的关键基因和关键酶 Table 5 Several key genes and key enzymes of SW synthesis were annotated to the original strain and D4 mutant strain by GO |
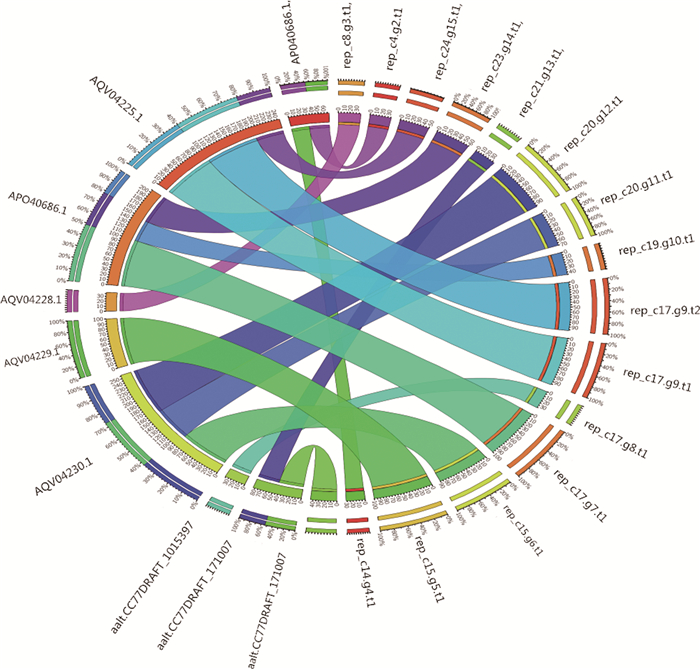
|
图 8 原始菌株Alternaria Section Undifilum oxytropis基因组关键蛋白注释结果 Figure 8 Annotated results of several key proteins of the original strain Alternaria Section Undifilum oxytropis genome |
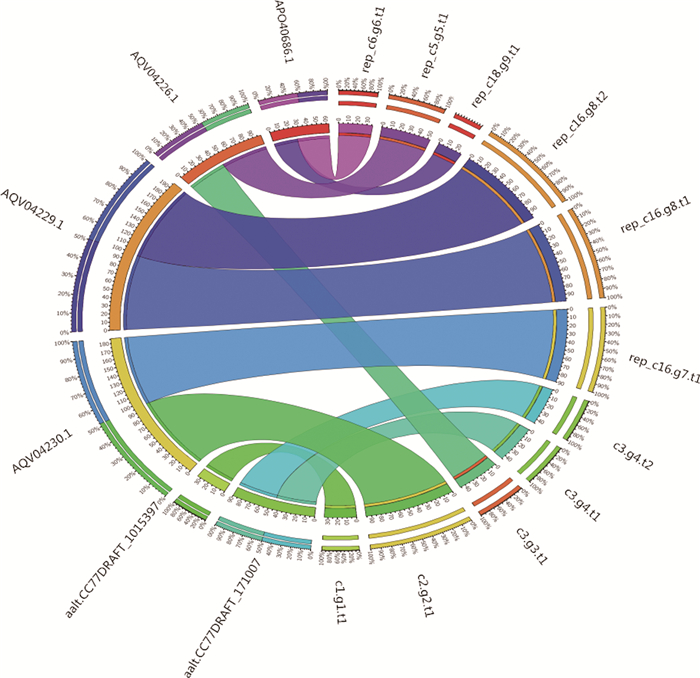
|
图 9 D4突变菌株基因组关键蛋白注释结果 Figure 9 Annotated results of several key proteins of D4 mutant strain |
由于原始菌株和D4突变株均与Alternaria oxytropis中SW生物合成主要基因、mycotoxins slaframine中SwnK的完整基因高度相似,充分证实了SwnK基因在D4突变株和原始菌株中对SW生物合成的重要性,参考Cook等[26]关于功能基因位置关系的研究,结合BLAST结果,对疯草内生真菌Alternaria Section Undifilum oxytropis中的相关基因结构及位置关系进行了预测(图 10)。
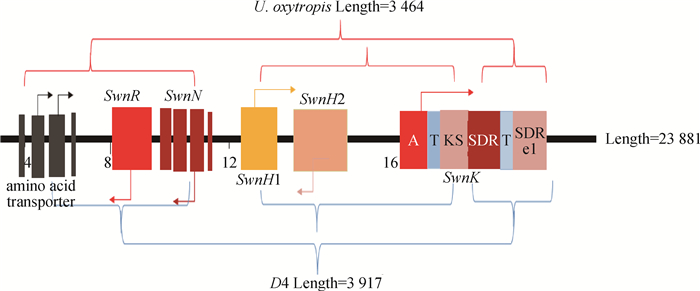
|
A代表一种具有腺苷酰化的多功能蛋白质;T代表磷酸泛酰巯基乙胺结合蛋白;KS代表b-ketoacyl合成酶;AT代表酰基转移酶;SDR代表转移酶;SDR e1代表硫酯还原酶 A represents a multifunctional protein withadenosylated; T represents phosphoacid sulfhydryl ethylamine binding protein; KS represents b-ketoacyl synthase; AT represents acyl transferase; SDR represents transferase; SDR e1 represents thioester reductase 图 10 SwnK基因在原始菌株与D4突变株中的基因结构及位置关系预测 Figure 10 Prediction of SwnK gene structure and location relationship between original strains and D4 mutant strain |
参考Cook等[26]和Lu等[18]对可能的SW生物合成路线预测结果,并基于以上试验结果,对Alternaria Section Undifilum oxytropis生物合成SW通路进行了预测,其通路应该是:SW生物合成的前体物质L-Lysine, 首先在酵母氨酸脱氢酶(APO40686.1)的作用下生成saccharopine, 再进一步在酵母氨酸脱氢酶的作用下生成L-2-aminoadipate-6-semialdehyde,在L-saccharopine oxidase酶的催化下生成5′-piperideine-6-L-carboxylate, 再经过pyrroline-5-carboxylate reductase的催化生成pipecolic acid, pipecolic acid在amino和SwnK酶的协同催化下,并在ATP参与和在Malonyl-CoA和SwnK、SwnR、SwnH的催化下生成中间产物1-Hydroxyin dolizidine, 最终经过SwnR、SwnH、NAD(P)H的催化生物合成SW(图 11)。
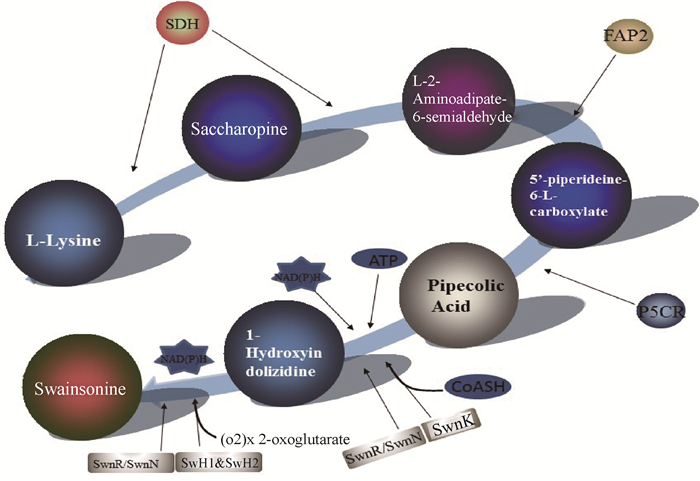
|
图 11 Alternaria Section Undifilum oxytropis产SW生物合成通路预测 Figure 11 Prediction of produced SW biosynthesis pathway in Alternaria Section Undifilum oxytropis |
参与苦马豆素合成和代谢的相关基因的功能验证,可以为在分子水平上阐释内生真菌中苦马豆素合成和代谢的调控机制提供依据。Mukherjee等[27]通过构建了一套Alternaria Section Undifilum oxytropis原生质体转化系统,筛选苦马豆素合成相关基因,确证已知的ITS、β、tublin、gpd和mtSUU等序列与苦马豆素合成无关。Mukherjee等[2 8]将酵母氨酸还原酶基因导入到转化系统中,通过基因干扰技术抑制了Alternaria Section Undifilum oxytropis中酵母氨酸还原酶基因的表达,导致该真菌中赖氨酸和酵母氨酸的消耗量增加,而苦马豆素及哌可酸的产量显著提升,从分子水平上进一步佐证了Alternaria Section Undifilum oxytropis中苦马豆素的合成路径可能与豆类丝核菌相似的假设,确定酵母氨酸还原酶基因在疯草内生真菌合成苦马豆素的过程中发挥着重要调控作用。近年来,李海利等[29-30]对棘豆属和黄芪属疯草的Alternaria Section Undifilum oxytropis蛋白质组进行了系统研究,利用二维蛋白质电泳、一维固相等电聚胶电泳与质谱联用等技术,共从棘豆属和黄芪属疯草的Alternaria Section Undifilum oxytropis中分离并鉴定出101种蛋白质,初步确定酵母氨酸还原酶、哌可酸氧化酶以及一些其他的可能与SW合成相关的蛋白酶及其结构,进一步确定各种蛋白酶在SW合成中的调控作用。
疯草内生真菌是一类有菌丝的真菌,在DNA提取过程中如何利用更好的方法破壁且对所提取的真菌DNA或者RNA基因链的完整性不造成影响至关重要。尽管真菌基因组的提取方法有很多,但由于提取过程中多糖和其他杂质均不宜去除,对后续的试验结果可能会造成影响及误差。本试验通过利用C1磁珠纯化法将对所提取的DNA大片段进行吸附纯化,从而达到了有效除杂和去除EDTA的目的。
高通量测序是目前应用较广的一种测序手段,其原理是预先将目的基因组打碎成小片段随后加特定的接头,通过扩增反应后加入到含有各种测序所需酶的油滴中,这种以油和微珠以及DNA构成的系统只含有一个DNA片段,再通过扩增、富集后高通量测序。本试验研究目的之一是通过检测诱变前、后菌株的基因组差异,寻找诱变菌株基因的位点变化,并结合分子生物学软件综合分析,研究诱变前、后SW生物合成过程中的关键基因和关键酶的变化。虽然二代测序时PGM测序片段较短,但由于其测序耗时短、测序精准,因此利用二代测序技术在本研究中具有更好的优势。
利用BLAST基因对比分析表明,Alternaria Section Undifilum oxytropis原始株、D4突变株均与Cook等[26]Alternaria oxytropis 的SW生物合成基因簇以及mycotoxins slaframine Swnk完整基因高度相似, 进一步证实了Swnk基因在D4突变株和原始菌株中对SW生物合成的重要性,结合基因位置结构、功能注释等,预测了疯草内生真菌中的基因结构、位置关系以及Alternaria Section Undifilum oxytropis生物合成SW通路,这为后续的Alternaria Section Undifilum oxytropis合成SW机制的阐明从分子水平上提供理论依据。
4 结论通过对Alternaria Section Undifilum oxytropis菌株进行化学诱变筛选、高通量测序,并利用生物信息学软件对诱变前、后菌株关键基因进行位置、功能注释分析,预测了Alternaria Section Undifilum oxytropis生物合成SW通路,为揭示Alternaria Section Undifilum oxytropis生物合成SW机制做出了新的探索尝试。
| [1] |
周启武, 赵宝玉, 路浩, 等. 中国西部天然草地疯草生态及动物疯草中毒研究与防控现状[J]. 中国农业科学, 2013, 46(6): 1280–1296.
ZHOU Q W, ZHAO B Y, LU H, et al. The research and control situation of ecology and animal poisoning of locoweed in western natural grassland of China[J]. Scientia Agricultura Sinica, 2013, 46(6): 1280–1296. DOI: 10.3864/j.issn.0578-1752.2013.06.023 (in Chinese) |
| [2] |
赵宝玉, 刘忠艳, 万学攀, 等. 中国西部草地毒草危害及治理对策[J]. 中国农业科学, 2008, 41(10): 3094–3103.
ZHAO B Y, LIU Z Y, WAN X P, et al. Damage and control of poisonous-weeds in Chinese western grassland[J]. Scientia Agricultura Sinica, 2008, 41(10): 3094–3103. DOI: 10.3864/j.issn.0578-1752.2008.10.024 (in Chinese) |
| [3] | COOK D, RALPHS M H, WELCH K D, et al. Locoweed poisoning in livestock[J]. Soc Range Manage, 2009, 31(1): 16–21. |
| [4] |
黄鑫, 梁剑平, 高旭东, 等. 苦马豆素的来源、药理作用及检测方法研究进展[J]. 畜牧兽医学报, 2016, 47(6): 1075–1085.
HUANG X, LIANG J P, GAO X D, et al. Research advance on sources, pharmacological effects and detection methods of swainsonine[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(6): 1075–1085. (in Chinese) |
| [5] | COOK D, GARDNER D R, MARTINEZ A, et al. Screening for swainsonine among South American Astragalus species[J]. Toxicon, 2017, 139: 54–57. DOI: 10.1016/j.toxicon.2017.09.014 |
| [6] | WOUDENBERG J H C, GROENEWALD J Z, BINDER M, et al. Alternaria redefined[J]. Stud Mycol, 2013, 75: 171–212. DOI: 10.3114/sim0015 |
| [7] | BRAUN K, ROMERO J, LIDDELL C, et al. Production of swainsonine by fungal endophytes of locoweed[J]. Mycol Res, 2003, 107(8): 980–988. DOI: 10.1017/S095375620300813X |
| [8] | LU H, QUAN H Y, ZHOU Q W, et al. Endogenous fungi isolated from three locoweed species from rangeland in western China[J]. Afr J Microbiol Res, 2017, 11(5): 155–170. DOI: 10.5897/AJMR |
| [9] |
卢围, 路浩, 赵宝玉, 等. 小花棘豆产苦马豆素内生真菌的筛选与鉴定[J]. 畜牧兽医学报, 2011, 42(3): 429–436.
LU W, LU H, ZHAO B Y, et al. Screening and identification of swainsonsine-producing endophytic fungi from Oxytropis. flabra[J]. Acta Veterinaria et Zootechnica Sinica, 2011, 42(3): 429–436. (in Chinese) |
| [10] |
余永涛, 李金荣, 赵清梅, 等. 直立黄芪中产苦马豆素真菌的分离与鉴定[J]. 畜牧兽医学报, 2018, 49(8): 1770–1780.
YU Y T, LI J R, ZHAO Q M, et al. Isolation and identification of swainsonine-producing fungi from Astragalus adsurgens pall[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(8): 1770–1780. (in Chinese) |
| [11] | WU C C, HAN T S, LU H, et al. The toxicology mechanism of endophytic fungus and swainsonine in locoweed[J]. Environ Toxicol Pharmacol, 2016, 47: 38–46. DOI: 10.1016/j.etap.2016.08.018 |
| [12] |
路浩, 李国中, 杨晓雯, 等. 甘肃棘豆内生真菌种群多样性[J]. 中国兽医学报, 2014, 34(7): 1094–1099.
LU H, LI G Z, YANG X W, et al. Diversity of endophytic fungi from Oxytropis kansuensis[J]. Chinese Journal of Veterinary Science, 2014, 34(7): 1094–1099. (in Chinese) |
| [13] |
薛瑞旭, 权海云, 任祯慧, 等. 疯草内生真菌Undifilum oxytropis次级代谢产物分析[J]. 动物医学进展, 2016, 37(7): 64–70.
XUE R X, QUAN H Y, REN Z H, et al. Analysis of secondary metabolites of Undifilum Oxytropis from locoweeds[J]. Progress in Veterinary Medicine, 2016, 37(7): 64–70. DOI: 10.3969/j.issn.1007-5038.2016.07.013 (in Chinese) |
| [14] |
周启武, 于龙凤, 路浩, 等. 小花棘豆和变异黄芪内生真菌显微分布及定量检测[J]. 微生物学报, 2014, 54(5): 572–581.
ZHOU Q W, YU L F, LU H, et al. Microscopic distribution and quantitative detection of endophytic fungus Undifilum oxytropis from Oxytropis glabra DC and Astragalus variabilis[J]. Acta Microbiologica Sinica, 2014, 54(5): 572–581. (in Chinese) |
| [15] |
张蕾蕾, 余永涛, 何生虎, 等. 不同因素对疯草内生真菌合成苦马豆素的影响[J]. 畜牧兽医学报, 2015, 46(1): 163–173.
ZHANG L L, YU Y T, HE S H, et al. Influence of different factors on swainsonine production in fungal endophyte from locoweed[J]. Acta Veterinaria et Zootechnica Sinica, 2015, 46(1): 163–173. (in Chinese) |
| [16] |
权海云, 任祯慧, 路浩, 等. 苦马豆素研究最新进展[J]. 中国兽医学报, 2017, 37(8): 1633–1640.
QUAN H Y, REN Z H, LU H, et al. Recent research progress on swainsonine[J]. Chinese Journal of Veterinary Science, 2017, 37(8): 1633–1640. (in Chinese) |
| [17] |
萨如拉, 卢萍. 内生真菌苦马豆素生物合成研究进展[J]. 绿色科技, 2018(8): 227–229.
SA R L, LU P. Research progress of swainsonine biosynthesis in endophytic fung[J]. Journal of Green Science and Technology, 2018(8): 227–229. (in Chinese) |
| [18] | LU H, QUAN H Y, REN Z H, et al. The genome of Undifilum oxytropis provides insights into swainsonine biosynthesis and locoism[J]. Sci Rep, 2016, 6: 30760. DOI: 10.1038/srep30760 |
| [19] | REN Z H, SONG R J, WANG S, et al. The biosynthesis pathway of swainsonine, a new anticancer drug from three endophytic fung[J]. J Microbiol Biotechnol, 2017, 27(11): 1897–1906. DOI: 10.4014/jmb.1709.09003 |
| [20] | SINGH D, KAUR G. Optimization of different process variables for the production of an indolizidine alkaloid, swainsonine from Metarhizium anisopliae[J]. J Basic Microbiol, 2012, 52(5): 590–597. DOI: 10.1002/jobm.v52.5 |
| [21] |
童军卫, 刘补兴, 胡小铭, 等. 苦马豆素对胶质瘤细胞U87凋亡及HSP90/AKT通路的影响[J]. 实用药物与临床, 2018, 21(3): 241–245.
TONG J W, LIU B X, HU X M, et al. Influence of swainsonine on glioma cell U87 apoptosis and HSP90/AKT pathway[J]. Practical Pharmacy and Clinical Remedies, 2018, 21(3): 241–245. (in Chinese) |
| [22] |
郝宝成, 武凡琳, 邢小勇, 等. 苦马豆素抗牛病毒性腹泻病毒的研究[J]. 中国农业科学, 2014, 47(1): 170–181.
HAO B C, WU F L, XING X Y, et al. Study on inhibitory effect of the swainsonine from alkaloid of Astragalus strictus Grah. ex bend on bovine viral diarrhea virus[J]. Scientia Agricultura Sinica, 2014, 47(1): 170–181. DOI: 10.3864/j.issn.0578-1752.2014.01.018 (in Chinese) |
| [23] | SHIRNALLI G G, KAUSHIK M S, KUMAR A, et al. Isolation and characterization of high protein and phycocyanin producing mutants of Arthrospira platensis[J]. J Basic Microbiol, 2018, 58(2): 162–171. DOI: 10.1002/jobm.v58.2 |
| [24] |
白晓南.变异黄芪内生真菌的诱变及SW合成突变菌株的筛选[D].银川: 宁夏大学, 2017.
BAI X N. Mutagenesis of Astragalus variabills Bunge endophytic fungi and screening of SW mutant strains[D]. Yinchuan: Ningxia University, 2017. (in Chinese) http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y3286335 |
| [25] | SANDESH KAMATH B, VIDHYAVATHI R, SARADA R, et al. Enhancement of carotenoids by mutation and stress induced carotenogenic genes in Haematococcus pluvialis mutants[J]. Bioresour Technol, 2008, 99(18): 8667–8673. DOI: 10.1016/j.biortech.2008.04.013 |
| [26] | COOK D, DONZELLI B G G, CREAMER R, et al. Swainsonine biosynthesis genes in diverse symbiotic and pathogenic fungi[J]. G3, 2017, 7(6): 1791–1797. DOI: 10.1534/g3.117.041384 |
| [27] | MUKHERJEE S, DAWE A L, CREAMER R. Development of a transformation system in the swainsonine producing, slow growing endophytic fungus, Undifilum oxytropis[J]. J Microbiol Methods, 2010, 81(2): 160–165. DOI: 10.1016/j.mimet.2010.02.015 |
| [28] | MUKHERJEE S, DAWE A L, CREAMER R. Potential role for saccharopine reductase in swainsonine metabolism in endophytic fungus, Undifilum oxytropis[J]. Fungal Biol, 2012, 116(8): 902–909. DOI: 10.1016/j.funbio.2012.05.007 |
| [29] |
李海利.产苦马豆素疯草内生真菌蛋白质双向电泳及蛋白质组学研究[D].杨凌: 西北农林科技大学, 2012.
LI H L. Two dimensional electrophoresis and proteomics analysis of Producing swainsonine endophytic fungi proteins from locoweed[D]. Yangling: Northwest Agriculture and Forestry University, 2012. (in Chinese) |
| [30] | LI H L, YU Y Y, GAO R, et al. Analysis of secreted proteins from Undifilum cinereum by two dimensional gel electrophoresis and liquid chromatography-mass spectrometry/mass spectrometry[J]. J Anim Vet Adv, 2012, 11(11): 1881–1889. DOI: 10.3923/javaa.2012.1881.1889 |



