2. 江苏省动物重要疫病与人兽共患病防控协同创新中心, 扬州 225009
2. Jiangsu Co-innovation Center for Prevention and Control of Important Animal Infectious Diseases and Zoonosis, Yangzhou 225009, China
禽流感病毒(AIV)是一种能够导致急性呼吸系统疾病的病原体,它不但能够感染禽类,给养禽业带来巨大经济损失;还能够跨种传播感染并致死哺乳动物(包括人类),给公共卫生带来极大威胁[1]。AIV是一种有包膜结构的负链RNA病毒,其基因组包含8个RNA片段编码至少17种病毒蛋白,包括10种最初鉴定的蛋白质(PB2、PB1、PA、HA、NP、NA、M1、M2、NS1、NS2)和7种新发现的病毒蛋白PB1-F2、PB1-N40、PA-X、PA-N155、PA-N182、M42和NS3。PA蛋白是病毒RNA聚合酶复合物的第三个亚基,它在AIV的生命周期和发病机制中具有多重作用。PA蛋白的N-末端具有核酸内切酶活性、帽结合和启动子结合的能力,从而影响病毒转录和复制[2];PA蛋白的位点突变有助于AIV跨越宿主屏障、增强AIV的宿主适应性[3-4];此外,PA的关键位点能影响病毒的毒力[5-6];并且PA蛋白与宿主蛋白的“shut-off”有关[7-8]。然而PA蛋白在AIV生命周期和发病机制中发挥其作用的潜在机制在很大程度上仍是未知的。
流感病毒自身并不具有复制能力,感染宿主后必须借助宿主细胞系统进行复制。许多研究已经证实流感病毒依赖于宿主细胞蛋白来维持其生命周期和致病性[9-11],而蛋白之间的相互作用是维持宿主和病毒之间联系的关键方式,并且目前已经鉴定出能与流感病毒PA蛋白相互作用的宿主因子。HAX1是一种具有抗凋亡功能的细胞质蛋白,它能够与PA的核定位信号域结合,在病毒感染的细胞中,HAX1可以阻止PA的核积累来抑制病毒复制,这表明HAX1-PA相互作用是宿主用来限制病毒感染的防御机制之一[12]。hCLE是与PA蛋白相互作用的另一个宿主因子,PA的两个区域(氨基酸残基493—512和557—574)被证实与hCLE相互作用[13],在流感病毒感染期间,hCLE-PA相互作用能够增加病毒聚合酶活性,促进病毒RNA转录和复制,提高病毒滴度和病毒颗粒产生[14]。PA也与MCM复合体有相互作用,MCM复合体是病毒基因组复制的宿主调节剂[15]。此外,RanBP5[16]、AIFMI[17]、NPM[18]和RIG-I[19]等也被发现与PA相互作用并影响AIV的生命周期。然而,考虑到PA蛋白在流感病毒生命周期中的多重效应,其他与PA蛋白相互作用的宿主因子仍需要被鉴定。
目前,虽然在禽流感病毒基因变异、蛋白结构和功能等研究方面有了显著进展,但从病毒-宿主蛋白质互作的角度解析禽流感病毒的致病机制及宿主拮抗流感病毒感染的机制研究依然较少。以禽流感病毒-宿主蛋白的相互作用为突破口,是解析禽流感病毒致病机制和宿主拮抗病毒感染机制的另一思路。本研究通过筛选与致病性差异的H5N1 AIV有相互作用的人类细胞蛋白,可能从宿主方面阐释H5N1 AIV PA蛋白调控病毒生命周期及致病能力方面提供新的靶点。
1 材料与方法 1.1 毒株与细胞本研究所用的主要由PA基因决定的致病性差异的一对模式病毒为本实验室分离保存,分别为对小鼠表现为高致病性的CK10病毒: A/Chicken/Jiangsu/k0402/2010(小鼠半数致死剂量为0.33 log10 EID50)和对小鼠表现为低致病性的GS10病毒:A/Goose/Jiangsu/k0403/2010(小鼠半数致死剂量> 6.32 log10EID50)[20]。A549细胞购自中国科学院上海细胞研究所。
1.2 免疫沉淀(IP)提取细胞蛋白:将细胞上清弃去后,用预冷的PBS洗两遍,然后将残留PBS吸干,加入600 μL含浓度1 mmol·L-1 PMSF的RIPA细胞裂解液(碧云天),冰上裂解,并用摇床轻轻摇晃20 min,使细胞彻底裂解,收集于指形管中,然后在4 ℃下12 000 r·min-1离心15 min,取上清备用。
蛋白质与抗体结合:在获得的细胞蛋白质中加入总量为5 μL的PA兔多克隆抗体(GeneTex),对照组则加入总量为5 μL的非特异性兔多克隆抗体(GeneTex),试验组与对照组各三个重复,然后在4 ℃摇床轻摇过夜。
抗体与agarose结合:取30 μL Protein A+G agarose(Sigma)分别加入对照组与试验组的蛋白质与抗体混合物中,4 ℃摇床轻摇5 h。
洗脱:将充分作用后的样品以12 000 r·min-1瞬离,弃掉上清,加入预冷的PBS,轻轻颠倒混匀,12 000 r·min-1瞬离,弃上清,如此重复操作5次,最后用Glycline-HCl(pH = 3.0)洗脱,进行质谱分析。
1.3 质谱分析(LC-MS/MS)溶液内酶解:吸取已制备好的样品(约30 μg),加入30 μL STD buffer,沸水浴5 min,冷却至室温。加入200 μL UA buffer(8 mol·L-1 Urea,150 mmol·L-1 Tris-HCl pH8.5)混匀,转入30 kDa超滤管离心。加入200 μL UA buffer离心,弃滤液。加入100 μL IAA(50 mmol·L-1 IAA in UA),振荡1 min,避光室温孵育30 min,离心。加入100 μL UA buffer,离心,重复2次。加入100 μL 25 mmol·L-1 NH4HCO3,离心,重复2次。加入40 μL 25 mmol·L-1 NH4HCO3同时加入Trypsin,酶切过夜后离心。再加入40 μL 25 nmol·L-1 NH4HCO3,离心,酸化。
毛细管高效液相色谱方法:A液为0.1%甲酸的水溶液,B液为0.1%甲酸的乙腈水溶液(乙腈为84%)。色谱柱以95%的A液平衡后,样品由自动进样器上样至Trap柱。色谱梯度:0~50 min,A液线性梯度从4%到50%;50~54 min,B液线性梯度从50%到100%;54~60 min,B液维持在100%。
质谱数据采集:多肽和多肽碎片的质量电荷比按照下列方法采集:每次全扫描(full scan)后采集10个碎片图谱(MS2 scan)。
数据分析:质谱测试原始文件(raw file)用Mascot2.2软件检索相应的数据库,最后得到鉴定的蛋白质结果。数据库搜索参数如下,Database: uniprot Taxonomy,Human(145758);Enzyme: Trypsin;Dynamical modifications: Oxidation (M);Fixed modifications: Carbamidomethyl (C);Max Missed Cleavages: 2;ProteomicsTools: 3.1.6;Filter by score ≥20。
1.4 生物信息学分析软件GO注释使用Database for Annotation Visualization and Integrated Discovery (DAVID) (版本6.7);细胞通路分析使用KEGG通路数据库分析。
1.5 免疫共沉淀(co-immunoprecipitation, Co-IP)以1个MOI剂量的CK10和GS10病毒分别感染A549细胞,PBS感染作为对照。36 h后收集细胞蛋白,每个组分别使用PA与eEF1A1抗体进行IP试验,所得免疫复合物用eEF1A1及PA抗体进行Western blot试验进行分析验证。
1.6 Western blot样品加入上样缓冲液后沸水煮5 min变性,12% SDS-PAGE中电泳分离后,并将其转印到聚偏二氟乙烯(PVDF)膜上,120 V转印1 h。转印完毕后,用5%脱脂奶37 ℃封闭1 h,用合适的抗体进行孵育,室温2 h,随后,将膜用含0.05% Tween-20的TBST洗涤4次,每次10 min,再用合适的酶标二抗室温孵育1 h,再次将膜用含0.05% Tween-20的TBST洗涤4次,最后用增强的化学发光(ECL)系统来检测印迹蛋白质。
2 结果 2.1 筛选与PA蛋白有相互作用的宿主细胞蛋白为了有效地从AIV感染的细胞中鉴定与PA相互作用的宿主蛋白,测定了PA蛋白的峰值表达点。以1个MOI的剂量用CK10病毒感染A549细胞,在感染后12、24、36、48和60 h收获细胞蛋白,然后用PA抗体进行Western blot分析,同时以β-actin蛋白作为对照。结果表明,在CK10病毒感染A549过程中,PA表达水平逐渐升高,在感染后36 h左右达到最高[21]。因此,收集病毒感染细胞36 h后的细胞蛋白,并用PA抗体或作为对照的无关抗体进行免疫沉淀;得到的蛋白复合物通过LC-MS / MS分析,结果显示,与对照组相比(三个重复试验中至少出现两次的蛋白即视为存在相互作用),检测到278种与CK10病毒PA蛋白特异性沉淀的宿主细胞蛋白,287种与GS10病毒PA蛋白特异性沉淀的宿主细胞蛋白。而与GS10病毒相比,有160种宿主蛋白能够与CK10病毒PA蛋白特异性结合(表 1)。
|
|
表 1 特异性与CK10病毒PA蛋白互作的宿主蛋白(部分蛋白) Table 1 Host proteins specifically interact with CK10 PA protein (selected proteins) |
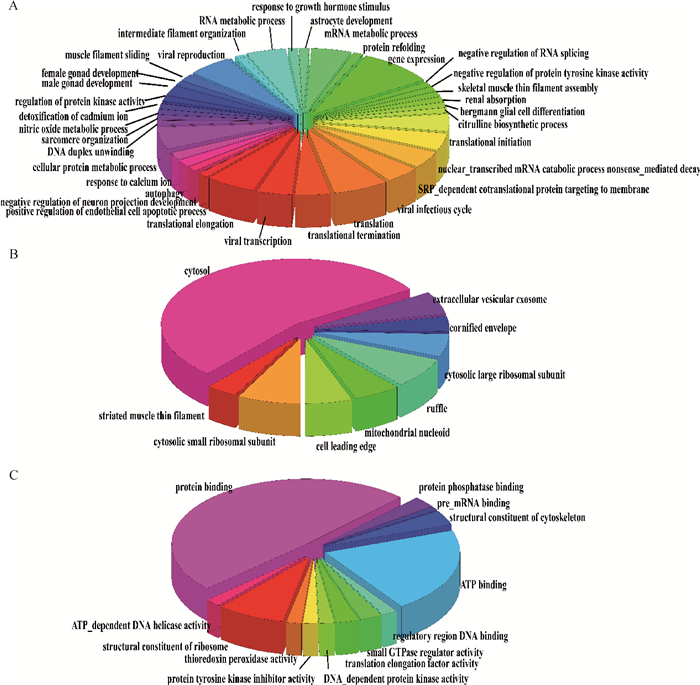
为了揭示与PA蛋白相互作用的差异宿主蛋白的功能,将所筛选的CK10与GS10病毒PA相互作用差异的160种宿主蛋白质进行GO注释分析。如图 1所示,GO注释分为三个方面,包括生物学进程(biological process)、细胞组成(cellular component)和分子功能(molecular function)。生物学进程分析结果显示:这160种宿主蛋白主要参与基因表达、翻译、病毒感染和病毒转录等生物学进程(图 1A),表明与流感病毒的感染息息相关;细胞组成分析结果显示,与PA蛋白相互作用的宿主蛋白主要分布于细胞质(图 1B);基于分子功能的分析结果表明,与PA相互作用的宿主蛋白主要发挥结合功能(图 1C)。
|
A.生物学进程;B.细胞组成;C.分子功能 A. Biological process; B. Cellular component; C. Molecular function 图 1 与PA相互作用差异宿主蛋白的GO功能注释分析 Figure 1 The GO annotation of the different proteins interacting with PA |
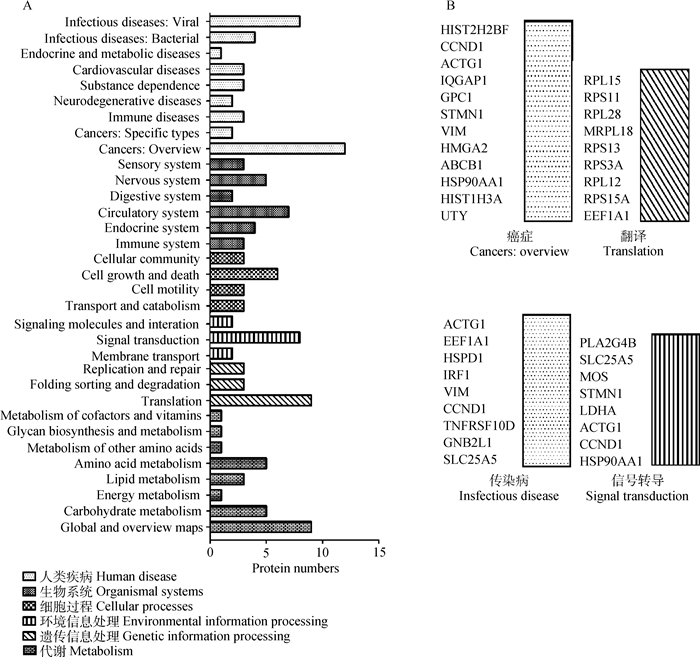
通过KEGG数据库对所筛选到的与PA相互作用的差异宿主蛋白进行细胞通路分析,发现其中的57种蛋白参与了126条细胞通路。主要涵盖了六种类型的细胞通路,包括人类疾病(human diseases)、遗传信息处理(genetic information processing)、生物系统(organismal systems)、代谢(metabolism)、细胞过程(cellular processes)和环境信息处理(environmental information processing)(图 2A);其主要参与的细胞通路为翻译、传染病、癌症和信号转导(图 2B)。
|
A.KEGG通路聚类分析;B.参与最多的四个通路的蛋白名称 A. Classification of the enriched KEGG pathways of the identified proteins; B. Name of the identified proteins related with the top four KEGG pathways 图 2 与PA相互作用差异宿主蛋白的KEGG通路分析 Figure 2 The KEGG pathway analysis of the different proteins interacting with PA |
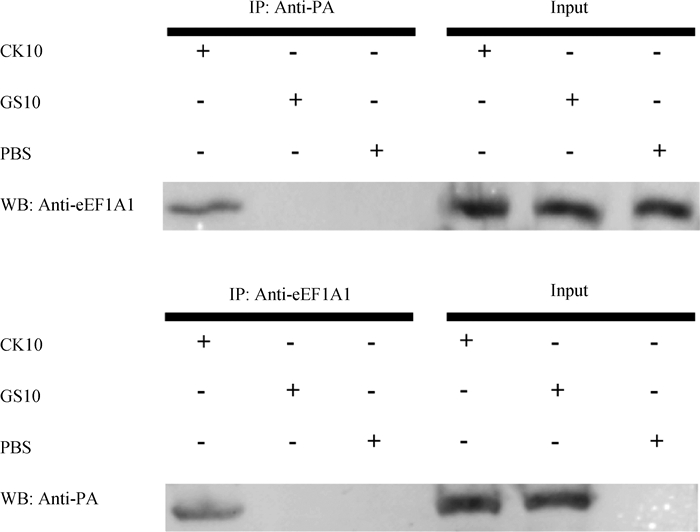
采用IP结合LC-MS/MS的方法,本研究筛选到eEF1A1可能特异性与CK10病毒PA存在相互作用。随后利用Co-IP进行了进一步的验证。分别以1个MOI剂量的CK10和GS10病毒或PBS作为对照直接感染A549细胞,感染病毒36 h后收集细胞蛋白,分别用PA和eEF1A1抗体进行IP试验,所得免疫沉淀物通过Western blot试验进行验证。如图 3所示,eEF1A1能够与CK10病毒PA蛋白相互作用,但不与GS10病毒PA蛋白相互作用。
|
图 3 Co-IP鉴定eEF1A1特异性地与CK10病毒PA蛋白相互作用 Figure 3 Identification of eEF1A1 interacts with CK10 PA protein specifically |
自1997年在亚洲发现第一个H5N1流感病例,H5N1亚型流感病毒已经迅速地蔓延到不同大洲的60多个国家和地区,给养禽业和人类健康带来了相当大的损害。流感病毒可以逃逸宿主免疫应答反应并导致持续感染。然而,直到现在,AIV感染的机制尚未完全阐明。病毒感染宿主细胞后,可通过与宿主细胞因子的相互作用,利用宿主细胞的转录翻译系统,在细胞中存活并不断地繁殖[22]。PA是流感病毒RNA聚合酶的一个亚基,具有多重作用:(1)在病毒转录和复制中起重要作用[15];(2)有助于增强AIV在哺乳动物宿主中的适应性[3, 23];(3)影响流感病毒的毒力[5-6];(4)与宿主细胞蛋白的“shut-off”有关[7-8];(5)流感病毒感染过程中,参与宿主的免疫调节[24]。此外,PA基因通过核糖体移码能够编码翻译PA-X蛋白,PA-X在病毒的生命周期和致病机制中起着重要的作用[25]。
鉴于PA蛋白在流感病毒生命周期和致病机制中的重要作用,挖掘与流感病毒PA蛋白相互作用宿主细胞蛋白对于研究病毒感染的潜在机制和开发新的抗病毒药物非常重要。而本研究中通过一对主要由PA决定的小鼠致病性差异显著的模式病毒筛选与PA相互作用差异宿主蛋白,更能挖掘出影响禽流感病毒致病性的宿主因子。
许多技术已被用于筛选蛋白间的相互作用,如亲和纯化结合质谱(AP-MS)和酵母双杂交(Y2H),与Y2H相比,AP-MS的使用更为广泛,因为它可以揭示天然状态下蛋白质之间的相互作用。在本研究中,应用IP偶联LC-MS / MS技术,筛选在H5N1流感病毒感染的A549细胞中与PA相互作用的宿主蛋白,这相比于使用PA表达质粒转染细胞后进行IP偶联LC-MS / MS相比,不仅在病毒复制期间保留蛋白质的天然构象,而且能够探索可能与PA间接相互作用的宿主细胞蛋白。
通过上述方法,共筛选到与致病性差异H5N1流感病毒的PA存在相互作用的160种差异人类细胞蛋白。为了进一步探索这些差异宿主细胞蛋白与病毒之间相互作用的生物学意义,对这些宿主蛋白进行了生物信息学分析。基于GO注释分析结果表明,所筛选的宿主细胞蛋白与病毒感染、翻译和复制高度相关(图 2)。
通过KEGG通路分析,160个所筛选的差异宿主细胞蛋白中有57种蛋白参与了126条细胞通路。其中参与最多且与病毒感染密切相关的为翻译相关途径,涉及到2条细胞通路以及9种宿主蛋白(图 3)。因此,我们推测翻译途径在流感病毒感染过程中发挥着至关重要的作用。因为在流感病毒进入宿主细胞后,病毒核糖核蛋白复合物和病毒RNA(vRNPs)被运送到细胞核内,在细胞核中,病毒基因组转录成mRNA,然后又被转运回细胞质并翻译成病毒蛋白,随后NP、PB1、PB2和PA蛋白将重新进入细胞核与病毒RNA形成新的RNP复合物[26]。因此,通过将与PA互作宿主细胞蛋白的KEGG通路分析,进一步证实了PA在病毒复制中的关键作用。其次还有传染病相关途径,包括14条通路和9个所筛选的蛋白(图 3)。在这9种蛋白中,ACTG1和TNFRSF10D与流感病毒感染途径高度相关,因此我们推测这两个蛋白可能是造成本研究中两株禽流感病毒致病性差异的主要宿主因子。同时免疫系统和信号转导通路也富集较多差异PA互作宿主蛋白,包括抗原的加工和呈递、NOD样受体信号通路、PI3K-Akt信号通路、MAPK信号通路和Jak-STAT信号通路等。已有的研究结果表明,PI3K-Akt信号通路在流感病毒感染过程中由NS1激活[27],发挥着抑制病毒复制的作用[28];而MAPK信号通路在流感病毒感染期间能够调节宿主炎性应答反应,从而影响病毒复制[29]。本研究的结果将进一步为研究禽流感病毒对宿主免疫应答的影响提供新的靶标蛋白。
利用Co-IP试验,对质谱结果进行了进一步验证,发现宿主蛋白eEF1A1能够特异性的与CK10病毒PA蛋白结合。eEF1A1不仅是一种翻译因子,它还有多种效用,包括调节细胞骨架、伴侣蛋白活性以及调节细胞增殖和细胞死亡[30]。以前的研究表明,eEF1A1与p53蛋白相互作用[31]。P53蛋白也与流感病毒相互作用,抑制p53蛋白表达能够增强机体先天和适应性免疫系统抵抗流感病毒感染[32],但是p53发挥此作用的机制仍未可知。因此,我们推测本研究中所鉴定的CK10病毒PA相互作用宿主蛋白eEF1A1可能与p53一起在流感病毒感染过程中发挥着重要作用,当然这需要进一步的试验进行验证。
4 结论在此研究中,通过比较两株小鼠致病性差异显著的H5N1禽流感病毒PA蛋白相互作用的人类细胞蛋白,筛选到160种差异相互作用的人类细胞蛋白。通过GO注释和KEGG通路分析,结果显示这些蛋白在病毒感染和病毒复制过程中发挥重要作用,此外Co-IP试验验证了宿主蛋白eEF1A1能够特异性地与CK10病毒PA蛋白相互作用。这为揭示与流感病毒毒力因子PA蛋白相关的分子机制提供了帮助,同时也将加速从宿主层面阐明流感病毒致病机制的步伐。
| [1] | WEBSTER R G, BEAN W J, GORMAN O T, et al. Evolution and ecology of influenza A viruses[J]. Microbiol Mol Biol Rev, 1992, 56(1): 152–179. |
| [2] | HARA K, SCHMIDT F I, CROW M, et al. Amino acid residues in the N-terminal region of the PA subunit of influenza A virus RNA polymerase play a critical role in protein stability, endonuclease activity, cap binding, and virion RNA promoter binding[J]. J Virol, 2006, 80(16): 7789–7798. DOI: 10.1128/JVI.00600-06 |
| [3] | SEYER R, HRINCIUS E R, RITZEL D, et al. Synergistic adaptive mutations in the hemagglutinin and polymerase acidic protein lead to increased virulence of pandemic 2009 H1N1 influenza A virus in mice[J]. J Infect Dis, 2012, 205(2): 262–271. DOI: 10.1093/infdis/jir716 |
| [4] | SUN Y P, XU Q, SHEN Y, et al. Naturally occurring mutations in the PA gene are key contributors to increased virulence of pandemic H1N1/09 influenza virus in mice[J]. J Virol, 2014, 88(8): 4600–4604. DOI: 10.1128/JVI.03158-13 |
| [5] | HULSE-POST D J, FRANKS J, BOYD K, et al. Molecular changes in the polymerase genes (PA and PB1) associated with high pathogenicity of H5N1 influenza virus in mallard ducks[J]. J Virol, 2007, 81(16): 8515–8124. DOI: 10.1128/JVI.00435-07 |
| [6] | SONG J S, FENG H P, XU J, et al. The PA protein directly contributes to the virulence of H5N1 avian influenza viruses in domestic ducks[J]. J Virol, 2011, 85(5): 2180–2188. DOI: 10.1128/JVI.01975-10 |
| [7] | DESMET E A, BUSSEY K A, STONE R, et al. Identification of the N-terminal domain of the influenza virus PA responsible for the suppression of host protein synthesis[J]. J Virol, 2013, 87(6): 3108–3118. DOI: 10.1128/JVI.02826-12 |
| [8] | LLOMPART C M, NIETO A, RODRIGUEZ-FRANDSEN A. Specific residues of PB2 and PA influenza virus polymerase subunits confer the ability for RNA polymerase Ⅱ degradation and virus pathogenicity in mice[J]. J Virol, 2014, 88(6): 3455–3463. DOI: 10.1128/JVI.02263-13 |
| [9] | HALE B G, JACKSON D, CHEN Y H, et al. Influenza A virus NS1 protein binds p85β and activates phosphatidylinositol-3-kinase signaling[J]. Proc Natl Acad Sci U S A, 2006, 103(38): 14194–14199. DOI: 10.1073/pnas.0606109103 |
| [10] | GACK M U, ALBRECHT R A, URANO T, et al. Influenza A virus NS1 targets the ubiquitin ligase TRIM25 to evade recognition by the host viral RNA sensor RIG-I[J]. Cell Host Microbe, 2009, 5(5): 439–449. DOI: 10.1016/j.chom.2009.04.006 |
| [11] | GAO S J, WU J X, LIU R Y, et al. Interaction of NS2 with AIMP2 facilitates the switch from ubiquitination to sumoylation of M1 in influenza A virus-infected cells[J]. J Virol, 2015, 89(1): 300–311. DOI: 10.1128/JVI.02170-14 |
| [12] | HSU W B, SHIH J L, SHIH J R, et al. Cellular protein HAX1 interacts with the influenza A virus PA polymerase subunit and impedes its nuclear translocation[J]. J Virol, 2013, 87(1): 110–123. DOI: 10.1128/JVI.00939-12 |
| [13] | HUARTE M, SANZ-EZQUERRO J J, RONCAL F, et al. PA subunit from influenza virus polymerase complex interacts with a cellular protein with homology to a family of transcriptional activators[J]. J Virol, 2001, 75(18): 8597–8604. DOI: 10.1128/JVI.75.18.8597-8604.2001 |
| [14] | RODRIGUEZ A, PÉREZ-GONZÉLEZ A, NIETO A. Cellular human CLE/c14orf166 protein interacts with influenza virus polymerase and is required for viral replication[J]. J Virol, 2011, 85(22): 12062–12066. DOI: 10.1128/JVI.00684-11 |
| [15] | KAWAGUCHI A, NAGATA K. De novo replication of the influenza virus RNA genome is regulated by DNA replicative helicase, MCM[J]. EMBO J, 2007, 26(21): 4566–4575. DOI: 10.1038/sj.emboj.7601881 |
| [16] | DENG T, ENGELHARDT O G, THOMAS B, et al. Role of ran binding protein 5 in nuclear import and assembly of the influenza virus RNA polymerase complex[J]. J Virol, 2006, 80(24): 11911–11919. DOI: 10.1128/JVI.01565-06 |
| [17] | BRADEL-TRETHEWAY B G, MATTIACIO J L, KRASNOSELSKY A, et al. Comprehensive proteomic analysis of influenza virus polymerase complex reveals a novel association with mitochondrial proteins and RNA polymerase accessory factors[J]. J Virol, 2011, 85(17): 8569–8581. DOI: 10.1128/JVI.00496-11 |
| [18] | MAYER D, MOLAWI K, MARTINEZ-SOBRIDO L, et al. Identification of cellular interaction partners of the influenza virus ribonucleoprotein complex and polymerase complex using proteomic-based approaches[J]. J Proteome Res, 2007, 6(2): 672–682. DOI: 10.1021/pr060432u |
| [19] | LI W Z, CHEN H J, SUTTON T, et al. Interactions between the influenza A virus RNA polymerase components and retinoic acid-inducible gene I[J]. J Virol, 2014, 88(18): 10432–10447. DOI: 10.1128/JVI.01383-14 |
| [20] | HU J, ZHAO K, LIU X, et al. Two highly pathogenic avian influenza H5N1 viruses of clade 2. 3. 2. 1 with similar genetic background but with different pathogenicity in mice and ducks[J]. Transbound Emerg Dis, 2013, 60(2): 127–139. DOI: 10.1111/tbed.2013.60.issue-2 |
| [21] | GAO Z, HU J, LIANG Y Y, et al. Generation and comprehensive analysis of host cell interactome of the PA Protein of the highly pathogenic H5N1 avian influenza virus in mammalian cells[J]. Front Microbiol, 2017, 8: 739. DOI: 10.3389/fmicb.2017.00739 |
| [22] | LAMKANFI M, DIXIT V M. Manipulation of host cell death pathways during microbial infections[J]. Cell Host Microbe, 2010, 8(1): 44–54. DOI: 10.1016/j.chom.2010.06.007 |
| [23] | SAKABE S, OZAWA M, TAKANO R, et al. Mutations in PA, NP, and HA of a pandemic (H1N1) 2009 influenza virus contribute to its adaptation to mice[J]. Virus Res, 2011, 158(1-2): 124–129. DOI: 10.1016/j.virusres.2011.03.022 |
| [24] | HUANG C H, CHEN C J, YEN C T, et al. Caspase-1 deficient mice are more susceptible to influenza A virus infection with PA variation[J]. J Infect Dis, 2013, 208(11): 1898–1905. DOI: 10.1093/infdis/jit381 |
| [25] | HU J, MO Y Q, WANG X Q, et al. PA-X decreases the pathogenicity of highly pathogenic H5N1 influenza A virus in avian species by inhibiting virus replication and host response[J]. J Virol, 2015, 89(8): 4126–4142. DOI: 10.1128/JVI.02132-14 |
| [26] | BERRI F, LÊ V B, JANDROT-PERRUS M, et al. Switch from protective to adverse inflammation during influenza:Viral determinants and hemostasis are caught as culprits[J]. Cell Mol Life Sci, 2014, 71(5): 885–898. DOI: 10.1007/s00018-013-1479-x |
| [27] | GAUR P, MUNJAL A, LAL S K. Influenza virus and cell signaling pathways[J]. Med Sci Monit, 2011, 17(6): RA148–RA154. |
| [28] | EHRHARDT C, MARJUKI H, WOLFF T, et al. Bivalent role of the phosphatidylinositol-3-kinase (PI3K) during influenza virus infection and host cell defence[J]. Cell Microbiol, 2006, 8(8): 1336–1348. DOI: 10.1111/cmi.2006.8.issue-8 |
| [29] | LEE C J, LIAO C L, LIN Y L. Flavivirus activates phosphatidylinositol 3-kinase signaling to block caspase-dependent apoptotic cell death at the early stage of virus infection[J]. J Virol, 2005, 79(13): 8388–8399. DOI: 10.1128/JVI.79.13.8388-8399.2005 |
| [30] | ABBAS W, KUMAR A, HERBEIN G. The eEF1A proteins:at the crossroads of oncogenesis, apoptosis, and viral infections[J]. Front Oncol, 2015, 5: 75. |
| [31] | BLANCH A, ROBINSON F, WATSON I R, et al. Eukaryotic translation elongation factor 1-alpha 1 inhibits p53 and p73 dependent apoptosis and chemotherapy sensitivity[J]. PLoS One, 2013, 8(6): e66436. DOI: 10.1371/journal.pone.0066436 |
| [32] | MUÑOZ-FONTELA C, PAZOS M, DELGADO I, et al. p53 serves as a host antiviral factor that enhances innate and adaptive immune responses to influenza A virus[J]. J Immunol, 2011, 187(12): 6428–6236. DOI: 10.4049/jimmunol.1101459 |



