非编码RNA(non-coding RNA, ncRNA)是指一大类不编码蛋白质,却具有生物活性的RNA[1],这类RNA曾一度被认为是转录的“暗物质”或“噪音”[2-3],但是随着研究的不断深入,越来越多的研究者证实了非编码RNA在转录水平上发挥着广泛的生物学作用[4]。非编码RNA根据链的长度可划分为短链非编码RNA和长链非编码RNA(lncRNA)。lncRNA是非编码RNA中重要的组成部分,其长度一般大于200 nt[5]。目前,根据lncRNA及其相关蛋白编码基因之间的关系将其分为反义lncRNA(antisense lncRNA)、双向lncRNA(bidirectional lncRNA)、内含子lncRNA(intronic lncRNA)、正义重叠区lncRNA(sense overlapping lncRNA)和基因间隔区(lincRNA)与加工转录本[6-8];根据功能,lncRNA可分为信号因子、引导因子、诱饵因子和支架因子[9]。不同类型的lncRNAs具有不同的作用。目前lncRNA在动物繁殖方面的生物学研究较少。lncRNA在动物早期生殖细胞形成、早期胚胎的着床和发育以及有关激素调节中扮演着必不可少的角色[10]。科学家们通过RNA-Seq对人、小鼠、牛等受精卵与受精前的配子、不同发育阶段的早期胚胎间的比较,发现大量与不同胚胎发育时期相关的lncRNAs[11-12],如Meg3与JARID2基因互作从而招募PRC2以反式作用抑制胚胎发育相关基因表达[13]。Chen等[14]在小鼠胚胎发育不同阶段的卵巢中发现了24个lncRNA差异表达,在相同胎龄的雌雄生殖器官中发现147个lncRNA差异表达,在卵巢中表达最高的oncRNA3是泛酸激酶1。Roeszler等[15]研究发现,在母鸡沉默lnc-MHM会造成卵巢不对称发育。Brown等[16]在对果蝇和小鼠卵巢lncRNA研究中发现了大量与启动子相关的反义lncRNAs,这些lncRNAs可能调控其同源基因的转录激活。Macaulay等[17]利用共聚焦透射电子显微镜和RNA-Seq发现,牛卵母细胞周围的卵丘细胞为成年母牛的卵母细胞运输大量的营养和物质,包括mRNA和lncRNA,由于卵母细胞及其周围细胞细胞质间的信息传递和物质运输方式有限,差异表达的lncRNA可能对卵子的发生具有重要作用。
目前在猪卵泡发育上,对于基因表达调控的研究主要集中在蛋白质编码基因上,而对参与猪卵泡发育的非编码RNA的研究相对较少,这遗漏了大量的遗传信息。本课题组前期通过高通量测序技术获得了梅山和杜洛克猪中等卵泡lncRNA 3 554个,其中已注释的为1 997个,未注释的为1 557个[18]。lncRNA-ENSSSCT00000018610是从梅山猪和杜洛克猪中等卵泡差异lncRNA数据库中获得的已注释长链非编码RNA。
为了对lncRNA-ENSSSCT00000018610进行深入的研究,本试验采用3′和5′ Rapid Amplification of cDNA Ends (RACE)技术扩增得到其全长序列,利用Real-time PCR对其进行组织表达谱分析,并对其在梅山猪和杜洛克猪的各级卵泡中的相对表达量进行检测,揭示了其在两个猪种不同组织和各级卵泡中的表达规律,有助于探究该lncRNA在卵泡发育过程中的作用机理,为深入研究猪种间排卵数差异的分子机理提供了科学依据。
1 材料与方法 1.1 试验样品采集试验选取同一胎次、发情正常、繁殖表现符合品种特征的梅山和杜洛克成年母猪各3头,相同条件下饲喂,每天对其发情状况进行观测,以发情出现静立反应时记做发情周期第0天,发情周期第14天沿耳缘静脉注射兽用氯前列烯醇注射液1支·头-1,注射后第4天屠宰,取不同级别卵泡(按直径分类,S卵泡:1.0~3.0 mm;M1卵泡:3.1~5.0 mm;M2卵泡:5.1~7.0 mm;L卵泡:7.0 mm以上)于液氮中冻存,同时取卵巢、下丘脑、心、肝、脾、肺、肾、小肠、肌肉、子宫、垂体、黄体、输卵管,用锡纸包裹,液氮速冻后于-80 ℃保存备用。
1.2 主要试剂与仪器主要试剂:TRIzol试剂(Invitrogen, Carlsbad, CA, USA);反转录试剂盒PrimerScriptTM RT regagent Kit(TaKaRa);LightCycler 480 SYBR Green、八连管(Roche);SMARTer® RACE cDNA Amplification Kit、SeqAmp DNA Polymerse、NucleoSpin Gel and PCR Clean-Up Kit、In-Fusion HD Cloning Kit、Stellar Competent Cells(Clontech);DNA Maker、西班牙进口Agarose琼脂糖、营养琼脂、胰蛋白胨、酵母提取物(北京天根)等。
主要仪器:超低温冰箱(-80 ℃)、恒温水浴锅、高压灭菌锅、移液枪、PCR仪、超净工作台、NanoDrop 2000、LightCycler 480实时荧光定量PCR仪、分析天平、高速离心机、电热恒温培养箱、水平电泳槽、电泳仪;GelDoc X R型凝胶成像系统等。
1.3 lncRNA-ENSSSCT00000018610保守性分析和编码潜能预测根据lncRNA-ENSSSCT00000018610在Ensembl上注释的序列信息,采用Phast Cons Score工具对lncRNA进行保守性评估,并用Coding-Potential Assessment Tool(CAPT)[19]对lncRNA-ENSSSCT00000018610编码能力进行预测。
1.4 引物设计与合成根据lncRNA-ENSSSCT00000018610部分已知核苷酸序列,用Primer Primer 5.0软件设计引物,分别用于实时荧光定量PCR、5′和3′ RACE PCR扩增,用于RACE克隆的特异性引物在5′末端添加15 bp序列“GATTACGCCAAGCTT”用于后续试验的连接测序,所有试验所需引物见表 1,引物序列由新疆乌鲁木齐市昆泰锐公司合成。
|
|
表 1 lncRNA-ENSSSCT00000018610及内参引物序列 Table 1 The primers for lncRNA-ENSSSCT00000018610 and internal reference gene |
取两品种猪的各级卵泡和不同组织100 mg于液氮中研磨,TRIzol法分别提取样本总RNA,用2%琼脂糖凝胶电泳和NanoDrop 2000验证总RNA的完整性、浓度及纯度。-80 ℃保存备用。实时荧光定量PCR所用cDNA的合成分别按照TaKaRa反转录试剂盒说明书进行操作,5′和3′ RACE PCR扩增参照SMARTer®RACE cDNA第一链合成说明书进行操作,获得的cDNA置于-20 ℃保存备用。
1.6 lncRNA-ENSSSCT00000018610全长克隆5′ RACE PCR反应:试验以5′卵泡cDNA为模板,Long和5′ GSP分别为上、下游引物,采用Touch Down PCR对lncRNA-ENSSSCT00000018610进行5′序列扩增。反应体系:5′RACE Ready cDNA 2.5 μL,10×UPM(Long) 5 μL,5′GSP 1 μL,PCR Grade H2O 15.5 μL,2×SeqAmp Buffer 25 μL,SeqAmp DNA Polymerse 1 μL。反应条件:95 ℃ 5 min;(95 ℃ 30 s,72 ℃ 3 min)5个循环;(95 ℃ 30 s,70 ℃ 30 s,72 ℃ 3 min)10个循环;(95 ℃ 30 s,62 ℃ 30 s,72 ℃ 3 min)20个循环。1.2%琼脂糖凝胶电泳检测,将出现的明显条带胶回收,并连接pUC 19 Linearized Vector转化并进行菌液测序。
3′ RACE PCR反应:试验以3′卵泡cDNA为模板,Long和3′GSP分别为上、下游引物,采用Touch Down PCR对lncRNA-ENSSSCT00000018610进行3′序列扩增。反应体系:3′ RACE Ready cDNA 2.5 μL,10×UPM (Long)5 μL,3′GSP 1 μL,PCR Grade H2O 15.5 μL,2×SeqAmp Buffer 25 μL,SeqAmp DNA Polymerse 1 μL。反应条件:95 ℃ 5 min;(95 ℃ 30 s,72 ℃ 3 min)5个循环;(95 ℃ 30 s,70 ℃ 30 s,72 ℃ 3 min)15个循环;(95 ℃ 30 s,62 ℃ 30 s,72 ℃ 3 min)25个循环。1.2%琼脂糖凝胶电泳检测,将出现的明显条带胶回收,并连接pUC 19 Linearized Vector转化并进行菌液测序。
1.7 猪lncRNA-ENSSSCT00000018610组织表达谱分析试验以各组织与卵泡cDNA为模板,GAPDH为内参,对lncRNA-ENSSSCT00000018610进行实时荧光定量PCR检测以分析其在猪不同组织中的表达情况。反应体系:SYBR® Green I PCR Mix 10 μL,上、下游引物各1 μL,cDNA模板2 μL,加ddH2O至20 μL。反应条件:95 ℃预变性5 min;95 ℃变性20 s,56 ℃退火30 s,72 ℃延伸30 s。
1.8 lncRNA-ENSSSCT00000018610靶基因预测试验通过cis(co-location)和trans(co-expression)两种方式对lncRNA-ENSSSCT00000018610靶基因进行预测。搜索lncRNA上游和下游100 kb内的编码基因作为顺式作用;反式作用是指通过表达水平鉴定彼此,并选择具有Pearson相关系数|r|>0.95的结果[20-21]。
1.9 数据处理以2-△△ Ct法[22]计算lncRNA-ENSSSCT00000018610在各组织及梅山猪和杜洛克猪各级卵泡中的相对表达量,用SPSS17.0软件中One-Way ANOVA方法分析比较其在两猪种内不同级别卵泡及品种间同级别卵泡表达水平的差异。
2 结果 2.1 lncRNA-ENSSSCT00000018610的保守性分析和编码潜能预测lncRNA-ENSSSCT00000018610位于猪基因组16号染色体,具有5个外显子,但均不具有编码潜能,对其保守性进行总体评估,保守性评分=0(表 2),符合lncRNA具有不保守性特征。此外,利用Coding-Potential Assessment Tool(CAPT)法对lncRNA-ENSSSCT00000018610编码能力进行预测,结果表明,lncRNA-ENSSSCT00000018610的编码能力预测结果为0.159 346 863,该数据比已使用的人lncRNA编码能力阈值0.364[23]要小,这进一步说明lncRNA-ENSSSCT00000018610不具有编码蛋白的潜能。
|
|
表 2 lncRNA-ENSSSCT00000018610序列保守性评估 Table 2 Conservative evaluation of lncRNA-ENSSSCT00000018610 sequence |
5′ RACE获得一条长度约为1 000 bp的明亮条带(图 1A),3′ RACE获得一条长度超过1 000 bp的明亮条带(图 1B),克隆测序分别获得长度为1 027、1 343 bp的序列。

|
M. DNA相对分子质量标准;5′. 5′ RACE PCR电泳条带; 3′. 3′ RACE PCR电泳条带 M. Marker 10000; 5′. 5′ RACE PCR electrophoresis band; 3′. 3′ RACE PCR electrophoresis band 图 1 lncRNA-ENSSSCT00000018610 5′-RACE(A)和3′-RACE(B)扩增结果电泳产物图 Figure 1 The electrophoresis of PCR products of 5′-RACE(A)and 3′-RACE (B) of lncRNA-ENSSSCT00000018610 |
5′ RACE得到的1 027 bp序列中有940 bp与已知序列相同,获得5′端未知序列74 bp。3′ RACE得到的1 343 bp左右序列中有1 269 bp与已知序列相同,且3′末端有明显的Poly A尾巴,获得3′端75 bp的未知序列。通过5′和3′ RACE扩增结果拼接,总共获得lncRNA-ENSSSCT00000018610的全长序列1 731 bp(图 2)。

|
图中第一行蓝色大写字母序列为lncRNA-ENSSSCT00000018610的高通量测序结果文件序列,第二行两端绿色大写字母序列分别为5′和3′ RACE末端克隆获得序列,中间为5′与3′ RACE拼接序列。第三行小写字母序列为RACE克隆得到的lncRNA-ENSSSCT00000018610 cDNA序列 The first row of blue capital letters in the figure is the lncRNA-ENSSSCT00000018610 high-throughput sequencing result file sequence. The second row of green capital letters are the sequence of the 5′ and 3′ RACE clones, and the middle is 5′ and 3′ RACE splicing sequence. The third row of lowercase letters sequence is the lncRNA-ENSSSCT00000018610 cDNA sequence cloned from RACE 图 2 5′与3′ RACE测序序列比对与拼接结果 Figure 2 The result of 5′ and 3′ RACE sequence comparison and splicing |
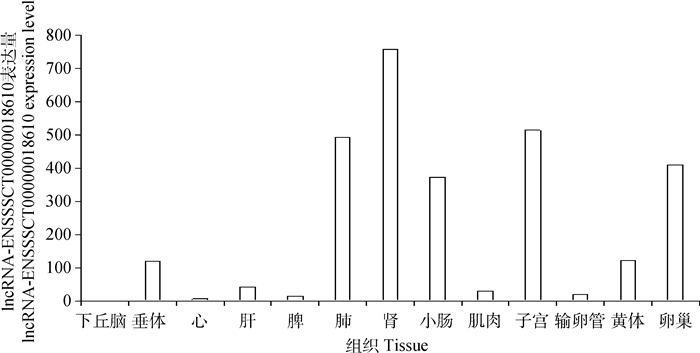
试验采用RT-PCR技术对lncRNA-ENSSSCT00000018610在猪不同组织中的表达情况作了分析,结果表明,其在下丘脑、垂体、心、肝、脾、肺、肾、小肠、肌肉、子宫、输卵管、黄体、卵巢中均有不同程度的表达(图 3)。
|
图 3 lncRNA-ENSSSCT00000018610在猪不同组织中的表达情况 Figure 3 Tissues expression profile of lncRNA-ENSSSCT00000018610 in pigs |
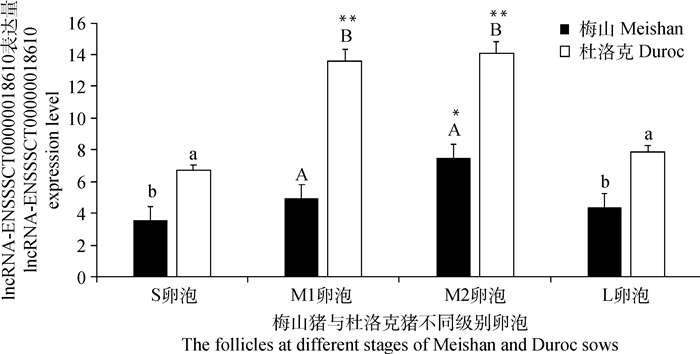
试验采用RT-PCR技术检测了梅山与杜洛克猪不同级别卵泡中lncRNA-ENSSSCT0000-0018610的表达情况(图 4)。结果表明,lncRNA-ENSSSCT00000018610在梅山猪M2卵泡中的表达量高于S、M1、L卵泡(P < 0.05),在杜洛克猪M1、M2卵泡中的表达量极显著高于S和L卵泡(P < 0.01),提示该lncRNA可能与卵泡发育有关。在杜洛克猪S、L卵泡中的表达量分别显著高于在梅山猪S、L卵泡中的表达量(P < 0.05),在杜洛克猪的M1、M2卵泡中的表达量分别极显著高于在梅山猪的M1、M2卵泡中的表达量(P < 0.01)。
|
同一品种不同级别卵泡间,**表示差异极显著(P < 0.01);*表示差异显著(P < 0.05)。不同品种同级卵泡间,肩标大写字母不同表示差异极显著(P < 0.01);肩标小写字母不同表示差异显著(P < 0.05) Among the different follicles in the same breed, ** mean extremely significant difference(P < 0.01);* mean significant difference(P < 0.05). In the same follicles of different breeds, the different capital letters mean extremely significant difference(P < 0.01); the different small letters mean significant difference(P < 0.05) 图 4 lncRNA-ENSSSCT00000018610在梅山和杜洛克母猪各级卵泡中的相对表达量 Figure 4 The relative expression of lncRNA-ENSSSCT00000018610 in follicles at different stages of Meishan and Duroc sows |
本研究通过构建蛋白编码基因的位置关系(co-location),以及与蛋白编码基因表达相关性(co-expression)预测得到lncRNA-ENSSSCT00000018610有多个潜在靶基因(表 3)。
|
|
表 3 lncRNA-ENSSSCT00000018610靶基因预测 Table 3 lncRNA-ENSSSCT00000018610 target genes prediction |
TNIP1是通过lncRNA-ENSSSCT00000018610位置关系(co-location)预测得到的潜在靶基因,KEGG富集分析发现,TNIP1被富集在NF-κB信号通路上,GO分析发现其参与19种生物学过程;CYP2J2、SCARB1、IBSP等基因是通过lncRNA-ENSSSCT00000018610表达相关性(co-expression)预测得到的潜在靶基因,它们分别被富集在卵巢类固醇激素通路和PI3K-Akt信号通路,参与猪卵巢类固醇激素合成、细胞生长分化以及卵泡发育等生物学过程。这提示,lncRNA-ENSSSCT00000018610可能通过对其靶基因的调控间接参与猪卵泡发育。
3 讨论lncRNA在各类生命活动中发挥着重要的调节作用,受到了更多科学家的关注。目前lncRNA的研究多停留在芯片检测和高通量测序层面上,对其机理的探究更是局限于人类的疾病、免疫及模式生物上,而在重要的经济动物生长发育及繁殖上,lncRNA的研究相对滞后,仍然处于摸索阶段。
随着基因芯片和测序技术的不断发展,对lncRNA的获取和鉴定方法也在不断更新,不同的研究者提出了不同的研究方法和鉴定流程,主要是高通量测序技术和基于lncRNA特征的计算机预测鉴定。高通量测序技术被广泛应用于非编码RNA研究,lncRNA测序能够通过rRNA去除法对含有和不含有PolyA尾巴的RNA一同富集建库测序,使得数据不浪费,从而能够鉴定到更多的lncRNA[23-24],这也使得lncRNA测序在更多的研究中应用。基于lncRNA特征的计算机预测包括CPAT、CPC、PhyloCSF等软件[25]。CPAT能从大量的候选转录物中迅速确认出编码和非编码记录,它用了一种全新的序列匹配的方法。CPAT预测方法采用逻辑回归模型,使用4种序列特征:ORF长度、覆盖率特征、Fickett Score和六聚体偏好性特征[11]。就软件的预测性能而言,CPAT (敏感性0.96,异性:0.97)精度更优于CPC (敏感性0.99,异性:0.74)、PhyloCSF (敏感性0.90,异性:0.63)[26]等方法,此外CPAT运算速度比其他软件快大约4个数量级[27]。因此这个运算速度快、方便且精度高的编码能力预测软件被研究者所认可。本试验所用的lncRNA是通过高通量测序技术结合计算机软件分析获得的,高通量测序结果显示,lncRNA-ENSSSCT00000018610在梅山和杜洛克猪中等卵泡中的表达差异极显著,加之使用CPAT对其进行编码能力预测,结果显示其编码能力分值为0.159,比已使用的人长链非编码RNA筛选阈值0.364[28]小。lncRNA-ENSSSCT00000018610含有一个长度为360 nt的ORF,编码120个氨基酸,而许多研究比较深入的lncRNA,如HOTAIR家族和H19都存在长度大于100 aa的ORF[29],这些ORF极有可能不翻译或翻译效率很低,也可能翻译出来的蛋白质会被快速的降解。所以尽管lncRNA-ENSSSCT00000018610的ORF >100 aa,其也不具有编码能力或者编码能力很弱。当然,也有一些RNA在最初的研究中被鉴定为lncRNA,但随着研究的不断深入,发现其也有编码能力[30],这些都需要更进一步验证。
RACE(rapid amplification of cDNA ends)是一种快速克隆cDNA末端的技术,此方法被广泛应用于未知碱基序列基因的克隆。该方法基于PCR技术,步骤少、耗时短,只需知道RNA内部很短的一小段cDNA序列就能快速克隆出基因的未知序列。RACE技术包括3′和5′末端序列扩增,完整且纯度高的总RNA和高质量3′和5′RACE ready cDNA是RACE成功的基础,而根据已知核酸序列设计出的优良特异性引物是RACE成功的决定性因素。本试验通过RACE技术成功克隆出了lncRNA-ENSSSCT00000018610全长为1 731 bp的cDNA。这为进一步研究lncRNA-ENSSSCT00000018610的生物学功能奠定了基础。
本研究通过在杜洛克猪不同组织中对lncRNA-ENSSSCT00000018610进行的荧光定量PCR发现,这个lncRNA在杜洛克猪各组织中广泛表达且范围波动较大,说明它具有组织表达特异性,其在子宫和卵巢中的较高表达可能提示,lncRNA-ENSSSCT00000018610可能参与母猪生殖活动过程。在梅山猪和杜洛克猪不同级别卵泡中的定量PCR显示,lncRNA-ENSSSCT00000018610在梅山猪中等卵泡M1、M2中的表达量均极显著低于杜洛克猪的M1和M2卵泡,与高通量测序结果一致,这说明了高通量测序具有一定的准确性。
此外,本研究通过构建lncRNA与蛋白编码基因的位置关系(co-location)预测得到其潜在靶基因TNIP1(又名ABIN-1),可通过结合A20调节对NF-κB信号的抑制作用,与细胞的生长分化以及动物胚胎发育相关[31],并且是一个影响长白猪产仔性状的候选基因[32];lncRNA与蛋白编码基因表达相关性(co-expression)预测得到其潜在靶基因CYP2J2、SCARB1、HSD3B1、HSD17B2、HSD17B7参与卵巢类固醇激素合成信号传导,参与卵泡发育及排卵过程,COL11A2、FGFR1、IBSP参与PI3K-Akt信号通路,而PI3K-Akt通路在哺乳动物卵泡发生和排卵的过程中起着关键的调控作用[33-34]。因此,lncRNA-ENSSSCT00000018610可能通过对其靶基因的调控作用间接参与卵泡发育过程,但lncRNA-ENSSSCT00000018610与预测得到的蛋白质编码基因之间的靶向关系仍需进一步验证。
4 结论lncRNA-ENSSSCT00000018610是一个不具编码潜能的非编码RNA,RACE克隆获得其全长序列1 731 bp。lncRNA-ENSSSCT00000018610在猪肾、子宫、卵巢中表达水平相对较高。lncRNA-ENSSSCT00000018610在梅山猪M2卵泡中的表达量显著高于S、M1、L卵泡,在杜洛克猪M1、M2卵泡中的表达量极显著高于S和L卵泡,在杜洛克猪S、L卵泡中的表达量分别显著高于梅山猪,在杜洛克猪M1、M2卵泡中的表达量分别极显著高于梅山猪。靶基因预测结果说明,lncRNA-ENSSSCT00000018610可能通过对其靶基因的调控作用间接参与卵泡发育过程。该试验对lncRNA-ENSSSCT00000018610的研究为进一步揭示其在猪卵泡发育中的作用机制提供了重要参考依据。
| [1] | MERCER T R, DINGER M E, MATTICK J S. Long non-coding RNAs:insights into functions[J]. Nat Rev Genet, 2009, 10(3): 155–159. DOI: 10.1038/nrg2521 |
| [2] | YANG L, DUFF M O, GRAVELEY B R, et al. Genomewide characterization of non-polyadenylated RNAs[J]. Genome Biol, 2011, 12(2): R16. DOI: 10.1186/gb-2011-12-2-r16 |
| [3] | PONTING C P, OLIVER P L, REIK W. Evolution and functions of long noncoding RNAs[J]. Cell, 2009, 136(4): 629–641. DOI: 10.1016/j.cell.2009.02.006 |
| [4] | VERGARA I A, ERHO N, TRICHE T J, et al. Genomic "Dark Matter" in prostate cancer:exploring the clinical utility of ncRNA as biomarkers[J]. Front Genet, 2012, 3: 23. |
| [5] |
吴昊, 张运海, 凌英会. 非编码RNA在胚胎发育过程中的作用[J]. 畜牧兽医学报, 2017, 48(1): 1–7.
WU H, ZHANG Y H, LING H Y. The progress of recent advances in the ncRNA-mediated regulation during embryogenesis[J]. Acta Veterinaria et Zootechnica Sinica, 2017, 48(1): 1–7. (in Chinese) |
| [6] | NAKAYA H I, AMARAL P P, LOURO R, et al. Genome mapping and expression analyses of human intronic noncoding RNAs reveal tissue-specific patterns and enrichment in genes related to regulation of transcription[J]. Genome Biol, 2007, 8(3): R43. DOI: 10.1186/gb-2007-8-3-r43 |
| [7] | STRUHL K. Transcriptional noise and the fidelity of initiation by RNA polymerase Ⅱ[J]. Nat Struct Mol Biol, 2007, 14(2): 103–105. DOI: 10.1038/nsmb0207-103 |
| [8] | DERRIEN T, JOHNSON R, BUSSOTTI G, et al. The GENCODE v7 catalog of human long noncoding RNAs:analysis of their gene structure, evolution, and expression[J]. Genome Res, 2012, 22(9): 1775–1789. DOI: 10.1101/gr.132159.111 |
| [9] |
杨峰, 易凡, 曹慧青, 等. 长链非编码RNA研究进展[J]. 遗传, 2014, 36(5): 456–468.
YANG F, YI F, CAO H Q, et al. The emerging landscape of long non-coding RNAs[J]. Hereditas, 2014, 36(5): 456–468. (in Chinese) |
| [10] | YAN L Y, YANG M Y, GUO H S, et al. Single-cell RNA-Seq profiling of human preimplantation embryos and embryonic stem cells[J]. Nat Struct Mol Biol, 2013, 20(9): 1131–1139. DOI: 10.1038/nsmb.2660 |
| [11] | HAMAZAKI N, UESAKA M, NAKASHIMA K, et al. Gene activation-associated long noncoding RNAs function in mouse preimplantation development[J]. Development, 2015, 142(5): 910–920. DOI: 10.1242/dev.116996 |
| [12] | CABALLERO J, GILBERT I, FOURNIER E, et al. Exploring the function of long non-coding RNA in the development of bovine early embryos[J]. Reprod Fertil Dev, 2015, 27(1): 40–52. DOI: 10.1071/RD14338 |
| [13] | KANEKO S, SON J, SHEN S S, et al. PRC2 binds active promoters and contacts nascent RNAs in embryonic stem cells[J]. Nat Struct Mol Biol, 2013, 20(11): 1258–1264. DOI: 10.1038/nsmb.2700 |
| [14] | CHEN H J, PALMER J S, THIAGARAJAN R D, et al. Identification of novel markers of mouse fetal ovary development[J]. PLoS One, 2012, 7(7): e41683. DOI: 10.1371/journal.pone.0041683 |
| [15] | ROESZLER K N, ITMAN C, SINCLAIR A H, et al. The long non-coding RNA, MHM, plays a role in chicken embryonic development, including gonadogenesis[J]. Dev Biol, 2012, 366(2): 317–326. DOI: 10.1016/j.ydbio.2012.03.025 |
| [16] | BROWN J B, BOLEY N, EISMAN R, et al. Diversity and dynamics of the Drosophila transcriptome[J]. Nature, 2014, 512(7515): 393–399. DOI: 10.1038/nature12962 |
| [17] | MACAULAY A D, GILBERT I, CABALLERO J, et al. The gametic synapse:RNA transfer to the bovine oocyte[J]. Biol Reprod, 2014, 91(4): 90. |
| [18] |
马力鹏.杜洛克猪与梅山猪中等猪卵泡差异表达lncRNA和mRNA的鉴定研究[D].石河子: 河子大学, 2017.
MA L P. Identification of differentially expressed lncRNA and mRNA between medium follicles in Duroc and Meishan Pig[D].Shihezi: Shihezi University, 2017.(in Chinese) http://kns.cnki.net/KCMS/detail/detail.aspx?filename=GWXK201702029&dbname=CJFD&dbcode=CJFQ |
| [19] | WANG L G, PARK H J, DASARI S, et al. CPAT:coding-potential assessment tool using an alignment-free logistic regression model[J]. Nucleic Acids Res, 2013, 41(6): e74. DOI: 10.1093/nar/gkt006 |
| [20] | ZHOU Z Y, LI A M, ADEOLA A C, et al. Genome-wide identification of long intergenic noncoding RNA genes and their potential association with domestication in pigs[J]. Genome Biol Evol, 2014, 6(6): 1387–1392. DOI: 10.1093/gbe/evu113 |
| [21] | HUNG T, CHANG H Y. Long noncoding RNA in genome regulation:prospects and mechanisms[J]. RNA Biol, 2010, 7(5): 582–585. DOI: 10.4161/rna.7.5.13216 |
| [22] | LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the method[J]. Methods, 2001, 25(4): 402–408. DOI: 10.1006/meth.2001.1262 |
| [23] |
林海军.胚胎中lncRNA的预测及其在胚胎发育过程中的功能分析[D].南昌: 南昌大学, 2015.
LIN H J.The prediction and functional analysis of lncRNA in embryonic development process[D].Nanchang: Nanchang University, 2015.(in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10403-1015627930.htm |
| [24] | WANG Y Y, XUE S Y, LIU X R, et al. Analyses of long non-coding RNA and mRNA profiling using RNA sequencing during the pre-implantation phases in pig endometrium[J]. Sci Rep, 2016, 6: 20238. DOI: 10.1038/srep20238 |
| [25] | LIANU M, LI W Q, TIAN H, et al. Sequential expression of long noncoding RNA as mRNA gene expression in specific stages of mouse spermatogenesis[J]. Sci Rep, 2014, 4: 5966. |
| [26] | WASHIETL S, HOFACKER I L, STADLER P F. Fast and reliable prediction of noncoding RNAs[J]. Proc Natl Acad Sci U S A, 2015, 102(7): 2454–2459. |
| [27] |
王羽.基于序列-结构信息的长非编码RNA预测方法[D].西安: 西安电子科技大学, 2014.
WANG Y.Method for lncRNA prediction based on sequence-structure information[D].Xi'an: Xidian University, 2014.(in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10701-1015437579.htm |
| [28] |
贾纯琰, 季小阳, 白雪, 等. 长链非编码RNA的调控机制及其在家畜中的预测方法[J]. 中国畜牧兽医, 2017, 44(7): 1895–1905.
JIA C Y, JI X Y, BAI X, et al. Regulatory mechanisms of long noncoding RNAs and forecasting methods in livestock[J]. China Animal Husbandry & Veterinary Medicine, 2017, 44(7): 1895–1905. (in Chinese) |
| [29] | DINGER M E, PAND K C, MERCER T R, et al. Differentiating protein-coding and noncoding RNA:challenges and ambiguities[J]. PLoS Comput Biol, 2008, 4(11): e1000176. DOI: 10.1371/journal.pcbi.1000176 |
| [30] | PAULI A, VALEN E, SCHIER A F, et al. Identifying(non-)coding RNAs and small peptides:challenges and opportunities[J]. Bioessays, 2015, 37(1): 103–112. DOI: 10.1002/bies.201400103 |
| [31] |
周晓宁, 何小梅, 贾新正, 等.ABIN-1基因5个SNP对长白母猪产仔数的影响[C]//第十二次全国畜禽遗传标记研讨会.南京: 中国畜牧兽医学会, 2010.
ZHOU X N, HE X M, JIA X Z, et al.Effect of 5 SNPs of ABIN-1 gene on litter size in Landrace Sows[C]//Twelfth National Symposium on Livestock and Poultry Genetic Markers.Nanjing: Chinese Animal Husbandry and Veterinary Association, 2010.(in Chinese) |
| [32] |
欧阳宏佳, 方梅霞, 周晓宁, 等.猪胚胎发育相关基因ABIN-1的cDNA克隆及表达[C]//中国动物遗传育种研究进展——第十五次全国动物遗传育种学术讨论会.杨凌: 中国畜牧兽医学会, 2009.
OUYANG H J, FANG M X, ZHOU X N, et al.cDNA cloning and expression of porcine embryonic development related gene ABIN-1[C]//Progress in Animal Genetics and Breeding in China: the 15th National Conference on Animal Genetics and Breeding Collection.Yangling: Chinese Animal Husbandry and Veterinary Association, 2009.(in Chinese) |
| [33] | SHI F, LAPOLT P S. Relationship between FoxO1 protein levels and follicular development, atresia, and luteinization in the rat ovary[J]. J Endocrinol, 2003, 179(2): 195–203. DOI: 10.1677/joe.0.1790195 |
| [34] |
丁威.二花脸猪卵泡形成和早期发育的特性及其相关通路的研究[D].南京: 南京农业大学, 2010.
DING W.Study on characteristics and signaling pathways involved in follicular formation and early development of erhualian pigs[D].Nanjing: Nanjing Agricultural University, 2010.(in Chinese) http://cdmd.cnki.com.cn/article/cdmd-10307-1012271046.htm |



