铜绿假单胞菌(Pseudomonas aeruginosa)是一种革兰阴性菌,广泛存在于不同的环境中,可以从人和动物各种生物体中分离出来,并且对外界环境具有较高的耐受性,因此,铜绿假单胞菌可以在各种生活及医院环境中生存,经常导致人和动物术后伤口感染,且烧伤后感染可造成死亡[1]。铜绿假单胞菌可引起皮肤病、肺炎、尿路感染、血液感染、慢性支气管炎以及骨髓炎等严重疾病[2]。
大肠杆菌(Escherichia coli)通常存在于人和动物肠道中,可以导致人兽共患病,也是临床上最容易产生耐药性的革兰阴性菌[3]。在兽医上大肠杆菌会引起牛、猪和鸡等动物患上仔猪水肿、白痢、黄痢以及气囊炎、腹膜炎、心包炎、卵巢炎等疾病,给养殖业带来极大损失。同时,人类群体感染大肠杆菌会引发腹泻、胆囊炎、尿道炎、结肠炎和败血症等病症[4]。
近年来,由于抗生素在世界范围内的广泛使用,铜绿假单胞菌和大肠杆菌的耐药性也逐渐增强,特别是对β-内酰胺类抗生素的耐药性日趋严重,多重耐药菌也开始出现[5]。因此,采用新技术开发新型抗菌药物十分有必要。
青霉素结合蛋白(penicillin binding proteins,PBPs),即黏肽合成酶,是一种对细菌的生长繁殖发挥着重要作用的蛋白,存在于细菌的细胞膜上,具有羧肽酶和转肽酶的活性,是细菌细胞壁肽聚糖合成过程中不可或缺的一种酶,也是青霉素等β-内酰胺类抗生素作用的关键靶位[6]。按相对分子质量区分,PBPs分为高相对分子质量黏肽合成酶(HMM)和低相对分子质量黏肽合成酶(LMM)[7]。铜绿假单胞菌黏肽合成酶PBP3和大肠杆菌黏肽合成酶PBP1b均为HMM青霉素结合蛋白,是细菌生长繁殖和细胞分裂过程中至关重要的蛋白,是合成细菌细胞壁肽聚糖的不可缺少的必需酶[8-9]。大肠杆菌PBP4、PBP5均为LMM黏肽合成酶,这两种PBPs不是维持细菌生长和繁殖的关键蛋白,但具有羧肽酶活性,对稳定蛋白质和肽聚糖的结构也有一定作用[10-12]。由于PBPs在肽聚糖合成中的重要作用,在新型抗菌药物的开发上,PBPs是一个十分有吸引力的靶点[13]。
铜绿假单胞菌和大肠杆菌对β-内酰胺类抗生素的主要耐药机制之一是β-内酰胺酶的产生[14-16]。青霉素等β-内酰胺类抗生素含有特殊结构β-内酰胺环,通过抑制细菌细胞壁PBPs的合成,使细菌裂解死亡来发挥抗菌作用,而β-内酰胺酶的产生导致β-内酰胺环被破坏,进而使抗生素失效,细菌产生耐药性[17-18]。此外,抗菌药物作用靶位的改变,即PBPs发生改变,以及细菌外膜渗透性降低和外膜主动泵出系统也会使细菌产生耐药性。
先导化合物是指本身具有一定活性,但同时由于较大的毒性或活性仍然有待提高,需要采取一定的措施对其结构进行改善和优化再进行实际应用的药物[19]。随着生物学和计算机技术的发展,通过计算机进行虚拟筛选从而发现先导化合物已成为新药研发的一种常用方法。虚拟筛选是利用高性能计算对大型化合物数据库进行分析,从而选择出可能的候选药物的一种技术[20-21]。在虚拟筛选的过程中,使用分子对接的方法找到具有活性的化合物是一种常用的手段。分子对接就是将大量的小分子依次放入到受体蛋白的活性位点,按照形状互补和能量互补等原则不断调整和优化受体和配体的位置以及自身构象,使二者在结构、空间和电荷上均达到最佳的匹配模式[22]。对比高通量的筛选技术,采用虚拟筛选不仅成本低,可行性更高,而且可以在更短时间内发现结构新颖的有潜力的先导化合物[23]。
根据青霉素结合蛋白与β-内酰胺类抗生素的结合原理,本研究选择铜绿假单胞菌PBP3以及大肠杆菌PBP1b、PBP4、PBP5为靶点,采用分子对接技术在数目庞大的小分子化合物的数据库中进行虚拟筛选,以期望找到与靶点亲和力高的先导化合物。之后,通过有机合成制备先导化合物,并验证其抗菌活性。
1 材料与方法 1.1 材料 1.1.1 DOCK程序、ZINC数据库和Chimera程序DOCK程序由美国加利福尼亚大学Kuntz等开发,本研究选用DOCK6.5版本。ZINC数据库为筛选所用的小分子化合物的来源(http://zinc.docking.orghttp://zinc.docking.org),选择其中的drug-like子数据库,化合物数量约580万。Chimera程序由加州大学旧金山分校建立,是一个交互式分子模型显示和分析程序,本研究中主要用于处理受体和配体以及显示对接结果。
1.1.2 菌种与培养基铜绿假单胞菌标准株CMCC10104、大肠杆菌标准株CMCC44102来自中国医学微生物菌种保藏中心,枯草芽胞杆菌(Bacillus subtilis)、单增李斯特菌(Listeria monocytogenes)、金黄色葡萄球菌(Staphylococcus aureus)来自浙江工商大学食品与生物学院;铜绿假单胞菌PBP3和大肠杆菌PBP1b的基因工程菌株为本实验室所保存;质控菌株为大肠杆菌标准株CMCC44102;MH和LB肉汤培养基购自杭州微生物试剂有限公司。
1.2 主要试剂与设备三聚氯氰(2,4,6-三氯-1,3,5三嗪)、N-N二异丙基乙胺(DIPEA)购于上海阿拉丁生化科技有限公司;甘氨酸购于上海麦克林生化科技有限公司;丙酮购于浙江三鹰化学试剂有限公司;二氯甲烷、乙酸乙酯购于无锡市展望化工试剂股份有限公司;盐酸、石油醚、氢氧化钠购于天津市永大试剂公司。旋转蒸发仪IKA RV10购于德国艾卡公司,JJ-CT-IFD洁净工作台(苏州市金净净化设备科技有限公司),LDEX-50KB立式压力蒸汽灭菌锅(上海申安医疗器械厂)。
1.3 虚拟筛选 1.3.1 生成受体和配体文件以铜绿假单胞菌黏肽合成酶PBP3为例,从protein data bank网站(http://www.pdb.org/pdb/home/home.do)上搜索并下载3PBQ文件(3PBQ.pdb),即铜绿假单胞菌青霉素结合蛋白PBP3与亚胺培南结合的复合物(亚胺培南结合在PBP3活性位点上)。运行Chimera程序,删除该复合物中的配体亚胺培南,保留受体PBP3,再对受体进行去溶剂、加氢、加电荷以及修改错误的氨基酸命名等操作,并保存为mol2格式的受体文件。再对受体进行去氢,保存其pdb文件。然后,再将该复合物删除受体PBP3,仅保留配体亚胺培南,同样进行加氢和加电荷等处理,保存为mol2格式文件。大肠杆菌黏肽合成酶PBP1b、PBP4、PBP5依上述方法分别下载3FWM、2EXA、3MZE文件,复合物中所含配体分别为默诺霉素、法罗培南和头孢西丁。对这些受体和配体如上处理,然后保存为相应文件。
1.3.2 球集的生成受体分子表面分布着很多凹槽,这些凹槽投射出的阴影称之为球集。选择距离亚胺培南、默诺霉素、法罗培南和头孢西丁等配体结合位置半径5 Å范围内的球集作为对接时的靶点,对不符合要求的球集进行删除处理,并生成盒子文件,用于约束后续分子对接中配体的结合位点。
1.3.3 Grid文件的生成在分子对接时,需要对受体和配体之间的相互作用能及结合能进行高性能计算,即进行dock的能量打分,包括对分子间静电引力,氢键作用、疏水作用和范德华力等相互作用能进行评价。在本阶段生成打分用的Grid文件。
1.3.4 分子对接分子对接就是将小分子化合物逐个放入到受体蛋白中的靶点部位,按照形状互补和能量互补等原则不断优化受体和配体的构象,使二者达到最佳的匹配模式。在本研究中,是将化合物数据库中的小分子分别与铜绿假单胞菌PBP3、大肠杆菌PBP1b、PBP4和PBP5对接,对接部位即前面选择的靶点球集,并进行位置和构象的不断调整和能量打分。
1.3.5 第一轮筛选第一轮筛选中,受体的设置选择刚性,配体选择柔性,并用Grid score对受体和配体之间的亲和力进行评价,对接过程中受体与数据库中的每个小分子都会获得最佳的空间匹配模式和最好的结合构象,Grid score会按照分数由大到小的顺序排列,选取其中分数好并且结构也比较好的化合物进入第二轮筛选。
1.3.6 第二轮筛选第二轮筛选使用Amber score,对上一轮筛选得到的得分较高的每个小分子化合物进行更细致的打分,在这一步中,受体也被设置为柔性,MD与能力优化等参数均使用默认值。结束计算后,按照Amber score分数大小进行排列,从中选取分数较好并且结构新颖的化合物进行有机合成。
1.4 合成先导化合物 1.4.1 2,4-二氯-6-哌啶基-1,3,5-均三嗪的合成[24]取三聚氯氰5.0 g(27.10 mmol)于100 mL二氯甲烷中搅拌溶解,再取50 mL二氯甲烷,加入2.68 mL(27.12 mmol)哌啶和5.2 mL(29.85 mmol)DIPEA,在0 ℃条件下将溶有三聚氯氰的二氯甲烷于1 h内逐滴加入其中,同时维持整个反应在0 ℃下进行,2 h后结束反应,溶液用浓盐酸(2×100 mL)、水(100 mL)分别进行水洗,分层过滤,取二氯甲烷层,用旋转蒸发仪去除二氯甲烷,干燥得到淡黄色固体粉末,产率81%。硅胶柱层析(石油醚:乙酸乙酯=9:1)。
1.4.2 2,4-二甘氨酸-6-哌啶基-1,3,5-均三嗪的合成[25]取上一步合成得到的2,4-二氯-6-哌啶基-1,3,5-均三嗪粉末4.66 g(20 mmol)溶解于丙酮中,再取3.45 g(46 mmol)甘氨酸和6.6 g(165 mmol)NaOH溶解于50 mL水中,于1 h内在70 ℃条件下逐滴加入到丙酮溶液中,加热4 h后冷却到室温,并在室温下搅拌1 h,结束后用浓盐酸调节pH至2左右,将装置移到0~5 ℃的冰浴中保持20 min,析出固体,过滤,10 mL蒸馏水洗3次,10 mL丙酮洗2次,待丙酮挥发后置于70 ℃烘箱烘干,干燥纯化后得到白色固体粉末,产率62%。硅胶柱层析(二氯甲烷:甲醇=5:1;二氯甲烷:甲醇=1:3)。
反应流程如图 1所示。
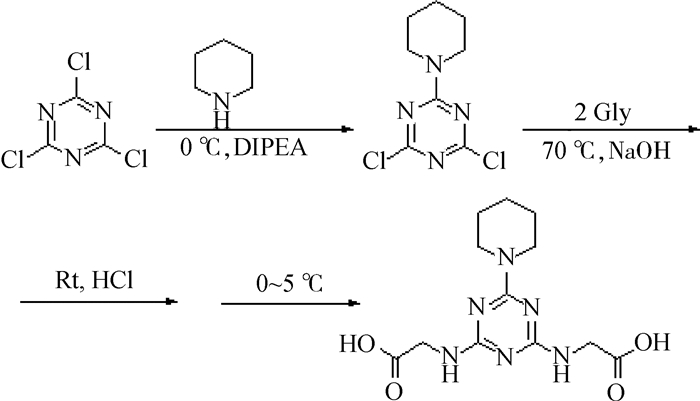
|
图 1 先导化合物合成流程图 Figure 1 Synthesis flow chart of lead compound |
将P. Aeruginosa、E. coli、B. subtilis、L. monocytogenes以及S. aureus加入到MH肉汤培养基中,37 ℃、180 r·min-1摇床培养过夜后稀释至0.5麦氏浊度(在良好的光照条件下将试管与0.5麦氏比浊管同时置于画有黑色平行线条的白色纸上进行对比),菌悬液浓度约为1×108 CFU·mL-1,待用。
1.5.2 不同浓度药物的制备将先导化合物、磺胺嘧啶分别用蒸馏水和DMSO溶解,配制成2 mg·mL-1的溶液,其中,向溶解情况不太好的溶液中加入0.2%的吐温80助溶;将氨苄青霉素钠用蒸馏水配制成0.8 mg·mL-1的溶液,待用。
1.5.3 最小抑菌浓度(MIC)的测定将配制好的先导化合物和磺胺嘧啶溶液分别在试管中用MH肉汤稀释为0、150、175、200、225、250和500 μg·mL-1,将氨苄青霉素钠同样用MH肉汤稀释为0、12.5、25、50、100、200 μg·mL-1,每管加入100 μL菌液,试验中设空白对照组,加10 mL的MH肉汤,不加菌悬液,将试管置于35 ℃的恒温培养箱中12 h培养,每种供试菌做三组平行试验。
1.6 先导化合物与PBPs的结合参照喻召武[26]的方法,将先导化合物携带的羧基转化为酰胺基,然后将转化后的产物与荧光染料NBD-Cl进行混合,制成荧光探针。以本实验室保存的PBP3和PBP1b基因工程菌株来制备PBP3和PBP1b蛋白,参照安艳冬[27]的方法诱导蛋白的表达,之后,高速离心,弃上清,菌体沉淀以50 mmoL·L-1 Tris-HCl缓冲液(pH=8.0)重悬,混匀后于超声破碎仪中破碎(超声时间3 s,间隔3 s,共100次,超声功率400 w),结束后在4 ℃、8 000 r·min-1条件下离心10 min,取上清液4 ℃保存。再取100 μL上清液加入200 μL荧光探针,并置于37 ℃恒温摇床中孵育2 h,使蛋白与荧光探针充分接触,然后取二者混合物100 μL并加入100 μL上样缓冲液,煮沸5 min,SDS-PAGE电泳,紫外光下观察荧光条带。
2 结果 2.1 受体与配体文件以铜绿假单胞菌PBP3为例,从protein data bank网站下载铜绿假单胞菌黏肽合成酶PBP3与亚胺培南复合物晶体结构(图 2a),对复合物进行除配体、去溶剂、加氢、加电荷等处理,得到单独的受体蛋白PBP3文件。然后进行配体准备工作,删除受体、加氢和加电荷等处理,得到配体文件。大肠杆菌黏肽合成酶的晶体结构PBP1b(图 2b)、PBP4(图 2c)、PBP5(图 2d)亦如上处理。
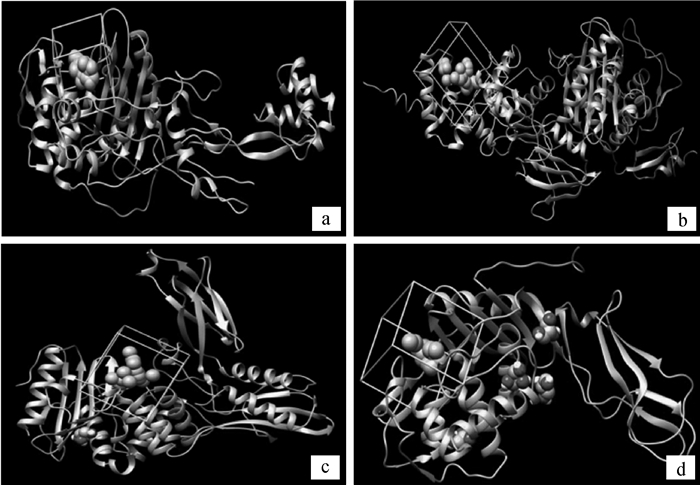
|
a、b、c、d展示了受体蛋白P. aeruginosa PBP3和E. coli PBP1b、PBP4、PBP5 Panels a, b, c and d represent the profiles of P. aeruginosa PBP3 and E. coli PBP1b, PBP4 and PBP5 图 2 铜绿假单胞菌PBP3及大肠杆菌PBP1b、PBP4、PBP5的晶体结构和靶点球集 Figure 2 Crystal structures and binding spheres of P. aeruginosa PBP3 and E. coli PBP1b, PBP4 and PBP5 |
以受体上原有配体的位置为基点,比如,以结合在铜绿假单胞菌PBP3上的亚胺培南的位置为基点,在sphgen程序中将筛选的球集范围定为5Å,筛选出21个球集作为对接时的靶点,同时在球集5Å的范围内设置一个盒子,以约束对接时的结合位点范围。大肠杆菌黏肽合成酶PBP1b、PBP4、PBP5均按上述方法处理,球集及盒子情况见图 2。
2.3 第一轮筛选在第一轮筛选中,使用Grid score对数据库中的小分子化合物与受体蛋白的亲和力进行了评价。经过计算,筛选出得分在-125.6 kJ·mol-1(-30 kcal·mol-1)以下的约6万个小分子化合物,进入第二轮筛选。
2.4 第二轮筛选使用Amber score作为第二轮筛选的打分方法,对上一步筛选得到的化合物进一步打分。Amber score的计算速度较慢,但优点是受体和配体均被设置为柔性,即可以自由变换本身构象,因而,二者可以经不断的改变而使结合更加契合和精准,所以Amber score的打分更加全面和精细。计算结束后,筛选出得分在-83.7 kJ·mol-1(-20 kcal·mol-1)以下的化合物约200个。经结构分析,发现很多化合物结构非常复杂,难以合成。所以,选取了其中1种得分较高、结构新颖并且易于合成的先导化合物用于后续研究,其得分情况如表 1所示。
|
|
表 1 筛选出的先导化合物的得分 Table 1 Scoring of selected lead compounds |
从表 1可以看出,在虚拟筛选过程中,化合物经第二轮筛选Amber score打分后获得较高的分数时,其在第一轮初筛时获得的Grid score的分数不一定会很高。同样的,在Grid score打分阶段获得高分的小分子化合物,当进入第二轮筛选经Amber score打分后,得到的最终分数也不一定会很高。这是由于在进行第二轮虚拟筛选时,配体和受体均为柔性,从而使二者的结合更加精准。而在第一轮初筛时,受体的设置是刚性,即在对接中受体的构象不允许发生变化,只有配体可以自由变化其构象,因而Grid score和Amber score的打分情况并不能完全吻合。虽然Grid score的打分不够精确,但是其运算速度快,可以快速剔除形态上和受体不匹配的化合物,大大提高了筛选的效率。而第二轮的Amber score打分精确,保证了筛选的精度。因此,结合Grid score和Amber score两轮打分,就可以从大量候选化合物中快速筛选出有潜力的先导化合物。
2.5 先导化合物与受体蛋白的结合使用chimera软件对先导化合物和受体蛋白PBP3、PBP1b、PBP4、PBP5的结合情况进行观察与分析,结果如图 3所示。
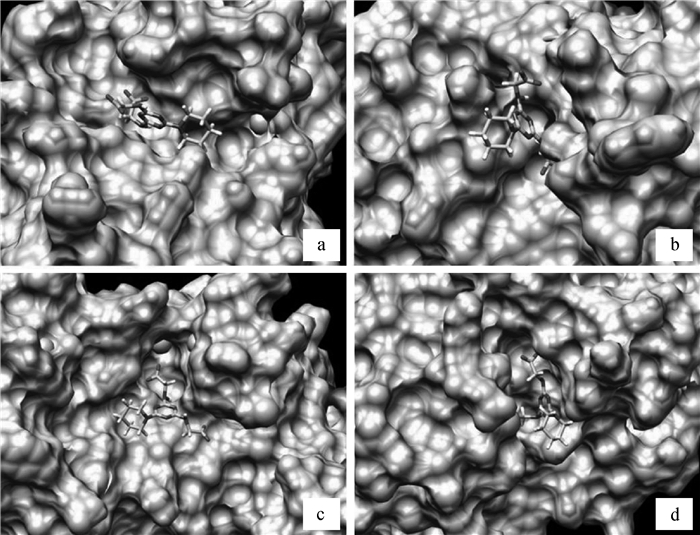
|
a、b、c、d展示了受体蛋白P. aeruginosa PBP3和E. coli PBP1b、PBP4、PBP5 Panels a, b, c and d represent the receptor protein of P. aeruginosa PBP3 and E. coli PBP1b, PBP4 and PBP5 图 3 筛选出的先导化合物与受体的结合情况 Figure 3 Binding of the lead compound with the receptors |
在虚拟筛选阶段下载pdb文件时,选择的分别是PBP3与亚胺培南((5R, 6S)-6-[(1R)-1-羟乙基]-3-[[2-[(亚氨基甲基)氨基]乙基]硫代]-7-氧代-1-氮杂双环[3.2.0]庚-2-烯-2-羧酸)、PBP1b与默诺霉素、PBP4与法罗培南([5R-[3(R^^),5Α,6Α(R^^)]]-6-(1-羟基乙基)-7-氧-3-(四氢-2-呋哺基)-4-硫-1-氮杂双环[3.2.0]庚-2-烯-2-羧酸)以及PBP5与头孢西丁(3-氨基甲酰基氧甲基-7-甲氧基-7-(2-噻吩乙酰胺基)-8-氧代-5-硫-1-杂氮双环[4.2.0]辛-2-烯-2-甲酸)的复合物文件,这些药物本身就结合在相应蛋白的活性位点上,在虚拟筛选时只是去除这些药物,然后将化合物放入药物原本的结合位置上。如图 3所示,可以看出筛选出的先导化合物均结合在受体蛋白铜绿假单胞菌PBP3、大肠杆菌PBP1b、PBP4和PBP5的凹槽处,即相应的活性位点上,而且受体与配体之间的形态匹配比较好。
此外,采用ligplot对受体蛋白和先导化合物之间的相互作用进行了进一步分析。从图 4可以看到,筛选出的这种先导化合物与受体之间普遍存在较多数目的疏水相互作用和氢键作用。其中,先导化合物与铜绿假单胞菌PBP3之间有4个氢键、2个疏水相互作用;与大肠杆菌PBP1b和PBP4之间有2个氢键、1个疏水作用以及4个氢键、2个疏水作用;与大肠杆菌PBP5之间相互作用最多,分别为4个氢键作用和6个疏水相互作用。这些较多的相互作用表明先导化合物与受体之间具有较高的亲和力。
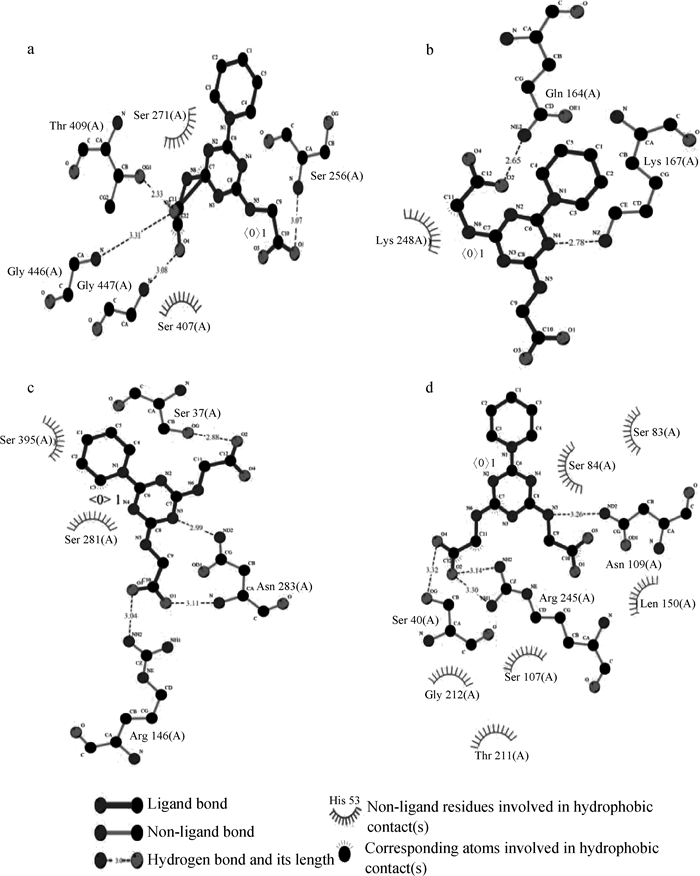
|
a、b、c、d展示了受体蛋白P. aeruginosa PBP3和E. coli PBP1b、PBP4、PBP5 Panels a, b, c and d represent the receptor protein of P. aeruginosa PBP3 and E. coli PBP1b, PBP4 and PBP5 图 4 筛选出的先导化合物与受体的相互作用 Figure 4 The interactions between the lead compound and the receptors |
根据虚拟筛选的结果,选择化合物ZINC00053282进行了有机合成(图 1)。之后,将先导化合物溶于氘带重水,并加入2%氢氧化钠,通过核磁验证了结构,碳谱和氢谱的结果分别如图 5、6所示。
|
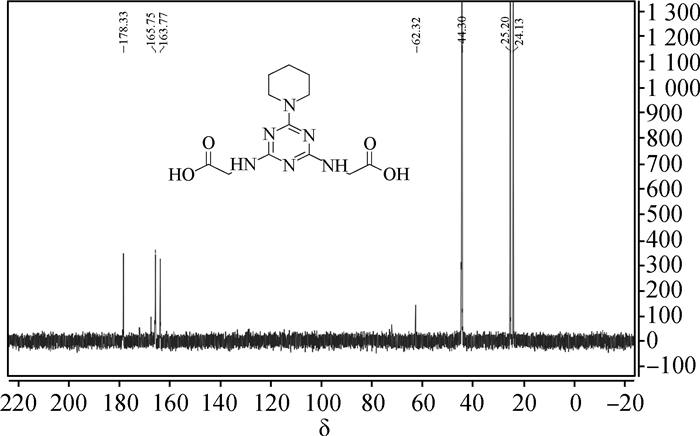
13C NMR (126 MHz, D2O) δ 178.33 (s), 165.75 (s), 163.77 (s), 62.32 (s), 44.30 (s), 25.20 (s), 24.13 (s) 图 5 先导化合物ZINC00053282碳谱图 Figure 5 13C NMR of lead compound ZINC00053282 |
|
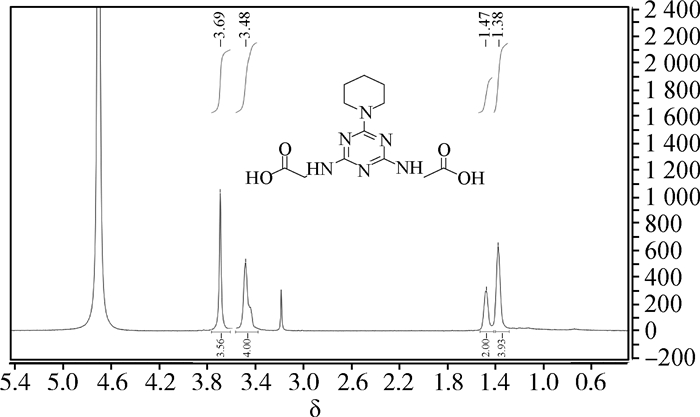
1H NMR (500 MHz, D2O) δ 3.69 (s, 4H), 3.48 (s, 4H), 1.47 (s, 2H), 1.38 (s, 4H) 图 6 先导化合物ZINC00053282氢谱图 Figure 6 1H NMR of lead compound ZINC00053282 |
分别测定了合成的先导化合物、磺胺嘧啶和氨苄青霉素钠对P. aeruginosa、E.coli、B.subtilis、L.monocytogenes以及S.aureus的最小抑菌浓度(MIC),结果见表 2。
|
|
表 2 先导化合物的最小抑菌浓度 Table 2 Minimum inhibitory concentration of lead compounds |
结果表明,合成的先导化合物对5种供试菌均具有抑菌活性,抑菌浓度在175~250 μg·mL-1之间,这说明合成出的先导化合物对革兰阳性菌和革兰阴性菌均有抑菌活性,属于广谱抗菌作用。同时,从表中可以看出先导化合物对P. aeruginosa和E. coli的抑菌效果比B. subtilis、L.monocytogenes和S. aureus的抑菌效果要好,分别为200和175 μg·mL-1,其中对S. aureus的抑菌活性最低,为250 μg·mL-1,对B. subtilis和L. monocytogenes的最小抑菌浓度均为225 μg·mL-1,这说明先导化合物对革兰阴性菌的抑菌活性要高于革兰阳性菌,其原因可能是我们在虚拟筛选过程中选择的靶点是革兰阴性菌的青霉素结合蛋白,也有可能是由于筛选出的先导化合物对革兰阴性菌的青霉素结合蛋白亲和力更高。此外,从表 2中可以看出,作为广谱抗菌药的磺胺嘧啶对5种菌的最小抑菌浓度均为500 μg·mL-1,说明先导化合物的广谱抑菌活性比磺胺嘧啶更高。虽然先导化合物抑菌效果不及氨苄青霉素钠(12.5 μg·mL-1),但是从结构上看,先导化合物不具有β-内酰胺环,而当前由于抗生素的β-内酰胺环结构被β-内酰胺酶水解,从而导致细菌产生耐药性的问题层出不穷,而我们合成的先导化合物没有这种结构,不会被β-内酰胺酶所水解,所以,可以考虑将这种先导化合物进行结构改造,进一步提高活性,用于耐药菌的治疗。
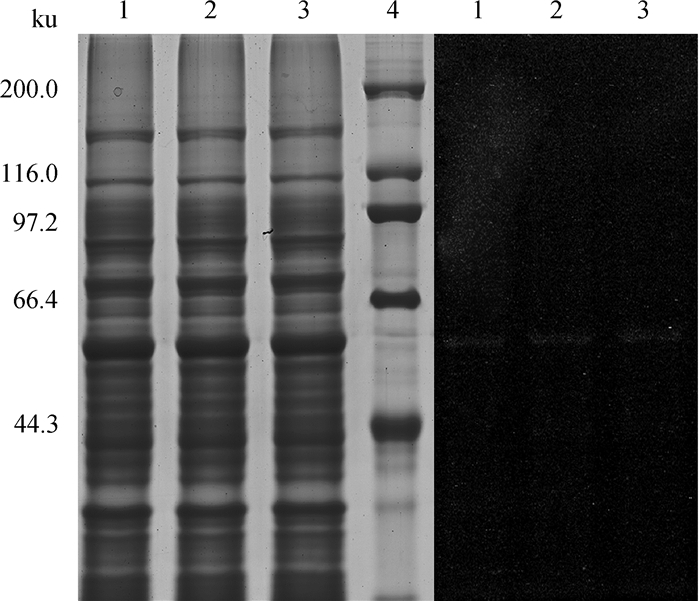
2.8 先导化合物与PBPs的结合目前,针对PBPs的药物开发通常都是通过探针的结合来推测作用机制。在本研究中,先将先导化合物和荧光试剂结合,制成荧光探针,然后与PBP3和PBP1b蛋白反应,经SDS-PAGE检测,得到的电泳图及荧光图分别如图 7和8所示。铜绿假单胞菌PBP3和大肠杆菌PBP1b的理论相对分子质量分别为60和80 ku,与图中所示一致。图 7中1~3为蛋白与荧光探针作用后的电泳条带,4为Marker蛋白,其中,对比荧光图可以看到,1~3泳道的条带在紫外下可以看到三条与之位置相对应的荧光条带,这证明先导化合物已经与PBP3相结合。
|
泳道1、2和3为蛋白与荧光探针作用后的电泳条带;4为相对分子质量标准(ku) Lane 1-3 represent electrophoretogram of PBP3 complex with fluorescent probe; Lane 4 represent marker 图 7 荧光探针与PBP3结合后的电泳图及荧光图 Figure 7 The electrophoretogram and fluorography of PBP3 combined with fluorescent probe |

|
泳道1、2为蛋白与荧光探针作用后的电泳条带;3为相对分子质量标准(ku) Lane 1-2 represent electrophoretogram of PBP1b complex with fluorescent probe; Lane 3 represent marker 图 8 荧光探针与PBP1b结合后的电泳图及荧光图 Figure 8 The electrophoretogram and fluorography of PBP1b combined with fluorescent probe |
同样的,图 8中1、2为蛋白与荧光探针作用后的电泳图,3为Mark蛋白,其中,泳道1、2中的条带与紫外下荧光图中两条荧光条带的位置相对应,同样证明先导化合物已与PBP1b结合,这说明筛选出的先导化合物可能是通过与PBP蛋白相结合,抑制其活性而发挥抗菌作用的。
3 讨论虚拟筛选是一种基于生物大分子的特定靶标,利用计算机进行辅助筛选,以找到具有活性的先导化合物的方法,当前虚拟筛选已经成为新药开发的重要手段,可以从几百万个分子中快速找出潜在的与靶蛋白具有相互作用的目标分子,大大地缩短了研究时间,提高了筛选效率。利用计算机辅助设计进行药物的大规模虚拟筛选,比传统的高通量筛选在速度、效率以及精准度上更加具有优势,已经成为替代高通量筛选的主要方法。目前,虚拟筛选的方法已取得了非常多的成就,比如,1999年用计算机虚拟筛选发现了3个活性较高的用于受体Muscarinic M3的拮抗剂分子[28];2002年通过虚拟筛选发现了多种能够抑制人碳酸酐酶的活性物质[29]。近年来,虚拟筛选在对阻断HIV Nef蛋白从而开发新型HIV药物[30],发现能够抑制神经氨酸酶流感病毒的药物[28],以及开发新型抗肿瘤药物[31]等方面均取得了重大的进展。
本研究针对当前人类和兽医临床上细菌对β-内酰胺类抗生素的耐药性问题,选择铜绿假单胞菌PBP3和大肠杆菌PBP1b、PBP4和PBP5为靶点,进行计算机辅助虚拟筛选,并对筛选出的编号为ZINC00053282的先导化合物进行了有机合成与抑菌活性研究,结果证明这种先导化合物对革兰阴性和阳性菌均有较好的抗菌作用,但对铜绿假单胞菌和大肠杆菌(175和200 μg·mL-1)的抑菌效果要比枯草芽胞杆菌、金黄色葡萄球菌和单增李斯特菌(225和250 μg·mL-1)更好,其原因可能是筛选的靶点来自于铜绿假单胞菌和大肠杆菌,也有可能是这种先导化合物相比革兰阳性菌,对革兰阴性菌更具有亲和力。
当然,作为先导化合物,目前的抗菌活性并不是很强,但是其结构新颖,与已有的β-内酰胺类等抗菌药物结构不同。在后续的研究中,经过进一步的结构改造,有望开发成为新型抗菌药物,用于耐药菌的治疗。
4 结论根据铜绿假单胞菌和大肠杆菌PBPs与β-内酰胺类抗生素的作用机制,以铜绿假单胞菌PBP3和大肠杆菌PBP1b、PBP4和PBP5等4个PBPs作为靶点,选择含有大量化合物的ZINC数据库来进行虚拟筛选,以期找到具有高亲和力的、能够抑制PBPs与底物结合,从而使细菌细胞壁无法合成而死亡的先导化合物。通过第一轮筛选,选择分值低于-125.6 kJ·mol-1的化合物,再以Amber score进行第二轮筛选,筛选出得分低于-83.7 kJ·mol-1的化合物约200个,经结构分析,选择编号为ZINC0053282的化合物进行了有机合成。经过核磁共振波谱仪的碳谱和氢谱验证,表明合成的化合物结构正确。抑菌试验的结果表明,合成的先导化合物抑菌效果较好,对革兰阴性和阳性菌都有效。由于该先导化合物结构新颖,没有β-内酰胺环,不会被β-内酰胺酶所降解,经进一步开发,可以用于对β-内酰胺类抗生素耐药的细菌的治疗。
| [1] | LIVERMORE D M. Has the era of untreatable infections arrived?[J]. J Antimicrob Chemother, 2009, 64(S1): i29–i36. |
| [2] | SUÁREZ C, PEÑA C, TUBAU F, et al. Clinical impact of imipenem-resistant Pseudomonas aeruginosa bloodstream infections[J]. J Infect, 2009, 58(4): 285–290. DOI: 10.1016/j.jinf.2009.02.010 |
| [3] |
韩伟, 张铁, 王春光, 等. 大肠埃希菌耐药机制研究进展[J]. 动物医学进展, 2006, 27(1): 51–53.
HAN W, ZHANG T, WANG C G, et al. Progress on the drug resistance of Escherichia coli[J]. Progress in Veterinary Medicine, 2006, 27(1): 51–53. (in Chinese) |
| [4] | VAN DEN BOGAARD A E, WILLEMS R, LONDON N, et al. Antibiotic resistance of faecal enterococci in poultry, poultry farmers and poultry slaughterers[J]. J Antimicrob Chemother, 2002, 49(3): 497–505. DOI: 10.1093/jac/49.3.497 |
| [5] | VANTARAKIS A, VENIERI D, KOMNINOU G, et al. Differentiation of faecal Escherichia coli from humans and animals by multiple antibiotic resistance analysis[J]. Lett Appl Microbiol, 2006, 42(1): 71–77. DOI: 10.1111/lam.2006.42.issue-1 |
| [6] | ZAPUN A, CONTRERAS-MARTEL C, VERNET T. Penicillin-binding proteins and β-lactam resistance[J]. FEMS Microbiol Rev, 2008, 32(2): 361–385. DOI: 10.1111/j.1574-6976.2007.00095.x |
| [7] |
熊亚莉, 范昕建. 青霉素结合蛋白研究进展[J]. 国外医药(抗生素分册), 2004, 25(5): 193–197.
XIONG Y L, FAN X J. Research progress of penicillin binding protein[J]. Foreign Medicine (Antibiotic Partition), 2004, 25(5): 193–197. (in Chinese) |
| [8] | CHEN W, ZHANG Y M, DAVIES C. Penicillin-binding protein 3 is essential for growth of Pseudomonas aeruginosa[J]. Antimicrob Agents Chemother, 2017, 61(1): 1616–1651. |
| [9] | PEPPER E D, FARRELL M J, FINKEL S E. Role of penicillin-binding protein 1b in competitive stationary-phase survival of Escherichia coli[J]. FEMS Microbiol Lett, 2006, 263(1): 61–67. DOI: 10.1111/fml.2006.263.issue-1 |
| [10] | MEBERG B M, PAULSON A L, PRIYADARSHINI R, et al. Endopeptidase penicillin-binding proteins 4 and 7 play auxiliary roles in determining uniform morphology of Escherichia coli[J]. J Bacteriol, 2004, 186(24): 8326–8336. DOI: 10.1128/JB.186.24.8326-8336.2004 |
| [11] | NELSON D E, YOUNG K D. Penicillin binding protein 5 affects cell diameter, contour, and morphology of Escherichia coli[J]. J Bacteriol, 2000, 182(6): 1714–1721. DOI: 10.1128/JB.182.6.1714-1721.2000 |
| [12] | GHOSH A S, YOUNG K D. Sequences near the active site in chimeric penicillin binding proteins 5 and 6 affect uniform morphology of Escherichia coli[J]. J Bacteriol, 2003, 185(7): 2178–2186. DOI: 10.1128/JB.185.7.2178-2186.2003 |
| [13] | ZERVOSEN A, SAUVAGE E, FRōRE J M, et al. Development of new drugs for an old target-the penicillin binding proteins[J]. Molecules (Basel, Switzerland), 2012, 17(12): 12478–12505. |
| [14] | JUAN C, MACIÁ M D, GUTIÉRREZ O, et al. Molecular mechanisms of β-lactam resistance mediated by AmpC hyperproduction in Pseudomonas aeruginosa clinical strains[J]. Antimicrob Agents Chemother, 2005, 49(11): 4733–4738. DOI: 10.1128/AAC.49.11.4733-4738.2005 |
| [15] | YORDANOV D, STRATEVA T. Pseudomonas aeruginosa-a phenomenon of bacterial resistance[J]. J Med Microbiol, 2009, 58(9): 1133–1148. DOI: 10.1099/jmm.0.009142-0 |
| [16] | SÁENZ Y, ZARAZAGA M, BRIÑAS L, et al. Antibiotic resistance in Escherichia coli isolates obtained from animals, foods and humans in Spain[J]. Int J Antimicrob Agents, 2001, 18(4): 353–358. DOI: 10.1016/S0924-8579(01)00422-8 |
| [17] | KONG K F, SCHNEPER L, MATHEE K. Beta-lactam antibiotics:From antibiosis to resistance and bacteriology[J]. APMIS, 2010, 118(1): 1–36. DOI: 10.1111/apm.2009.118.issue-1 |
| [18] |
张珍珍, 吴俊伟, 魏述永, 等. 动物源大肠杆菌超广谱β-内酰胺酶与头孢菌素酶基因型分析[J]. 畜牧兽医学报, 2009, 40(6): 898–903.
ZHANG Z Z, WU J W, WEI S Y, et al. Analysis of genotype of Escherichina coli-produacing ESBLs and Apmc β-lactamases isolated from farm animals[J]. Acta Veterinaria et Zootechnica Sinica, 2009, 40(6): 898–903. (in Chinese) |
| [19] |
叶德举, 罗小民, 沈建华, 等. 先导化合物的发现——整合计算机虚拟筛选、化学合成和生物测试方法[J]. 化学进展, 2007, 19(12): 1939–1946.
YE D J, LUO X M, SHEN J H, et al. Discovering potential drug leads via docking, synthesis and bioassay[J]. Progress in Chemistry, 2007, 19(12): 1939–1946. (in Chinese) |
| [20] | SHOICHET B K. Virtual screening of chemical libraries[J]. Nature, 2004, 432(7019): 862–865. DOI: 10.1038/nature03197 |
| [21] |
侯廷军. 计算机辅助药物分子设计方法研究[D]. 北京: 北京大学, 2002.
HOU T J. Study on computer-aided drug molecular design[D]. Beijing: Peking University, 2002. (in Chinese) |
| [22] | SPYRAKIS F, COZZINI P, KELLOGG G E, et al. Docking and scoring in drug discovery[M]//ABRAHAM D J, ROTELLA D P. Burger's medicinal chemistry and drug discovery. Canada: John Wiley & Sons, Inc., 2003: 601-684. |
| [23] | WASZKOWYCZ B, CLARK D E, GANCIA E. Outstanding challenges in protein-ligand docking and structure-based virtual screening[J]. Wiley Interdiscip Rev Comput Mol Sci, 2011, 1(2): 229–259. DOI: 10.1002/wcms.18 |
| [24] | IMPERIO D, GIOVENZANA G B, LAW G I, et al. Synthesis and comparative anion binding profiles of two di-aqua Eu(iii) complexes[J]. Dalton Trans, 2010, 39(41): 9897–9903. DOI: 10.1039/c0dt00773k |
| [25] | FUNFUENHA W, PHAKHODEE W, PATTARAWARAPAN M. Facile and efficient synthesis of C2-symmetrical 1, 3, 5-triazine polycarboxylate ligands under microwave irradiation[J]. Tetrahedron, 2014, 70(35): 5415–5419. DOI: 10.1016/j.tet.2014.06.127 |
| [26] |
喻召武. 靶向铜绿假单胞菌粘肽合成酶PBP3的抗菌先导化合物的虚拟筛选与活性研究[D]. 杭州: 浙江工商大学, 2016.
YU Z W. Virtual screening and activity studies of antimicrobial lead compounds targeting to penicillin-binding protein 3 of Pseudomonas aeruginosa[D]. Hangzhou: Zhejiang Gongshang University, 2016. (in Chinese) |
| [27] |
安艳冬. 大肠杆菌PBP1b和铜绿假单胞菌PBP3的表达纯化[D]. 杭州: 浙江工商大学, 2015.
AN Y D. Expression and purification of PBP1B from Escherichia coli and PPBP3 from Pseudomonas aeruginosa[D]. Hangzhou: Zhejiang Gongshang University, 2015. (in Chinese) |
| [28] |
吴可柱, 李昆, 李爱秀. 虚拟筛选技术与新药开发[J]. 武警医学院学报, 2011, 20(5): 415–419.
WU K Z, LI K, LI A X. Virtual screening and new drug discovery[J]. Journal of Armed Police Medical College, 2011, 20(5): 415–419. (in Chinese) |
| [29] | RECACHA R, COSTANZO M J, MARYANOFF B E, et al. Crystal structure of human carbonic anhydrase Ⅱ complexed with an anti-convulsant sugar sulphamate[J]. Biochem J, 2002, 361(3): 437–441. DOI: 10.1042/bj3610437 |
| [30] | GUIDO R V, OLIVA G, ANDRICOPULO A D. Virtual screening and its integration with modern drug design technologies[J]. Curr Med Chem, 2008, 15(1): 37–46. DOI: 10.2174/092986708783330683 |
| [31] | LIU A L, CAO H P, DU G H. Drug screening for influenza neuraminidase inhibitors[J]. Science in China Ser. C Life Sciences, 2005, 48(1): 1–5. DOI: 10.1360/062004-69 |

 图 1(Figure 1)
图 1(Figure 1)


