流感病毒为正黏病毒科流感病毒属的单股、负链、分节段RNA病毒,根据其基质蛋白(M)和核蛋白(NP),可分为A型(influenza A virus,IAV)、B型(influenza B virus,IBV)和C型(influenza C virus,ICV)流感病毒。IAVs有18个血凝素(HA)亚型和11个神经氨酸酶(NA)亚型,其不仅能感染野鸟和家禽,还可以感染人、猪、马、鲸等多种哺乳动物,引起感染动物的呼吸系统疾病。IBV只感染人类,主要引起人的季节性流行性感冒。ICV主要感染人类,常引起儿童的下呼吸道感染[1],郭元吉等[2]也曾从猪体内分离到该型流感病毒。
2011年,美国首次报道从表现流感症状的猪体内分离到D型流感病毒(influenza D virus,IDV),其与ICV在结构和基因组成上相似,但氨基酸相似性仅为50%左右,并且IDV抗体与IAV、IBV、ICV均无交叉反应性[3-4]。近几年来,多个国家或地区不断从猪、牛体内分离到IDVs,给畜牧业健康发展和公共卫生安全造成潜在威胁,作者就IDV病毒特性、流行病学、致病性、动物模型建立以及诊断和预防等方面进行如下综述。
1 D型流感病毒特性IDV基因组由7个片段组成,共编码PB2、PB1、P3、NP、HEF、M(M1和CM2)、NS(NEP和NS1)等9种蛋白[5],并且每个基因片段非编码区基因5′端(5′-AGCAGUAGCAAG-3′)和3′端(3′-C/UCGUAUUCGUC-5′)高度保守,几乎与ICV一致,但3′端第5位碱基不同,IDV为腺嘌呤(adenine,A),而ICV为胞嘧啶(cytosine,C)。这些非编码区基因之间反向互补形成的柄状结构在病毒粒子复制和包装过程中发挥关键作用,能够启动mRNAs的转录[6]。IAV和IBV病毒粒子表面均含有2个糖蛋白,即血凝素(HA)和神经氨酸酶(NA),而IDV同ICV一样表面只含有1个糖蛋白,即HEF。PB2、PB1和P3共同组成多聚酶复合体,NP蛋白和多聚酶复合体组成IDV的核糖核蛋白复合体(vRNPs)。ICV和IDV的M1蛋白是其外周膜蛋白的主要组成部分,都是通过RNA剪接后翻译而成,但剪接方式不同,ICV是在其mRNA上引入终止密码子,而IDV是在其外显子前面插入12个碱基;未剪接mRNA则翻译成p42蛋白前体。p42蛋白在其内部信号肽作用下裂解产生跨膜蛋白CM2,作为RNPs进入宿主细胞内进行复制的离子通道。NS通过剪接产生NS1和NEP,NEP是一种核输出蛋白,能介导RNPs输出细胞核[6-7]。
与IAV、IBV相比,IDV与ICV在结构和组成上较为接近,但氨基酸相似性仅为50%左右。分子进化分析表明IDV可能与ICV来自共同的祖先,也可能是由ICV进化而来。贝叶斯分子钟分析推断IDV大约在1971年前后进化成2个谱系,D/OK(D/swine/Oklahoma/1334/2011)谱系和D/660(D/bovine/Oklahoma/660/2013)谱系,但2个谱系之间存在交叉反应性[6]。采用邻近法(bootstrap值为1 000)对NCBI公布的21株IDVs HEF基因进行遗传进化分析(图 1)发现意大利、中国流行毒株属于D/OK谱系,而美国、墨西哥2个谱系均有流行(表 1)。但D/bovine/France/2986/2012、D/bovine/Ibaraki/7768/2016、D/bovine/Miyazaki/B22/2016均位于独立进化分支,与2个谱系进化距离较远,因此其谱系尚不能确定。Murakami等[8]对D/bovine/Ibaraki/7768/2016采用最大似然法(bootstrap值为500)绘制其遗传进化树发现除M基因外,其余6个基因片段均位于独立进化分支,因此推测其可能属于新的进化谱系。Collin等[9]通过HEF(IDV)序列比对和分子模型比较发现212位点可能影响其抗原性。D/OK谱系毒株HEF 212位点均为赖氨酸(lysine,K),而D/660谱系毒株212位点可能为精氨酸(arginine,R)或赖氨酸,但位于独立进化分支的D/bovine/France/2986/2012、D/bovine/Ibaraki/7768/2016、D/bovine/Miyazaki/B22/2016的212位点为丝氨酸(serine,S)(表 1),因此HEF 212位点是否与其抗原性变化有关还有待进一步研究证实[10]。Su等[6]采用贝叶斯马尔可夫连锁蒙特卡洛方法(Bayesian Markov chain Monte Carlo)对HEF(IDV)分析发现HEF(IDV)每个节点平均每年进化速率为1.54×10-3(95%置信区间:5.4×10-4~2.7×10-3),高于HEF(ICV)4.87×10-4(95%置信区间: 4.12×10-4~5.66×10-4),表明IDV比ICV更容易发生变异进化,因此应对IDV的变异进行持续监测[6, 11]。
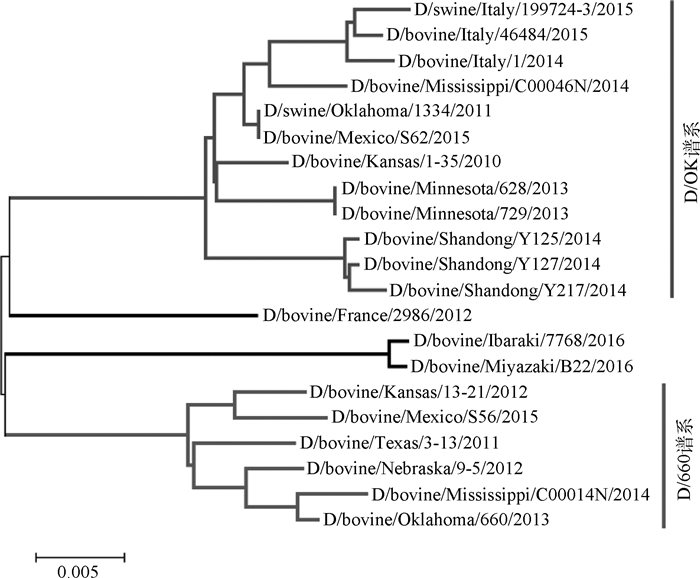
|
图 1 IDV HEF基因遗传进化分析 Figure 1 Phylogenetic tree of HEF gene of IDV |
|
|
表 1 不同IDV毒株HEF 212位点氨基酸、谱系及流行地区 Table 1 The amino acid of HEF 212 site, lineage and infected area of IDVs |
流感病毒表面糖蛋白在其宿主感染和致病性方面具有重要作用,因此目前研究较多的是IDV的HEF蛋白。我国学者Song等[12]对IDV表面唯一糖蛋白HEF生物学特性研究发现其具有与HEF(ICV)相似的受体识别、受体破坏及膜融合等功能。HEF由HEF1和HEF2两个亚基组成,分为受体结合区(receptor binding domain,R)、酯酶活性区(esterase domain,E,由E1、E′和E2三个亚功能区组成)以及融合活性区(fusion domain,F,分为F1、F2和F3三个亚功能区)。通过比较分析IDV和ICV HEF氨基酸序列及2.4 Å分辨率的三维结构,发现它们的整体结构及亚功能区结构和功能高度相似,其中E最保守,与ICV序列一致性分别为66.7%、68.8%和56.6%(E1、E′、E2),主链C原子叠合的均方根偏差(RMSD)分别为0.396 Å、0.390 Å和0.435 Å;R序列一致性为46.3%,RMSD为0.642 Å;F序列一致性分别为42.1%、41.2%、56.8%(F1、F2、F3),RMSD分别为0.567 Å、0.817 Å、1.445 Å,表明这两个蛋白的受体结合特性、受体破坏特性以及膜融合功能具有较高的相似性。由于F3含有融合肽,虽然其序列一致性较高,但RMSD为1.445 Å,所以推测IDV的膜融合功能可能与ICV有一定差异[12]。
IAVs通过特异结合唾液酸α2, 3-半乳糖(禽源)或唾液酸α2, 6-半乳糖(人源)而感染禽或人,部分亚型(如H1N1、H5N1、H7N9等)具有双受体结合特性,既可感染人也可感染禽。Song等[12]利用聚糖微阵列方法证实IDV能特异结合9-O-乙酰唾液酸(9-O-Ac-Sia),并且无论是α2, 3还是α2, 6连接的糖苷键9-O-乙酰唾液酸受体都能发生特异结合。与ICV不同的是,IDV仍能与C5乙酰化或糖基化修饰的9-O-Ac-Sia结合。通过解析HEF蛋白-受体复合物2.2 Å分辨率三维晶体结构发现IDV受体结合区域与ICV相似,靠近HEF1球状头部的顶端,位于170-环、190-环、230-螺旋、270-环以及5个氨基酸(F127、W185、Y231、F229以及F297)形成的一个浅腔内。不同的是HEF(ICV)带负电荷的D269位点和带正电荷K235位点形成盐桥促使270-环和230-螺旋相连形成封闭的受体结合区域,而IDV的T239和A273均为不带电荷氨基酸,因此270-环和230-螺旋没有连接,而是形成一个特征性的开放通道,所以IDV能结合不同宿主受体的多种多聚糖链,这可能与其宿主谱较为广泛有关[6, 10]。另外一个重要特征就是HEF(IDV)的127位氨基酸与IBV(第95位)相同,都为苯丙氨酸(phenylalanine,F),而HEF(ICV,第127位)和HA(IAV,第98位)为酪氨酸(tyrosine,Y)。虽然F127不能与9-O-Ac-Sia的乙酰基形成氢键,但是C4和T171之间至少可以形成2个氢键,有助于稳定受体构像避免平移或旋转,进而增加病毒与受体的亲和力[12]。
IAV和IBV的NA具有受体破坏特性, 能够裂解末端唾液酸使其与糖蛋白或糖脂脱离,释放病毒粒子[13];而ICV和IDV主要是通过其HEF介导病毒粒子的进入和释放。HEF不仅能够结合特异受体,还具有乙酰酯酶活性,水解受体9-O-Ac-Sia C9连接的乙酰基团使病毒粒子和受体解离。由S57、D356和H359组成的催化三联体是HEF受体裂解活性的关键位点,并且这三个位点高度保守,ICV和IDV完全一致。Song等[12]发现随着温度的降低,HEF脂酶活性也逐渐降低,但是在4 ℃条件下,仍具有较强的脂酶活性[10, 12]。
HA或HEF在体内蛋白酶作用下裂解是流感病毒入侵的第一步,ICV和IDV的HEF都只含有1个碱性裂解位点,呈低致病性分子特征,在蛋白酶作用下裂解为HEF1和HEF2两个蛋白亚基,序列分析表明ICV和IDV HEF2融合肽N末端前8个氨基酸完全一致(IFGIDDLI),并且都暴露在表面,而IAV和IBV HA2融合肽N末端插入一个负电腔洞中。融合蛋白的裸露可能与其亲脂性有关[10, 12-14]。
IAV热稳定性和耐酸能力对其跨种传播能力具有重要作用。Yu等[15]发现IDV较IAV、IBV、ICV具有较强的热稳定性和酸稳定性,53 ℃加热2 h或pH3.0环境下作用0.5 h IDV仍具有感染性,而其他流感病毒已完全失去感染活性。不同的细胞、组织甚至是动物体内pH不同,因此推测IDV可能会具有较广泛的组织嗜性和宿主感染性。反向遗传学研究表明HEF蛋白对IDV的热稳定性和耐酸能力起到关键作用,所以应对HEF蛋白与热稳定性、耐酸等特性之间的分子机制进一步研究,防止其发生变异引起跨种传播。
2 D型流感病毒流行病学IAVs以野鸟为自然贮存宿主,IBVs和ICVs以人为自然宿主,而目前普遍认为IDV的自然贮存宿主是牛,可以感染奶牛、肉牛、水牛以及黄牛等多种牛,6月龄犊牛最易感,主要引起牛呼吸系统疾病。猪、山羊、绵羊甚至人均有IDV阳性或抗体阳性的报道[6, 16-19]。实验条件下,IDV也可以感染豚鼠、雪貂等动物。IDV可以在雪貂、豚鼠或牛群中发生接触传播,空气传播能力有限,因此推测IDV的传播方式同其他流感病毒一样,主要通过水平接触传播[3, 20-21]。
2011年,IDV首次在美国俄克拉何马州分离到,随后美国的堪萨斯州、内布拉斯加州、得克萨斯州、明尼苏达州、密西西比州等多个州牛群中检测并分离到IDVs。根据IDV遗传进化关系,目前IDV主要分为D/OK和D/660两个谱系,但存在一定的交叉反应性。Luo等[19]挑选不同谱系的D/bovine/Mississippi/C00013N/2014 (D/13N)和D/bovine/Mississippi/C00046N/2014 (D/46N)作为检测抗原,对采集于2003—2004年内布拉斯加州40个养殖场的293份牛血清样品中IDV抗体回溯性调查,发现240份血清样品IDV血凝抑制抗体阳性(效价大于或等于1:40),阳性率高达81.9%,其中有3份血清样品只对D/13N呈阳性,5份样品只对D/46N阳性,同时发现对D/13N和D/46N血凝抑制阳性的113份样品的血凝抑制效价不同(log2平均值相差1.09±0.29)。以上监测结果证明IDV早在2003年已经在美国出现,并且IDV在流行过程中发生了抗原性变异,形成2个抗原群,但存在一定的交叉反应性。
White等[18]以D/bovine/Kansas/1-35/2010作为检测抗原对佛罗里达州35名健康的牛场工作人员IDV抗体进行检测,发现超过94%的牛场工作人员IDV抗体阳性(效价大于或等于1:40),其中32人血凝抑制(haemagglutination inhibition,HI)抗体阳性,效价为1:40~1:160;34人中和抗体阳性,效价为1:40~1:320,这些人曾患过发热性疾病或有病牛、病猪接触史。White等[18]对当地4名普通人(近10年没有牛接触史)的IDV血清学调查发现有3人HI抗体阳性,效价为1:40~1:80;2人中和抗体阳性,效价为1:40~1:80。Eckard[22]利用D/swine/Oklahoma/1334/2011 (D/OK)作为检测抗原对居住在奶牛养殖场附近的高危暴露人群的741份血清中IDV的HI抗体进行检测,发现仅有8人呈HI抗体弱阳性(效价大于等于1:40),阳性率约为1%。以上血清学结果表明IDV也可以感染人,尤其是牛场或猪场工作人员为易感人群,但其不会引发人致命性疾病。
除美国以外,法国[23]、意大利[24-25]、爱尔兰、日本[8, 26]、中国[27]、摩洛哥、贝宁、多哥以及肯尼亚[28]等多个国家或地区也有过IDV感染的相关报道。Salem等[28]利用血凝抑制方法检测肯尼亚地区单峰驼血清样品中的流感病毒抗体,发现其与IDV(D/bovine/Nebraska/9-5/2012)和ICV(C/Victoria/1/11)结合后都能抑制红细胞凝集。进一步研究发现血清经过四个凝集单位的ICV吸附处理后,IDV抗体滴度下降超过2个滴度,阳性率由99%降至8.2%,而经IDV吸附处理后,ICV抗体阳性率也由94%下降为9%。因此推测IDV可能会感染单峰驼并且与ICV发生抗体交叉反应,但是相关感染机制以及感染骆驼后与ICV发生的抗体交叉反应性还有待进一步研究。
3 D型流感病毒致病性及动物模型研究研究发现IDV感染是临床常见牛呼吸道疾病(BRD)的主要原因[6]。Ferguson等[20]将牛源IDV(D/bovine/Mississippi/C00046N/2014)人工感染犊牛后,能够引起感染犊牛的气管炎症,并出现干咳、流鼻涕等轻微呼吸道症状,证明IDV比较容易感染牛群且发生接触传播。为进一步研究IDV传播能力,Ferguson等[20]通过人工模拟自然状态下流感传播途径,利用2只雪貂接触感染牛群鼻分泌液污染物后并未检测到病毒,血清IDV抗体阴性。因此,虽然IDV能够感染并引起牛群的轻微呼吸系统疾病,但其通过排泄污染物传染给人的风险仍较低。
Hause等[3]将分离到的首株IDV(D/OK)人工感染雪貂和猪,评价其哺乳动物致病性和传播能力。D/OK分离株感染雪貂后未引起流感的临床症状和病理学变化,而且只能从感染组和接触组雪貂鼻洗液中检测到病毒,上呼吸道、下呼吸道、肺、小肠、肝、脾等组织均检测不到病毒,空气传播组雪貂虽未检测到IDV,但1只雪貂IDV抗体转阳,表明IDV不仅能感染雪貂,并且存在水平接触传播的可能性,同时具有有限的飞沫传播能力。同样,D/OK感染猪后也观察不到流感临床症状和病理学变化,但是猪感染后能通过鼻洗液持续排毒。与IAV相比,虽然IDV在雪貂和猪体内尤其是上呼吸道感染复制能力较低,但其有限的哺乳动物间接触传播能力给人类健康造成巨大的潜在威胁。
小鼠、豚鼠、雪貂及非人灵长动物等哺乳动物广泛用于A型流感病毒感染机制和宿主免疫反应等相关研究。IDV虽然能感染雪貂,但在雪貂体内复制能力有限。豚鼠属于哺乳纲动物,其气道高反应性和支气管淋巴组织等均与人类相似,并且与猪、雪貂相比具有体型小、容易操作、价格低廉等优势,因此Sreenivasan等[21]利用豚鼠构建IDV哺乳动物感染模型,IDV在豚鼠鼻甲骨、肺、呼吸道等组织内有效复制,并能在豚鼠间传播。IDV哺乳动物感染模型的建立有助于IDV感染和致病性等相关分子生物学特性研究。
4 D型流感病毒诊断和预防技术研究病毒分离培养是流感病毒诊断的金标准,Hause等[3]发现IDV的细胞嗜性较为广泛,能在猪睾丸细胞(ST)、肺癌细胞(A549)、犬肾细胞(MDCK)、非洲绿猴肾细胞(Marc-145)、人直肠癌细胞(HRT-18G)、乳仓鼠肾细胞(BHK-21)、猪肾细胞(PK-15)等多种细胞中生长繁殖,但在BHK-21和PK-15细胞中复制能力较差。因此IDV的诊断可以采用细胞分离法。
由于流感病毒其表面蛋白糖蛋白能与鸡红细胞发生凝集作用,因此,IDV血清学诊断主要采用血凝和血凝抑制方法,同时也可以采用酶联免疫吸附试验、补体结合试验等方法。
聚合酶链式反应(PCR)具有快速、准确、敏感等特点,因此IDV鉴别诊断可以采用此方法。研究表明流感病毒基因组中PB1基因序列最为保守。因此,目前大部分是基于IDV的PB1基因建立PCR检测方法[29]。Hause等[3]根据D/swine/1334/Oklahoma/2011分离株PB1基因(1 420~1 555 bp区间)设计引物,建立了IDV Taqman荧光定量PCR检测方法。Faccini等[30]根据GenBank公布的所有IDV分离株序列的PB1基因(14株)保守区域(在1 215~1 323 bp)设计引物,建立了两步法荧光定量PCR。
由于IDV发现时间较短,所以目前尚未有商品化疫苗和特异性治疗药物。Hause等[31]利用0.1%β-丙内酯将D/bovine/Kansas/162655/2012灭活后添加30% Emulsigen佐剂制备全病毒灭活疫苗,免疫牛后虽然能显著降低IDV排毒量(减少1~2 TCID50·mL-1),但不能对D/bovine/Kansas/162655/2012的攻击起到100%保护作用。因此建议应用广谱抗病毒药物辅以对症疗法预防和治疗IDV感染。
5 展望近几年来,IAVs(H1N1、H5N1、H5N8、H7N9等)和IBVs肆虐全球,尤其是2017年年初流感呈高发态势,给养殖业造成巨大损失的同时严重影响着人群健康。新发现的D型流感病毒感染宿主范围较广,不但可以感染猪、牛、羊、骆驼等,还能感染人,尤其是牛场工作人员为高危人群。虽然IDV致病力不高且哺乳动物间传播能力有限,但是其可能会通过呼吸道表面的毛细血管内皮细胞进入宿主循环系统,引起病毒血症,给人类健康造成严重威胁[27]。多项研究发现我国山东、广州等多地区存在IDV感染情况,且均属于D/OK谱系[17, 27]。IDV在我国是一直存在还是从他国传入,通过何种方式传入我国,这些问题仍有待研究。鉴于当前IDV流行范围不断扩大,并且在流行过程中较ICV容易发生变异,加之热稳定性好、耐酸,在自然环境中容易存活,因此应加强并且持续进行IDV流行病学调查,尤其是人群中IDV流行情况,同时对其遗传进化规律、致病分子机制、诊断方法以及疫苗研发等相关问题进一步研究。
| [1] | GOUARIN S, VABRET A, DINA J, et al. Study of influenza C virus infection in France[J]. J Med Virol, 2008, 80(8): 1441–1446. DOI: 10.1002/(ISSN)1096-9071 |
| [2] | GUO Y J, JIN F E, WANG P, et al. Isolation of influenza C virus from pigs and experimental infection of pigs with influenza C virus[J]. J Gen Virol, 1983, 64(1): 177–182. DOI: 10.1099/0022-1317-64-1-177 |
| [3] | HAUSE B M, DUCATEZ M, COLLIN E A, et al. Isolation of a novel swine influenza virus from Oklahoma in 2011 which is distantly related to human influenza C viruses[J]. PLoS Pathog, 2013, 9(2): e1003176. DOI: 10.1371/journal.ppat.1003176 |
| [4] | SHENG Z Z, RAN Z G, WANG D, et al. Genomic and evolutionary characterization of a novel influenza-C-like virus from swine[J]. Arch Virol, 2014, 159(2): 249–255. DOI: 10.1007/s00705-013-1815-3 |
| [5] | HAUSE B M, COLLIN E A, LIU R X, et al. Characterization of a novel influenza virus in cattle and swine:proposal for a new genus in the Orthomyxoviridae family[J]. mBio, 2014, 5(2): e00031–14. |
| [6] | SU S, FU X L, LI G R, et al. Novel influenza D virus:epidemiology, pathology, evolution and biological characteristics[J]. Virulence, 2017, 8(8): 1580–1591. DOI: 10.1080/21505594.2017.1365216 |
| [7] | PEKOSZ A, LAMB R A. Influenza C virus CM2 integral membrane glycoprotein is produced from a polypeptide precursor by cleavage of an internal signal sequence[J]. Proc Natl Acad Sci U S A, 1998, 95(22): 13233–13238. DOI: 10.1073/pnas.95.22.13233 |
| [8] | MURAKAMI S, ENDOH M, KOBAYASHI T, et al. Influenza D virus infection in herd of cattle, Japan[J]. Emerg Infect Dis, 2016, 22(8): 1517–1519. DOI: 10.3201/eid2208.160362 |
| [9] | COLLIN E A, SHENG Z Z, LANG Y K, et al. Cocirculation of two distinct genetic and antigenic lineages of proposed influenza D virus in cattle[J]. J Virol, 2015, 89(2): 1036–1042. DOI: 10.1128/JVI.02718-14 |
| [10] | WANG M Y, VEIT M. Hemagglutinin-esterase-fusion (HEF) protein of influenza C virus[J]. Protein Cell, 2016, 7(1): 28–45. DOI: 10.1007/s13238-015-0193-x |
| [11] | GATHERER D. Tempo and mode in the molecular evolution of influenza C[J]. PLoS Curr, 2010, doi: 10.1371/currents.RRN1199. (2010-12-07)[2018-04-17]. http://currents.plos.org/influenza/article/tempo-and-mode-in-the-molecular-209adpo20ccgj-3/. |
| [12] | SONG H, QI J X, KHEDRI Z, et al. An open receptor-binding cavity of hemagglutinin-esterase-fusion glycoprotein from newly-identified influenza D virus:basis for its broad cell tropism[J]. PLoS Pathogens, 2016, 12(1): e1005411. DOI: 10.1371/journal.ppat.1005411 |
| [13] | WEBSTER R G, BEAN W J, GORMAN O T, et al. Evolution and ecology of influenza A viruses[J]. Microbiol Rev, 1992, 56(1): 152–179. |
| [14] | ROSENTHAL P B, ZHANG X D, FORMANOWSKI F, et al. Structure of the haemagglutinin-esterase-fusion glycoprotein of influenza C virus[J]. Nature, 1998, 396(6706): 92–96. DOI: 10.1038/23974 |
| [15] | YU J S, HIKA B, LIU R X, et al. The hemagglutinin-esterase fusion glycoprotein is a primary determinant of the exceptional thermal and acid stability of influenza D virus[J]. mSphere, 2017, 2(4): e00254–17. |
| [16] | FERGUSON L, ECKARD L, EPPERSON W B, et al. Influenza D virus infection in Mississippi beef cattle[J]. Virology, 2015, 486: 28–34. DOI: 10.1016/j.virol.2015.08.030 |
| [17] | JIANG W M, WANG S C, PENG C, et al. Identification of a potential novel type of influenza virus in bovine in China[J]. Virus Genes, 2014, 49(3): 493–496. DOI: 10.1007/s11262-014-1107-3 |
| [18] | WHITE S K, MA W J, MCDANIEL C J, et al. Serologic evidence of exposure to influenza D virus among persons with occupational contact with cattle[J]. J Clin Virol, 2016, 81: 31–33. DOI: 10.1016/j.jcv.2016.05.017 |
| [19] | LUO J R, FERGUSON L, SMITH D R, et al. Serological evidence for high prevalence of influenza D viruses in cattle, Nebraska, United States, 2003-2004[J]. Virology, 2017, 501: 88–91. DOI: 10.1016/j.virol.2016.11.004 |
| [20] | FERGUSON L, OLIVIER A K, GENOVA S, et al. Pathogenesis of influenza D virus in cattle[J]. J Virol, 2016, 90(12): 5636–5642. DOI: 10.1128/JVI.03122-15 |
| [21] | SREENIVASAN C, THOMAS M, SHENG Z S, et al. Replication and transmission of the novel bovine influenza D virus in a guinea pig model[J]. J Virol, 2015, 89(23): 11990–12001. DOI: 10.1128/JVI.01630-15 |
| [22] | ECKARD L E. Assessment of the zoonotic potential of a novel bovine influenza virus[D]. Tennessee: The University of Tennessee, 2016. |
| [23] | DUCATEZ M, PELLETIER C, MEYER G. Influenza D virus in cattle, France, 2011-2014[J]. Emerg Infect Dis, 2015, 21(2): 368–371. |
| [24] | CHIAPPONI C, FACCINI S, DE MA TTIA A, et al. Detection of influenza D virus among swine and cattle, Italy[J]. Emerg Infect Dis, 2016, 22(2): 352–354. DOI: 10.3201/eid2202.151439 |
| [25] | FONI E, CHIAPPONI C, BAIONI L, et al. Influenza D in Italy:towards a better understanding of an emerging viral infection in swine[J]. Sci Rep, 2017, 7(1): 11660. DOI: 10.1038/s41598-017-12012-3 |
| [26] | HORIMOTO T, HIONO T, MEKATA H, et al. Nationwide distribution of bovine influenza D virus infection in Japan[J]. PLoS One, 2016, 11(9): e0163828. DOI: 10.1371/journal.pone.0163828 |
| [27] | ZHAI S L, ZHANG H, CHEN S N, et al. Influenza D virus in animal species in Guangdong Province, Southern China[J]. Emerg Infect Dis, 2017, 23(8): 1392–1396. DOI: 10.3201/eid2308.170059 |
| [28] | SALEM E, COOK E A J, LBACHA H A, et al. Serologic evidence for influenza C and D virus among ruminants and camelids, Africa, 1991-2015[J]. Emerg Infect Dis, 2017, 23(9): 1556–1559. DOI: 10.3201/eid2309.170342 |
| [29] | KISHIMOTO M, TSUCHIAKA S, RAHPAYA S S, et al. Development of a one-run real-time PCR detection system for pathogens associated with bovine respiratory disease complex[J]. J Vet Med Sci, 2017, 79(3): 517–523. DOI: 10.1292/jvms.16-0489 |
| [30] | FACCINI S, DE MATTIA A, CHIAPPONI C, et al. Development and evaluation of a new Real-Time RT-PCR assay for detection of proposed influenza D virus[J]. J Virol Methods, 2017, 243: 31–34. DOI: 10.1016/j.jviromet.2017.01.019 |
| [31] | HAUSE B M, HUNTIMER L, FALKENBERG S, et al. An inactivated influenza D virus vaccine partially protects cattle from respiratory disease caused by homologous challenge[J]. Vet Microbiol, 2017, 199: 47–53. DOI: 10.1016/j.vetmic.2016.12.024 |



