2. 新疆农业大学马产业研究院, 乌鲁木齐 830052
2. Institute of Horse Industry, Xinjiang Agricultural University, Urumqi 830052, China
辅肌动蛋白(ACTN)是近年来在细胞骨架与细胞运动研究中的热点蛋白。目前的研究结果表明,在哺乳动物中主要存在4种不同类型的辅肌动蛋白,其中辅肌动蛋白-2(ACTN2)和辅肌动蛋白-3(ACTN3)为骨骼肌Z线的主要组成部分,且这两种蛋白具有高度的相似性(81%序列一致)[1-3]。ACTN3基因仅在骨骼肌快肌纤维中表达,且其多态性与肌肉力量有关,可能依赖于其对纤维类型比例的控制功能,与纤维分布和肌肉力量有显著关联,在ACTN3基因敲除小鼠中三羧酸循环、线粒体电子信号传递链和脂肪酸氧化代谢途径中的关键酶一直较高[4-5]。与此同时,ACTN3蛋白缺乏对骨骼肌结构[6]、新陈代谢[7]及钙通路[8]等均会产生一定的影响。Gu等[9]通过对纯血马进行研究,结果显示,ACTN3基因在纯血马中有4个突变位点。Mata等[10]通过对法国骑乘马的研究表明,ACTN3基因的cDNA有8个突变位点,其中6个外显子突变位点均为同义突变。Thomas等[11]的研究结果表明,5个澳大利亚品种马中共发现34个突变位点,其中启动子g.1104G>A突变后,纯血马中未发现AA基因型,重挽型品种(夏尔马和克莱兹代尔马)中未发现GG基因型,其中纯血马为优秀的速度型运动马,而夏尔马和克莱兹代尔马则为挽用型,赵启南等[12]通过对蒙古马进行调教训练的研究也显示,ACTN3基因的表达上调,这些研究在一定程度上显示,ACTN3基因可能会对运动性能具有重要的意义。
伊犁马是我国知名运动马品种,其以哈萨克马为母本,通过与奥尔洛夫马、顿河马、阿哈捷金马等轻型马品种进行杂交培育而成,并与纯血马、新吉尔吉斯马等品种进行杂交改良[13],现为我国优良的国产骑乘马品种[14],具有良好的运动性能。现对伊犁马运动性能的研究主要集中在调教训练、血液生化等方面,对分子标记研究相对较少。因此,针对ACTN3基因的结构与功能,并将其运用于实际生产具有重大意义。
本研究以伊犁马为试验动物,通过测序法对ACTN3基因多态性位点进行筛选,并进行连锁不平衡及生物信息学分析,探究伊犁马ACTN3基因多态性,为伊犁马的早期运动选材及分子选育等提供相关理论支持。
1 材料与方法 1.1 试验材料以38匹健康伊犁马为研究对象,其中公马16匹,母马22匹,均为2016年伊犁马常态化赛事参赛马匹。通过枸橼酸钠采血管采集静脉血样5 mL,混匀后,置于-20℃保存。
1.2 引物设计与PCR扩增通过DNA提取试剂盒(生工生物工程(上海)股份有限公司)提取血液DNA。根据NCBI公布的纯血马ACTN3序列(HQ005425)采用Primer-BLAST对所有外显子进行引物设计,当相邻两个外显子之间间隔较小(小于350 bp)时,两个外显子通过同一对引物进行扩增(表 1)。引物由生工生物工程(上海)股份有限公司进行合成。通过降落PCR进行扩增,PCR体系(25 μL):上下游引物各0.5 μL,dNTP(10 mmol·L-1)0.5 μL,Taq Buffer 2.5 μL,Taq酶(5 U·μL-1)0.2 μL,ddH2O加至25 μL。PCR扩增程序:95 ℃预变性3 min;94 ℃变性30 s,63 ℃退火45 s,共计10个循环,每个循环降低0.5 ℃,72 ℃延伸1 min,94 ℃变性30 s,58 ℃退火45 s,72℃延伸1 min,30个循环,72 ℃修复延伸10 min。PCR扩增产物用1.5%TAE琼脂糖电泳进行检测。
|
|
表 1 扩增引物信息 Table 1 PCR primer information |
将所有扩增产物送至生工生物工程(上海)股份有限公司进行测序,并利用DNAStar软件对所测得结果进行校正,与马ACTN3基因序列进行对比(HQ005425),通过BLAST分析并确定突变位点。
1.4 伊犁马群体遗传结构分析通过haploview 4.2软件根据测序情况计算杂合度、有效等位基因数、多态信息含量、基因(型)频率及检测Hardy-Weinberg平衡状态(当P>0.05时判定处于Hardy-Weinberg平衡状态)。
1.5 连锁不平衡及单倍型分析通过HaploView 4.2软件对所测外显子区域突变位点进行连锁不平衡分析及单倍型构建。
1.6 生物信息学分析利用NCBI中的ORF find确定寻找马ACTN3基因的编码区,并进行氨基酸的翻译,利用BioXM 2.6对突变位点编码的氨基酸序列进行比对,通过在线软件RNAfold预测不同单倍体mRNA二级结构(http://rna.tbi.univie.ac.at/cgi-bin/RNAWebSuite/RNAfold.cgi)及最小自由能,运用在线软件sopm预测蛋白质二级结构(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)。
2 结果 2.1 伊犁马ACTN3基因测序及SNP鉴定所测序列与马ACTN3基因序列(HQ005425)具有高度同源性,可以确定PCR产物为马ACTN3的基因序列。通过对测序结果进行比对分析,结果显示,在本试验中ACTN3基因共发现有8个外显子突变位点,各突变位点的具体信息如表 2所示。其中位于第15外显子的T9059G突变为错义突变,突变使得ACTN3蛋白质的第594个氨基酸由甘氨酸变为半胱氨酸,其余各突变位点均为同义突变。
|
|
表 2 ACTN3基因外显子突变位点信息 Table 2 The information of SNPs of ACTN3 gene exon |
对检测出的突变位点的基因(型)频率及多态信息含量等参数进行计算,如表 3所示。在伊犁马中G3334A、A8608G、T9059G、C9091G和C11304T 5个突变位点均只检测到两种基因型,其余3个突变位点检测到3种基因型。G3334A、A6206G和A11517G的优势等位基因为G,G3334A的优势基因型为GG,A6206G和A11517G的优势基因型为AG。A8608G的优势等位基因为A,优势基因型为AA。T9059G的优势等位基因为T,优势基因型为TT。C9091G、C10660T和C11304T的优势等位基因为C,优势基因型为CC。根据多态信息含量(PIC)的分类,A6206G和A11517G属于中度多态(0.25 < PIC < 0.50),其余各突变均为低度多态(PIC < 0.25)。根据卡方检验的结果显示,所检测各位点均处于Hardy-Weinberg平衡状态(P>0.05)。
|
|
表 3 伊犁马ACTN3突变位点分析 Table 3 SNPs analysis of ACTN3 in Yili horse |
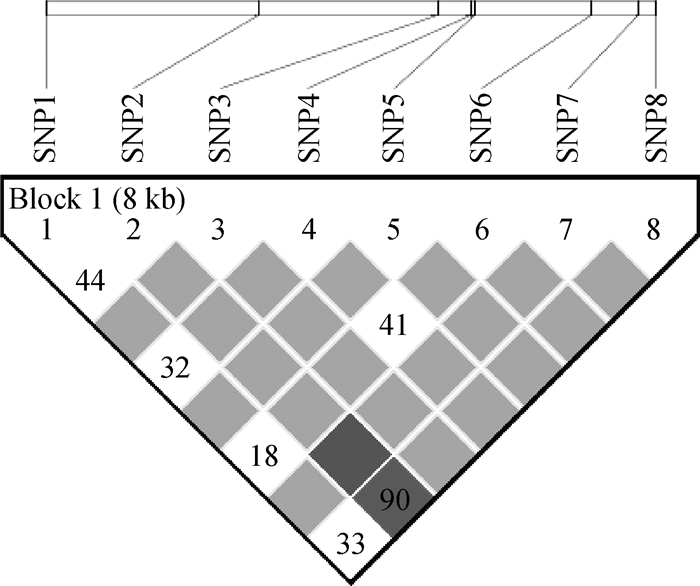
通过haploview 4.2软件构建伊犁马ACTN3外显子的连锁不平衡图谱,并进行连锁不平衡分析,如图 1和表 4所示。在各突变位点中存在不同程度的连锁,其中A6206G和A11517G间存在强的连锁不平衡(D’>0.75,r2>0.33),其余各突变位点间连锁关系相对较弱。通过haploview 4.2软件对各突变位点进行单倍型构建,结果如表 5所示,在伊犁马ACTN3基因外显子中共构建14个单倍型,各单倍型频率为0.012~0.477,其中优势单倍型为H1(GGATCCCG)。
|
图 1 ACTN3各位点连锁不平衡分析 Figure 1 Linkage disequilibrium analysis of ACTN3 SNPs |
|
|
表 4 ACTN3基因多态位点配对连锁不平衡分析 Table 4 Analysis of pairwise linkage disequilibrium of ACTN3 gene SNPs |
|
|
表 5 ACTN3基因单倍型频率分析 Table 5 Haplotype frequencies of ACTN3 gene |
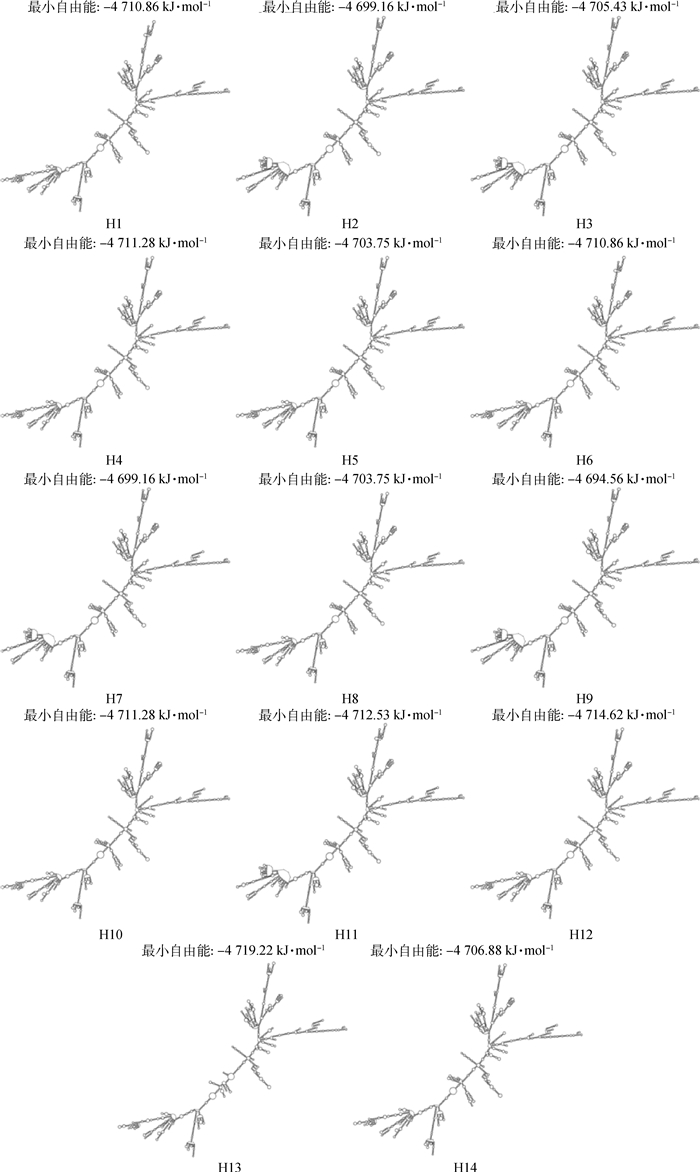
通过RNAfold对所构建14种单倍型的mRNA二级结构进行分析,如图 2所示。在所构建的单倍型中,各二级结构间存在一定程度的改变,且其最小自由能也存在一定的差异,最小自由能为-4 719.22~-4 694.56 kJ·mol-1,其中单倍型H9(GAAGCCCA)的二级结构最小自由能最大,单倍型H13(GGGTCCTA)的二级结构最小自由能最小。
|
图 2 ACTN3不同单倍型mRNA二级结构图 Figure 2 mRNA secondary structure of different haplotypes of ACTN3 |
通过sopm对不同基因型所编码ACTN3蛋白质的二级结构进行预测,结果如表 6所示。结果表明,T9059G为错义突变,由于氨基酸的变化使得ACTN3蛋白质的二级结构发生了改变。β折叠由5.65%变为5.88%;α螺旋由64.63%变为64.19%;无规卷曲度由19.29%变为19.40%;伸展度由10.42%变为10.53%。
|
|
表 6 突变前后ACTN3蛋白质二级结构分析 Table 6 Prediction of ACTN3 protein secondary structure before and after mutation |
ACTN3蛋白的主要功能之一是维持细胞骨架结构[15-16],在人基因中定位于11q13.1染色体,小鼠基因中定位于194.12 cM,且对人和小鼠的相关研究结果表明其对运动性能有着显著影响[17-21]。ACTN蛋白最重要的作用是结合肌动蛋白并使其链接成束[16],维持不同细胞特定的形态,并赋予细胞一定的韧性与强度。同时还能结合如胞质蛋白、膜受体和信号分子等蛋白分子,参与细胞活动,对肌动蛋白丝的形成与定位具有重要作用。Ebashi等[22]首次从兔肉中提取出α-辅肌动蛋白,并认定其为肌动蛋白和肌球蛋白体外结合的必需蛋白。
在ACTN3外显子中共鉴定出8个突变位点,其中3个突变位点与前人的研究结果一致[10-11],新发现5个多态位点(G3334A、A8608G、T9059G、C9091G和C10660T)。8个多态位点所对应编码的氨基酸分别处于ABD域、中心杆状域(RLs域)和“EF”手型域,ABD域的主要生理作用是维持Z线完整性及肌纤维组织[23],能与其它Z线蛋白[24]和磷脂、磷脂酰肌醇二磷酸[25-26]相交联。RLs域由4个血影蛋白组成[27],其二级结构显示为3个α螺旋[28],主要通过与K相关蛋白质相互作用影响糖酵解酶活性[29],“EF”手型域结合Ca2+,对肌动蛋白[30]及其它蛋白的动力学进行调节,从而影响肌纤维类型[31]。这表明,以上突变位点可能会影响到马ACTN3蛋白质结构与功能,影响肌纤维结构、信号传导及氧化代谢等,从而影响马匹的运动性能。
多数情况下,基因对性能的影响是多个突变共同作用的结果[32]。本研究通过对所检测到的8个多态位点进行连锁不平衡分析表明,各突变之间存在一定程度的连锁,并构建出14种单倍型。通过对不同单倍型mRNA进行二级结构分析显示,各单倍型间二级结构与最小自由能有一定的差异,其中单倍型H9(GAAGCCCA)的最小自由能最高,这表明单倍型H9(GAAGCCCA)的二级结构稳定性最低,而单倍型H13(GGGTCCTA)的最小自由能最低,因此其mRNA的二级结构最为稳定。
通过进一步对各突变导致的蛋白质二级结构进行分析,结果显示,所检测到的8个多态位点中有7个为同义突变,其氨基酸并未发生改变,其蛋白质结构与功能是否发生改变及其对运动性能的影响还有待于进一步的研究明确。而T9059G为错义突变,通过在线软件进行预测分析,结果显示,其mRNA最小自由能增大,稳定性降低,在蛋白质翻译过程中会导致第594号氨基酸由甘氨酸变为半胱氨酸,对二级结构的预测分析结果显示,其二级结构发生改变,这有可能会导致蛋白质三级结构改变,从而影响到氧化酶活性,使得马匹的运动性能受到影响。
4 结论在伊犁马ACTN3基因中共发现8个多态位点,共可组成14种单倍型,其中单倍型H1(GGATCCCG)为优势单倍型,不同单倍型mRNA的二级结构与最小自由能有一定的差异。在8个突变位点中,T9059G为错义突变,使得编码ACTN3蛋白质的二级结构发生改变,可能对马ACTN3蛋白质功能产生影响,从而影响马匹的运动性能,该突变位点可能是与马匹运动性能相关的重要功能位点。
| [1] | BEGGS A H, BYERS T J, KNOLL J H, et al. Cloning and characterization of two human skeletal muscle alpha-actinin genes located on chromosomes 1 and 11[J]. J Biol Chem, 1992, 267(13): 9281–9288. |
| [2] | DEL COSO J, VALERO M, SALINERO J J, et al. ACTN3 genotype influences exercise-induced muscle damage during a marathon competition[J]. Eur J Appl Physiol, 2017, 117(3): 409–416. DOI: 10.1007/s00421-017-3542-z |
| [3] | MARCHLER-BAUER A, LU S, ANDERSON J B, et al. CDD:a Conserved Domain Database for the functional annotation of proteins[J]. Nucleic Acids Res, 2011, 39(D1): D225–D229. |
| [4] | GINEVICENE V, JAKAITIENE A, AKSENOV M O, et al. Association analysis of ACE, ACTN3 and PPARGC1A gene polymorphisms in two cohorts of European strength and power athletes[J]. Biol Sport, 2016, 33(3): 199–206. DOI: 10.5604/20831862.1201051 |
| [5] | MIN S K, LIM S T, KIM C S. Association of ACTN3 polymorphisms with BMD, and physical fitness of elderly women[J]. J Phys Ther Sci, 2016, 28(10): 2731–2736. DOI: 10.1589/jpts.28.2731 |
| [6] | GARTON F C, NORTH K N. The Effect of heterozygosity for the ACTN3 null allele on human muscle performance[J]. Med Sci Sport Exer, 2016, 48(3): 509–520. DOI: 10.1249/MSS.0000000000000784 |
| [7] | The PLoS Genetics Staff. Correction:altered Ca2+ kinetics associated with α-actinin-3 deficiency may explain positive selection for ACTN3 null allele in human evolution[J]. PLoS Genet, 2015, 11(6): e1005312. DOI: 10.1371/journal.pgen.1005312 |
| [8] | HEAD S I, CHAN S, HOUWELING P J, et al. Altered Ca2+ kinetics associated with α-actinin-3 deficiency may explain positive selection for ACTN3 null allele in human evolution[J]. PLoS Genet, 2015, 11(2): e1004862. |
| [9] | GU J, MACHUGH D E, MCGIVNEY B A, et al. Association of sequence variants in CKM (creatine kinase, muscle) and COX4I2 (cytochrome c oxidase, subunit 4, isoform 2) genes with racing performance in thoroughbred horses[J]. Equine Vet J, 2010(38): 569–575. |
| [10] | MATA X, VAIMAN A, DUCASSE A, et al. Genomic structure, polymorphism and expression of the horse alpha-actinin-3 gene[J]. Gene, 2012, 491(1): 20–24. DOI: 10.1016/j.gene.2011.09.014 |
| [11] | THOMAS K C, HAMILTON N A, NORTH K N, et al. Sequence analysis of the equine ACTN3 gene in Australian horse breeds[J]. Gene, 2014, 538(1): 88–93. DOI: 10.1016/j.gene.2014.01.014 |
| [12] |
赵启南, 芒来, 白东义, 等. 蒙古马高负荷运动训练前后转录组差异表达分析[J]. 畜牧兽医学报, 2017, 48(6): 1007–1016.
ZHAO Q N, MANG L, BAI D Y, et al. Mongolian horses transcriptome differential expression analysis before and after a high load exercise training[J]. Acta Veterinaria et Zootechnica Sinica, 2017, 48(6): 1007–1016. (in Chinese) |
| [13] |
姚新奎, 韩国才.
马生产管理学[M]. 北京: 中国农业大学出版社, 2008.
YAO X K, HAN G C. Horse production and management[M]. Beijing: China Agricultural University Press, 2008. (in Chinese) |
| [14] |
中华人民共和国农业部. NY/T 2831-2015伊犁马[S]. 北京: 中国农业出版社, 2015.
The Ministry of Agriculture of the People's Republic of China. NY/T 2831-2015 Yili Horse[S]. Beijing: China Agriculture Press, 2015. (in Chinese) |
| [15] | DE MORAES V N, FERRARI G D, CHIARATTO T, et al. Association of genetic polymorphisms with physical capacities and body composition in older women[J]. Rev Bras Cineantropom Desempenho Hum, 2016, 18(1): 11–19. DOI: 10.5007/1980-0037.2016v18n1p11 |
| [16] | BAUMERT P, LAKE M J, STEWART C E, et al. Genetic variation and exercise-induced muscle damage:implications for athletic performance, injury and ageing[J]. Eur J Appl Physiol, 2016, 116(9): 1595–1625. DOI: 10.1007/s00421-016-3411-1 |
| [17] | PICKERING C, KIELY J. ACTN3:more than just a gene for speed[J]. Front Physiol, 2017, 8: 1080. DOI: 10.3389/fphys.2017.01080 |
| [18] | DIONÍSIO T J, THIENGO C R, BROZOSKI D T, et al. The influence of genetic polymorphisms on performance and cardiac and hemodynamic parameters among Brazilian soccer players[J]. Appl Physiol Nutri Metab, 2017, 42(6): 596–604. DOI: 10.1139/apnm-2016-0608 |
| [19] | KIKUCHI N, MIYAMOTO-MIKAMI E, MURAKAMI H, et al. ACTN3 R577X genotype and athletic performance in a large cohort of Japanese athletes[J]. Eur J Sport Sci, 2016, 16(6): 694–701. DOI: 10.1080/17461391.2015.1071879 |
| [20] | PAPADIMITRIOU I D, LUCIA A, PITSILADIS Y P, et al. ACTN3, R577X and ACE I/D gene variants influence performance in elite sprinters:a multi-cohort study[J]. Bmc Genomics, 2016, 17: 285. DOI: 10.1186/s12864-016-2462-3 |
| [21] | SETO J T, QUINLAN K G R, LEK M, et al. ACTN3 genotype influences muscle performance through the regulation of calcineurin signaling[J]. J Clin Invest, 2013, 123(10): 4255–4263. DOI: 10.1172/JCI67691 |
| [22] | EBASHI S, EBASHI F. α-actinin, a new structural protein from striated muscle:Ⅰ.Preparation and action on actomyosin-ATP interaction[J]. J Biochem, 1965, 58(1): 7–12. DOI: 10.1093/oxfordjournals.jbchem.a128167 |
| [23] | NORTH K. Why is α-actinin-3 deficiency so common in the general population? The evolution of athletic performance[J]. Twin Res Hum Genet, 2008, 11(4): 384–394. DOI: 10.1375/twin.11.4.384 |
| [24] | LIN Z, HIJIKATA T, ZHANG Z, et al. Dispensability of the actin-binding site and spectrin repeats for targeting sarcomeric α-actinin into maturing Z bands in vivo:implications for in vitro binding studies[J]. Dev Biol, 1998, 199(2): 291–308. DOI: 10.1006/dbio.1998.8920 |
| [25] | YANG N, GARTON F, NORTH K. α-actinin-3 and performance[J]. Med Sport Sci, 2009, 54: 88–101. DOI: 10.1159/000235698 |
| [26] | YIN H L, JANMEY P A. Phosphoinositide regulation of the actin cytoskeleton[J]. Annu Rev Physiol, 2003, 65(1): 761–789. DOI: 10.1146/annurev.physiol.65.092101.142517 |
| [27] | BARON M D, DAVISON M D, JONES P, et al. The sequence of chick alpha-actinin reveals homologies to spectrin and calmodulin[J]. J Biol Chem, 1987, 262(36): 17623–17629. |
| [28] | DJINOVIC-CARUGO K, GAUTEL M, YLÄNNE J, et al. The spectrin repeat:a structural platform for cytoskeletal protein assemblies[J]. FEBS Lett, 2002, 513(1): 119–123. DOI: 10.1016/S0014-5793(01)03304-X |
| [29] | HOGARTH M W, HOUWELING P J, THOMAS K C, et al. Evidence for ACTN3 as a genetic modifier of Duchenne muscular dystrophy[J]. Nat Commun, 2017, 8: 14143. DOI: 10.1038/ncomms14143 |
| [30] | TANG J H, TAYLOR D W, TAYLOR K A. The three-dimensional structure of α-actinin obtained by cryoelectron microscopy suggests a model for Ca2+-dependent actin binding 1[J]. J Mol Biol, 2001, 310(4): 845–858. DOI: 10.1006/jmbi.2001.4789 |
| [31] | GARTON F C, SETO J T, QUINLAN K G R, et al. α-Actinin-3 deficiency alters muscle adaptation in response to denervation and immobilization[J]. Hum Mol Genet, 2014, 23(7): 1879–1893. DOI: 10.1093/hmg/ddt580 |
| [32] |
轩俊丽, 马晓萌, 王慧华, 等. 绵羊季节性繁殖相关基因TSHR外显子多态性研究[J]. 畜牧兽医学报, 2016, 47(7): 1342–1353.
XUAN J L, MA X M, WANG H H, et al. Study on exon polymorphism of seasonal breeding related gene TSHR in sheep[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(7): 1342–1353. (in Chinese) |



