猪圆环病毒(porcine circovirus,PCV)是一种呈二十面体对称、无囊膜的单股环状负链DNA病毒。它是迄今为止发现的最小的动物病毒,成熟的病毒粒子平均直径约为17 nm[1-2]。PCV主要分为三种血清型,PCV1、PCV2和PCV3。PCV1基因组序列为1 759 bp,包含7个开放阅读框(ORFs),即ORF1~ORF7;PCV2基因组序列为1 767或1 768 bp,包含11个开放阅读框,即ORF1~ORF11[3]。各个开放阅读框之间大小相距甚多,正链上包括ORF1、ORF5、ORF7、ORF10,负链上包括ORF2、ORF3、ORF4、ORF6、ORF8、ORF9、ORF11,虽然圆环病毒非常短小,但是这些开放阅读框之间会有部分碱基重叠,这样就可以充分利用病毒有限的遗传物质;PCV3基因组序列为2 000 bp,目前只确定了ORF1和ORF2,对于它的更多的开放阅读框作用功能还没有定论[4-5]。ORF1主要负责与病毒复制相关蛋白的编码(Rep蛋白),ORF2主要负责病毒衣壳蛋白的编码(Cap蛋白)[6]。
PCV1虽普遍存在于猪群中,但是对猪没有致病性[7];PCV2被认为是断奶仔猪综合征(postweaning multisystemic wasting syndrome,PMWS)的主要病原,导致断奶仔猪生长缓慢,消瘦等,甚至一些呼吸道疾病、肠道疾病、繁殖障碍等也被证实与PCV2相关[8-10];PCV3是2016年新发现的猪环状病毒,与猪皮炎肾病综合征(porcine dermatitis and nephropathy syndrome)相关,且会导致母猪死亡率和流产率升高[11]。
为了解PCV3在健康猪群中的感染情况,本研究对广西某市屠宰场健康猪淋巴结进行PCR检测。对阳性样品进行全序列扩增并分析比对序列,进一步了解PCV3的遗传变异情况,为广西地区PCV3的研究提供科学依据。
1 材料与方法 1.1 材料 1.1.1 样品采集2017年6月—2017年8月来自广西某市屠宰场的淋巴结181份,进行PCV3检测。
1.1.2 主要试剂PCR用预混酶购自Vazyme;DH5α感受态细胞、DNA抽提试剂盒购自康为世纪公司;氨苄西林钠粉末购自Soarbio;pMD18-T载体、BanHⅠ内切酶、Hind Ⅲ内切酶、DNA MarkerDL2000购自TaKaRa;DNA纯化、质粒小提试剂盒购自OMEGA。
1.2 方法 1.2.1 引物设计PCV3的检测引物是根据PCV3基因上保守片段设计的一对引物命名为PCV3-2F/PCV3-1R,PCR扩增长度为649 bp;PCV3全基因引物分为PCVG2-F/G2-R和PCVG4-F/G4-R,PCR扩增长度分别是1 075和1 337 bp。引物使用终浓度为10 pmol·μL-1,稀释后-20 ℃保存。引物序列和片段长度如表 1所示。
|
|
表 1 本研究所用引物列表 Table 1 List of primers used in this study |
取适量送检组织病料置于碾钵中充分碾磨至糊状,加入2~3 mL灭菌PBS(pH 7.42)混匀,将混合液吸至干净的1.5 mL EP管中,置于-80 ℃冰箱反复冻融3次。按照DNA提取试剂盒说明书,提取病毒基因组DNA,提取的DNA置于-20 ℃保存备用。
1.2.3 DNA纯化回收及重组质粒鉴定DNA纯化回收按照试剂盒说明步骤操作,将胶回收产物与pMD18-T载体按其说明书进行连接,转化入DH5α感受态细胞。挑选单个菌落接种含有Amp(100 μg·mL-1)的LB培养基,放入37 ℃恒温摇床中以200 r·min-1培养12~14 h。按质粒小提试剂盒进行质粒抽提,用BamHⅠ、Hind Ⅲ内切酶对质粒酶切鉴定,将酶切鉴定正确的质粒送往上海杰李生物有限公司测序。
1.2.4 序列比对分析通过DNAStar软件编辑测序结果,将剪辑完成的核苷酸片段放到NCBI-Blast网站上进行比对,得到的结果进行初步分析。
2 结果 2.1 淋巴结中PCV3病原检测对于广西某市屠宰猪181份淋巴结进行PCV3的病原检测,PCV3电泳结果表明,阳性条带在650 bp左右,与预期片段相一致,如图 1。其中PCV3阳性共44份。
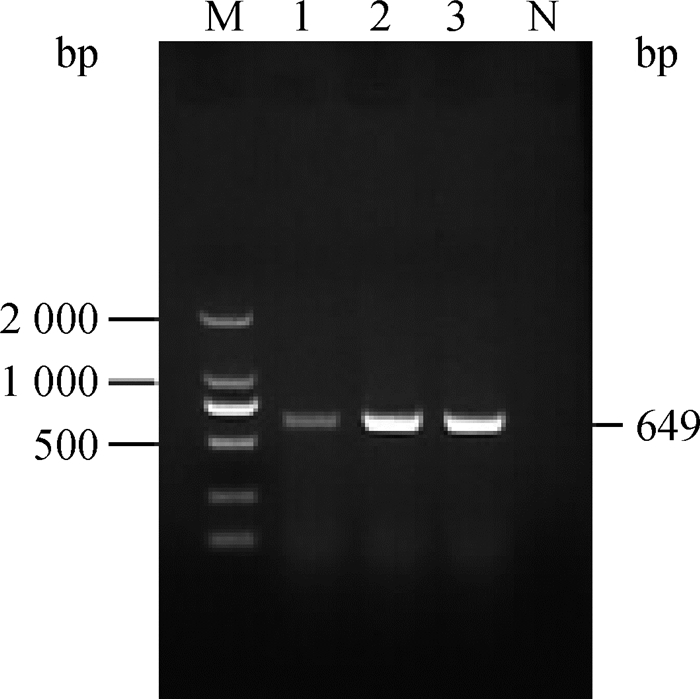
|
1~3.扩增的目的片段; N.阴性对照; M. DL2000 DNA相对分子质量标准 1-3.PCR product of fragments of PCV3; N. The negative sample; M. DNA marker (DL2000) 图 1 PCV3检测结果 Figure 1 The detecting results of PCV3 |
用引物PCVG2F/R、PCVG4F/R对已经确认为PCV3阳性的样品进行全基因扩增,分别得到长度约为1 100和1 300 bp的片段,与预期片段相一致,见图 2、3。
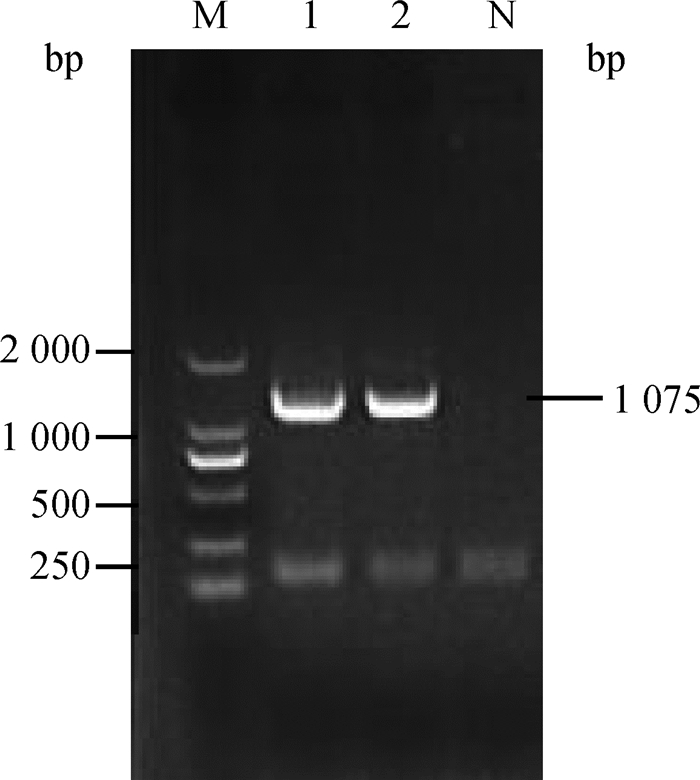
|
1、2.扩增的目的片段; N.阴性对照; M. DL2000 DNA相对分子质量标准 1, 2. PCR product of fragments of PCV3 genome; N.The negative sample; M. DNA marker (DL2000) 图 2 PCV3-G2的克隆 Figure 2 Clones of the PCV3-G2 |
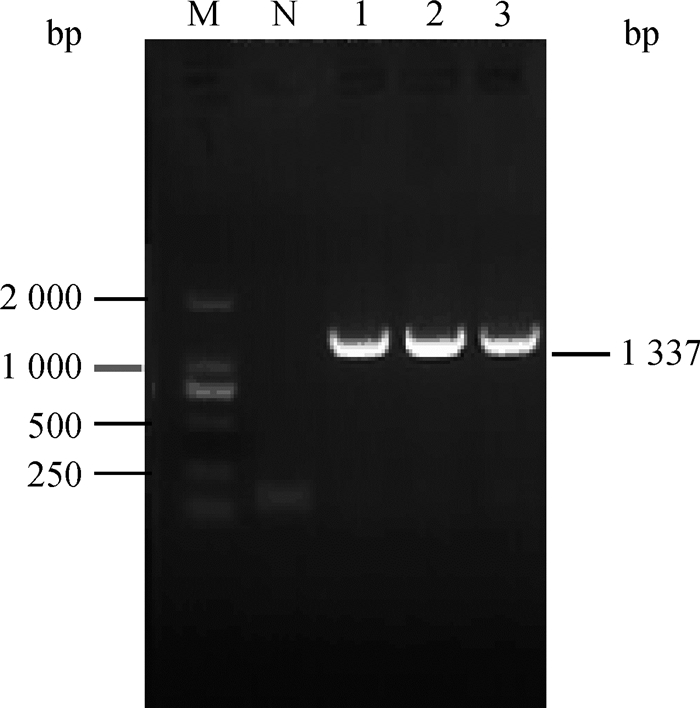
|
1~3.扩增的目的片段; N.阴性对照; M. DL2000 DNA相对分子质量标准 1-3. PCR product of fragments of PCV3 genome; N.The negative sample; M. DNA marker (DL2000) 图 3 PCV3-G4的克隆 Figure 3 Clones of the PCV3-G4 |
运用MEGA5.2软件将本研究的6株PCV3全基因核苷酸序列与国内外26株PCV3全基因序列共同建立遗传演化树,结果如图 4。从图中可以看出,世界各地的PCV3毒株主要分为两大分支,其中有4株(GXDX2-14、GXDX2-2、GXDX1-82和GXDX2-15)位于其中一个分支,GXDX2-4和GXDX2-1位于另外一个分支。其中GXDX2-15毒株虽然和GXDX2-14、GXDX2-2、GXDX1-82这三株位于同一大的分支,但是与他们的距离比较远,说明它们的亲缘关系可能有点远,其独立在一个小分支上,反而和另一大分支上的GXDX2-4距离比较近。
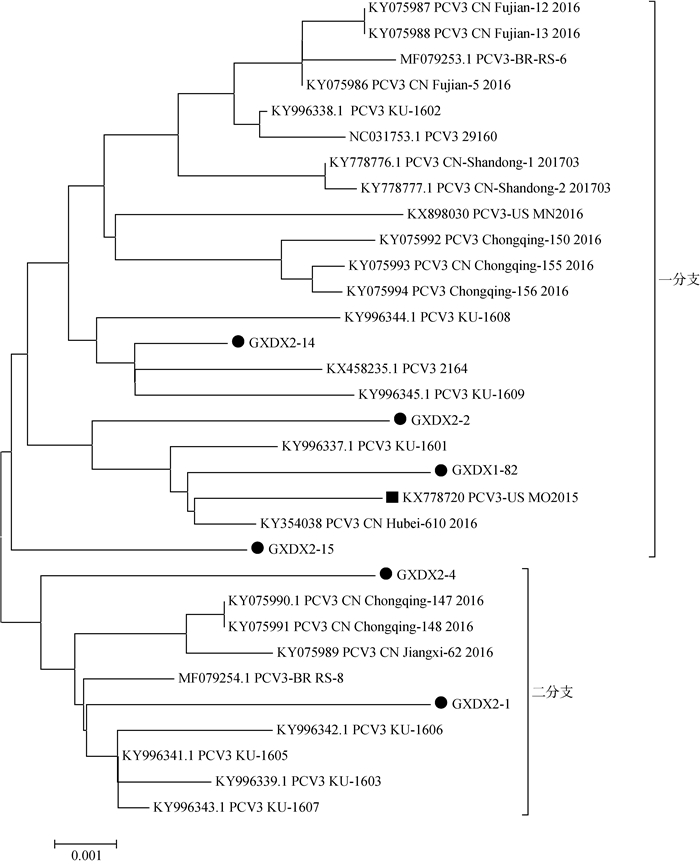
|
●.本研究所获的PCV3全序列;■.首条PCV3全序列 ●. Full sequence of the PCV3 obtained by this study; ■. The complete sequence of the first PCV3 图 4 PCV3全基因核苷酸序列的遗传演化树 Figure 4 Genetic phylogenetic tree of PCV3 complete gene nucleotide sequence |
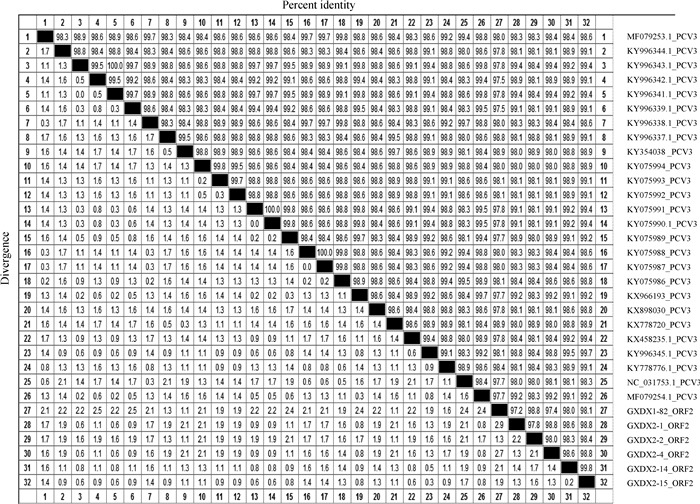
运用MegAlign软件将本研究所获的6株PCV3全基因序列与国内外26株PCV3全基因序列进行同源性比较,结果发现本研究得到的6株PCV3毒株之间相似性最高,在98.6%~99.4%,其中GXDX2-14与GXDX2-15相似性为99.4%,相似性较低的是GXDX1-82与GXDX2-1、GXDX1-82与GXDX2-4。与国内外其他26条PCV3毒株比对结果发现,毒株间相似性最高为99.6%(美国株KX458235.1与GXDX2-14),相似性最低为98.6%(美国株KX778720与GXDX2-4)。国内外PCV3全基因的相似性在98.6%~99.9%,说明目前世界各国的PCV3全基因序列相似性都比较高,遗传关系比较近。
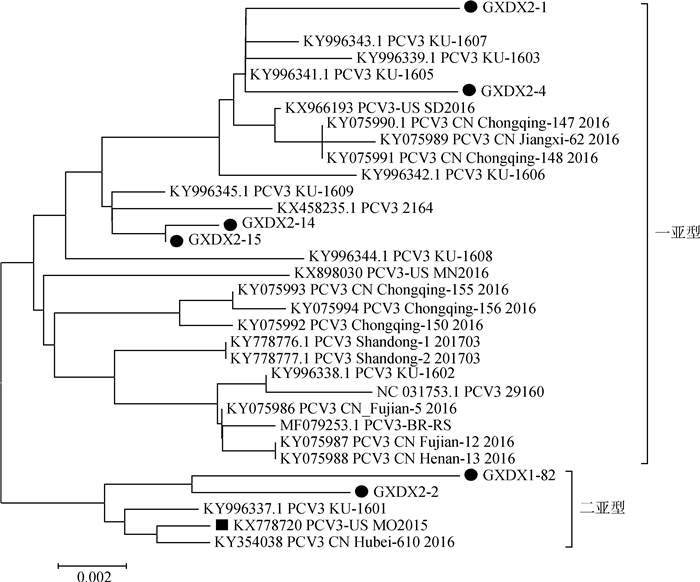
2.5 PCV3 ORF2基因核苷酸序列的遗传进化树分析对本研究6株毒株和国内外26株PCV3毒株的ORF2进行遗传演化的分析,主要是依据ORF2的遗传演化情况来对PCV3进行分型。ORF2基因与全基因生成的进化树有所区别,但从图 6中可以看出,还是主要分为两大分支。从图中可以发现GXDX2-1、GXDX2-4、GXDX2-14和GXDX2-15同处于一大分支上,其中GXDX2-14和GXDX2-15与美国株KX458235.1、韩国株KY996345.1同处于一个小的分支,GXDX2-1和GXDX2-4同处于一个小的分支, 在同一分支的还有三株韩国株KY996343.1、KY996339.1、KY996341.1,说明它们具有更近的亲缘性。GXDX2-14和GXDX2-15在一个小分支上,与它们遗传距离较近的有美国株KX458235.1、韩国株KY996345.1。GXDX1-82、GXDX2-2和美国株KX778720、韩国株KY996337.1、中国湖北株KY354038处于同一大分支上,分为二亚型,亲缘性较近;而GXDX1-82和GXDX2-2又同处于一个小的分支下,亲缘性更为接近。

|
图 5 国内外PCV3全基因核苷酸序列同源性分析 Figure 5 Homology analysis od nucleotide sequence of PCV3 whole gene at worldwide |
|
●.本研究所获的PCV3 ORF2序列;■.首次发现的PCV3 ORF2序列 ●. ORF2 sequence of the PCV3 obtained by this study; ■. ORF2 sequence of the first PCV3 图 6 PCV3 ORF2基因核苷酸序列的遗传进化树 Figure 6 Genetic evolution of the nucleotide sequence of the PCV3 ORF2 gene |
本研究中6株PCV3的ORF2基因序列与国内外26株PCV3的ORF2基因序列进行同源性比较(图 7),结果发现这6株PCV3毒株的ORF2基因序列之间相似性最高可达99.8%(GXDX2-14与GXDX2-15),相似性最低为97.2%(GXDX1-82与GXDX2-1)。这6株的ORF2基因序列与国内外其他26株ORF2基因序列比较结果显示,相似性为97.5%~99.7%。而国内外所有ORF2基因序列相似性比较来看,相似性最高为100%(如中国福建株KY075987和中国河南株KY075988)。
|
图 7 国内外PCV3 ORF2基因序列相似性比较 Figure 7 Comparison of PCV2 ORF2 gene homology at home and abroad |
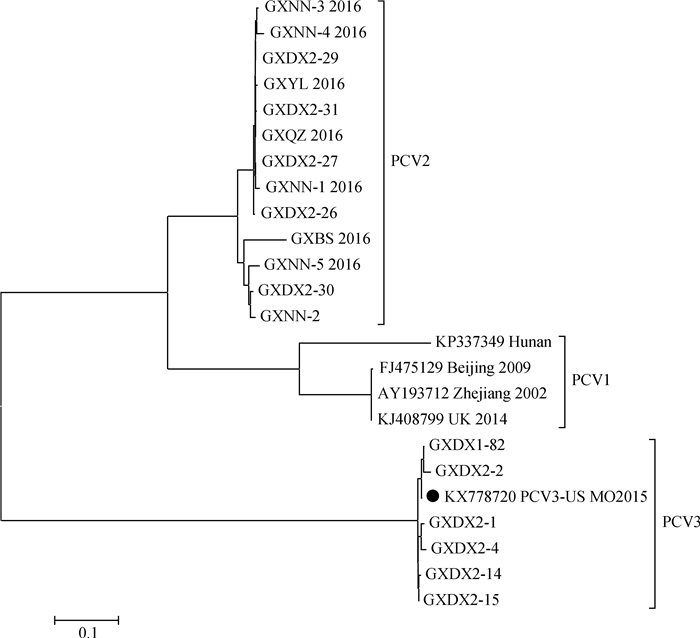
将本实验室已有的13条PCV2 ORF2基因和7条PCV3 ORF2基因以及从NCBI下载的PCV1 ORF2基因做成遗传进化树,如图 8。从图中可以看出,PCV1、PCV2和PCV3亲缘性都比较远。虽然PCV1和PCV2都在同大一分支上,但是在分支中距离相差很远。PCV3在另外一个大分支上,且明显与其他两个型距离更远。说明PCV1、PCV2和PCV3三个基因型遗传关系都比较远。
|
●.首次发现的PCV3序列 ●. Sequence of the first PCV3 图 8 PCVs ORF2基因的核苷酸序列遗传演化 Figure 8 Genetic evolution of the nucleotide sequence of the PCVs ORF2 |
图 9中1~4株为PCV1 Cap蛋白;5~17株为PCV2 Cap蛋白;18~23株为PCV3 Cap蛋白。PCV1 Cap蛋白与PCV2 Cap蛋白之间的相似性为67.2%~69.8%;PCV1 Cap蛋白与PCV3 Cap蛋白之间的相似性为26.1%~27.0%;PCV2 Cap蛋白与PCV3 Cap蛋白之间的相似性为26.5%~29.9%。据此推测PCV2与PCV3交叉保护性极低。

|
图 9 PCVs Cap蛋白的相似性比较 Figure 9 Comparison of PCVs Cap homology |
近年来,世界各地生猪养殖业中关于PCV2感染的报道依然层出不穷,其可导致猪免疫功能下降,生长发育受阻,对养猪业造成了巨大额度经济损失[12]。而在2016年出现的猪圆环病毒新的血清型PCV3,更是给猪圆环病毒的防控增加了难度[13-14]。目前在健康屠宰猪群中关于PCV2的隐性感染已经有所报道,而关于PCV3在健康屠宰猪群中的感染还未有报道。本研究通过调查广西某市健康屠宰猪群PCV3的感染情况,并对PCV3进行序列分析、比对,以了解病毒的遗传演化和流行情况,对疾病防控以及疫苗研究等具有重要的指导意义。
本研究克隆得到的6株PCV3全基因组序列长度均为2 000 bp,6株毒株之间相似性为98.6%~99.4%。与国内外其他26条毒株比对发现,相似性为98.6%~99.6%。无论是广西内PCV3全基因组的相似性比对,还是国内外PCV3全基因组的相似性比对,PCV3毒株之间相似性都非常高。通过构建PCV3全基因的遗传进化树可以发现,PCV3主要分为两大分支,本试验扩增得到的PCV3全基因在两大分支中都有分布。PCV3 ORF2基因的相似性为97.2%~100%,相比较PCV2而言,PCV3的相似性更高,更为保守。从PCV3 ORF2基因的遗传进化树来看,GXDX2-1、GXDX2-4、GXDX2-14和GXDX2-15同位于一亚型,GXDX1-82与GXDX2-2同位于二亚型。并且在一亚型中,似乎还可以分为两个基因型,但由于PCV3为新发现病毒,GenBank上的基因序列还不够多,无法对其下结论。
此外本研究构建了PCV1、PCV2和PCV3全基因核苷酸序列进化树,从进化树中可以看出, PCV1和PCV2处于同一大分支,而PCV3处于另一分支。通过对PCVs的Cap蛋白进行同源性分析,发现PCV2 Cap蛋白与PCV3 Cap蛋白之间的相似性仅为26.5%~29.9%,推测PCV2与PCV3交叉保护性极低。目前市场有众多商品化的PCV2疫苗,对PCV2的防控起到一定的作用,但已有的PCV2疫苗无法对PCV3提供有效的免疫保护作用[15],所以对PCV3的防控任重而道远。
4 结论扩增了6株猪圆环病毒3型全基因组序列,遗传进化分析可分为两大型,彼此之间较为保守。通过对PCVs Cap蛋白的同源性进行比对分析,推测PCV2与PCV3不具有交叉保护性。
| [1] | TISCHER I, GELDERBLOM H, VETTERMANN W, et al. A very small porcine virus with circular single-stranded DNA[J]. Nature, 1982, 295(5844): 64–66. DOI: 10.1038/295064a0 |
| [2] | FRANZO G, TUCCIARONE C M, DOTTO G, et al. International trades, local spread and viral evolution: The case of porcine circovirus type 2 (PCV2) strains heterogeneity in Italy[J]. Infect Genet Evol, 2015, 32: 409–415. DOI: 10.1016/j.meegid.2015.04.004 |
| [3] | MOROZOV I, SIRINARUMITR T, SORDEN S D, et al. Detection of a novel strain of porcine circovirus in pigs with postweaning multisystemic wasting syndrome[J]. J Clin Microbiol, 1998, 36(9): 2535–2541. |
| [4] | MAHÉ D, BLANCHARD P, TRUONG C, et al. Differential recognition of ORF2 protein from type 1 and type 2 porcine circoviruses and identification of immunorelevant epitopes[J]. J Gen Virol, 2000, 81: 1815–1824. DOI: 10.1099/0022-1317-81-7-1815 |
| [5] | LIU Q, WILLSON P, ATTOH-POKU S, et al. Bacterial expression of an immunologically reactive PCV2 ORF2 fusion protein[J]. Protein Expr Purif, 2001, 21(1): 115–120. DOI: 10.1006/prep.2000.1356 |
| [6] | NAWAGITGUL P, MOROZOV I, BOLIN S R, et al. Open reading frame 2 of porcine circovirus type 2 encodes a major capsid protein[J]. J Gen Virol, 2000, 81(Pt 9): 2281–2287. |
| [7] | TISCHER I, RASCH R, TOCHTERMANN G. Characterization of papovavirus-and picornavirus-like particles in permanent pig kidney cell lines[J]. Zentralbl Bakteriol Orig A, 1974, 226(2): 153–167. |
| [8] | HARMS P A, SORDEN S D, HALBUR P G, et al. Experimental reproduction of severe disease in CD/CD pigs concurrently infected with type 2 porcine circovirus and porcine reproductive and respiratory syndrome virus[J]. Vet Pathol, 2001, 38(5): 528–539. DOI: 10.1354/vp.38-5-528 |
| [9] | CHOI C, KIM J, KANG I J, et al. Concurrent outbreak of PMWS and PDNS in a herd of pigs in Korea[J]. Vet Rec, 2002, 151(16): 484–485. DOI: 10.1136/vr.151.16.484 |
| [10] | HINES R K, LUKERT P D. Porcine circovirus as a cause of congenital tremors in newborn pigs[C]//Proceedings of America Association Swine Prsctitioners. Chicago: America Association Swine Prsctitioners, 1994: 344-345. |
| [11] | PALINSKI R, PIÑEYRO P, SHANG P C, et al. A novel porcine circovirus distantly related to known circoviruses is associated with porcine dermatitis and nephropathy syndrome and reproductive failure[J]. J Virol, 2016, 91(1): e01879–16. |
| [12] | GRAU-ROMA L, FRAILE L, SEGALÉS J. Recent advances in the epidemiology, diagnosis and control of diseases caused by porcine circovirus type 2[J]. Vet J, 2011, 187(1): 23–32. DOI: 10.1016/j.tvjl.2010.01.018 |
| [13] | KU X, CHEN F, LI P, et al. Identification and genetic characterization of porcine circovirus type 3 in China[J]. Transbound Emerg Dis, 2017, 64(3): 703–708. DOI: 10.1111/tbed.2017.64.issue-3 |
| [14] | CHEN G H, MAI K J, ZHOU L, et al. Detection and genome sequencing of porcine circovirus 3 in neonatal pigs with congenital tremors in South China[J]. Transbound Emerg Dis, 2017, 64(6): 1650–1654. DOI: 10.1111/tbed.2017.64.issue-6 |
| [15] |
贺会利, 李军, 潘艳, 等. 广西首例猪圆环病毒3型的发现及其衣壳蛋白序列分析[J]. 南方农业学报, 2017, 48(8): 1499–1503.
HE H L, LI J, PAN Y, et al. The first report of porcine circovirus type 3 infection in Guangxi and sequence analysis of its capsid protein[J]. Journal of Southern Agriculture, 2017, 48(8): 1499–1503. (in Chinese) |



