猪丁型冠状病毒(porcine deltacoronavirus,PDCoV)是近年新发现的一种猪肠道病毒,该病毒属于尼多病毒目(Nidovirales),冠状病毒科(Coronaviridae),冠状病毒属(Coronavirus)[1]。早在2009年我国香港收集的猪直肠拭子中已被检测到,但未分离到该病毒[2-3]。之后在健康的野生鸟类,亚洲豹猫身上也发现了它的身影,也证实该病毒与猪携带的PDCoV有极高的亲缘性[4]。Woo等[1]提出的冠状病毒进化模型主张蝙蝠为α、β冠状病毒的最初基因源,而鸟类为γ、δ冠状病毒的最初基因源,因此δ冠状病毒有可能是由野生鸟类传给小型哺乳动物和家猪。PDCoV与PEDV、TGEV临床发病症状相似,能够引起哺乳仔猪的消化系统疾病,导致猪腹泻、呕吐和脱水,严重者多系统衰竭死亡[5]。目前该病毒已在美国[6]、加拿大[7]、韩国[8]、老挝[9]被检测到,感染的范围正逐步扩大,对养猪业构成严重的威胁。近年,国内的一些流行病学调查结果表明PDCoV在中国大陆的流行较为普遍,检测率高达30%[10-13],Dong等[14]在2004年存档的安徽样本中检出PDCoV CHN-AH-2004毒株,说明PDCoV早于首次发现时就已存在于中国猪群;吴美洲等[15]对2014年11月至2015年5月之间采集的我国3个省的10个猪场的64份样品进行了检测,PDCoV的检测率为23.4%;2015年3月Song等[16]对我国江西省进行调查,PDCoV的检测率已经达到33.1%。作为新发型传染性病原,需要高度重视和立即加强对PDCoV的研究,降低养猪业带来的损失和威胁。
PDCoV为有囊膜的单链正股RNA病毒,已测定的PDCoV全基因组序列大小约为25.4 kb,是目前已知冠状病毒中基因组最小的[16]。共编码4种结构蛋白,即纤突蛋白(S)、包膜蛋白(E)、膜蛋白(M)、核衣壳蛋白(N)和4种非结构蛋白。PDCoV的基因组构成和排列顺序:5′非编码区(5′UTR)、开放阅读框1a /1b、S、E、M、非结构蛋白NS6、N、非结构蛋白NS7、3′非编码区(3′UTR)[17]。目前PDCoV的蛋白功能研究较少,根据其他冠状病毒的研究结果推测PDCoV的S蛋白在诱导中和抗体[18]、病毒识别宿主和决定病毒毒力等方面[19]发挥重要的生物学功能。Thachil等[20]利用真核表达系统表达PDCoV的S蛋白,成功建立了检测PDCoV的ELISA方法,该方法的敏感性与特异性分别达到91%、95%,间接证实S蛋白具有良好的反应性。本研究克隆了PDCoV的S1基因,在生物信息学分析基础上,将抗原表位预测区域Sa插入大肠杆菌中进行原核表达,提纯蛋白免疫新西兰大白兔制备了兔抗Sa蛋白高免血清,证明原核表达的Sa蛋白具有良好的抗原性和免疫原性,为开展S蛋白的应用研究奠定了基础。
1 材料与方法 1.1 材料 1.1.1 质粒、菌株及实验动物大肠杆菌DH5α和Rosetta(DE3);pMD19-T质粒、pET22b(+)原核载体由实验室保存;PDCoV阳性病料由本实验室保存;两月龄雌性新西兰兔购自简阳达硕动物科技有限公司。
1.1.2 主要试剂、材料PrimeScript RT reagent Kit、HindⅢ、T4DNA ligase均购自大连宝生物工程有限公司;Mouse anti-His单克隆抗体、HRP-羊抗兔二抗、弗氏完全佐剂和弗氏不完全佐剂均购自Sigma公司;PVDF膜购自Millipore公司。BCA蛋白浓度测定试剂盒(P0012S)购自碧云天公司;普通DNA产物纯化试剂盒(DP204)购自天根生化科技有限公司;质粒提取试剂盒购自OMEGA公司。
1.2 方法 1.2.1 S1基因的克隆以本实验室保存的PDCoV阳性病料为材料,提取病毒总RNA。采用PrimeScript RT reagent Kit合成单链cDNA, 取1 μL产物采用克隆引物(参考CH/Sichuan/S27/2012;GenBank:KT266822.1)进行扩增,上游引物:5′-ATGCAGAGAGCTCTATTGATTATGAC-3′;下游引物:5′-ATAAAAGAAAGTAGGTGTGACAATAG-3′,目的片段大小为1 566 bp。PCR产物经纯化后克隆至pMD19-T载体,转化大肠杆菌DH5α中进行氨苄抗性筛选,重组大肠杆菌进行菌落PCR鉴定和序列测定。
1.2.2 生物信息学分析通过Edit Seq分析测序结果的开放阅读框、蛋白编码、蛋白分子量及等电点。用在线软件signalP3.0 ( http://www.cbs.dtu.dk/services/SignalP/)分析S1蛋白信号肽序列,软件分别运用神经网络模型(neural networks,NN)和隐马尔可夫模型(hiddenMarkov models,HMM)。利用在线软件TMHMM Server v. 2.0(http://genome.cbs.dtu.dk/services/TMH-MM-2.0/)分析S1蛋白跨膜结构。利用DNAStar软件中的Protean软件提供的模块,用Chou-Fasman和Gamier-Rob-son法分析S1蛋白的二级结构、用Kyte-Doolit-tle法分析S1的亲水性,用Karplus-Schulz法分析S1的柔性区域,用Emini法分析S1蛋白的表面可能性。利用DNAStar软件中的Protean提供的模块,采用Jameson-Wolf法分析鼠S1蛋白的B细胞表位。
1.2.3 Sa基因引物的设计及其片段的扩增参考生物信息软件预测结果,设计一对单酶切同源重组连接表达载体的特异性引物, 上游引物:5′-agtgcggccgcaagcttCATAAACCCACGAGTAATTCCA CC-3′, 下游引物:5′-gagctccgtcgacaagcttATCAGTGATTACACTAGAAGTGTCT-3′(HindⅢ),扩增Sa基因106—1 290 bp(36—430 aa), 目的片段为1 188 bp, 以pMD19-T-S1质粒为模板对Sa基因进行PCR扩增,PCR反应程序:98 ℃ 5 min;98 ℃ 15 s;57 ℃ 15 s;72 ℃ 40 s; 34个循环;72 ℃ 8 min,PCR产物经1%琼脂糖凝胶电泳鉴定。
1.2.4 Sa基因原核表达载体的构建与重组蛋白的表达PCR产物纯化后参照ClonExpressⅡ同源重组克隆试剂盒使用说明书与使用HindⅢ限制性内切酶处理的pET-22b(+)表达载体进行连接。连接转化大肠杆菌感受态细胞DH5α,阳性克隆菌落PCR鉴定和上海生工测序。质粒少量提取试剂盒提取测序正确的pET22b-Sa重组质粒转化大肠杆菌感受态Rosetta(DE3)菌株。挑取经PCR验证的阳性菌落,接种至6 mL含120 mg·L-1 Amp LB液体培养基的试管中37 ℃、220 r·min-1摇至OD600 nm=0.6,加IPTG至终浓度为0.8 mmol·L-1,37 ℃、220 r·min-1诱导表达5 h。于诱导表达后取菌液5 mL,离心得菌体,加入SDS-PAGE上样缓冲液, 煮沸8 min,12%SDS-PAGE分析,同时取空质粒菌液诱导5 h的产物作为对照。
参考上述方法,取未诱导阳性菌液1 mL接种至200 mL含120 mg·L-1 Amp LB液体培养基的500 mL三角瓶中,37 ℃、220 r·min-1培养至OD600 nm=0.6~0.8, 加入IPTG至终浓度为0.8 mmol·L-1, 诱导表达5 h;12 000 r·min-1离心10 min,菌体用50 mmol·L-1 Tris-HCl进行重悬,采用超声破碎的方法,离心后收集上清和沉淀。样品经SDS-PAGE电泳后,转至聚偏二氟乙烯(PVDF)膜上,经5%脱脂乳封闭,以Mouse anti-His单克隆抗体(1:5 000)为一抗,羊抗鼠HRP-IgG(1:7 000)为二抗,通过DAB底物显色进行Western blot鉴定。
1.2.5 Sa蛋白的纯化对含有目的蛋白的上清液,12 000 r·min-1离心10 min,之后采用0.22 μm滤膜过滤、超滤柱浓缩、Ni柱亲合层析纯化的方法进行纯化,纯化后的蛋白质进行SDS-PAGE电泳鉴定。
1.2.6 兔抗Sa蛋白多克隆抗体的制备使用BCA蛋白定量试剂盒测定Sa蛋白浓度(3.938 mg·mL-1),并把蛋白浓度定至1 mg·mL-1,与等体积的弗氏完全佐剂或弗氏不完全佐剂乳化,免疫新西兰兔(表 1),制备兔抗Sa蛋白多克隆抗体。免疫前耳静脉采集血液,间接ELISA测定抗体效价。四免10 d后经心脏采血,分离血清,于-20 ℃保存备用。
|
|
表 1 免疫程序 Table 1 Immune program |
以纯化的Sa蛋白为包被抗原,37 ℃1 h包被ELISA板(0.2 μg·孔), PBST洗涤3次(5 min·次-1, 下同)后用5%的脱脂奶粉37 ℃封闭1.5 h, PBST洗涤3次, 以兔抗Sa蛋白多克隆抗体为一抗(1:1 000~1:12 800), 37 ℃孵育1 h, PBST洗涤3次, 加入HRP标记羊抗兔IgG(1:5 000), 37 ℃孵育1 h, PBST洗涤3次后加入TMB显色底物, 显色10 min后加入终止液, 酶标仪检测OD值。以免疫血清OD450 nm值与阴性对照血清OD450 nm的比值≥2.1为阳性判定标准,判定制备的兔抗Sa蛋白多克隆抗体的最低检出效价。
1.2.8 兔抗Sa蛋白多克隆抗体免疫活性的鉴定将纯化得到的蛋白样品经SDS-PAGE电泳分离后,转印到PVDF膜上, 然后用5%脱脂奶粉于37 ℃封闭1 h,TBST漂洗3次,用采集到的兔多抗血清稀释(1:200)后作为一抗孵育过夜,TBST洗3次,HRP-IgG(羊抗兔)二抗(1:7 000倍)孵育1 h,TBST洗涤3次,最后加入底物(DAB),反应5~15 min,观察显色情况;对PVDF膜上的条带进行照相及分析。
2 结果 2.1 PDCoV-S1基因的克隆利用克隆型特异性引物对PDCoV-S1基因进行RT-PCR,扩增出了大小约为1 566 bp的cDNA片段,其长度与预期大小相一致(图 1)。
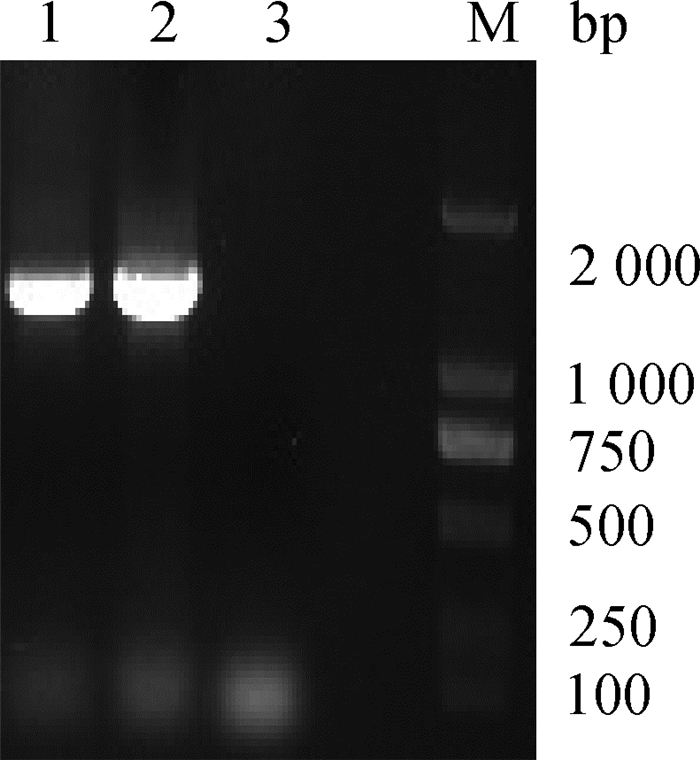
|
M. DL2000 DNA相对分子质量标准;1~2. S1基因扩增产物;3.阴性对照 M.DNA marker DL2000;1-2. RT-PCR product of S1 gene; 3. Control 图 1 S1基因的RT-PCR扩增 Figure 1 Amplification of S1 gene by RT-PCR |
本研究克隆得到PDCoV的S1基因(GenBank No.:KY398009)。经Edit Seq分析表明, S1的cDNA序列含有一个完整1 566 bp的开放阅读框, 编码522个氨基酸, 碱基组分分别为A共418个碱基(26.7%),C共379的碱基(24.2%),G共275个碱基(17.6%),T共494个碱基(31.5%)。氨基酸的等电点为6.15,蛋白质的相对分子质量为57.8 ku。通过与CH/Sichuan/S27/2012毒株S1基因进行比对,仅有碱基位点突变存在,没有碱基的插入与缺失。
2.2.2 S1蛋白跨膜结构的分析SignalP4.1软件分析信号肽切割位点,预测方法为神经网络(neural network),结果显示(图 2A)该蛋白序列含有一个信号肽切割位点,位于19、20位氨基酸之间(KFA19-D20D)。将推导的蛋白氨基酸序列运用TMHMM server软件预测跨膜区,分析结果(图 2B)表明该蛋白非跨膜蛋白。
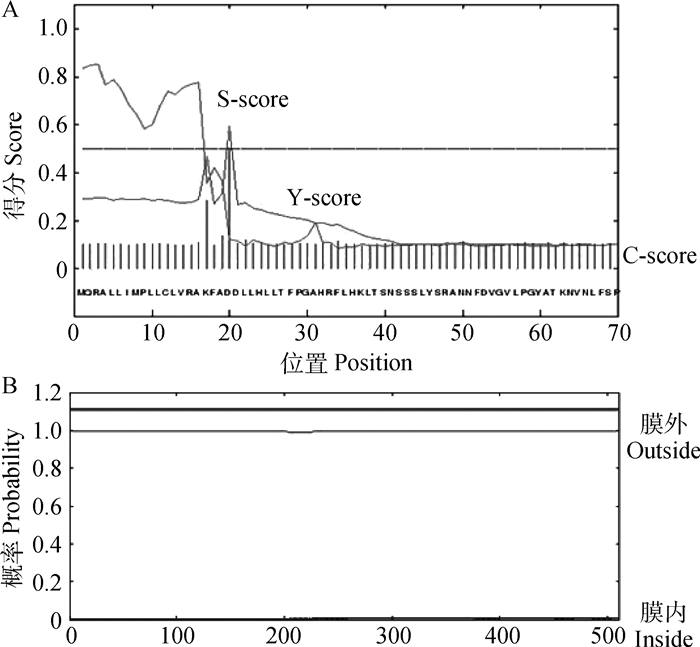
|
图 2 S1蛋白的信号肽分析(A)和跨膜区域分析(B) Figure 2 Signal peptide analysis of S1 protein(A) and transmembrane domain analysis (B) |
Chou-Fasman法预测S1蛋白α螺旋结果显示,S1蛋白骨架可形成多个α螺旋,位置分别在第18—20、36—40、231—240、289—290、300—302、321—324、341—344区段;用Gamier-Robson法预测的α螺旋结构较Chou-Fasman方法预测的数量少,但结果基本一致(图 3A)。Kyte-Doolittle法分析S1的亲水性结果显示,S1蛋白分子亲水性区域的分布不均匀,亲水区域占整个蛋白的多数部分,为亲水蛋白(图 3B)。Karplus-Schulz法分析S1的柔性区域结果显示,S1蛋白含有较多的柔性区域,主要集中在蛋白第19—21、36—50、58—64、68—76、82—88、94—98、106—111、123—124、136—138、146—150、158、168—170,174—177、182—185、191—196、201—204、225—228、242—243、249—255、260—263、270—274、282—286、290—294、298—303、320区域(图 3C)。Emini法对S1蛋白表面可能性预测的结果显示,S1蛋白的氨基酸表面可能性区域主要是第2—7、10—12、46—50、55—63、84—89、120—124、130—135、180—191区段,其他区段展示的表面可能性较小或表现为负值(图 3D)
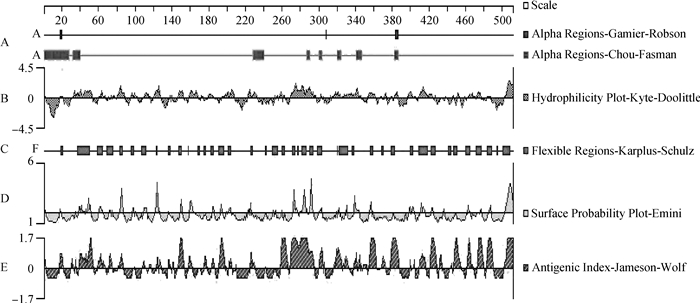
|
A.蛋白α螺旋结构的预测(Chou-Fasman法与Gamier-Robson法);B.蛋白亲水性的预测(Kyte-Doolittle方法);C.蛋白的柔性区域的预测(Karplus-Schulz方法);D.蛋白表面可能性的预测(Emini方法);E.蛋白潜在抗原表位的预测(Jameson-Wolf方法) A. α-Helix prediction (Gamier-Robson and Chou-Fasman models); B. Hydrophilicity prediction (Kyte-Doolittle model); C. Flexible regions prediction (Karplus-Schulz model); D. Surface probability prediction (Emini model); E. Antigenic prediction (Jameson-Wolf model) 图 3 S1蛋白的二级结构分析 Figure 3 The secondary structure prediction of S1 protein |
Jameson-Wolf法分析S1蛋白潜在的抗原表位位点结果显示,S1蛋白存在15个潜在的B细胞抗原表位,主要集中于40—460 aa的位置。与CH/Sichuan/S27/2012株S1蛋白相比,都具有15个抗原,存在抗原表位氨基酸的变异:37—40 aa由KPTS变为KLTS(图 3E)。
2.3 重组原核表达载体pET22b-Sa的鉴定随机挑取数个Rosetta(DE3)单菌落,经PCR验证其在1 200 bp左右出现特异条带(图 4),其结果与预期相符。菌液扩大培养后抽提质粒送生工生物(上海)股份有限公司测序, 测序结果与目的基因一致,说明外源基因已成功转化至大肠杆菌受体菌株中。
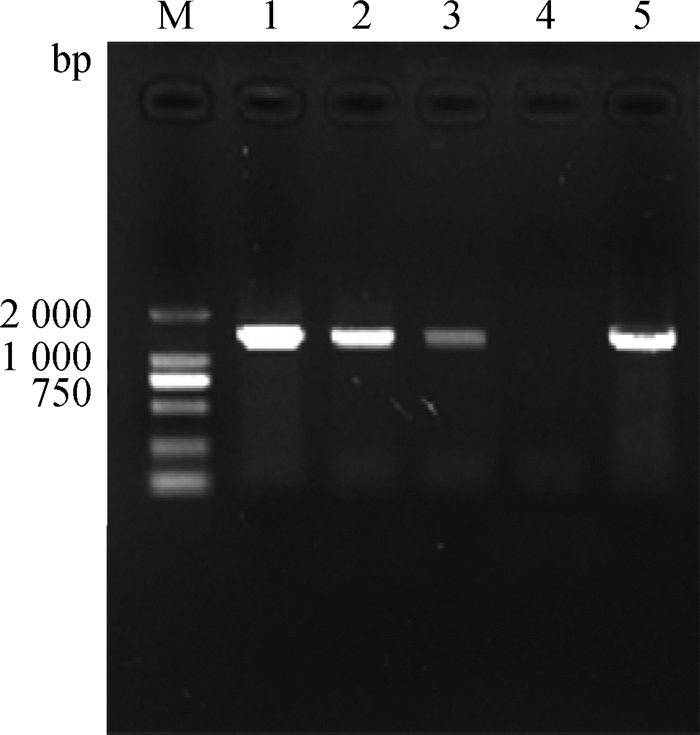
|
M. DL2000 DNA相对分子质量标准;1~3. Rosetta菌液样品;4.阴性对照;5.阳性对照 M. DL2000 DNA marker; 1-3. Bacteria samples; 4. Negative control; 5. Positive control 图 4 PDCoV Sa基因鉴定结果 Figure 4 Identification results of PDCoV Sa gene |
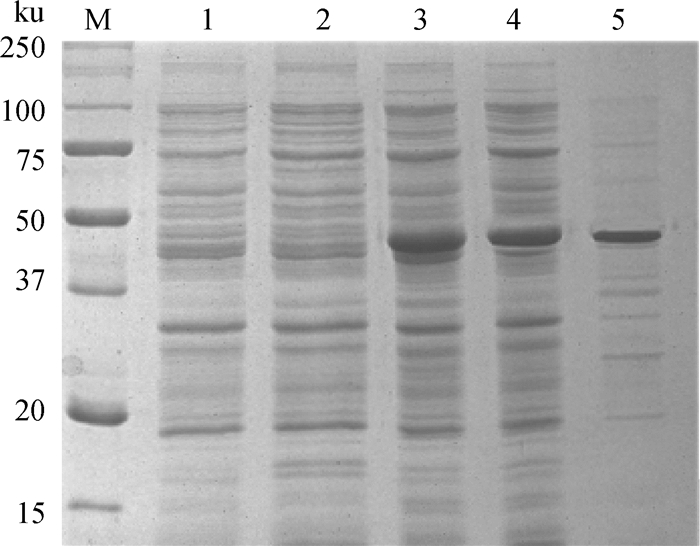
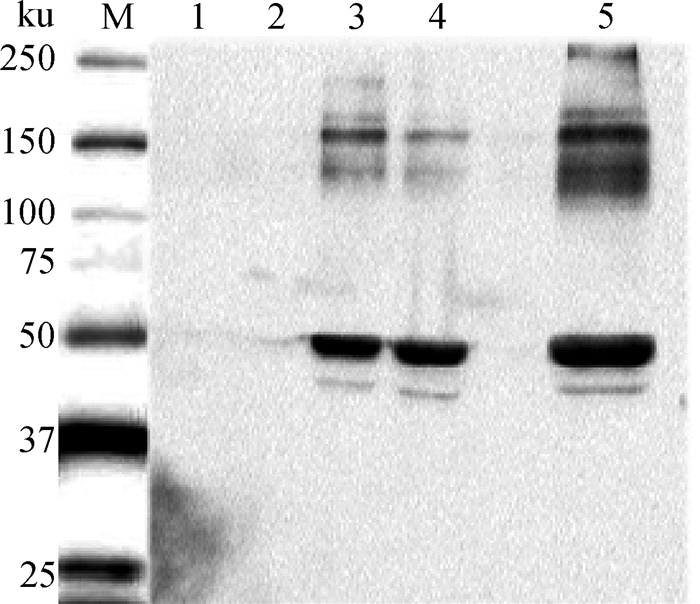
诱导后,目的蛋白在pET22b-Sa上清与包涵体中均有表达,相对分子质量约为46 ku(图 5),将重组Sa蛋白进行Western blot检测,结果显示,pET22b-Sa上清蛋白和包涵体蛋白均在46 ku处有一条目的条带(图 6),说明重组蛋白能与Anti-His-tag反应。
|
M.蛋白质相对分子质量标准;1.空载2.诱导前;3.诱导后全菌;4.上清;5.包涵体 M. Protein marker; 1:Rosetta (DE3) light; 2:pET22b-Sa not induced; 3. All bacteria after induction; 4. The supernatant fluid protein; 5. Inclusion body protein 图 5 重组蛋白质可溶性分析 Figure 5 Soluble analysis of Recombinant protein |
|
M.蛋白质相对分子质量标准;1.空载;2.诱导前;3.诱导后包涵体;4.诱导后上清;5.诱导后全菌 M. Protein marker; 1. Rosetta (DE3) light; 2. pET22b-Sa not induced; 3. Inclusion body protein; 4. The supernatant fluid protein; 5. All bacteria after induction 图 6 Sa蛋白的Western blot鉴定(His单克隆抗体) Figure 6 Detection of recombinant protein Sa by Western blot(His mAb) |
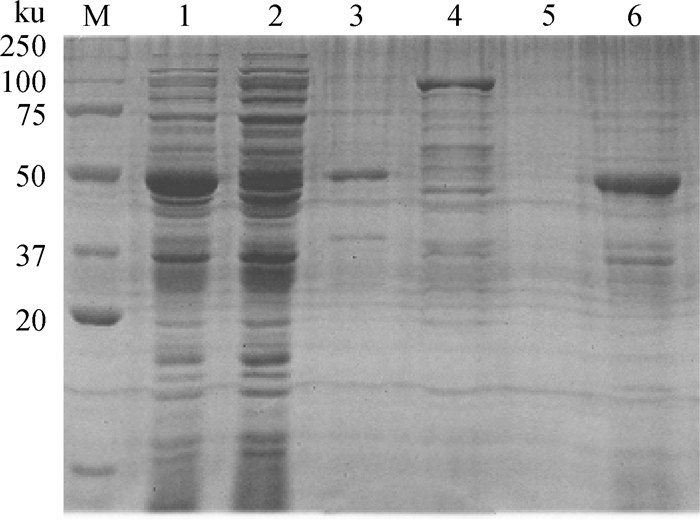
将上清蛋白离心超滤之后经Ni亲和层析柱纯化得到纯度较高的Sa蛋白(图 7)。
|
M.蛋白质相对分子质量标准;1.全菌;2.上清;3.包涵体;4.洗脱峰1;5.洗脱峰2;6.纯化后蛋白 M. Protein marker; 1. The total bacteria liquid; 2. Protein in solution; 3. Inclusion body protein; 4. Elution peak for the first time; 5. The second elution peak; 6. Purified protein 图 7 重组蛋白的纯化结果 Figure 7 Purification of recombinant protein |
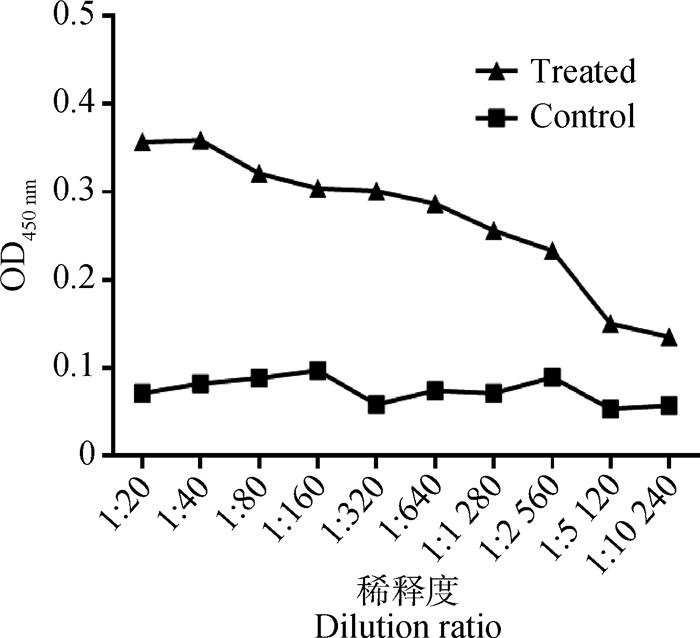
兔抗S1蛋白的血清进行间接ELISA检测时,免疫血清OD450 nm值与阴性对照血清OD450 nm的比值≥2.1为阳性判定标准,经计算,此研究制备的兔抗血清效价为1:10 240(图 8), 为了检验所制备抗体的特异性,利用纯化后的pET22b-Sa重组蛋白,经Western blot检测发现所制备的阳性抗血清组在46 ku处存在特异条带;同时,以阴性血清作为阴性对照,未见条带(图 9),这说明所制备的抗血清具有较好的特异性。
|
图 8 间接ELISA方法检测兔抗S1蛋白多克隆抗体的效价 Figure 8 In-ELISA analysis of positive samples |
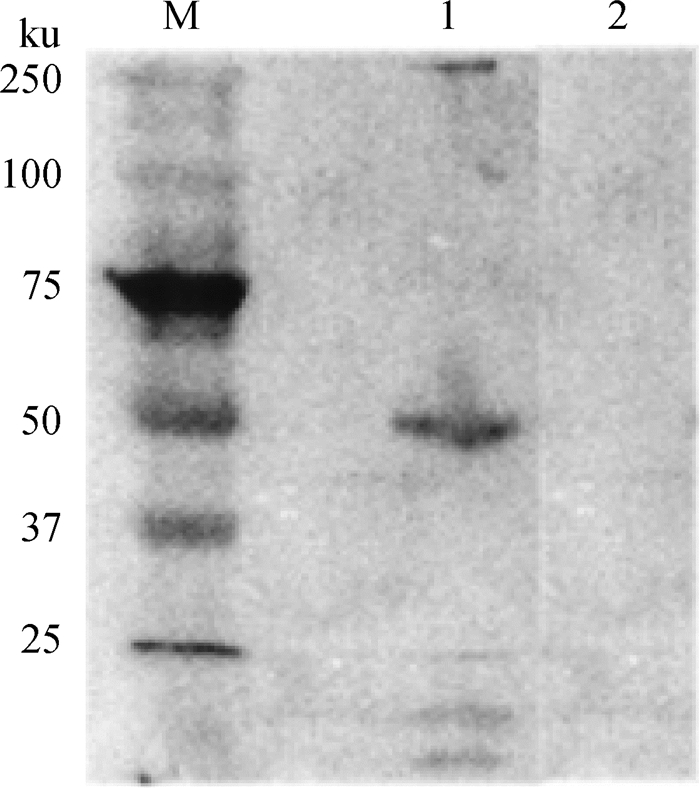
|
M.蛋白质相对分子质量标准;1.阳性血清;2.阴性血清 M. Protein marker; 1. Positive antiserum; 2. Negative antiserum 图 9 多克隆抗体特异性鉴定 Figure 9 Polyclonal antibody specificity identification |
PDCoV作为一种新发型病毒性腹泻的致病原,可单独感染仔猪,也可与PEDV、TGEV、PoRV混合感染[21],引起患病仔猪的死亡,给养殖业带来巨大的损失。S蛋白作为冠状病毒的纤突糖蛋白,在病毒侵袭宿主,决定病毒毒力等方面发挥作用[22]。目前对于PDCoV S基因及功能的研究还十分有限,根据其他猪冠状病毒的研究报道得知,冠状病毒S蛋白缺乏水解位点不能被切割,N端的抗原表位区S1区主要是病毒与细胞受体结合的功能区[23],C端的膜融合区域S2区与膜融合有关[24]。可以推测PDCoV的S1蛋白包含主要的中和位点以及刺激宿主细胞产生中和抗体的能力[3]。针对PDCoV S1基因在不同毒株间变异性较大的特点[25],通过对S1基因的序列测定,可以作为诊断鉴别不同毒株的依据。本研究成功扩增出了S1基因,序列全长1 566 bp,编码522个氨基酸,通过与NCBI公布的CH/Sichuan/S27/2012株S1基因相比,虽然在基因序列方面,两株病毒S1基因存在较多点突变,但在B细胞抗原表位预测结果显示,两蛋白在抗原表位上基本一致没有差异,除此之外,它们的糖基化位点也基本一致,证明该病毒在四川的流行并没有发生变异。由结果“2.2.4 ”对PDCoV CHN-SC2015株S1蛋白的抗原表位预测结果发现该蛋白的抗原表位主要集中Sa区域,综合其他冠状病毒属成员[26]如PEDV的S蛋白抗原表位分布在不含信号肽切割位点的非跨膜区域,最终选择了CHN-2015株S1蛋白的36—430 aa残基用以表达,该蛋白的氨基酸序列均位于S1区域并且不含有信号肽切割位点;目前对于PDCoV S基因及蛋白的结构与功能尚不明确,PDCoV的检测技术还不完善,因此对S蛋白深入研究在该病的治疗和预防等方面有应用前景。本研究中证实Sa(36—430 aa)蛋白存在抗原表位,但不能定义PDCoV抗原表位的准确位置,需要进一步通过试验验证。
大肠杆菌表达系统具有操作简单、周期短、价格低、获得量高等优点,且人们对其基因背景、表达特征的了解比较清楚。虽然存在一些缺陷,如没有翻译后加工的功能,以及表达的蛋白没有足够的生物学活性,但表达产物在与功能活性无关的免疫性质研究中较为理想,故仍然是目前最受欢迎的异源表达系统之一[27]。有研究指出,糖基化蛋白不易于原核表达,根据宿主对密码子的偏爱性优化cDNA密码子,提高靶蛋白表达水平[28]。试验中pET22b-Sa表达载体转入Rosetta感受态细胞进行诱导表达的表达量不是很高,可能与病毒蛋白本身特性有关,可考虑密码子优化或更换能优于pET-22b(+)的载体用以表达病毒蛋白。在运用间接ELISA的方法检测多抗血清的效价时,Sa蛋白包被时间不够导致测定的多抗效价明显偏低,所以在测定多抗效价过程中应该提前优化测定的条件,防止出现数值的偏差。
通过SDS-PAGE分析证实了重组蛋白主要以上清的形式表达, 使用该重组蛋白作为免疫原免疫健康雌性新西兰大白兔, 成功制备了抗Sa蛋白多克隆抗体, 并且以该抗体为一抗进行蛋白质印迹分析, 证实了Sa蛋白的相对分子质量为46 ku, 以上这些研究结果一方面证明了S1基因抗原表位预测区Sa蛋白具有抗原性, 另一方面也为我们将来进一步研究Sa蛋白的结构和功能奠定了坚实的基础。
4 结论克隆了PDCoV的S1基因,在生物信息学分析基础上,将抗原表位预测区域Sa插入大肠杆菌中进行原核表达,提纯蛋白免疫新西兰大白兔制备了兔抗Sa蛋白高免血清,证明原核表达的Sa蛋白具有良好的抗原性和免疫原性,间接确定该区域存在PDCoV的抗原表位,为开展S蛋白的应用研究奠定了基础。
| [1] | WOO P C Y, LAU S K P, LAM C S F, et al. Discovery of seven novel mammalian and avian coronaviruses in the genus Deltacoronavirus supports bat coronaviruses as the gene source of Alphacoronavirus and Betacoronavirus and avian coronaviruses as the gene source of Gammacoronavirus and Deltacoronavirus[J]. J Virol, 2012, 86(7): 3995–4008. DOI: 10.1128/JVI.06540-11 |
| [2] | SU M J, LI C Q, GUO D H, et al. A recombinant nucleocapsid protein-based indirect enzyme-linked immunosorbent assay to detect antibodies against porcine deltacoronavirus[J]. J Vet Med Sci, 2016, 78(4): 601–606. DOI: 10.1292/jvms.15-0533 |
| [3] | ZHANG J Q. Porcine deltacoronavirus:Overview of infection dynamics, diagnostic methods, prevalence and genetic evolution[J]. Virus Res, 2016, 226: 71–84. DOI: 10.1016/j.virusres.2016.05.028 |
| [4] | WOO P C Y, HUANG Y, LAU S K P, et al. Coronavirus genomics and bioinformatics analysis[J]. Viruses, 2010, 2(8): 1804–1820. DOI: 10.3390/v2081803 |
| [5] | JUNG K, HU H, EYERLY B, et al. Pathogenicity of 2 porcine deltacoronavirus strains in gnotobiotic pigs[J]. Emerg Infect Dis, 2015, 21(4): 650–654. DOI: 10.3201/eid2104.141859 |
| [6] | MCCLUSKEY B J, HALEY C, ROVIRA A, et al. Retrospective testing and case series study of porcine delta coronavirus in U.S. swine herds[J]. Prev Vet Med, 2016, 123: 185–191. DOI: 10.1016/j.prevetmed.2015.10.018 |
| [7] | MA Y M, ZHANG Y, LIANG X Y, et al. Origin, evolution, and virulence of porcine deltacoronaviruses in the United States[J]. mBio, 2015, 6(2): e00064. |
| [8] | LEE J H, CHUNG H C, NGUYEN V G, et al. Detection and phylogenetic analysis of porcine deltacoronavirus in Korean swine farms, 2015[J]. Transbound Emerg Dis, 2016, 63(3): 248–252. DOI: 10.1111/tbed.2016.63.issue-3 |
| [9] | LORSIRIGOOL A, SAENG-CHUTO K, TEMEEYASEN G, et al. The first detection and full-length genome sequence of porcine deltacoronavirus isolated in Lao PDR[J]. Arch Virol, 2016, 161(10): 2909–2911. DOI: 10.1007/s00705-016-2983-8 |
| [10] |
龙云凤, 王毅谦, 姜珊, 等. 猪δ冠状病毒研究进展[J]. 亚洲兽医病例研究, 2017, 6(2): 21–28.
LONG Y F, WANG Y Q, JIANG S, et al. Research progress on porcine deltacoronavirus[J]. Asian Case Reports in Veterinary Medicine, 2017, 6(2): 21–28. (in Chinese) |
| [11] | CHEN F Z, ZHU Y X, WU M Z, et al. Full-length genome characterization of Chinese porcine deltacoronavirus strain CH/SXD1/2015[J]. Genome Announc, 2015, 3(5): e01284–15. |
| [12] | WANG Y W, YUE H, FANG W H, et al. Complete genome sequence of porcine deltacoronavirus strain CH/Sichuan/S27/2012 from Mainland China[J]. Genome Announc, 2015, 3(5): e00945–15. |
| [13] |
陈建飞, 王潇博, 焦贺勋, 等. 国内首株猪德尔塔冠状病毒(Porcine deltacoronavirus)的分离鉴定[J]. 中国预防兽医学报, 2016, 38(3): 171–174.
CHEN J F, WANG X B, JIAO H X, et al. Isolation and identification of the first Porcine deltacoronavirus strain in China[J]. Chinese Journal of Preventive Veterinary Medicine, 2016, 38(3): 171–174. (in Chinese) |
| [14] | DONG N, FANG L R, ZENG S L, et al. Porcine deltacoronavirus in Mainland China[J]. Emerg Infect Dis, 2015, 21(12): 2254–2255. DOI: 10.3201/eid2112.150283 |
| [15] |
吴美洲, 陈芳洲, 朱银杏, 等. 丁型冠状病毒我国猪源株的遗传变异分析[J]. 中国兽医科学, 2016, 46(6): 689–694.
WU M Z, CHEN F Z, ZHU Y X, et al. Genetic variation analysis of porcine deltacoronavirus in China[J]. Chinese Veterinary Science, 2016, 46(6): 689–694. (in Chinese) |
| [16] | SONG D, ZHOU X, PENG Q, et al. Newly emerged porcine Deltacoronavirus associated with diarrhoea in swine in China: Identification, prevalence and full-length genome sequence analysis[J]. Transbound Emerg Dis, 2015, 62(6): 575–580. DOI: 10.1111/tbed.2015.62.issue-6 |
| [17] | WANG L Y, HAYES J, SARVER C, et al. Porcine deltacoronavirus: Histological lesions and genetic characterization[J]. Arch Virol, 2016, 161(1): 171–175. DOI: 10.1007/s00705-015-2627-4 |
| [18] | SUO S Q G W, REN Y D, LI G X, et al. Immune responses induced by DNA vaccines bearing Spike gene of PEDV combined with porcine IL-18[J]. Virus Res, 2012, 167(2): 259–266. DOI: 10.1016/j.virusres.2012.05.007 |
| [19] | GÓMEZ N, WIGDOROVITZ A, CASTAÑÓN S, et al. Oral immunogenicity of the plant derived spike protein from swine-transmissible gastroenteritis coronavirus[J]. Arch Virol, 2000, 145(8): 1725–1732. DOI: 10.1007/s007050070087 |
| [20] | THACHIL A, GERBER P F, XIAO C T, et al. Development and application of an ELISA for the detection of porcine deltacoronavirus IgG antibodies[J]. PLoS One, 2015, 10(4): e124363. |
| [21] | ZHAI S L, WEI W K, LI X P, et al. Occurrence and sequence analysis of porcine deltacoronaviruses in southern China[J]. Virol J, 2016, 13: 136. DOI: 10.1186/s12985-016-0591-6 |
| [22] | MAHY B W J. Virology: Molecular biology of the coronaviruses[J]. Nature, 1983, 305(5934): 474–475. DOI: 10.1038/305474a0 |
| [23] | BRIAN D A, BARIC R S. Coronavirus genome structure and replication[M]//ENJUANES L. Coronavirus Replication and Reverse Genetics. Berlin Heidelberg: Springer, 2005. |
| [24] | LEE H K, YEO S G. Cloning and sequence analysis of the nucleocapsid gene of porcine epidemic diarrhea virus Chinju99[J]. Virus Genes, 2003, 26(2): 207–212. DOI: 10.1023/A:1023447732567 |
| [25] | CHEN Q, LI G W, STASKO J, et al. Isolation and characterization of porcine epidemic diarrhea viruses associated with the 2013 disease outbreak among swine in the United States[J]. J Clin Microbiol, 2014, 52(1): 234–243. DOI: 10.1128/JCM.02820-13 |
| [26] | SUN D B, FENG L, SHI H Y, et al. Identification of two novel B cell epitopes on porcine epidemic diarrhea virus spike protein[J]. Vet Microbiol, 2008, 131(1-2): 73–81. DOI: 10.1016/j.vetmic.2008.02.022 |
| [27] |
解庭波. 大肠杆菌表达系统的研究进展[J]. 长江大学学报:自然科学版, 2008, 5(3): 77–82.
XIE T B. Research progress on E. coli expression system[J]. Journal of Yangtze University:Natural Science Edition, 2008, 5(3): 77–82. (in Chinese) |
| [28] |
苏鹏, 龚国利. 优化大肠杆菌表达外源蛋白的研究进展[J]. 生物技术通报, 2017, 33(2): 16–23.
SU P, GONG G L. Research progress on optimizing the expression of exogenous proteins in Escherichia coli[J]. Biotechnology Bulletin, 2017, 33(2): 16–23. (in Chinese) |



