2. 天津农学院动物科学与动物医学学院, 天津 300384
2. College of Animal Science and Veterinary Medicine, Tianjin Agricultural University, Tianjin 300384, China
随着我国生猪养殖集约化的持续推进,养猪业逐渐进入微利时代,成本控制成为该时期一个鲜明特征。在养猪生产中,饲料投入约占总养殖成本的70%,因此,饲料利用效率的微小提升就会带来饲养成本的大幅下降[1-2]。因此,饲料利用效率越来越受到人们的重视。目前,对猪的生长性状和瘦肉率、产仔数等繁殖性状的选择已经取得明显进展,饲料利用效率现已成为猪育种中重要选择目标性状之一。饲料利用效率评价指标主要包括饲料增重比(feed/gain ratio, F/G)、增重饲料比(gain/feed ratio, G/F)和剩余采食量(residual feed intake,RFI)。从统计学角度来说,直接利用比值性状制定选择指数是不尽合理的[3]。RFI是畜禽实际采食量与用于维持和增重所需要的预测采食量之差,能够反映动物本身由遗传背景决定的代谢差异。同时,多项研究表明,RFI具有中等遗传力[4-6],是可遗传的。因此,RFI更加适合作为饲料利用效率的评估指标用于畜禽遗传选育。随着电子饲喂设备的更新换代以及采食量测定系统的不断发展,RFI现已应用于一些有关饲料利用效率的研究中[1, 7-9]。
估计RFI需要大量的平均日采食量数据,虽然电子饲喂设备的更新换代以及采食量测定系统的不断发展使得采食量数据收集更加便利,但是依然昂贵费时。因此,鉴定与RFI相关的分子标记十分必要。应用重测序技术,Liu等[10]检测出46个与科宝鸡RFI显著相关的SNPs。Seabury等[11]利用混合模型进行GWAS分析,在安格斯牛中定位到7个高效QTLs,在海福特牛中鉴定出4个高效QTLs。目前,科研人员对猪RFI分子机理也开展了许多工作。Fan等[12]通过关联分析发现,FTO上p.Ala198Ala和TCF7L2上c.646+514A>G与RFI显著关联。Sanchez等[13]在大白猪SSC6上检测到一个与RFI相关的QTL。Onteru等[14]通过GWAS方法在大白猪SSC2、3、5、7、8、9、14和15上检测到了与RFI显著关联的SNPs。Mauch等[9]在SSC1、5、6、14和16上定位到与RFI相关的QTLs。Bai等[15]以军牧1号白猪为研究对象,利用GWAS方法检测到12个与RFI显著关联的SNPs。2014年Do等[16]对杜洛克猪的饲料利用效率开展研究,认为HLCS是猪RFI性状的重要候选基因。但目前针对HLCS的研究较少,在猪中未见相关报道,其对猪剩余采食量的影响尚不明确。
HLCS基因位于人21号染色体长臂22区,全长HLCS蛋白包含726个氨基酸,预测分子量为81 ku。猪HLCS基因位于13号染色体上,包含12个外显子。全羧化酶合成酶能够催化生物素与羧化酶及组蛋白结合[17-19],生物素化的羧化酶在糖原异生、脂肪酸合成、氨基酸等代谢途径中发挥关键作用[20],生物素化的组蛋白可抑制基因转录[21]。本研究以猪HLCS基因为研究对象,检测其组织表达情况,筛选多态性,并探讨多态位点与RFI性状之间的关系。
1 材料与方法 1.1 试验材料试验所用猪为美系杜洛克,均来自河南省雏鹰农牧集团股份有限公司尉氏分公司,3头杜洛克猪在100 kg左右时进行屠宰,迅速收集心、肝、脾、肾、脂肪、肌肉、小肠和胃底组织于冻存管中,并尽快置于液氮罐中,用于组织表达分析。利用美国奥斯本自动喂料系统测定1 292头美系杜洛克猪的每日采食量和每日体重,并采集所有试验中涉及个体的耳组织。90日龄开始测定,体重达100 kg时结束,并在此时用Aquila Vet兽用B超仪测定猪的背膘厚及眼肌面积。试验中RFI计算参考前人方法[22]:
RFI=ADFI-(b1×(OnBW-30)+ b2×(OffBW-100)+ b3×MetamidBW + b4×ADGA + b5×OffBFA),式中,ADFI指平均日采食量;OnBW指初测体重;OffBW指结测体重;MetamidBW指中间代谢体重,计算公式为((OnBW+OffBW)/2)0.75;ADGA代表 90~180天之间的平均日增重校正值;OffBFA指测试结束时10~11肋背膘厚校正到100 kg体重时的值。100 kg背膘厚和30~100 kg ADGA的校正公式来源于加拿大遗传改良计划。
1.2 主要试剂Taq DNA聚合酶、dNTP混合物、动物组织总RNA提取试剂盒均购自天根生化科技有限公司(北京);反转录试剂盒(PrimeScriptTM RT Reagent Kit)和荧光定量染料(SYBR® Premix Ex TaqTM Ⅱ)均购于TaKaRa公司(大连)。
1.3 DNA和RNA的提取用酚-仿法从耳组织样品中提取基因组DNA,利用Nano Drop 2000核酸定量仪测定每头猪耳样DNA的浓度,A260 nm/A280 nm比值为1.7~2.2为合格样品。使用动物组织总RNA提取试剂盒提取总RNA,提取过程全按照试剂盒说明书操作。提取的RNA经IMPLEN超微量分光光度计检测合格方可用于后续试验。按照反转录试剂盒说明书将mRNA反转录成cDNA。
1.4 引物设计参考HLCS基因mRNA序列(GenBank登录号为XM_021070965.1),使用NCBI数据库中的Primer-BLAST设计普通PCR和荧光定量PCR引物,GAPDH作为内参基因,引物信息见表 1。引物均由英潍捷基(上海)贸易有限公司合成。
|
|
表 1 普通PCR和荧光定量PCR引物 Table 1 Primers for PCR amplifications and real-time PCR for porcine HLCS |
利用ABI QuantStudioTM 7 Flex实时荧光定量PCR仪检测HLCS在心、肝、脾、肾、脂肪、肌肉、小肠和胃底组织中的表达量。反应总体积为20 μL:2×SYBR® Premix Ex Taq Ⅱ 10 μL,上下游引物(10 μmol·L-1)各0.4 μL,50×ROX Reference Dye Ⅱ 0.4 μL,模板2 μL,ddH2O 6.8 μL。PCR程序:95 ℃预变性30 s;95 ℃变性5 s,60 ℃ 34 s,40个循环;反应结束后分析熔解曲线。采用2-ΔΔCt法计算目的基因相对表达量。使用SAS9.2软件中的ANOVA程序进行方差分析。
1.6 PCR扩增HLCS基因扩增反应体系为25 μL:10×Taq Buffer 2.5 μL,dNTP(10 mmol·L-1) 0.5 μL,ddH2O 18.5 μL,模板2 μL,上下游引物(10 μmol·L-1)各0.5 μL,DNA聚合酶(2.5 U·μL-1) 0.5 μL。PCR反应条件:94 ℃预变性3 min;94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸1 min,35个循环;72 ℃延伸10 min;4 ℃保存。反应产物用1.5%琼脂糖凝胶电泳检测,条带明亮且长度符合预期的产物交由北京诺赛基因组研究中心有限公司进行Sanger测序。运用SeqMan软件对测序结果进行分析。
1.7 统计分析计算各变异位点等位基因频率、基因型频率、多态信息含量、群体杂合度及Hardy-Weinberg平衡检验。使用SAS9.2软件中的GLM程序,配合下列模型进行最小二乘方差分析,比较RFI在各基因型之间的差异。使用的模型:
| $ {Y_{ijkl}} = \mu + {G_i} + {S_j} + {B_k} + {P_l} + {e_{ijkl}} $ |
其中,Yijkl为性状测定值;μ为群体均值;Gi为基因型效应;Sj为性别效应;Bk为测定批次效应;Pl为胎次效应;eijkl为随机误差。
2 结果 2.1 猪HLCS基因组织表达谱分析荧光定量PCR检测HLCS基因mRNA在杜洛克猪多种组织中的表达情况如图 1所示。HLCS基因在所检测的8种组织中均有表达,其中,在脂肪组织中表达量最高,肝次之,在心和肌肉中痕量表达。方差分析显示,HLCS在脂肪组织中表达量显著高于除肝以外的其它组织(P<0.05),在肝中的表达显著高于除脂肪和脾以外的其它组织(P<0.05),在心和肌肉中的表达则显著低于除胃底以外的其它所有组织(P<0.05)。
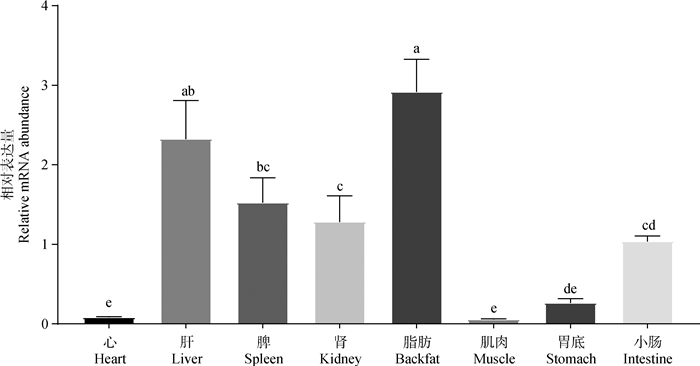
|
不同组织间,不同字母表示差异显著(P<0.05),相同字母表示不显著(P>0.05) The different letters indicate significant difference (P < 0.05), the same letter indicate no significant difference (P > 0.05) among different tissues 图 1 HLCS在杜洛克猪各组织中的表达水平 Figure 1 Relative mRNA abundance of the porcine HLCS in 8 tissues of Duroc pigs |
在1 292头美系杜洛克中挑选RFI具有明显差异的个体各10头组成高、低组。高组个体RFI均值为(0.528±0.077) kg,低组个体RFI均值为(-0.545±0.059) kg。利用PCR扩增和测序技术,在HLCS编码区共发现5个SNPs(图 2),其中,c.1173C>T和c.1408G>C位于外显子4,c.1976A>G位于外显子6,c.2160G>A位于外显子7,c.2325G>A位于外显子8。其中,c.1408G>C和c.1976A>G为非同义突变位点,前者使得丙氨酸转变为脯氨酸,后者导致天冬氨酸转变为甘氨酸。
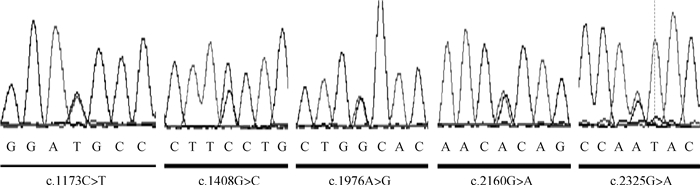
|
图 2 猪HLCS基因SNPs位点示意图 Figure 2 Schematic representation of SNPs in porcine HLCS |
用288头杜洛克个体针对c.1408G>C和c.1976A>G位点进行基因分型,分别成功分型226头和241头,各位点的基因型频率、等位基因频率及群体遗传特性见表 2。从表 2可以看出,2个多态位点的杂合度均接近0.5,说明它们的纯合度和杂合度基本相同。PIC值显示,这2个位点在此杜洛克群体中的多态性较高,遗传变异比较大。c.1408G>C和c.1976A>G的优势等位基因均为G,基因频率分别为0.513和0.575;优势等位基因型均为杂合基因型。卡方检验显示,c.1408G>C在所选择的杜洛克猪群中未达到Hardy-Weinberg平衡(P<0.05),而c.1976A>G处于Hardy-Weinberg平衡(P>0.05)。
|
|
表 2 HLCS基因多态位点c.1408G>C和c.1976A>G的基因型和等位基因频率及群体遗传特性 Table 2 The genotype and allele frequency and the population genetic characteristics of c.1408G > C and c.1976A > G in HLCS |
通过对c.1408G>C和c.1976A>G多态位点与RFI性状进行群体关联分析发现,前者基因型间的RFI没有显著差异(P>0.05),但表现出GG基因型个体的RFI比CC基因型个体低的趋势;后者与RFI显著关联(P<0.5),AA基因型个体的RFI显著高于GG基因型个体(表 3)。2个位点上的有利等位基因型比不利等位基因型分别低0.018 1和0.063 4 kg。
|
|
表 3 HLCS多态位点基因型与RFI的关联分析 Table 3 Association of genotypes at the two polymorphism sites with RFI |
生物素是生物体新陈代谢不可或缺的物质,它是生物素依赖性羧化酶的辅助因子。生物素依赖性羧化酶主要有丙酰辅酶A羧化酶、丙酮酸羧化酶、甲基巴豆酰辅酶A羧化酶和2种乙酰辅酶A羧化酶,这些羧化酶在糖异生、脂肪酸合成及氨基酸分解代谢中催化关键反应[23]。新合成的生物素依赖性羧化酶不具有酶活性,需在HLCS催化下与生物素以共价键结合后才具有活性[24]。目前,针对HLCS的研究主要集中在人的羧化酶缺乏症上。羧化酶缺乏症主要分为两种,一种是生物素酶缺乏(BTD)所致,一种是全羧化酶合成酶缺乏(HLCSD)所致。HLCS活性下降,导致生物素无法与生物素依赖的羧化酶结合,从而影响羧化酶活性,使脂肪酸合成、糖异生及氨基酸的分解代谢发生障碍,这些过程与机体新陈代谢密切相关[25]。临床上,羧化酶缺乏的儿童会表现出发育迟缓的症状[26]。研究表明,代谢通路(metabolic pathways)与猪RFI性状显著关联,并且代谢过程中各因子的变异会引起RFI的变化[27];而HLCS基因就位于代谢通路中。鉴于HLCS在新陈代谢中的重要作用及Do等[16]的研究结果,本研究分析了猪HLCS基因的多态性,并进一步分析了猪HLCS基因与RFI的关系。
本研究首先检测了猪HLCS基因在多种组织中的表达情况,发现其在脂肪组织和肝中表达量较高。在人类各相应组织中(不包括肌肉),HLCS也均表达,只是肾中表达量最高,肝次之,心最低,脂肪与肝接近[28]。这些结果预示着HLCS可能在脂质合成代谢过程中发挥重要作用。Lkhagvador等[29]利用RFI高、低组在猪的脂肪组织和肝中鉴定出大量差异表达基因,表明脂质合成代谢与RFI存在密切关系;但Lkhagvador等[29]鉴定出的差异表达基因并不包含HLCS。同时,本实验室前期对RFI高、低组杜洛克垂体组织RNA-seq数据的差异表达分析中,也未捕获到HLCS基因。由此推测,HLCS并不是通过表达量的改变影响RFI,而可能通过改变蛋白结构或功能来影响RFI。
在人类的全羧化酶缺乏症(holocarboxylase synthetase deficiency,HCSD)研究中均在患者的HLCS基因上检测到错义突变[25, 30-33]。R508W和V550M发生在生物素结合区域,是HCSD患者HLCS基因上常见突变,可导致HLCS与生物素结合能力下降[25]。Esaki等[34]利用定点突变的方法在HLCS基因上引入Q699R变异,发现重组HLCS蛋白与生物素的亲和力显著下降。这些研究结果进一步验证了上述猜测。猪HLCS基因上也存在诸多变异,截止到2017年12月2日,NCBI的dbSNP数据库[35]共收录了177个位于猪HLCS基因cDNA序列上的突变位点,其中166个为SNPs,11个为Indels,但是这些变异的生物学意义还未得到诠释。本研究中共发现5个SNPs,除c.1173C>T外均已存在于NCBI的dbSNP数据库中,其中c.1408G>C和c.1976A>G可导致错义突变。对c.1408G>C和c.1976A>G错义突变在杜洛克群体中分型,两者的优势等位基因均为G。根据Botstein等[36]提出的衡量多态信息含量标准,本研究中的2个错义突变位点多态信息含量(PIC)均为中度多态,表明2个位点具有作为育种标记的潜力,适合进行关联分析。通过对c.1408G>C和c.1976A>G多态位点与RFI性状进行群体关联分析发现,c.1408G>C基因型间的RFI没有显著差异(P>0.05),而c.1976A>G与RFI显著关联(P<0.05),GG基因型更有利。使用SMART(http://smart.embl-heidelberg.de/)分析猪HLCS氨基酸序列,发现c.1976A>G位于BPL_N结构域上。虽然该结构域功能未知,但其氨基酸序列变化更易改变蛋白结构及功能,进而影响猪RFI。本研究不仅为开展RFI性状分子选育提供了有价值的育种标记,同时也为进一步探究HLCS基因影响猪RFI的分子调控机理奠定了研究基础。
4 结论本研究表明,HLCS基因在猪的多种组织中均表达,且在与新陈代谢密切相关的肝、脂肪等组织中高表达。在猪HLCS基因编码区共检测到5个多态位点,包括2个错义突变位点c.1408G>C和c.1976A>G。对2个错义突变位点在杜洛克群体进行关联分析,发现c.1976A>G与杜洛克猪RFI存在显著关联。这些结果表明,猪HLCS基因上错义突变位点c.1976A>G可作为猪饲料利用效率性状选育的潜在分子标记。
| [1] |
蒲蕾, 岳慧洁, 张龙超, 等. 猪饲料利用效率的研究进展[J]. 中国农业大学学报, 2016, 21(4): 77–85.
PU L, YUE H J, ZHANG L C, et al. Current progress in feed utilization efficiency of pig[J]. Journal of China Agricultural University, 2016, 21(4): 77–85. (in Chinese) |
| [2] |
邓昌彦. 影响养猪生产成本的主要因素及其控制对策[J]. 农业新技术(今日养猪业), 2005(3): 48–50.
DENG C Y. The main factors tha affect the cost of pig production and its control countermeasures[J]. New Agricultural Technology (Pigs Today), 2005(3): 48–50. (in Chinese) |
| [3] |
杨运清, 王忠新, 尹炳北. 比值性状的指数选择方法探讨[J]. 畜牧兽医学报, 2000, 31(3): 198–202.
YANG Y Q, WANG Z X, YIN B B. Study on the index selection method for ratio trait[J]. Acta Veterinaria et Zootechnica Sinica, 2000, 31(3): 198–202. (in Chinese) |
| [4] | GILBERT H, BIDANEL J P, GRUAND J, et al. Genetic parameters for residual feed intake in growing pigs, with emphasis on genetic relationships with carcass and meat quality traits[J]. J Anim Sci, 2007, 85(12): 3182–3188. DOI: 10.2527/jas.2006-590 |
| [5] | HOQUE M A, KADOWAKI H, SHIBATA T, et al. Genetic parameters for measures of residual feed intake and growth traits in seven generations of Duroc pigs[J]. Livest Sci, 2009, 121(1): 45–49. DOI: 10.1016/j.livsci.2008.05.016 |
| [6] | MAUCH E, SER O N, YOUNG J, et al. Diet by genotype interaction in yorkshire pigs divergently selected for feed efficiency[R]. IOWA State University, 2017. |
| [7] | GILBERT H, BILLON Y, BROSSARD L, et al. Divergent selection for residual feed intake in the growing pig[J]. Animal, 2017, 11(9): 1427–1439. DOI: 10.1017/S175173111600286X |
| [8] | WILLIAMS K, WEIGEL K, COBLENTZ W, et al. 0321 effect of diet energy level and genomic residual feed intake on dairy heifer performance[J]. J Anim Sci, 2016, 94(S5): 154. |
| [9] | MAUCH E D, SERÃO N V, YOUNG J M, et al. 0391 genomic regions associated with residual feed intake of divergently selected lines of Yorkshire pigs when fed a low-energy, high-fiber diet[J]. J Anim Sci, 2016, 94(S5): 189. |
| [10] | LIU J, LIU R, WANG J, et al. Exploring genomic variants related to residual feed intake in local and commercial chickens by whole genomic resequencing[J]. Genes (Basel), 2018, 9(2): E57. DOI: 10.3390/genes9020057 |
| [11] | SEABURY C M, OLDESCHULTE D L, SAATCHI M, et al. Genome-wide association study for feed efficiency and growth traits in U.S. beef cattle[J]. BMC Genomics, 2017, 18: 386. DOI: 10.1186/s12864-017-3754-y |
| [12] | FAN B, LKHAGVADORJ S, CAI W, et al. Identification of genetic markers associated with residual feed intake and meat quality traits in the pig[J]. Meat Sci, 2010, 84(4): 645–650. DOI: 10.1016/j.meatsci.2009.10.025 |
| [13] | SANCHEZ M P, TRIBOUT T, IANNUCCELLI N, et al. A genome-wide association study of production traits in a commercial population of Large White pigs:Evidence of haplotypes affecting meat quality[J]. Genet Sel Evol, 2014, 46: 12. DOI: 10.1186/1297-9686-46-12 |
| [14] | ONTERU S K, GORBACH D M, YOUNG J M, et al. Whole genome association studies of residual feed intake and related traits in the pig[J]. PLoS One, 2013, 8(6): e61756. DOI: 10.1371/journal.pone.0061756 |
| [15] | BAI C, PAN Y, WANG D, et al. Genome-wide association analysis of residual feed intake in Junmu No.1 White pigs[J]. Anim Genet, 2017, 48(6): 686–690. DOI: 10.1111/age.2017.48.issue-6 |
| [16] | DO D N, OSTERSEN T, STRATHE A B, et al. Genome-wide association and systems genetic analyses of residual feed intake, daily feed consumption, backfat and weight gain in pigs[J]. BMC Genet, 2014, 15: 27. |
| [17] | WOLF B. Disorders of biotin metabolism[M]//SCRIVER C R, BEAUDET A L, SLY W S, et al. The metabolic and molecular basis of inherited disease. New York: McGraw-Hill, 2001: 3935-3956. |
| [18] | STANLEY J S, GRIFFIN J B, ZEMPLENI J. Biotinylation of histones in human cells:Effects of cell proliferation[J]. FEBS J, 2001, 268(20): 5424–5429. |
| [19] | KOBZA K, CAMPOREALE G, RUECKERT B, et al. K4, K9 and K18 in human histone H3 are targets for biotinylation by biotinidase[J]. FEBS J, 2005, 272(16): 4249–4259. DOI: 10.1111/j.1742-4658.2005.04839.x |
| [20] | ZEMPLENI J, WIJERATNE S S K, HASSAN Y I. Biotin[J]. BioFactors, 2009, 35(1): 36–46. DOI: 10.1002/biof.v35:1 |
| [21] | PESTINGER V, WIJERATNE S S K, RODRIGUEZ-MELENDEZ R, et al. Novel histone biotinylation marks are enriched in repeat regions and participate in repression of transcriptionally competent genes[J]. J Nutr Biochem, 2011, 22(4): 328–333. DOI: 10.1016/j.jnutbio.2010.02.011 |
| [22] | GORBACH D, CAI W, DEKKERS J C M, et al. Large-scale SNP association analyses of residual feed intake and its component traits in pigs[C]//Proceedings of the 9th World Congress on Genetics Applied to Livestock Production. Leipzig, Germany: German Society for Animal Science, 2010: 265. |
| [23] | LEÓN-DEL-RÍO A, VALADEZ-GRAHAM V, GRAVEL R A. Holocarboxylase synthetase:A moonlighting transcriptional coregulator of gene expression and a cytosolic regulator of biotin utilization[J]. Annu Rev Nutr, 2017, 37: 207–223. DOI: 10.1146/annurev-nutr-042617-104653 |
| [24] | MOCK D M. Biotin:From nutrition to therapeutics[J]. J Nutr, 2017, 147(8): 1487–1492. DOI: 10.3945/jn.116.238956 |
| [25] |
郑宏, 卢婷婷, 陆相朋, 等. 全羧化酶合成酶缺乏症1例临床及基因分析[J]. 临床儿科杂志, 2017, 35(8): 605–608.
ZHENG H, LU T T, LU X P, et al. The clinical and genetic features of holocarboxylase synthetase deficiency in a male patient[J]. Journal of Clinical Pediatrics, 2017, 35(8): 605–608. (in Chinese) |
| [26] | ELREFAI S, WOLF B. Disorders of biotin metabolism[M]//ROSENBERG R N, PASCUAL J N. Rosenberg's Molecular and Genetic Basis of Neurological and Psychiatric Disease. 5th ed. San Diego, CA: Elsevier, 2015: 531-539. |
| [27] | DO D N, STRATHE A B, OSTERSEN T, et al. Genome-wide association and pathway analysis of feed efficiency in pigs reveal candidate genes and pathways for residual feed intake[J]. Front Genet, 2014, 5: 307. |
| [28] | FAGERBERG L, HALLSTRÖM B M, OKSVOLD P, et al. Analysis of the human tissue-specific expression by genome-wide integration of transcriptomics and antibody-based proteomics[J]. Mol Cell Proteomics, 2014, 13(2): 397–406. DOI: 10.1074/mcp.M113.035600 |
| [29] | LKHAGVADOR J S, QU L, CAI W, et al. Gene expression profiling of the short-term adaptive response to acute caloric restriction in liver and adipose tissues of pigs differing in feed efficiency[J]. Am J Physiol Regul Integr Comp Physiol, 2010, 298(2): R494–R507. DOI: 10.1152/ajpregu.00632.2009 |
| [30] |
李端, 刘丽, 李秀珍, 等. 中国人多种羧化酶缺陷症患儿4例及其父母基因突变分析[J]. 中华儿科杂志, 2006, 44(11): 865–868.
LI D, LIU L, LI X Z, et al. Gene mutation analysis in four Chinese patients with multiple carboxylase deficiency[J]. Chinese Journal of Pediatrics, 2006, 44(11): 865–868. DOI: 10.3760/j.issn:0578-1310.2006.11.018 (in Chinese) |
| [31] |
张豪正, 王广新. HLCS基因突变致全羧化酶合成酶缺乏症一例的临床和遗传学特点[J]. 中华肥胖与代谢病电子杂志, 2017, 3(2): 82–85.
ZHANG H Z, WANG G X. Clinical and genetic characteristics of holocarboxylase synthetase deficiency in a case[J]. Chinese Journal of Obesity and Metabolic Diseases (Electronic Edition), 2017, 3(2): 82–85. (in Chinese) |
| [32] | HUI J, LAW E, CHUNG C, et al. The first reported HLCS gene mutation causing holocarboxylase synthetase deficiency in a Vietnamese patient[J]. World J Pediatr, 2012, 8(3): 278–280. DOI: 10.1007/s12519-011-0301-9 |
| [33] |
刘舒, 刘畅, 曾玉坤, 等. 一个罕见的羧化全酶合成酶缺乏症家系的临床分析与基因诊断[J]. 中国优生与遗传杂志, 2015, 23(10): 35–37, 45.
LIU S, LIU C, ZENG Y K, et al. Clinical and gene mutation analysis in a family of holocarboxylase synthetase deficiency[J]. Chinese Journal of Birth Health & Heredity, 2015, 23(10): 35–37, 45. (in Chinese) |
| [34] | ESAKI S, MALKARAM S A, ZEMPLENI J. Effects of single-nucleotide polymorphisms in the human holocarboxylase synthetase gene on enzyme catalysis[J]. Eur J Hum Genet, 2012, 20(4): 428–433. DOI: 10.1038/ejhg.2011.198 |
| [35] | SHERRY S T, WARD M H, KHOLODOV M, et al. dbSNP:The NCBI database of genetic variation[J]. Nucleic Acids Res, 2001, 29(1): 308–311. DOI: 10.1093/nar/29.1.308 |
| [36] | BOTSTEIN D, WHITE R L, SKOLNICK M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. Am J Hum Genet, 1980, 32(3): 314–331. |



