2. 农业部草牧业创新重点实验室, 兰州 730020;
3. 甘肃省肉羊繁育生物技术工程实验室, 民勤 733300
2. Key Laboratory of Grassland Livestock Industry Innovation of Ministry of Agriculture, Lanzhou 730020, China;
3. Sheep Breeding Biotechnology Engineering Laboratory of Gansu Province, Minqin 733300, China
随着基因组学和生物信息学的不断发展,基因组结构变异研究的不断深入,学者们挖掘出越来越多的基因组结构变异类型。拷贝数变异(Copy number variations, CNV)成为继简单重复序列(Simple sequence repeat, SSR)和单核苷酸多态性(Single nucleotide polymorphism, SNP)之后一种新的基因组结构变异类型。2006年R.Redon等[1]结合SNP基因分型和微阵列比较基因组杂交技术(Array-based comparative genomic hybridization, aCGH)对270个人的全基因组进行了CNV鉴定,构建了人的第一张CNV图谱,并将获得的大量CNV数据储存在基因组变异数据库中(The Database of Genomic Variants, DGV)。随后的研究发现,CNV覆盖比例可达到人类基因组的12%[2]。科学家们对许多模式动物,如果蝇[3]、黑猩猩[4]、小鼠[5]的CNV研究发现,CNV和不同类型疾病的发生具有很强的关联性。在牛上,2010年G.E.Liu等[6]利用aCGH技术第一次在全基因组水平分析了17个品种共计90头牛的全基因组CNV,发现一些特异性的拷贝数变异区(Copy number variation regions, CNVRs)与不同生产类型的牛品种密切相关。因此,本文对牛全基因组CNV的研究进展进行全面总结,以期为牛CNV今后的研究提供一定的指导信息。
1 CNV的定义及形成机制DNA序列的复杂变异为物种表型的变异奠定了分子基础,从单个碱基到染色体畸变,变异类型分为单核苷酸多态性(SNP)、寡核苷酸的插入缺失(Indels)、拷贝数变异(CNV)、基因组结构变异(SV)4种形式[7]。J.L.Freeman和S.W.Scherer等[8-9]研究表明,拷贝数变异(CNV)是基因组结构变异(SV)的重要组成部分,主要指1 kb~5 Mb的DNA片段拷贝数改变,这些片段的缺失、重复、倒位和易位统称为CNV。随着新一代测序技术和相应运算程序的结合发展,越来越小的具有拷贝数突变性质的DNA片段被识别,CNV的定义也随之发生改变。2011年,R.E.Mills等[10]将CNV定义为个体之间基因组序列上>50 bp片段的插入、缺失及其它复杂变异。
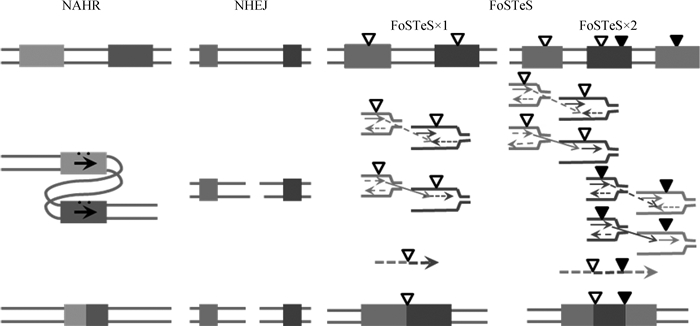
染色体结构的改变主要表现为同源重组(Homologous recombination, HR)和非等位同源重组(Non-allelic homologous recombination, NAHR)两种机制[11]。NAHR是产生CNV的重要机制,主要发生在减数分裂时期的DNA损伤修复过程中,往往位于同源重复区(Segmental duplication, SD)或低拷贝的重复区内(Low copy repeats, LCRs)[12]。同源序列的倒位[13]、重复或缺失都会造成特定基因拷贝数的改变进而导致染色体结构的改变[14]。此外,基于DNA损伤及错误重复修复的非同源末端连接(Non-homologous end-joining, NHEJ)、重复叉停滞及模板交换(Fork stalling and template switching, FoSTeS)和微同源序列介导重复和重组(Microhomology-mediated replication-dependent recombination, MMBIR)也是产生CNV的重要途径[15]。NHEJ与NAHR的形成特征不同,它是在两个重组末端之间不具有高同源序列的基础上仍然能够发生DNA断端连接,导致染色体重排的发生(图 1)。
2 CNV在牛全基因组上的分布特点CNV在牛基因组上的分布特点在一定程度上反映了CNV的形成机制及不同牛种的选择压和驯化历史特点。牛基因组上CNV的分布具有非随机性。通过比对不同染色体上总CNV长度与其染色体长度的比值发现CNV的丰度在不同染色体中存在显著差异,其中含CNV丰度较高的染色体为5、15、18、27、29和X[17]。研究同时发现,这些染色体上富含片段重复序列(Segmental duplication, SD),说明牛基因组中的SD与高频率CNV存在极显著正相关[18],也证实SD是拷贝数变异发生的热点区域和主要诱导形式[6, 19-20]。牛基因组上CNV的长度在100~200 kb范围内所占的比例最高。此外,不同牛品种的CNV数量和在染色体上的分布位置也存在很大的差异,暗示CNV数量和分布位置的差异与不同牛品种的生存环境及受到的选择压密切相关[21]。G.E.Liu等[22]研究表明,瘤牛和非洲普通牛种的CNV丰度远大于欧洲普通牛种,这种差异主要是由不同牛种的分化程度、历史有效群体大小、基因渗入和选择压所导致[23]。
3 全基因组CNV研究方法随着基因组学与生物信息学的快速发展,各类基因组CNV检测技术和相应运算程序的运用使全基因CNV的研究更加全面深入。目前检测全基因组CNV的方法主要有两大类、一类是以核苷酸互补杂交为基础的比较基因组杂交(aCGH)和SNP芯片检测方法,另一类是以测序为基础的新一代直接测序技术(Next-generation sequencing, NGS)[24]。研究者们利用3种全基因组CNV检测方法对牛基因组CNV进行了大量的研究,并取得了很大的进展(表 1)。
|
|
表 1 牛基因组CNV研究汇总 Table 1 Summary of CNV surveys performed in cattle |
比较基因组杂交(CGH)是首次应用于全基因组范围内检测CNV的有效方法。其原理是在一张有固定探针的特制芯片上用不同的荧光素分别标记试验样本和对照样本并进行共杂交,通过特定仪器检测试验样本和对照样本之间荧光比率的变化来确定试验样本的相对拷贝数变化。aCGH的分辨率主要取决于制备芯片过程中固定在芯片上探针的密度和片段大小,为了识别更多更有效的CNV,科学家通过增加芯片中探针密度及制备更短的探针,结合更高的信噪比,不断提高CNV的检测效率。根据芯片制作过程中探针的来源可细分为细菌人工染色体CGH芯片(Bacterial artificial chromosome-comparative genomic hybridization, BCA-CGH)和寡核苷酸探针CGH芯片(Oligo-CGH)。其中BCA-CGH芯片具有较高的信噪比,但分辨率较低,很难检测到50 kb以下的CNV信号,制备芯片的代价昂贵,费时费力。Oligo-CGH芯片则具有高精度、高分辨率、易制备的优点[29]。
3.2 SNP芯片技术检测全基因组CNVCNV也可以通过SNP芯片技术检测,SNP芯片不需要同时使用两个样本的DNA(试验组和对照组)和探针进行双杂交,通过比较测试样本的信号强度与其他个体的强度确定每个位点的相对基因组拷贝数[30]。与CGH相比,SNP芯片的优点在于鉴定CNV的同时还可判定其基因型,还可以显示杂合性缺失[30]。但芯片上的探针都是以已知的CNV所构建,导致这种以核苷酸杂交为基础的CNV检测方法通常具有一些系统性的缺陷。比如不能检测小片段的CNV、很难检测位于SD区域中的CNV及发掘新的、无偏差的复杂突变[24]。
3.3 新一代测序技术检测全基因组CNV新一代测序技术是将研究个体测序序列与参考基因组比对并确定CNV,克服了微阵列杂交技术的固有缺陷,对基因组CNV的检测具有很大优势[19]。首先,新一代测序技术相较于基于探针杂交的方法具有更高的分辨率和灵敏度;其次,应用测序技术可以鉴定位于SD序列内的复杂结构变异,从而进一步促进芯片技术的升级[31];最后,通过测序技术检测CNV的费用持续下降,各种算法不断成熟使得研究范围和样本量不断扩大[32]。目前,新一代测序技术已广泛应用于牛基因组CNV的研究中[17, 25, 33-34]。
4 CNV的遗传效应CNV作为基因组结构变异的主要形式之一,在不同物种基因组上普遍存在。与SNP相比,CNV所涉及的核苷酸数目远远大于SNP,故其会造成基因组上大片段基因序列改变,进而导致基因结构的改变和基因表达量的变化[35]。例如,通过重复或缺失DNA片段对单个或多个基因的结构造成改变进而导致基因表达的增强或减弱,甚至丧失基因的功能[7, 16, 19]。2004年J.Sebat等[36]首次在人基因组研究中发现了大量的拷贝数多态性(Copy number polymorphisms,CNPs),证实CNV基因参与调节神经功能、细胞生长、物质代谢和疾病发生。大量的研究报道CNV在人类疾病易感性方面扮演着重要角色,比如CCL3L1基因和HIV、AIDS[37],FCGR3B基因和血管球性肾炎[38],DEFB4A基因和克罗恩病[39]等。随后人们进一步对其他物种,包括家禽[40]、猪[41-42]、狗[43]、绵羊[44]、山羊[45]、马[46]、牦牛[47]等全基因组CNV的研究,发现涉及CNV的基因与复杂性状及环境适应性相关,暗示CNV是揭示动物经济性状和分析群体遗传结构的重要工具。
4.1 CNV与牛数量性状的相关关系对人类和啮齿类动物的研究提出,CNV在生物体的健康发育、疾病易感性评估、物种的演化等方面扮演重要的角色[16, 48]。目前,大量的研究报道了牛基因组上CNV和牛数量性状密切相关,并将CNV作为研究复杂性状的一种分子标记应用于全基因组关联性分析(GWAS)中。M.Mielczarek等[7]研究表明,在牛基因组拷贝数变异类型中缺失型的比率远远大于重复型,大部分的缺失断点依靠cGMP蛋白激酶的表达,而大多数扩增断点鉴定为TRAC基因,在生物免疫过程中扮演重要角色。Y.H.Gao等[17]利用全基因组重测序技术研究4头高乳脂、乳蛋白和4头低乳脂、乳蛋白荷斯坦牛的CNV,发现差异CNVRs主要涉及脂质和蛋白的代谢,并鉴定10个候选基因作为早期选育的分子标记。A.M.Duran等[49]利用Illumina芯片技术对荷斯坦奶牛CNV与牛奶体细胞数评分做了关联性分析,通过SV 8.3.1和PENNCNV-CNVRULER两种不同的检测方法分别鉴定到7个和11个候选基因作为全基因组选择的分子标记。Y.L.Hou等[50]通过对高、低剩余采食量(Residual feed intake, RFI)荷斯坦奶牛做全基因组CNV检测,探讨CNV和饲料利用效率的关系,发现高饲料利用率奶牛的特异CNV与炎症反应和免疫应答相关,而低饲料利用率奶牛的特异CNV与骨骼和器官发育相关。
4.2 CNV和牛种群体遗传结构的关系基因组结构的变异是研究物种群体遗传结构的重要资源。CNV为物种的驯化、选择和品种形成提供重要的分子标记。CNV作为继SNP和SSR之后研究基因组结构变异的一个新视角,能够从不同角度探究物种群体遗传结构的问题[6, 51]。通过研究不同遗传背景、地理分布牛品种基因组的特异CNVRs,揭示牛种群体遗传结构差异的遗传学基础[52-55]。L.Z.Zhang等[53]分析12个不同父系、母系起源的中国牛种CNVRs分布特点,并对不同起源牛种的CNVRs做聚类分析,确定了中国牛种之间的起源进化和群体结构。L.Y.Xu等[52]利用高密度SNP芯片分析了世界范围内8个牛种的CNV,结果显示,不同牛种CNV与牛种地理分布和群体结构具有很强的关联性,与先前基于SNP研究群体结构的结果相一致。
4.3 CNV对基因表达的剂量效应分析人类基因组中有近一半的CNVRs与蛋白编码区相重叠,CNV导致基因结构和剂量的改变,对物种进化、表型变异和疾病发生等产生重要影响[36]。大量的研究结果表明CNV和基因表达剂量存在非常复杂的关系[56-58]。研究者将这种复杂的关系归结为“剂量效应”。“剂量效应”是指结构基因或者调控序列在基因组上的拷贝数目发生改变,从而引起相应基因表达水平和蛋白或者表型变化的现象[59]。通过分析CNV基因和其mRNA及蛋白表达水平的关系,将剂量效应分为剂量敏感型、剂量不敏感型CNV。将剂量敏感型CNV中能引起其mRNA及蛋白表达水平降低甚至失活的一类称为剂量反转型[56]。由于物种在长期的进化过程中形成的蛋白互作机制可以抵制剂量敏感型CNV对基因结构和剂量的改变[60],因此绝大多数涉及CNV的基因与其mRNA及蛋白表达水平没有显著关系,表现为低水平的敏感性,即剂量不敏感型。
5 牛Y染色体CNV研究牛Y染色体的雄性特异区(Male specific region of Y chromosome, MSY)在减数分裂过程中不与X染色体进行同源重组,而且含有大量重复序列的回文结构,导致很难获得Y染色体的基因连锁图谱和准确的序列信息[61]。目前,牛全基因组CNV的研究仅局限在常染色体,而Y染色体CNV的研究只针对特定的多拷贝基因家族,这些多拷贝基因家族往往特异或主要地在睾丸中表达,暗示这些多拷贝基因和公牛精子发生、繁殖力密切相关[62],其拷贝数变异可作为公牛繁殖力早期选育的有效分子标记。
通过研究杂种牛和纯种牛Y染色体上与精子发生相关基因的拷贝数变异能够为解释牛种父系起源和繁殖力差异提供理论基础。A.Mandal等[63]检测了杂种牛(普通牛×瘤牛)和瘤牛Y染色体上4个与精子发生相关基因的绝对拷贝数,发现SRY基因(Sex determining region on Y chromosome)拷贝数和TSPY基因(Testis-specific protein Y-encoded, TSPY)拷贝数在杂种牛中显著高于瘤牛,并结合前人对于不同牛种Y染色体长度显著差异的研究,推断TSPY拷贝数的增加使得杂种牛的Y染色体增长[64]。G.W.Zhang等[65]通过研究HSFY、ZNF280BY、PRAMEY和TSPY在牦牛和犏牛中拷贝数的差异来揭示犏牛雄性不育的分子机制,发现在犏牛中TSPY基因的拷贝数显著高于牦牛,认为TSPY基因拷贝数的异常增多会导致雄性犏牛的不育。
X.P.Yue等[66-67]对15个牛品种共460头公牛的PRAMEY基因(Preferentially expressed antigen in melanoma, Y-linked)、ZNF280BY基因(Zinc finger protein 280B, Y-linked)和HSFY基因(Heat-shock transcription factor, Y-linked)的拷贝数变异进行了分析,发现3个基因的拷贝数在品种间和品种内都存在显著差异,其中普通牛血统牛种的PRAMEY和ZNF280BY中值拷贝数显著低于瘤牛血统的牛种,而HSFY的中值拷贝数在普通牛血统的牛种中显著高于瘤牛血统的牛种。这3个基因的CNV与荷斯坦公牛繁殖性状的关联分析发现,PRAMEY拷贝数和阴囊周径(Scrotal circumference, SC)、相对阴囊周径(Relative length of scrotal circumference, RLSC)、不返情率(Non-return rate, NRR)、正常精子比例(Percentage of normal sperm, PNS)呈显著负相关[67];ZNF280BY和HSFY拷贝数与SC和RLSC呈负相关,ZNF280BY基因与公牛授精后受配母牛受孕率(Sir conception rate, SCR)呈正相关[66]。C.K.Hamilton等[68]分析了14个牛品种中TSPY基因拷贝数变异情况,发现TSPY基因的拷贝数在种间和种内也存在显著差异,并发现TSPY基因拷贝数与56天NRR成正相关,而与其mRNA表达水平呈显著负相关[57]。
6 牛全基因组CNV研究存在的问题与展望 6.1 牛全基因组CNV研究存在的问题全基因组CNV的研究不断地应用于牛GWAS、群体遗传结构分析等方面,成为牛种重要经济性状选择和起源进化分析的有效工具。但目前的研究仍然受到一定的限制:第一,CNV的鉴定还没有统一性的标准,使得不同研究的结果不能进行有效的整合分析;第二,目前使用的牛参考基因组仅为普通牛种(海福特牛),这导致研究瘤牛品种全基因组尤其是Y染色体CNV时会造成一定程度的偏差;第三,目前许多全基因组CNV研究只是筛选了大量候选基因及区域,但并未深入研究其功能和作用机理。
6.2 牛全基因组CNV研究展望CNV作为基因组结构变异的重要来源为物种复杂性状的研究提供分子基础。通过对不同牛种基因组CNV的研究,构建完整的全基因组CNV遗传图谱,为牛种基因组数据库做进一步的补充。将牛种特异性的CNV与性状及选择压相关联,比较不同牛种之间的CNV,揭示品种间的遗传变异,为优良性状选育和遗传资源多样性保护奠定基础。此外,通过结合CNV和SNP为牛种全基因组关联性分析(GWAS),选择重要经济性状的候选基因提供有力支撑,从而加快育种进程和提高遗传改良效果。
| [1] | REDON R, ISHIKAWA S, FITCH K R, et al. Global variation in copy number in the human genome[J]. Nature, 2006, 444(7118): 444–454. DOI: 10.1038/nature05329 |
| [2] | MACDONALD J R, ZIMAN R, YUEN R K, et al. The database of genomic variants:a curated collection of structural variation in the human genome[J]. Nucleic Acids Res, 2014, 42(D1): 986–992. DOI: 10.1093/nar/gkt958 |
| [3] | GRICE S J, LIU J L, WEBBER C. Synergistic interactions between Drosophila orthologues of genes spanned by de novo human CNVs support multiple-hit models of Autism[J]. PLoS Genet, 2015, 11(3): e1004998. DOI: 10.1371/journal.pgen.1004998 |
| [4] | MUCHMORE E A. Chimpanzee models for human disease and immunobiology[J]. Immunol Rev, 2010, 183(1): 86–93. |
| [5] | HIROI N, TAKAHASHI T, HISHIMOTO A, et al. Copy number variation at 22q11.2:from rare variants to common mechanisms of developmental neuropsychiatric disorders[J]. Mol Psychiatry, 2013, 18(11): 1153–1165. DOI: 10.1038/mp.2013.92 |
| [6] | LIU G E, HOU Y L, ZHU B, et al. Analysis of copy number variations among diverse cattle breeds[J]. Genome Res, 2010, 20(5): 693–703. DOI: 10.1101/gr.105403.110 |
| [7] | MIELCZAREK M, FR SZCZAK M, GIANNICO R, et al. Analysis of copy number variations in Holstein-Friesian cow genomes based on whole-genome sequence data[J]. J Dairy Sci, 2017, 100(7): 5515–5525. DOI: 10.3168/jds.2016-11987 |
| [8] | FREEMAN J L, PERRY G H, FEUK L, et al. Copy number variation:new insights in genome diversity[J]. Genome Res, 2006, 16(8): 949–961. DOI: 10.1101/gr.3677206 |
| [9] | SCHERER S W, LEE C, BIRNEY E, et al. Challenges and standards in integrating surveys of structural variation[J]. Nat Genet, 2007, 39: S7–S15. DOI: 10.1038/ng2093 |
| [10] | MILLS R E, WALTER K, STEWART C, et al. Mapping copy number variation by population-scale genome sequencing[J]. Nature, 2011, 470(7332): 59–65. DOI: 10.1038/nature09708 |
| [11] | HASTINGS P J, LUPSKI J R, ROSENBERG S M, et al. Mechanisms of change in gene copy number[J]. Nat Rev Genet, 2009, 10(8): 551–564. DOI: 10.1038/nrg2593 |
| [12] | BAILEY J A, GU Z P, CLARK R A, et al. Recent segmental duplications in the human genome[J]. Science, 2002, 297(5583): 1003–1007. DOI: 10.1126/science.1072047 |
| [13] | SMITH C E, LLORENTE B, SYMINGTON L S. Template switching during break-induced replication[J]. Nature, 2007, 447(7140): 102–105. DOI: 10.1038/nature05723 |
| [14] | BAUTERS M, VAN ESCH H, FRIEZ M J, et al. Nonrecurrent MECP2 duplications mediated by genomic architecture-driven DNA breaks and break-induced replication repair[J]. Genome Res, 2008, 18(6): 847–858. DOI: 10.1101/gr.075903.107 |
| [15] | LIU P F, CARVALHO C M B, HASTINGS P J, et al. Mechanisms for recurrent and complex human genomic rearrangements[J]. Curr Opin Genet Dev, 2012, 22(3): 211–220. DOI: 10.1016/j.gde.2012.02.012 |
| [16] | ZHANG F, GU W L, HURLES M E, et al. Copy number variation in human health, disease, and evolution[J]. Annu Rev Genom Hum Genet, 2009, 10(1): 451–481. DOI: 10.1146/annurev.genom.9.081307.164217 |
| [17] | GAO Y H, JIANG J P, YANG S H, et al. CNV discovery for milk composition traits in dairy cattle using whole genome resequencing[J]. BMC Genomics, 2017, 18: 265. DOI: 10.1186/s12864-017-3636-3 |
| [18] | 刘欣, 王立刚, 王立贤. 拷贝数变异及其研究进展[J]. 畜牧兽医学报, 2010, 41(8): 927–931. |
| [19] | PIROOZNIA M, GOES F S, ZANDI P P. Whole-genome CNV analysis:advances in computational approaches[J]. Front Genet, 2015, 6: 138. |
| [20] | LIU G E, VENTURA M, CELLAMARE A, et al. Analysis of recent segmental duplications in the bovine genome[J]. BMC Genomics, 2009, 10: 571. DOI: 10.1186/1471-2164-10-571 |
| [21] | DA SILVA J M, FERNANDA G P, DA SILVA L O, et al. Genome-wide copy number variation (CGNV) detection in Nelore cattle reveals highly frequent variants in genome regions harboring QTLs affecting production traits[J]. BMC Genomics, 2016, 17: 454. DOI: 10.1186/s12864-016-2752-9 |
| [22] | LIU G E, BROWN T, HEBERT D A, et al. Initial analysis of copy number variations in cattle selected for resistance or susceptibility to intestinal nematodes[J]. Mamm Genome, 2011, 22(1-2): 111–121. DOI: 10.1007/s00335-010-9308-0 |
| [23] | UPADHYAY M, DA SILVA V H, MEGENS H J, et al. Distribution and functionality of copy number variation across european cattle populations[J]. Front Genet, 2017, 8: 108. DOI: 10.3389/fgene.2017.00108 |
| [24] | MASSAIA A, XUE Y L. Human Y chromosome copy number variation in the next generation sequencing era and beyond[J]. Hum Genet, 2017, 136(5): 591–603. DOI: 10.1007/s00439-017-1788-5 |
| [25] | BICKHART D M, HOU Y L, SCHROEDER S G, et al. Copy number variation of individual cattle genomes using next-generation sequencing[J]. Genome Res, 2012, 22(4): 778–790. DOI: 10.1101/gr.133967.111 |
| [26] | ZHANG L Z, JIA S G, YANG M J, et al. Detection of copy number variations and their effects in Chinese bulls[J]. BMC Genomics, 2014, 15: 480. DOI: 10.1186/1471-2164-15-480 |
| [27] | SASAKI S, WATANABE T, NISHIMURA S, et al. Genome-wide identification of copy number variation using high-density single-nucleotide polymorphism array in Japanese Black cattle[J]. BMC Genet, 2016, 17: 26. |
| [28] | KARIMI K, ESMAILIZADEH A, WU D D, et al. Mapping of genome-wide copy number variations in the Iranian indigenous cattle using a dense SNP data set[J]. Anim Prod Sci, 2017. DOI: 10.1071/AN16384 |
| [29] | YLSTRA B, VAN DEN IJSSEL P, CARVALHO B, et al. BAC to the future! or oligonucleotides:a perspective for micro array comparative genomic hybridization (array CGH)[J]. Nucleic Acids Res, 2006, 34(2): 445–450. DOI: 10.1093/nar/gkj456 |
| [30] | CARTER N P. Methods and strategies for analyzing copy number variation using DNA microarrays[J]. Nat Genet, 2007, 39(7S): S16–S21. DOI: 10.1038/ng2028 |
| [31] | ALKAN C, COE B P, EICHLER E E. Genome structural variation discovery and genotyping[J]. Nat Rev Genet, 2011, 12(5): 363–376. DOI: 10.1038/nrg2958 |
| [32] | CHEN Y, ZHAO L, WANG Y, et al. SeqCNV:a novel method for identification of copy number variations in targeted next-generation sequencing data[J]. BMC Bioinformatics, 2017, 18: 147. DOI: 10.1186/s12859-017-1566-3 |
| [33] | ZHAN B J, FADISTA J, THOMSEN B, et al. Global assessment of genomic variation in cattle by genome resequencing and high-throughput genotyping[J]. BMC Genomics, 2011, 12: 557. DOI: 10.1186/1471-2164-12-557 |
| [34] | BOUSSAHA M, ESQUERRE D, BARBIERI J, et al. Genome-wide study of structural variants in bovine holstein, montbéliarde and normande dairy breeds[J]. PLoS One, 2015, 10(8): e0135931. DOI: 10.1371/journal.pone.0135931 |
| [35] | HOU Y L, LIU G E, BICKHART D M, et al. Genomic regions showing copy number variations associate with resistance or susceptibility to gastrointestinal nematodes in Angus cattle[J]. Funct Integr Genomic, 2012, 12(1): 81–92. DOI: 10.1007/s10142-011-0252-1 |
| [36] | SEBAT J, LAKSHMI B, TROGE J, et al. Large-scale copy number polymorphism in the human genome[J]. Science, 2004, 305(5683): 525–528. DOI: 10.1126/science.1098918 |
| [37] | GONZALEZ E, KULKARNI H, BOLIVAR H, et al. The influence of CCL3L1 gene-containing segmental duplications on HIV-1/AIDS susceptibility[J]. Science, 2005, 307(5714): 1434–1440. DOI: 10.1126/science.1101160 |
| [38] | AITMAN T J, DONG R, VYSE T J, et al. Copy number polymorphism in Fcgr3 predisposes to glomerulonephritis in rats and humans[J]. Nature, 2006, 439(7078): 851–855. DOI: 10.1038/nature04489 |
| [39] | FELLERMANN K, STANGE D E, SCHAEFFELER E, et al. A chromosome 8 gene-cluster polymorphism with low human beta-defensin 2 gene copy number predisposes to Crohn disease of the colon[J]. Am J Hum Genet, 2006, 79(3): 439–448. DOI: 10.1086/505915 |
| [40] | ZHANG H, DU Z Q, DONG J Q, et al. Detection of genome-wide copy number variations in two chicken lines divergently selected for abdominal fat content[J]. BMC Genomics, 2014, 15: 517. DOI: 10.1186/1471-2164-15-517 |
| [41] | ZHOU L S, LI J, YANG J, et al. Genome-wide mapping of copy number variations in commercial hybrid pigs using a high-density SNP genotyping array[J]. Russ J Genet, 2016, 52(1): 85–92. DOI: 10.1134/S1022795415120145 |
| [42] | WANG Y N, TANG Z L, SUN Y Q, et al. Analysis of genome-wide copy number variations in Chinese indigenous and western pig breeds by 60 K SNP genotyping arrays[J]. PLoS One, 2014, 9(9): e106780. DOI: 10.1371/journal.pone.0106780 |
| [43] | JUNG S H, YIM S H, OH H J, et al. De novo copy number variations in cloned dogs from the same nuclear donor[J]. BMC Genomics, 2013, 14: 863. DOI: 10.1186/1471-2164-14-863 |
| [44] | MA Q, LIU X X, PAN J F, et al. Genome-wide detection of copy number variation in Chinese indigenous sheep using an ovine high-density 600 K SNP array[J]. Sci Rep, 2017, 7: 912. DOI: 10.1038/s41598-017-00847-9 |
| [45] | FONTANESI L, MARTELLI P L, BERETTI F, et al. An initial comparative map of copy number variations in the goat (Capra hircus) genome[J]. BMC Genomics, 2010, 11: 639. DOI: 10.1186/1471-2164-11-639 |
| [46] | KADER A, LIU X X, DONG K Z, et al. Identification of copy number variations in three Chinese horse breeds using 70K single nucleotide polymorphism BeadChip array[J]. Anim Genet, 2016, 47(5): 560–569. DOI: 10.1111/age.12451 |
| [47] | ZHANG X, WANG K, WANG L Z, et al. Genome-wide patterns of copy number variation in the Chinese yak genome[J]. BMC Genomics, 2016, 17: 379. DOI: 10.1186/s12864-016-2702-6 |
| [48] | GIRIRAJAN S, CAMPBELL C D, EICHLER E E. Human copy number variation and complex genetic disease[J]. Annu Rev Genet, 2011, 45(1): 203–226. DOI: 10.1146/annurev-genet-102209-163544 |
| [49] | DURAN A M, ROMÁN PONCE S I, RUIZ LÓPEZ F J, et al. Genome-wide association study for milk somatic cell score in holstein cattle using copy number variation as markers[J]. J Anim Breed Genet, 2017, 134(1): 49–59. DOI: 10.1111/jbg.2017.134.issue-1 |
| [50] | HOU Y L, BICKHART D M, CHUNG H, et al. Analysis of copy number variations in Holstein cows identify potential mechanisms contributing to differences in residual feed intake[J]. Funct Integr Genomic, 2012, 12(4): 717–723. DOI: 10.1007/s10142-012-0295-y |
| [51] | STRILLACCI M G, COZZI M C, GORLA E, et al. Genomic and genetic variability of six chicken populations using single nucleotide polymorphism and copy number variants as markers[J]. Animal, 2017, 11(5): 737–745. DOI: 10.1017/S1751731116002135 |
| [52] | XU L Y, HOU Y L, BICKHART D M, et al. Population-genetic properties of differentiated copy number variations in cattle[J]. Sci Rep, 2016, 6: 23161. DOI: 10.1038/srep23161 |
| [53] | ZHANG L Z, JIA S G, PLATH M, et al. Impact of parental Bos taurus and Bos indicus origins on copy number variation in traditional chinese cattle breeds[J]. Genome Biol Evol, 2015, 7(8): 2352–2361. DOI: 10.1093/gbe/evv151 |
| [54] | ROSSE I C, ASSIS J G, OLIVEIRA F S, et al. Whole genome sequencing of Guzerá cattle reveals genetic variants in candidate genes for production, disease resistance, and heat tolerance[J]. Mamm Genome, 2017, 28(1-2): 66–80. DOI: 10.1007/s00335-016-9670-7 |
| [55] | BICKHART D M, XU L Y, HUTCHISON J L, et al. Diversity and population-genetic properties of copy number variations and multicopy genes in cattle[J]. DNA Res, 2016, 23(3): 253–262. DOI: 10.1093/dnares/dsw013 |
| [56] | ZHOU J, BERNARDO L, DOPMAN E B, et al. Copy-number variation:the balance between gene dosage and expression indrosophila melanogaster[J]. Genome Biol Evol, 2011, 3: 1014–1024. DOI: 10.1093/gbe/evr023 |
| [57] | HAMILTON C K, VERDUZCO-GÓMEZ A R, FAVETTA L A, et al. Testis-specific protein Y-encoded copy number is correlated to its expression and the field fertility of Canadian Holstein bulls[J]. Sex Dev, 2012, 6(5): 231–239. DOI: 10.1159/000338938 |
| [58] | STRANGER B E, FORREST M S, DUNNING M, et al. Relative impact of nucleotide and copy number variation on gene expression phenotypes[J]. Science, 2007, 315(5813): 848–853. DOI: 10.1126/science.1136678 |
| [59] | HARAKSINGH R R, SNYDER M P. Impacts of variation in the human genome on gene regulation[J]. J Mol Biol, 2013, 425(21): 3970–3977. DOI: 10.1016/j.jmb.2013.07.015 |
| [60] | DOPMAN E B, HARTL D L. A portrait of copy-number polymorphism in Drosophila melanogaster[J]. Proc Natl Acad Sci U S A, 2007, 104(50): 19920–19925. DOI: 10.1073/pnas.0709888104 |
| [61] | LIU W S, DE LEÓN F A P. Mapping of the bovine Y chromosome[J]. Electron J Biol, 2007, 3: 5–12. |
| [62] | LIU W S, CHANG T C. Y chromosome-linked genes implicated in spermatogenesis in cattle[M]//JUENGEL J L, MIYAMOTO A, PRICE C, et al. Reproduction in Domestic Ruminants ⅤⅢ. Leicestershire, England: Context Products Ltd, 2014: 239-255. |
| [63] | MANDAL A, SHARMA A. Variations in the length of the Y chromosome and the seminal attributes of Karan Fries bulls[J]. Vet Res Commun, 2003, 27(7): 567–575. DOI: 10.1023/A:1026083321377 |
| [64] | MUKHERJEE A, DASS G, JAGAN M G, et al. Absolute copy number differences of Y chromosomal genes between crossbred (Bos taurus×Bos indicus) and Indicine bulls[J]. J Anim Sci Biotechnol, 2013, 4: 15. DOI: 10.1186/2049-1891-4-15 |
| [65] | ZHANG G W, GUAN J Q, LUO Z G, et al. A tremendous expansion of TSPY copy number in crossbred bulls (Bos taurus ×Bos grunniens)[J]. J Anim Sci, 2016, 94(4): 1398–1407. DOI: 10.2527/jas.2015-9983 |
| [66] | YUE X P, DECHOW C, CHANG T C, et al. Copy number variations of the extensively amplified Y-linked genes, HSFY and ZNF280BY, in cattle and their association with male reproductive traits in Holstein bulls[J]. BMC Genomics, 2014, 15: 113. DOI: 10.1186/1471-2164-15-113 |
| [67] | YUE X P, CHANG T C, DEJARNETTE J M, et al. Copy number variation of PRAMEY across breeds and its association with male fertility in Holstein sires[J]. J Dairy Sci, 2013, 96(12): 8024–8034. DOI: 10.3168/jds.2013-7037 |
| [68] | HAMILTON C K, FAVETTA L A, DI MEO G, et al. Copy number variation of testis-specific protein, Y-encoded (TSPY) in 14 different breeds of cattle (Bos taurus)[J]. Sex Dev, 2009, 3(4): 205–213. DOI: 10.1159/000228721 |