猪流行性腹泻(porcine epidemic diarrhea, PED)是由猪流行性腹泻病毒(porcine epidemic diarrhea virus, PEDV)引起的猪的一种高度接触性肠道传染病[1],以排水样粪便、呕吐、脱水和食欲下降、精神沉郁为主要特征。对刚出生的仔猪危害最大。该病于1971年在英国首次暴发,随后瑞士、法国、中国、日本、朝鲜和美国等国家均发生该病,呈地方流行性[2-5]。PEDV是有囊膜的RNA病毒,属于冠状病毒科α冠状病毒属成员。PEDV基因组全长28 kb左右,包括7个开放阅读框,它们分别编码四个结构蛋白(S、E、M和N)及三个非结构蛋白(pp1a、pp1ab和ORF3)。PEDV S蛋白由1 383个氨基酸组成,是最大的结构蛋白。S蛋白是糖蛋白,位于病毒粒子表面[6]。有研究表明S蛋白能够激活宿主产生中和抗体。已经发现四个中和表位(COE:aa 499—638,SS2:aa 748—755,SS6:aa 764—771,2C10:aa 1 368—1 374)[7-9]。因此,S蛋白在PEDV遗传演化中具有重要作用。
2010年以前,PED在中国呈散发状态。但是,2010年以后,新的变异毒株在中国许多地区出现,导致刚出生小猪严重腹泻,给我国养猪业造成巨大的经济损失。PEDV变异毒株能够引起1~10日龄仔猪几乎100%的感染率和80%~100%的致死率[10-11]。河南是中国中部养猪最多的省份,但是近些年部分猪场发生PED,导致大规模的仔猪死亡。因此,本研究调查了最近河南地区不同PEDV分离株S基因的改变情况,以便进一步了解该地区PEDV的流行病学,为疾病的防控提供理论依据。
1 材料与方法 1.1 病料河南省各地区疑似暴发PEDV的自然发病猪,采集发病猪肠道组织及内容物,无菌处理,于-80 ℃冻存备用。
1.2 细胞和菌体所用宿主菌E. coli DH5α感受态细胞由信阳农林学院牧医工程学院传染病实验室保存并提供。
1.3 主要试剂病毒DNA/RNA提取试剂盒购自Magen公司;反转录试剂盒购于Promega公司;RT-PCR相关试剂盒购自TOYOBO生物科技有限公司和Vazyme公司;DNA Marker DL2000等购自TaKaRa公司;胶回收试剂盒购自Promega公司;EB替代染料,;pEASY-Blunt Simple Cloning Vector和Trans-5α感受态细胞均购自北京全式金生物技术有限公司。
1.4 引物设计与合成根据GenBank中发布的CV777和近些年发布的PEDV变异毒株序列保守区域设计4对特异性引物,将S基因分成4个片段(S1、S2、S3、S4)进行扩增,每两段之间有一部分重叠区域,引物由上海生工生物工程公司合成。引物信息见表 1。
|
|
表 1 PEDV S基因扩增引物序列 Table 1 Sequences of amplified PEDV S gene |
将处理的病料根据Magen公司产品试剂盒进行病毒核酸的提取,然后根据TaKaRa反转录试剂盒,以四个片段的下游引物进行病毒RNA的反转录扩增合成cDNA。运用TaKaRa高保真酶扩增S基因,扩增产物用1%琼脂糖凝胶电泳鉴定条带大小。
1.6 基因克隆和序列分析PCR扩增产物通过1%琼脂糖凝胶电泳鉴定后,使用Promega公司胶回收试剂盒对目的片段进行胶回收,然后连接到北京全式金生物有限公司的pEASY-Blunt Simple Cloning Vector上,经过转化挑菌鉴定后送往测序公司进行测序。将测序正确的序列运用Lasergene的SeqMan软件进行序列拼接,随后用于Lasergene的Meglian软件和Mega5.0对全长S基因进行序列比对分析和构建系统进化树。
将所获得的PEDV S基因核苷酸序列均上传至GenBank。
2 结果 2.1 S基因核苷酸序列38株PEDV临床样品信息和S基因扩增片段大小见表 2,S基因在GenBank中的编号为KY211038到KY211075。
|
|
表 2 临床样品信息和扩增片段大小 Table 2 Clinical sample information and amplification fragment size |
运用MEGA5.2软件对S基因的氨基酸序列进行进化分析。如图 1所示,扩增的PEDV S基因片段均属于Group2,DR13毒株和CV777毒株属于Group1。Group2又进一步的被分为五个亚组,ZMD10/11S、PDS1/2S、NY9S和XY5S这几个PEDV S基因片段与GD-1、CHGD-01亲缘性较近,属于Subgroup2;LY1S、NY1/5S、XY2S与变异毒株BJ-2011-1、AH2012、AJ1102亲缘关系近,属于Subgroup3;扩增的S基因片段ZMD3/4/5/7/8/9S、XY1/4/6S、NY2/3/4/6/7/10S和LH2S与USA Colorado 2013变异毒株亲缘关系较近,属于Subgroup4;其他扩增的S基因片段与PEDV CH ZMDZY 11有更近的亲缘关系,属于Subgroup5。
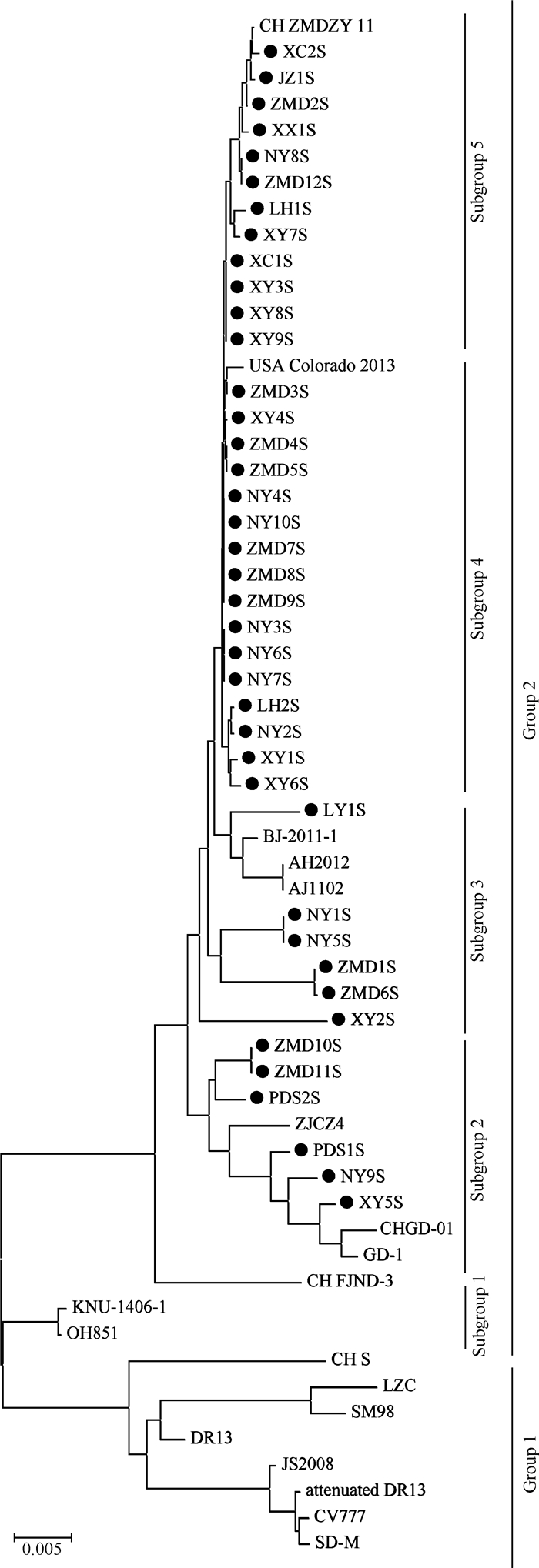
|
图 1 PEDV S基因的进化树分析 Figure 1 Phylogenetic tree analysis of PEDV S gene |
为了深入了解S基因的特性,运用MegAlign软件对所有扩增的S基因编码的S蛋白的氨基酸序列与参考毒株进行比对分析。结果显示:扩增的S基因片段之间核苷酸序列的相似性在96.0%~100%;编码的氨基酸序列的相似性在94.7%~100%;扩增的S基因片段与参考毒株之间核苷酸序列的相似性为84.6%~100%;编码的氨基酸序列的相似性为79.3%~100%。与经典毒株CV777、LZC相比,所有扩增的S基因编码的S蛋白在第55—58氨基酸位点均有5个氨基酸的插入,在第159—160氨基酸位点有2个氨基酸的缺失。与其他分离的PEDV不同的是,NY1/5S的S蛋白在第358—363氨基酸位点有6个氨基酸的插入,XY6S的S蛋白在第393—394氨基酸位点有2个氨基酸的插入。除了经典毒株CV777与LZC,所有扩增的S基因编码的S蛋白在第55—58氨基酸位点均有4个氨基酸的插入,在第159—160位氨基酸位点均有2个氨基酸的缺失。然而,扩增的XY4S、ZMD4/5/7/8/9S、NY4/8/10S S基因编码的S蛋白在第1 362—1 366氨基酸位点有5个氨基酸的缺失,XY3/8/9S、XC1S在第161位以及第1 362—1 366位氨基酸位点有6个氨基酸的缺失。XY4S、ZMD4/5/7/8/9S、NY4/8/10S、XY3/8/9S与XC1的S蛋白与其他毒株相比在C端有11个氨基酸的缺失。
PEDV的S蛋白是一个含有四个中和表位的一型糖蛋白。四个中和表位:COE(499—638 aa)、SS2(748—755 aa)、SS6(764—771 aa)与2C10(1 368—1 374 aa)。所有扩增的S基因片段的中和表位与代表毒株进行分析比对,结果显示:氨基酸的替换主要位于COE与2C10两个区域,中和表位SS2与SS6两个区域相对比较保守。在中和表位COE区域,LY1S有三个位点的氨基酸突变,分别如下:562位的赖氨酸突变为天冬氨酸,607位的丝氨酸突变为甘氨酸,608位的甘氨酸突变为颉氨酸。NY1S与NY5S有两个相同的突变,即:538位的苯丙氨酸突变为丝氨酸,608位的甘氨酸突变为颉氨酸。XY2S有四个突变,即:580位的赖氨酸突变为精氨酸,592位的丙氨酸突变为脯氨酸,607位的丝氨酸突变为天冬氨酸,608位的甘氨酸突变为丝氨酸。XC2、XX1S、LH1S与XY7S毒株有三个相同的突变,分别如下:520位的组氨酸突变为精氨酸,526位的异亮氨酸突变为苏氨酸,583位的赖氨酸突变为天冬酰胺。JZ1S有两个氨基酸突变,即:526位的异亮氨酸突变为苏氨酸,583位的赖氨酸突变为天冬酰胺。在中和表位2C10区域,很多扩增的S基因编码的S蛋白在1 371氨基酸位点或1 375氨基酸位点有一个突变。ZMD4/11S、LY1S、NY1/5S、XY2S与ZMD3S均在1 375氨基酸位点由丙氨酸突变为颉氨酸。XY3/4/8/9S、ZMD4/5S、NY4/8/10S、ZMD7/8/9S与XC1S毒株均在1 371氨基酸位点由谷氨酰胺突变为亮氨酸。然而,所有扩增的S基因编码的S蛋白在SS2与SS6两个中和表位没有一个氨基酸突变。
3 讨论 3.1 关于PEDV S基因扩增测序目前,PEDV在中国流行并且很多学者已经报道PEDV变异毒株正在不断出现,经典毒株制作的PEDV弱毒疫苗可能对于新出现的PEDV变异毒株不具有完全的保护能力[12-15]。本研究中,2014年至2015年中国中部河南省各地区猪场腹泻病料中,扩增到38株PEDV的S全基因片段并测序。将扩增的S基因与参考毒株进行遗传演化分析。结果表明:所有扩增的S基因与新分离的变异毒株CH/ZMDZY/11、AH2012、USA Colorado 2013以及2011年后中国和美国分离的毒株亲缘关系很近,均属于Group2。遗传演化分析结果与先前的报道相似[16-17],证明现今在河南省境内流行的PEDV毒株为变异毒株。
3.2 关于PEDV S基因进化树分析另外,Group2又进一步被分为五个亚组,分别为Subgroup1、Subgroup2、Subgroup3,Subgroup4和Subgroup5。扩增的ZMD10/11S、PDS1/2S、NY9S、XY5S S基因片段与从中国南部分离出的GD-1、CHGD-01毒株亲缘相近,属于Subgroup2。LY1S、NY1/5S和XY2S属于Subgroup3,与中国北部、东部、中部分离的变异毒株BJ-2011-1、AH2012、AJ1102亲缘关系相近。进一步分析得出,ZMD3/4/5/7/8/9S、XY1/4/6S、NY2/3/4/6/7/10S与LH2S毒株与USA Colorado 2013毒株亲缘关系相近,属于Subgroup4[18]。其他扩增的S基因片段与中国中部分离的CH ZMDZY 11毒株亲缘关系相近,属于Subgroup5[19]。这些结果表明,本次从河南省扩增的38株PEDV S基因片段经历了不同的变异演化。
3.3 关于PEDV S蛋白氨基酸序列比对分析对于中国PEDV野毒毒株S基因全长进行演化分析,将会有助于更好地了解中国河南省PEDV毒株的遗传演化关系。分析结果表明:所有扩增的PEDV S基因片段都有一个共同的重要特点,即编码的S蛋白有氨基酸的插入和缺失。扩增的38株PEDV S基因编码的S蛋白在第55—58位氨基酸位点均有4个氨基酸的插入,在第159—160位氨基酸位点均有2个氨基酸的缺失,与之前的研究报道相似[20]。
3.4 关于PEDV S蛋白免疫原性分析PEDV的S蛋白属于Ⅰ型糖蛋白,包括四个中和表位,分别为COE(499—638 aa)、SS2(748—755 aa)、SS6(764—771 aa)与2C10(1 368—1 374 aa)。序列比对分析结果表明,扩增的38株PEDV S基因编码的S蛋白在中和表位SS2与SS6是保守的,未发生突变。与经典毒株CV777相比,在中和表位COE区域,XY2S、XC2、XX1S、LH1S、XY7S、LY1S和JZ1S毒株均有氨基酸突变。在中和表位2C10区域,扩增的38株PEDV S基因中有20株编码的S蛋白有1个氨基酸突变。另外,XC2S、XX1S、LH1S与XY7S四株与美国分离的USA Colorado 2013毒株有相似的氨基酸突变[12]。有7株与BJ-2011-1、USA Colorado 2013、CH/ZMDZY/11相似,第1 375位的丙氨酸突变为颉氨酸[7, 12, 20]。PEDV的S蛋白是一个位于病毒表面的糖蛋白,并且诱导宿主产生中和抗体[6, 20]。这些扩增的S基因编码的S蛋白的中和表位氨基酸的突变是否会影响机体中和抗体的产生,有待进一步的研究。
4 结论扩增河南地区2014—2015年采集病料中的38株猪流行性腹泻病毒(PEDV)的S基因并测序。分析显示所有PEDV S基因扩增片段与近年来中国和美国分离的变异毒株亲缘关系较近;扩增的PEDV S基因编码蛋白中存在氨基酸的插入和缺失;部分编码蛋白序列在C端有11个氨基酸的缺失;许多编码蛋白序列的中和表位COE和2C10区域存在氨基酸突变。
| [1] |
殷震, 刘景华.
动物病毒学[M]. 2版. 北京: 科学出版社, 1997: 681-688.
YIN Z, LIU J H. Animal virology[M]. 2nd ed. Beijing: Science Press, 1997: 681-688. (in Chinese) |
| [2] | BRIDGEN A, DUARTE M, TOBLER K, et al. Sequence determination of the nucleocapsid protein gene of the porcine epidemic diarrhoea virus confirms that this virus is a coronavirus related to human coronavirus 229E and porcine transmissible gastroenteritis virus[J]. J Gen Virol, 1993, 74(Pt 9): 1795–1804. |
| [3] | CHANG S H, BAE J, KANG T J, et al. Identification of the epitope region capable of inducing neutralizing antibodies against the porcine epidemic diarrhea virus[J]. Mol Cells, 2002, 14(2): 295–299. |
| [4] | CHEN J F, WANG C B, SHI H Y, et al. Molecular epidemiology of porcine epidemic diarrhea virus in China[J]. Arch Virol, 2010, 155(9): 1471–1476. DOI: 10.1007/s00705-010-0720-2 |
| [5] | CHEN X, YANG J X, YU F S, et al. Molecular characterization and phylogenetic analysis of porcine epidemic diarrhea virus (PEDV) samples from field cases in Fujian, China[J]. Virus Genes, 2012, 45(3): 499–507. DOI: 10.1007/s11262-012-0794-x |
| [6] |
张强敏, 郭福生, 尹燕博, 等. 猪流行性腹泻病毒分子生物学特征[J]. 中国病毒学, 2002, 17(4): 381–384.
ZHANG Q M, GUO F S, YIN Y B, et al. Molecular biological traits of Porcine epidemic diarrhea virus[J]. Virologica Sinica, 2002, 17(4): 381–384. (in Chinese) |
| [7] | GAO Y Y, KOU Q W, GE X N, et al. Phylogenetic analysis of porcine epidemic diarrhea virus field strains prevailing recently in China[J]. Arch Virol, 2013, 158(3): 711–715. DOI: 10.1007/s00705-012-1541-2 |
| [8] | GE J W, LIU D Q, LI Y J. Construction of recombinant lactobacilli expressing the core neutralizing epitope (COE) of porcine epidemic diarrhea virus and a fusion protein consisting of COE and Escherichia coli heat-labile enterotoxin B, and comparison of the immune responses by orogastric immunization[J]. Can J Microbiol, 2012, 58(11): 1258–1267. DOI: 10.1139/w2012-098 |
| [9] | HUANG Y W, DICKERMAN A W, PIÑEYRO P, et al. Origin, evolution, and genotyping of emergent porcine epidemic diarrhea virus strains in the United States[J]. mBio, 2013, 4(5): e00737–13. |
| [10] |
刘孝珍, 陈建飞, 时洪艳, 等. 2011年猪流行性腹泻病毒的遗传变异分析[J]. 中国预防兽医学报, 2012, 34(3): 180–183.
LIU X Z, CHEN J F, SHI H Y, et al. Genetic variation analysis of porcine epidemic diarrhea virus isolated in 2011[J]. Chinese Journal of Preventive Veterinary Medicine, 2012, 34(3): 180–183. (in Chinese) |
| [11] | LEE D K, PARK C K, KIM S H, et al. Heterogeneity in spike protein genes of porcine epidemic diarrhea viruses isolated in Korea[J]. Virus Res, 2010, 149(2): 175–182. DOI: 10.1016/j.virusres.2010.01.015 |
| [12] | MARTHALER D, JIANG Y, OTTERSON T, et al. Complete genome sequence of porcine epidemic diarrhea virus strain USA/Colorado/2013 from the United States[J]. Genome Announc, 2013, 1(4): e00555–13. |
| [13] | PARK S J, MOON H J, YANG J S, et al. Sequence analysis of the partial spike glycoprotein gene of porcine epidemic diarrhea viruses isolated in Korea[J]. Virus Genes, 2007, 35(2): 321–332. DOI: 10.1007/s11262-007-0096-x |
| [14] | PENSAERT M B, DE BOUCK P. A new coronavirus-like particle associated with diarrhea in swine[J]. Arch Virol, 1978, 58(3): 243–247. DOI: 10.1007/BF01317606 |
| [15] | SONG D, PARK B. Porcine epidemic diarrhoea virus: a comprehensive review of molecular epidemiology, diagnosis, and vaccines[J]. Virus Genes, 2012, 44(2): 167–175. DOI: 10.1007/s11262-012-0713-1 |
| [16] | SPAAN W, CAVANAGH D, HORZINEK M C. Coronaviruses: structure and genome expression[J]. J Gen Virol, 1988, 69(Pt 12): 2939–2952. |
| [17] | SUN D B, FENG L, SHI H Y, et al. Identification of two novel B cell epitopes on porcine epidemic diarrhea virus spike protein[J]. Vet Microbiol, 2008, 131(1-2): 73–81. DOI: 10.1016/j.vetmic.2008.02.022 |
| [18] | SUN R Q, CAI R J, CHEN Y Q, et al. Outbreak of porcine epidemic diarrhea in suckling piglets, China[J]. Emerg Infect Dis, 2012, 18(1): 161–163. |
| [19] | TEMEEYASEN G, SRIJANGWAD A, TRIPIPAT T, et al. Genetic diversity of ORF3 and spike genes of porcine epidemic diarrhea virus in Thailand[J]. Infect Genet Evol, 2014, 21: 205–213. DOI: 10.1016/j.meegid.2013.11.001 |
| [20] | WANG X M, NIU B B, YAN H, et al. Complete genome sequence of a variant porcine epidemic diarrhea virus strain isolated in central China[J]. Genome Announc, 2013, 1(1): e00243–12. |



