2. 西南民族大学 青藏高原研究院, 成都 610041
2. Institute of Qinghai-Tibetan Plateau, Southwest Minzu University, Chengdu 610041, China
沙门菌属于革兰阴性菌,在自然界分布极广,且菌属型别繁多,抗原复杂[1],是能引起人和多种动物致病的病原菌。在世界范围内,由沙门菌导致的食源性疾病一直排在细菌性食物中毒的首位[2]。牦牛沙门菌病,也称为牦牛副伤寒(“小牛瘟”),沙门菌主要在牦牛肠道生长繁殖,可通过肠道上皮细胞进入体内,并通过血液循环,导致全身感染,引起腹泻、发热及肠道炎症反应[3]。本病在我国牦牛繁衍区广泛流行,幼龄牦牛和成年牦牛均能感染发病和造成死亡。其中对幼龄牦牛危害最为严重,致死率高,严重地危害着牦牛业的健康发展[4-6]。目前,尚无针对牦牛沙门菌病的有效疫苗,且不同血清型间无交叉免疫保护力,同时抗生素等药物治疗极易诱使沙门菌产生耐药性,使得牦牛沙门菌病始终未能得到有效防控。此外,对于沙门菌免疫机制的研究多集中于鸡、鼠等常规实验动物,目前对于牦牛沙门菌的研究多集中于病原体本身[7-8],对牦牛机体抵御沙门菌感染的相关免疫机制的研究尚未见任何报道。
同时,对沙门菌免疫机制的研究多为单一基因或通路。由于宿主抵御病原体感染的免疫反应是一个复杂的作用机制,涉及许多分子及其信号通路,而这些分子及其信号通路又受许多基因的网络调控,所以仅从单个或少数基因层面是无法深入挖掘机体抗感染免疫信息的,而在组学层面上加以描述才能更加有效地理解其中的作用机制[9]。近年来,随着高通量测序技术的不断发展,在功能基因组学层面上对感染后的组织或细胞进行差异表达基因,已逐渐被公认为是鉴定机体抗感染免疫相关基因及其调控网络的一个有效工具[10-11]。同时随着近期牦牛全基因组的释放[12],为牦牛的相关研究提供了可操作的蓝图。
鉴于此,本研究选取了高致病性的牦牛源御成门沙门菌作为模式病原体[13],利用转录组学高通量技术筛选牦牛外周免疫器官脾感染沙门菌后的差异表达基因,并通过关联分析鉴定出牦牛抗沙门菌感染相关免疫基因及其调控网络,为进一步了解牦牛抗感染相关分子免疫机制及牦牛重大疫病的分子抗病育种提供基础。
1 材料与方法 1.1 材料牦牛源御成门沙门菌swun3736菌株由西南民族大学生命科学与技术学院动物医学实验室于2012年从牦牛粪便中分离, 血清型经四川省动物疫病预防控制中心鉴定。健康1月龄犊牦牛由西南民族大学青藏基地提供。
1.2 菌株培养将御成门沙门菌菌株于37 ℃条件下,在SS平板划线分离培养,挑选单菌落在LB液体培养基中,37 ℃培养至对数生长期后,4 ℃保存备用。
1.3 动物感染与样本采集选取18只犊牛,用牛属ELISA(赛默飞公司)试剂盒对其进行常见疫病细菌(沙门菌、巴氏杆菌、大肠杆菌、布氏杆菌、结核杆菌)和病毒(腹泻-黏膜病病毒、轮状病毒)的相关抗体检测,排除携带上述病原体的可能性。根据预试验症状,选择12、24、48 h三个时间取样。将18只犊牛分为试验组和对照组(各9头),分别于颈静脉注射10 mL沙门菌菌液(约1×108CFU)和生理盐水,然后分别在感染后12、24、48 h三个时间点随机选取试验组和对照组各3只犊牛,采集其脾样本,放入液氮中保存。
1.4 RNA提取及转录组测序用TRIzol(Life Technologies公司,美国)试剂提取脾样本RNA,提取的RNA使用DNAse I(TaKaRa公司,大连)消化DNA后,用Oligotex mRNA小量提取试剂盒(Qiagen公司,德国)分离纯化mRNA,然后使用Nanodrop ND-1000分光光度计对RNA的质量和浓度进行测定。用打断试剂在恒温混匀仪中对纯化的mRNA进行片段化,以打断的mRNA为模板合成一链cDNA,然后配制二链合成反应体系合成二链cDNA,纯化和配对末端修复后,在cDNA的3′末端加上碱基“A”并连接接头,进行片段大小选择后用PCR扩增;构建好的cDNA文库经Agilent Bioanalyzer 2100及ABI StepOnePlus Real-Time PCR System质检合格后,在Illumina HiSeqTM 2500平台上对文库进行测序。
1.5 转录组数据分析对Hiseq 2500测序产生的原始读数进行QC测试后,通过去除接头序列、空序列及低质量测序序列(≤Q20质量值),将原始读数过滤为净测序序列(Clean reads)。使用HISAT2软件将净测序序列比对到牦牛基因组序列上,根据每条read在基因组上的位置统计染色体的测序深度。使用Bowtie2软件将净测序序列比对到参考基因序列上,对测序的整体质量进行评估。应用Blast2 GO程序将比对上的基因与GO(Gene ontology)数据库进行比对,然后应用WEGO程序对这些基因涉及的主要生物学功能进行GO分类注释。通过与KEGG(Kyoto encyclopedia of genes and genomes)数据库比对,对这些基因涉及的信号通路或代谢途径(Pathway)进行分析。最后进行差异基因网络互作分析,对18个样本的所有差异基因进行Soft power值的筛选,然后通过以TOM矩阵进行距离的划分,得到基因之间的距离,再依照距离进行模块的划分,根据WGCNA权重网络分析得到各模块内基因间网络关系,通过Cytoscape进行网络图绘制。
2 结果 2.1 RNA高通量测序基本数据分析本研究对试验组和对照组三个时间点共计18个样本进行测序并通过质控。核苷酸组成分析显示,转录组数据总GC含量为54.62%。碱基组成和质量分析表明,原始测序数据的碱基组成情况良好。高质量测序序列的平均比例为97.12%,此结果说明测序质量良好。将Clean reads比对到参考基因上,平均映射比率为80.17%,且测序序列均匀地覆盖在参考基因上,说明此次序的整体质量良好、测序数据随机性良好,可用于后续的分析。
2.2 差异表达基因的统计试验组与对照组三个时间点差异基因筛选统计显示,12 h筛选出77个差异表达基因,其中48个为上调,29个为下调。24 h筛选出134个差异表达基因,其中55个表现为上调,79个表现为下调。48 h筛选出202个差异表达基因,其中82个表现为上调,120个表现为下调。
2.3 差异表达基因GO功能及pathway显著性富集分析GO分析表明(表 1),试验组与对照组12 h差异表达基因涉及生物学过程、细胞组分及分子功能3个大类36个小类,在生物过程类别中,最为富集的是细胞过程,其次是代谢过程、生物调节及生物过程的调控。试验组与对照组24 h差异表达基因涉及生物学过程、细胞组分及分子功能3个大类44个小类,在生物过程类别中,最为富集的是细胞过程,其次是单一有机体过程及生物调节。试验组与对照组48 h差异表达基因涉及生物学过程、细胞组分及分子功能3个大类46个小类,在生物过程类别中,最为富集的是细胞过程,其次是单一有机体过程及生物调节。
|
|
表 1 各时间点生物学过程前十富集GO分类 Table 1 Classification of the first ten enriched GO before the biological process |
KEGG注释结果表明,试验组与对照组12 h差异基因涉及96条通路, 富集前20的通路中,与感染、免疫相关的通路有5条,富集前3的通路与能量代谢有关。试验组与对照组24 h差异基因涉及164条通路, 富集前20的通路中,与感染、免疫相关的通路有9条,其他通路多与疾病相关。试验组与对照组48 h差异基因涉及188条通路,富集前20的通路中,与感染、免疫相关的通路有3条,有6条通路与癌症相关,其他通路多与疾病相关(表 2)。
|
|
表 2 试验中主要富集通路及其相关差异基因 Table 2 The enrichment pathway and its associated differential genes in this experiment |
WGCNA分析显示,处于网络枢纽的基因共66个,处于网络中心的基因是SCOC和NOCT(图 1)。
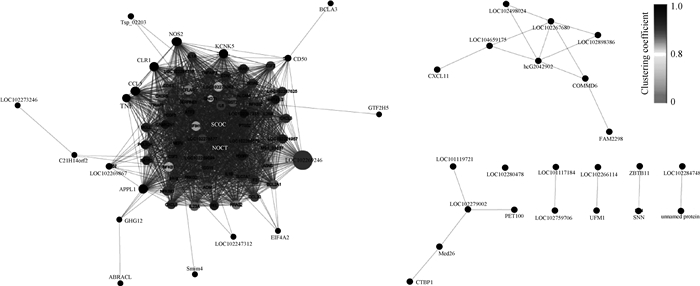
|
图 1 基因网络图 Figure 1 Gene network diagram |
脾免疫是动物抵御外界病原体感染重要机制,在机体抗感染免疫中具有重要地位。本研究以牦牛源御成门沙门菌为模式病原体,采用转录组高通量测序技术RNA-Seq对感染牦牛脾组织进行转录组测序,筛选出差异表达基因,在组学层面上探讨了牦牛脾细胞与御成门沙门菌的互作机制。
3.1 差异表达基因的GO分析试验组和对照组进行差异表达基因的GO分析显示,注射沙门菌12 h后,生物学过程的富集类别多与细胞对于病原的反应相关,分子功能中最为富集的是绑定类别及催化类别,推测绑定类别及催化作用在该时期的机体抗感染机制中有着重要的作用。
24 h后,宿主发生炎症反应,生物过程类别与12 h时几乎一致,但增加了细胞杀伤。分子功能类别增加了4个与信号传递相关的类别,推测该时期细胞的凋亡及信号传递十分重要。
48 h后,试验组临床表现和病变较对照组更为剧烈,功能类别与24 h的分析没有太大区别,但差异基因数量显著增加,分子功能类别增加了趋化因子活性和核酸结合转录因子活性,缺少了结构分子活性,推测该时期分子的趋化作用及核酸转录对机体抗感染机制非常重要。
3.2 差异表达基因的KEGG分析差异表达基因KEGG分析显示,注射沙门菌12 h后,与感染、免疫相关的富集通路有TGF-β信号通路、PI3K-Akt信号通路、NOD-like受体信号通路、吞噬体、细胞因子-细胞因子受体相互作用。其中,TGF-β信号通路、NOD-like受体信号通路所涉及的差异基因都表现为下调,包括Rbx1、RANTES、BMP。Rbx1是E3泛素连接酶的重要组成部分,通过促进细胞基质的及时降解来激活E3泛素连接酶并参与泛素化过程,同时泛素连接酶E3还涉及多种疾病的发病机制[14]。RANTES是趋化性细胞因子CC亚族成员,能趋化或刺激多种白细胞。这些基因的下调推测是受到了沙门菌的抑制,具体机制还不明确,有待进一步研究。PI3K-Akt信号通路和吞噬体涉及的差异基因表现为上调,这些差异基因包括Gβγ和F-actin。Gβγ与Gα亚基组成的异三聚体G蛋白能传递G蛋白偶联受体(GPCR)的信号,从而调节激素、神经递质、趋化因子等的生理反应[15-16]。F-actin是细胞骨架的成分之一,对CD4+T细胞形态的维持、物质的运输、细胞的运动,以及病毒侵染等过程具有重要作用[17]。这些基因的上调,说明宿主的免疫细胞开始激活分化。
沙门菌感染的一个重要特征是肠道炎症[3],此时通路炎性肠病也有富集表现,其涉及的差异基因为TCR、NOD2,TCR和NOD2都是重要的天然免疫受体。TCR/CD3复合物能诱导T细胞激活、刺激诱导NF-κB信号通路,此时大部分TCR上调,是感染初期宿主发现沙门菌入侵后的快速免疫应答。有少量TCR表现为下调,猜测可能是沙门菌为躲避免疫系统监视而产生的抑制作用。
此外,本阶段最为富集的通路为氧化磷酸化通路、非酒精性脂肪肝通路和心肌收缩通路,这三条通路涉及的差异基因较为相似,主要是细胞色素c氧化酶,其中COX3和COX7c表现为下调,其余差异基因都表现为上调。细胞色素c氧化酶上调能促进线粒体的氧化磷酸化作用,而氧化磷酸化系统是细胞能量产生的最终生化途径,能为部分免疫细胞的分化提供能量。大量研究表明,病原体感染宿主细胞后对宿主能量代谢的影响主要是对氧化磷酸化作用的抑制。COX3和COX7c的下调,推测是受到了沙门菌的抑制。
24 h后,与感染、免疫相关的富集通路有细胞因子受体相互作用、TNF信号通路、趋化因子信号通路、Toll-like受体信号通路、沙门菌感染、NOD-like受体信号通路、NF-κB信号通路、Jak-STAT信号通路、细胞凋亡。分析它们所涉及的差异基因,发现此时参与免疫的主要是趋化因子。趋化因子能让细胞迁移至炎性部位,且能与其受体结合参与多种病理过程,对免疫细胞分化有重要作用[18]。其中,涉及到大多数通路的是IL-8、IL-1β、TNF-α、IL-6等炎性因子,这些炎性因子都表现为上调。IL-1能激活抗原递呈细胞、促进T、B细胞生长及免疫球蛋白的合成[19]。IL-6、IL-8能调节中性粒细胞趋化因子的生成,促使中性粒细胞迁移至感染局部,参与炎症反应[20-21]。IL-1β和TNF-α可以诱导NF-κB通路,促进炎性因子的分泌,IL-1和TNF-α还能激活凋亡通路,诱导细胞凋亡[22]。这些炎性因子的上调,说明宿主正处于炎症阶段,以此抵抗沙门菌。
有研究表明,Toll样受体信号通路中的TLR4能识别革兰阴性菌并影响IL-1、IL-6、IL-8、TNF-α等炎性因子的释放[23-24],但此时试验组与对照组相比,TLR4并无明显差异,而该通路的下游基因IL-1、IL-6、IL-8、TNF-α等都显示为上调,推测TLR4可能在此阶段之前发生过变化,但本次研究并未捕捉到。在24 h阶段,CCL2也涉及多个富集通路,CCL2是在IL-8、TNF-α等炎症因子的刺激下分泌,并积极诱导免疫细胞参与炎症反应的趋化因子[25]。本阶段IL-8、TNF-α都表现为上调,但CCL2却表现为下调,推测沙门菌对其有抑制作用,具体机制有待进一步研究。
48 h后,与感染、免疫相关的富集通路有PI3K-Akt信号通路、吞噬体等。其中PI3K-Akt信号通路和吞噬体最为富集且涉及的差异基因众多。这两条通路涉及的差异基因都包含了TLR2,TLR2是TLRs家族的成员之一,是表达于天然免疫细胞、内皮细胞和上皮细胞等表面最重要模式识别受体,能刺激炎症因子的合成,参与机体免疫[26],此时TLR2表现为下调,推测沙门菌感染对其有抑制作用,具体机制有待进一步研究。此外,PI3K-Akt信号通路涉及的差异基因还包括IL6、G-CSF、IL2α、NF-κB、PDGF-B、PDGFRA、CSF-1R等,除CSF-1R表现为下调外,其它基因都表现为上调,其中,NF-κB基因对机体免疫十分重要。NF-κB基因可在TLRs识别到细菌入侵后被激活[27],其主要作用是对免疫和炎症作用的调控,PI3K-Akt信号通路中的促炎症因子IL6、G-CSF、IL2α等都在NF-κB的调控范围之内。IL6、G-CSF、IL2α等促炎症因子还可以促进炎症的进一步发生[28-29]。此外,IL-1β、TNF-α还可以通过激活或抑制NF-κB的活性来维持整个炎症作用有效、有限的发生,以达到适度炎症促进免疫的目的,避免过度炎症引发自身的免疫疾病或炎症相关疾病[30-31]。吞噬体所涉及的差异基因还包括大量的整合素,这是一类广泛分布于细胞表面的黏附分子受体,其主要功能为细胞的信号转导与活化、细胞的生长和分化、炎症、创伤愈合等[32]。然而,此时这些整合素大部分显示为下调,其具体机制有待进一步研究。
此外,48 h阶段的富集通路中出现了许多与癌变相关的通路,如病毒致癌、前列腺癌、非小细胞肺癌、黑色素、膀胱癌、急性骨髓性白血病等,这些癌症相关通路大多都包含NF-κB基因,NF-κB和其信号通路中的大部分激活因子被认为在肿瘤的发生发展中有重要作用[33]。
此时氧化磷酸化通路的差异基因QCR10、COX1、COX7B、COX7C全部显示为下调,推测此时宿主的能量代谢系统可能受到了抑制。
3.3 WGCNA共表达网络分析WGCNA共表达网络分析显示,IL-1A、IL-1B、IL-2RA、IL-6、IL-23A、TNF、CXCR5、CXCL3、CXCL8、CCL5、CCL20、CSF等都显示为富集,这些基因可以在前文KEGG分析的富集通路中找到,验证了KEGG分析的结果。SCOC和NOCT处于网络图的中心,说明这两个基因与其他富集基因的相关性最高。SCOC是一种高尔基体蛋白,通过与ARL1相互作用参与高尔基体运输,SCOC能促进自噬体的形成,自噬可以维持细胞稳态,恢复细胞代谢功能[34]。NOCT是一种脱腺苷酶,通过参与代谢脂肪基因转录后的昼夜节律调节牵涉脂质代谢的许多方面。NOCT在脂质代谢、脂肪形成、葡萄糖稳态、炎症和骨生成中发挥重要作用[35]。SCOC和NOCT的富集说明细胞代谢功能在整个免疫过程中起着重要作用。
4 讨论以牦牛源御成门沙门菌为模式病原体,用RNA-Seq测序技术对感染牦牛的脾转录组进行分析,筛选出牦牛感染御成门沙门菌后脾细胞的差异表达基因,在组学层面上探讨了牦牛脾细胞与沙门菌的分子互作机制,为进一步研究高原动物的免疫机制提供了依据。
| [1] |
黄素珍, 杜元钊, 张艳红. 检测肠炎沙门氏菌ELISA方法的建立与应用研究[J]. 中国预防兽医学报, 2006, 28(2): 196–200.
HUANG S Z, DU Y Z, ZHANG Y H. Establishment and application of ELISA method for detecting Salmonella SPP[J]. Chinese Journal of Preventive Veterinary Medicine, 2006, 28(2): 196–200. (in Chinese) |
| [2] |
陈建林, 刘明辉. 细菌性食物中毒流行趋势及预防对策[J]. 中国卫生检验杂志, 2002, 12(4): 481.
CHEN J L, LIU M H. The epidemic trend and preventive measures of bacterial food poisoning[J]. Chinese Journal of Health Laboratory Technology, 2002, 12(4): 481. (in Chinese) |
| [3] | RAGHUNATHAN A, REED J, SHIN S, et al. Constraint-based analysis of metabolic capacity of Salmonella typhimurium during host-pathogen interaction[J/OL]. Bmc Syst Biol, 2009(3): 38. [2018-02-06]. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2678070/pdf/1752-0509-3-38.pdf. |
| [4] |
樊万庚, 张长德, 齐厉生, 等. 牦牛副伤寒调查研究报告[J]. 中国兽医杂志, 1981, 7(5): 26–27.
FAN W G, ZHANG C D, QI L S, et al. Investigation report on calf paratyphoid fever[J]. Chinese Journal of Veterinary Medicine, 1981, 7(5): 26–27. (in Chinese) |
| [5] |
黄南州畜牧兽医科学研究所. 牛副伤寒氧化铝双价菌苗试验报告[J]. 青海畜牧兽医杂志, 1982(4): 30-35.
Huangnan State Animal Husbandry Department. Test report of the aluminium oxide bivalent bacterial seedlings on bovine paratyphoid[J]. Chinese Qinghai Journal of Animal and Veterinary Sciences, 1982(4): 30-35. (in Chinese) |
| [6] |
左永让. 牦牛副伤寒的诊断报告[J]. 青海畜牧兽医杂志, 1985(5): 24–25.
ZUO Y R. Diagnosis report of yak paratyphoid fever[J]. Chinese Qinghai Journal of Animal and Veterinary Sciences, 1985(5): 24–25. (in Chinese) |
| [7] | YUE H, ZHU X X, ZHANG B, et al. Emergence of the virulence plasmid in Salmonella onarimon and Salmonella blegdam from yak[J]. Vet Microbiol, 2013(1): 162. |
| [8] |
储倩, 朱晓霞, 岳华, 等. 川西北牦牛沙门氏菌的健康带菌调查及药敏实验[J]. 四川畜牧兽医, 2011(1): 24–25, 28.
CHU Q, ZHU X X, YUE H, et al. Epidemiological investigation and drug sensitive test of Salmonella in yak from northwest Sichuan[J]. Sichuan Animal & Veterinary Sciences, 2011(1): 24–25, 28. (in Chinese) |
| [9] | MACHUGH D E, GORMLEY E, PARK S D, et al. Gene expression profiling of the host response to Mycobacterium bovis infection in cattle[J]. Transbound Emerg Dis, 2009, 56(6-7): 204–214. DOI: 10.1111/jva.2009.56.issue-6-7 |
| [10] | RISMANI-YAZDI H, HAZNEDAROGLU B Z, BIBBY K, et al. Transcriptome sequencing and annotation of the microalgae Dunaliella tertiolecta: pathway description and gene discovery for production of next-generation biofuels[J/OL]. Bmc Genomics, 2011, 12: 148. [2018-02-06]. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3061936/pdf/1471-2164-12-148.pdf. |
| [11] | RADFORD A D, CHAPMAN D, DIXON L, et al. Application of next-generation sequencing technologies in virology[J]. J Gen Virol, 2012, 93. |
| [12] | QIU Q, ZHANG G, MA T, et al. The yak genome and adaptation to life at high altitude[J]. Nat Genet, 2012, 44(8): 946–949. DOI: 10.1038/ng.2343 |
| [13] |
雷占东, 张斌, 岳华, 等. 一株牦牛源御成门沙门菌对小鼠的致病性[J]. 畜牧兽医学报, 2015, 46(2): 339–343.
LEI Z D, ZHANG B, YUE H, et al. The study on pathogenicity of a yak Salmonella onarimon isolates in mice[J]. Acta Veterinaria et Zootechnica Sinica, 2015, 46(2): 339–343. (in Chinese) |
| [14] | JIA L, BICKEL J S, WU J, et al. RBX1(RING box protein 1) E3 ubiquitin ligase is required for genomic integrity by modulating DNA replication licensing proteins[J]. J Biol Chem, 2011, 286(5): 3379–3386. DOI: 10.1074/jbc.M110.188425 |
| [15] | KHAN S M, SLENO R, GORA S, et al. The expanding roles of Gβγ subunits in G protein-coupled receptor signaling and drug action[J]. Pharmacol Rev, 2013, 65(2): 546–577. |
| [16] | YAN J, MIHAYLOV V, XU X, et al. A Gβγ effector, ElmoE, transduces GPCR signaling to the actin network during chemotaxis[J]. Dev Cell, 2012, 22(1): 92–103. DOI: 10.1016/j.devcel.2011.11.007 |
| [17] | BURKHARDT J K, CARRIZOSA E, SHAFFER M H. The actin cytoskeleton in T cell activation[J]. Annu Rev Immunol, 2008, 26: 233–259. DOI: 10.1146/annurev.immunol.26.021607.090347 |
| [18] |
马小彤. 趋化因子与肿瘤和炎症[J]. 白血病·淋巴瘤, 2005, 14(5): 287–290.
MA X T. Chemokines with tumors and inflammation[J]. Journal of Leukemia & Lymphoma, 2005, 14(5): 287–290. (in Chinese) |
| [19] | HURME M, SANTTILA S. IL-1 receptor antagonist (IL-1Ra) plasma levels are co-ordinately regulated by both IL-1Ra and IL-1beta genes[J]. Eur J Immunol, 2015, 28(8): 2598–2602. |
| [20] | HIRANO T. Interleukin 6 and its receptor:ten years later[J]. Int Rev Immunol, 1998, 16(3-4): 249–284. DOI: 10.3109/08830189809042997 |
| [21] | AHMED N S, ALKHAYAT A M, ABDULLAH F T. Interleukins IL-6, IL8, IL10 and tumor necrosis factor TNF expression in human infected with Giardiaduodenalis[J]. Am J Med Med Sci, 2015, 5(1): 15–19. |
| [22] | WANG L, DU F, WANG X. TNF-alpha induces two distinct caspase-8 activation pathways[J]. Cell, 2008, 133(4): 693–703. DOI: 10.1016/j.cell.2008.03.036 |
| [23] | POLTORAK A, HE X, SMIRNOVA I, et al. Defective LPS signaling in C3H/HeJ and C57BL/l0ScCr mice:mutations in Tlr4 gene[J]. Science, 1998, 282(5396): 2085–2088. DOI: 10.1126/science.282.5396.2085 |
| [24] | HOSHINO K, TAKEUCHI O, KAWAI T, et al. Cutting edge:Toll-like receptor 4(TLR4)-deficient mice are hyporesponsive to lipopolysaccharide:evidence for TLR4 as the Lps gene product[J]. J Immunol, 1999, 162(7): 3749–3752. |
| [25] | QIAN B Z, LI J, ZHANG H, et al. CCL2 recruits inflammatory monocytes to facilitate breast-tumour metastasis[J]. Nature, 2011, 475(7355): 222–225. DOI: 10.1038/nature10138 |
| [26] | AKIRA S, TAKEDA K, KAISHO T. Toll-like receptors:critical proteins linking innate and acquired immunity[J]. Nat Immunol, 2001, 2(8): 675–680. DOI: 10.1038/90609 |
| [27] | KAWAI T, AKIRA S. The role of pattern-recognition receptors in innate immunity:update on Toll-like receptors[J]. Nat Immunol, 2010, 11(5): 373–384. DOI: 10.1038/ni.1863 |
| [28] | LUSTER A D. Chemokines-chemotactic cytokines that mediate inflammation[J]. N Engl J Med, 1998, 338(7): 436–445. DOI: 10.1056/NEJM199802123380706 |
| [29] | FELDMANN M, BRENNAN F M, ELLIOTT M J, et al. TNF alpha is an effective therapeutic target for rheumatoid arthritis[J]. Ann N Y Acad Sci, 1995, 766: 272–278. DOI: 10.1111/nyas.1995.766.issue-1 |
| [30] | LI Q T, VERMA I M. NF-κB regulation in the immune system[J]. Nat Rev Immunol, 2002(2): 725–734. |
| [31] | BLONSKA M, SHAMBHARKAR P B, KOBAYASHI M, et al. TAK1 is recruited to the tumor necrosis factor-α (TNF-α) receptor 1 complex in a receptor-interacting protein (RIP)-dependent manner and cooperates with MEKK3 leading to NF-κB activation[J]. J Biol Chem, 2005, 280(52): 43056–43063. DOI: 10.1074/jbc.M507807200 |
| [32] | MOSER M, LEGATE K R, ZENT R, et al. The tail of integrins, talin, and kindlins[J]. Science, 2009, 324(5929): 895–899. DOI: 10.1126/science.1163865 |
| [33] | PCRKINS N D. The diverse and complex roles of NF-κB subunits in cancer[J]. Nat Rev Cancer, 2012, 12(2): 121–132. DOI: 10.1038/nrc3204 |
| [34] | JOACHIM J, WIRTH M, MCKNIGHT N C, et al. Coiling up with SCOC and WAC:two new regulators of starvation-induced autophagy[J]. Autophagy, 2012, 8(9): 1397–1400. DOI: 10.4161/auto.21043 |
| [35] | STUBBLEFIELD J J, TERRIEN J, GREEN C B, et al. Nocturnin:at the crossroads of clocks and metabolism[J]. Trends Endocrinol Metab, 2012, 23(7): 326–333. DOI: 10.1016/j.tem.2012.03.007 |



