嵌杯病毒科(Caliciviridae)病毒粒子一般呈球形或近球形,核衣壳呈二十面体对称,直径27~40 nm。电镜观察检查时可见病毒粒子内部的高密度区为许多明亮的相互交叉的线条,而低密度区是7个光线比较暗的凹陷,中央有1个,周围存在6个,整体观察构成六芒星状的图像,其形态如同一个具有似花边杯状或花萼状,故此又称杯状病毒。嵌杯病毒为单股、正链RNA病毒,其基因组大小约为7.5 kb。嵌杯病毒与微RNA病毒非常接近,以前归类于微RNA病毒科。目前,嵌杯病毒科分为5个属:水疱疹病毒属(Vesivirus)、兔病毒属(Lagovirus)、纽布病毒属(Nebovirus)和2个人类的嵌杯病毒属,即诺瓦病毒属(Norovirus)和札幌病毒属(Sappovirus)[1]。
水貂嵌杯病毒(mink calicivirus, MCV)是在美国的一个有出血性肺炎史却一直未确定病原的水貂养殖场首次检测到[2]。此种普通MCV通常不表现消化道的临床症状,可能表现为肺炎等呼吸道症状。2000年,M.Guo等报道了一种能引起水貂肠炎的嵌杯病毒,被命名为水貂肠道嵌杯病毒(mink enteric calicivirus, MEC)[3]。MEC与MCV的病毒颗粒形态没有差别,但是这两种病毒的RNA多聚酶基因分析却存在较大差异。MEC的RNA多聚酶区域与人札幌病毒和猪嵌杯病毒的氨基酸相似性在64%~71%,与水疱疹病毒相似性在40%~51%,与诺瓦病毒氨基酸相似性为29%~33%。而MCV的RNA聚合酶的氨基酸序列与水疱疹病毒的相似性最高,达到58%~81%,与诺瓦病毒的相似性在43%~51%;与兔病毒属病毒的相似性在35%~37%;与诺瓦样病毒相似性在27%~35%。结果表明MCV与水疱疹病毒亲缘关系最近,MEC所属的科属同札幌病毒的亲缘关系最近。
国内外对感染水貂的嵌杯病毒研究较少,我国仅徐贵财2007年在大连地区的某发病水貂养殖场检测到该病原[4],对病原特性和基因组特性进行研究,并报道了世界上唯一的水貂嵌杯病毒全基因组序列[5]。本文对在青岛地区检测的一株水貂感染的嵌杯病毒的基因组进行高通量测序和分析,以期深入了解我国水貂感染的嵌杯病毒的分子生物学特性。
1 材料与方法 1.1 材料和仪器Ion PGM Template OT2 400 Kit、Ion PGM Sequencing 400 Kit V2、E-Gel SizeSelect 2% Agarose、Ion 316 Chip Kit V2、Dynabeads MyOne Streptavidin C1 Beads、Qubit核酸浓度测定仪及配套试剂均购自Life technologies公司;超净工作台(美国Forma Scientific);移液器(Eppendorf);孵化器(德州诚信孵化设备有限公司);高速台式离心机(德国Heraeus Biofuge primoR);PCR扩增仪(Perkin Elmeter Gen Amp PCR System 9600)。
1.2 核酸提取自青岛市某养貂场2016年春季采集的粪便样品中检测到水貂嵌杯病毒(Mink/China/2/2016)[6],将该份样品充分混匀后,经高速离心和过滤去除大分子物质和细菌,使用QIAamp Viral RNA Mini Kit(Qiagen,德国)提取病毒核酸,-80 ℃保存。
1.3 高通量测序(NGS)文库构建提取的病毒核酸采用中国动物卫生与流行病学中心报道的方法[6]构建NGS文库。具体方法:8 μL病毒RNA加入1 μL 100 μmol·L-1引物A15N6和2 μL无核酸酶的水进行混合,72 ℃ 5 min,然后RNA引物混合物放在冰上孵育至少3 min;混合物加入4 μL 5×first-strand buffer, 1 μL dNTP (100 μmol·L-1), 2 μL DTT (0.1 mol·L-1), 1 μL RNaseOUTTM Recombinant Ribonuclease Inhibitor (40 U·μL-1)和1 μL SuperScript® Ⅲ Reverse Transcriptase (200 U·μL-1) (Invitrogen, USA), 25 ℃反应15 min,42 ℃反应30 min,70 ℃ 15 min终止反应;反应产物中再加入1 μL RNase H (TaKaRa, Japan),37 ℃反应20 min;采用DynaMagTM-2 Magnet和Agencourt® AMPure® XP Reagent (Beckman Coulter, USA)纯化合成的第一链cDNA;纯化的第一链cDNA中加入引物B15N6,70 ℃ 5 min;再加入1 μL Klenow fragment (5 U) (NEB, USA), 5 μL 10 × NEBuffer 2, 2 μL dNTP (100 μmol·L-1)和1 μL DTT (0.1 mol·L-1),37 ℃反应30 min;最后采用1 × Phusion High-Fidelity Buffer, 10 μmol·L-1引物A30和B30(表 1), 0.5 U Phusion High-Fidelity DNA Polymerase (NEB, USA),进行PCR扩增。PCR产物采用E-Gel® SizeSelectTM Agarose Gel进行电泳,筛选和回收约450 bp的扩增产物作为NGS文库。
|
|
表 1 引物与引物序列 Table 1 Primer and its sequence |
将NGS文库稀释为26 pmol·L-1,应用Ion PGM Template OT2 400 Kit对DNA文库进行测序前的样品处理。处理后的样品加至Ion 316芯片,置于PGM测序仪进行测序。
1.5 测序数据分析NGS测序数据经质控后,采用CLC genomics workbench 8.5.1 (Qiagen, Germany)将测序得到的reads参考GenBank中已发表的水貂嵌杯病毒MCV-DL/2007/CN全基因序列[5](GenBank登录号:NC_019712)进行mapping拼接,并分析拼接完成的基因组中的ORF,注释后的序列提交GenBank。
1.6 基因组特性分析采用MEGA 6.0[7]将Mink/China/2/2016的全基因与衣壳蛋白氨基酸序列分别与GenBank中嵌杯病毒科的代表毒株(表 2)的全基因与衣壳蛋白氨基酸序列进行比对和进化分析,并对这些毒株的衣壳蛋白氨基酸序列之间的p-distance进行分析。同时,将Mink/China/2/2016的RdRP氨基酸序列通过BLAST比对,分析该病毒是否属于MEC。
|
|
表 2 基因比较分析所使用的嵌杯病毒毒株 Table 2 The viruses used in the genome comparison |
芯片上样率为73%,测序文库得到质量较高的序列数据测序数据。CLC genomics workbench 8.5.1拼接得到水貂嵌杯病毒中国株的全基因大小为8 427 bp,GenBank登录号为MF677852。Mink/China/2/2016基因组具有与其他嵌杯病毒相似的结构和基因顺序,其5′端有帽子结构,与结合蛋白VPg共价结合,其3′端为聚A尾,基因组(图 1)中主要包含3个ORF。ORF1(14—5 851)编码1 946个氨基酸的多聚蛋白,包括AAA-like蛋白、RNA解旋酶(RNA helicase)、Walker A模体、Walker B模体、ATP结合位点、多肽酶(peptidase)、蛋白酶(protease)和RNA依赖的RNA多聚酶(RdRP);ORF2(5 857—7 899)编码包含衣壳蛋白(coat protein)的681个氨基酸;ORF3编码59个氨基酸。
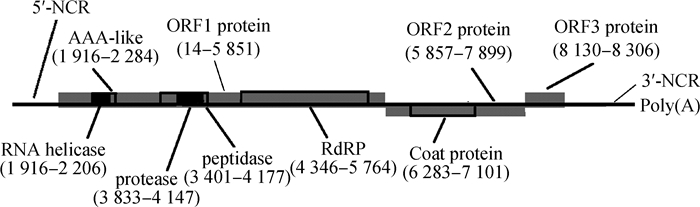
|
图 1 Mink/China/2/2016基因组结构 Figure 1 Genome structure of Mink/China/2/2016 |
Mink/China/2/2016与19株代表性毒株(表 2)的全基因组进化分析结果见图 2,衣壳蛋白氨基酸序列的进化分析见图 3,各毒株衣壳蛋白氨基酸序列之间的p-distance进行分析结果见图 4。全基因序列进化分析与衣壳蛋白氨基酸序列进化分析结果一致,说明仅分析衣壳蛋白氨基酸序列就能够反映嵌杯病毒的遗传进化关系。Mink/China/2/2016与已报道全基因的1株水貂嵌杯病毒(MCV DL/2007/CN)亲缘关系最近,两株水貂嵌杯病毒与VESV、WCV、FCV、CaCV归为同一分支。此分支各毒株衣壳蛋白氨基酸序列间的p-distance均小于0.7,该属衣壳蛋白氨基酸序列与其他属间的p-distance大于0.7,按照ICTV第九次病毒分类报告中嵌杯病毒科中各属的分类标准,应将水貂嵌杯病毒与VESV、WCV、FCV、CaCV归为水疱疹病毒属。BLAST结果显示,Mink/China/2/2016为MCV,其RdRP氨基酸序列与MCV较近,与MEC较远。
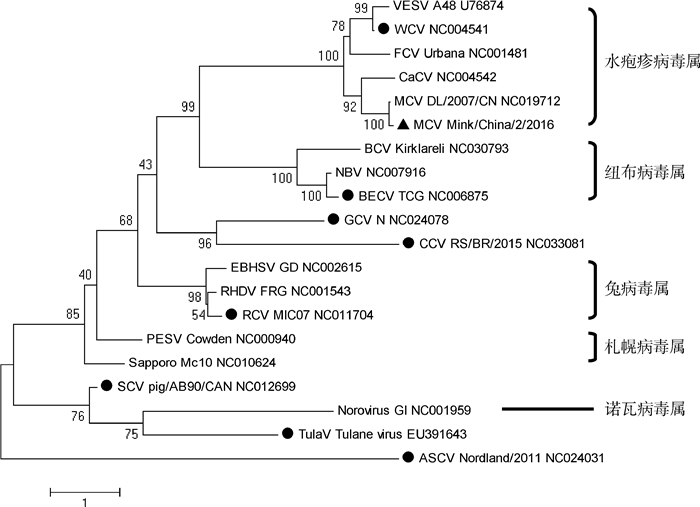
|
▲.水貂嵌杯病毒株Mink/China/2/2016;●. ICTV未确定属的嵌杯病毒 ▲. Mink calicivirus isolate Mink/China/2/2016;●. Caliciviruses which had not classified by ICTV 图 2 嵌杯病毒全基因进化分析 Figure 2 Genome evolution analysis of caliciviruses |
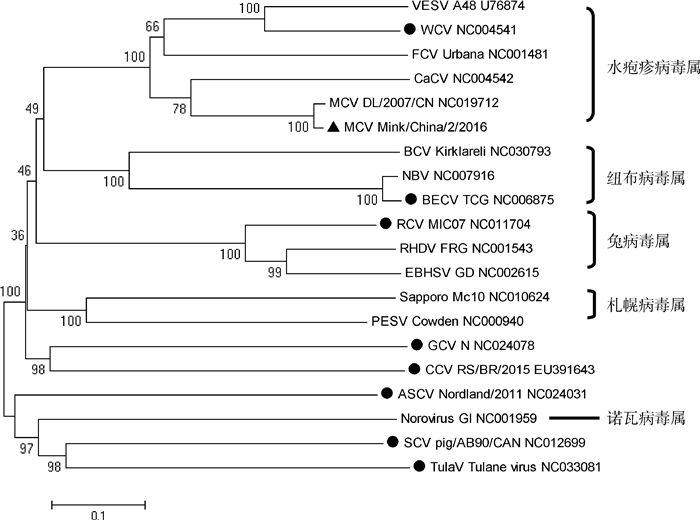
|
▲.水貂嵌杯病毒中国株Mink/China/2/2016;●. ICTV未确定属的嵌杯病毒 ▲. Mink calicivirus isolate Mink/China/2/2016;●. Caliciviruses which had not classified by ICTV 图 3 嵌杯病毒衣壳蛋白氨基酸序列进化分析 Figure 3 Capsid protein amino acid evolution analysis of caliciviruses |
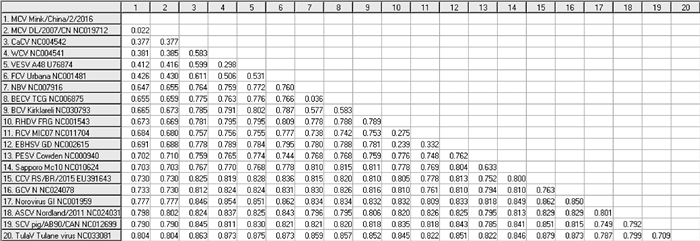
|
图 4 Mink/China/2/2016与嵌杯病毒代表毒株衣壳蛋白氨基酸序列分析 Figure 4 P-distance analysis of the capsid protein amino acid sequence of Mink/China/2/2016 and the representative caliciviruses |
同时,对表 2中8种ICTV未分类的嵌杯病毒分析发现:WCV归为水疱疹病毒属;BECV归为纽布病毒属;GCV和CCV归为一个单独的新属;RCV归为兔病毒属;ASCV、SCV和TulaV按照ICTV分类标准不属于任一已知的属。
3 讨论嵌杯病毒可引起包括人类、猫、猪、海洋哺乳动物、兔、野兔、牛、犬、爬行类、两栖类和昆虫等多个物种的呼吸道病、水疱、坏死性肝炎和胃肠炎等疾病。嵌杯病毒的感染常常持久,症状可能不明显、轻微,也可能严重[8]。对人、猪、猫、兔和猪等动物感染的嵌杯病毒的研究报道较多,但对水貂感染的嵌杯病毒的报道国内外仅有3篇文献对此进行描述和研究。J. F. Evermann等[2]最先从水貂分离到嵌杯病毒,并发现分离到的水貂杯状病毒具有非常特殊的生物学特性,能够与人O型红细胞发生凝集,不能凝集鸡、鹅、兔、猪、牛、山羊、绵羊、犬、大鼠、豚鼠的红细胞,该生物学特性的发现是本科属继兔出血热病毒之后第二个具有能够凝集红细胞特性的病毒。该毒株还具有广泛的细胞嗜性、血凝特性以及感染细胞表现出来得迅速致成CPE现象等,对于该病毒的研究将具有非常重要的意义。之后,M. Guo等[3]又发现了能引起水貂肠炎的新型嵌杯病毒MEC,并认为MCV属嵌杯病毒科札幌病毒属。2011年,B. C. Yang等[5]报道了我国的一株MCV的全基因组。
为深入了解水貂嵌杯病毒的特性,本文从我国青岛地区水貂养殖场检测到一株嵌杯病毒,并采用高通量测序方法获得该病毒的全基因,也是世界上第二个报道的MCV全基因序列。从全基因分析结果来看,在青岛检测到的毒株与2011年报道的大连株全基因序列相似性极高,都属于水疱疹病毒属成员。编码的3个ORF的大小相同,仅由于氨基酸序列变化而存在微小的差异。说明MCV在我国的不同区域和不同时间的流行毒株变异非常小,若研发防控此病的疫苗将在不同地区均具适用性。
本文还对现在已发现的所有19种动物嵌杯病毒进行了遗传进化分析,为分析8种ICTV未分类的嵌杯病毒(WCV、BECV、GCV、CCV、ASCV、SCV、RCV和TulaV)的分类地位提供基础。根据分析结果推测,WCV和BECV分别属于水疱疹病毒属和纽布病毒属。而禽源的两种病毒GCV和CCV在进化树上属同一分支,且两者衣壳蛋白氨基酸序列p-distance小于0.7,应属同一个属,但与其他嵌杯病毒亲缘关系较远。ASCV、SCV和TulaV衣壳蛋白氨基酸序列p-distance均大于0.7,但在进化树中与诺瓦病毒属关系较近,是否可归为诺瓦病毒有待进一步研究。
4 结论报道了世界上第二株水貂感染的嵌杯病毒全基因,同时对嵌杯病毒各属的进一步分类提供基础数据,有利于了解我国流行的水貂嵌杯病毒的特性,进一步明确其与其他嵌杯病毒的进化关系。
| [1] |
陆承平.
兽医微生物学[M]. 5版. 北京: 中国农业出版社, 2013.
LU C P. Veterinary microbiology[M]. 5th ed. Beijing: China Agriculture Press, 2013. (in Chinese) |
| [2] | EVERMANN J F, SMITH A W, SKILLING D E, et al. Ultrastructure of newly recognized caliciviruses of the dog and mink[J]. Arch Virol, 1983, 76(3): 257–261. DOI: 10.1007/BF01311109 |
| [3] | GUO M, EVERMANN J F, SAIF L J. Detection and molecular characterization of cultivable caliciviruses from clinically normal mink and enteric caliciviruses associated with diarrhea in mink[J]. Arch Virol, 2001, 146(3): 479–493. DOI: 10.1007/s007050170157 |
| [4] |
徐贵财. 水貂杯状病毒分离鉴定与生物学特性研究[D]. 北京: 中国农业科学院, 2009.
XU G C. Isolation and characterization of a calicivirus from mink[D]. Beijing: Chinese Academy of Agricultural Sciences, 2009. (in Chinese) |
| [5] | YANG B C, WANG F X, ZHANG S Q, et al. Complete genome sequence of a mink calicivirus in China[J]. J Virol, 2012, 86(24): 13835. DOI: 10.1128/JVI.02582-12 |
| [6] | QIU Y, CHEN J M, WANG T, et al. Detection of viromes of RNA viruses using the next generation sequencing libraries prepared by three methods[J]. Virus Res, 2017, 237: 22–26. DOI: 10.1016/j.virusres.2017.05.003 |
| [7] | TAMURA K, STECHER G, PETERSON D, et al. MEGA6:molecular evolutionary genetics analysis version 6.0[J]. Mol Biol Evol, 2013, 30(12): 2725–2729. DOI: 10.1093/molbev/mst197 |
| [8] |
MARKEY P J, LEONARD F C, FITZPATRICK E S, 等. 兽医微生物及所致传染病[M]. 2版. 陈继明, 马洪超, 陆承平, 译. 北京: 中国农业出版社, 2015.
MARKEY P J, LEONARD F C, FITZPATRICK E S, et al. Veterinary microbiology and microbial disease[M]. 2nd ed. CHEN J M, MA H C, LU C P, trans. Beijing: China Agriculture Press, 2015. (in Chinese) |



