2. 重庆市畜牧科学院, 荣昌 402460
2. Chongqing Academy of Animal Science, Rongchang 402460, China
中国是世界最大的猪肉生产和消费国,猪肉产量占世界50%以上。目前,为了降低对环境的污染,在控制养殖规模的前提下,又要不影响市场供给量,就需提高猪的生产水平和健康状况。饲养动物对环境的适应性是一个十分重要的问题,没有良好的适应性,生产性能再好的遗传潜力也不能充分表现,甚至不能生存[1-2]。随着养猪业规模化、集约化程度的不断提高,环境温度被认为是影响猪健康和生产力的最重要的外界环境影响因素,猪可通过物理和化学调节来适应外界温度的变化[3]。据报道,中国从加拿大、美国、英国和日本等国引进的猪种在夏季或冬季死亡率较高,并且高温或低温环境会导致母猪生殖失败,如分娩率低和发情期延迟等[4],主要原因是外来猪种对中国不同地区环境的适应性差,进而影响到中国以外来猪种为主的养猪业生产水平。而中国地方猪种由于世世代代长期生活在当地的自然条件下,形成了对当地环境的良好适应性,在极端气候条件下也能表现出良好的生长发育和生产性能。适应性是机体抗逆性和抗病性的表现,是有一定的遗传基础,受到各类基因的调控。因此,了解中国地方猪种高、低温环境下基因表达情况对理解猪的环境适应性具有一定的实践意义,可为生产上选择性提高猪的环境适应性奠定理论基础,也为中国地方猪种资源的保护及开发提供理论依据。
动物驯化是人类历史上最重要的事件之一,使人类从狩猎和采集的生活方式过渡到更安定的生活方式。大约在一万年前,猪被驯养[5]。从那时起,猪就开始受到自然选择和人工选择的综合影响,导致外貌、生育率、生长、适口性和当地适应性等表型出现显著地分化[6]。当前,中国是家猪遗传资源的主要拥有国家,占全球所有品种的1/3以上(约100种)。经过长期的自然选择和人工选育,中国地方猪种已进化出了极强的适应性来适应中国广阔的、复杂的地理生态环境[6]。中国高低纬度和不同海拔地区的地方猪种对冷热环境温度具有明显的体温调节机制。M.Z.Li等[7]通过对藏猪和四川盆地特有家猪品种从基因组水平进行了基因选择性清除分析,结果揭示了不同海拔环境猪适应性的分子机理,并鉴定出藏猪高海拔适应性的快速进化基因268个,家猪肌肉生长、脂肪沉积、免疫基因等受到人工强烈选择的基因516个。H.Ai等[8]在全基因组范围内系统地鉴别出了与不同纬度环境适应性相关的219个候选基因位点,揭示了中国南方猪和北方猪适应不同环境温度的分子机制。关于同一地区(像重庆夏季酷热、冬季严寒的极端气候地区)猪对不同环境温度的分子适应性研究还未见报道,因此,本研究比较分析了重庆地区低、高温月份猪能量代谢相关的分子表达水平,以期了解猪适应不同环境温度的分子变化情况,为阐明猪适应性进化的遗传基础作前期研究。
1 材料与方法 1.1 试验设计本研究选取荣昌猪为试验对象,按月份记录6月龄(180 d)猪的月增长体重,记录周期为1~12月,并利用全自动温湿度记录仪,采集试验场所的气温数据。所有猪参照NRC[9](1998)猪营养标准饲喂于西南大学畜牧实训基地,并且对猪舍温度不进行人为控制,比如地暖、水帘和风机等。
1.2 样品采集在低温(1月)和高温(8月)各选取12头猪进行屠宰,宰前24 h禁食、供水,宰后立即采集大脑、心、肝、肺、肾、眼肌和腰肌放入液氮中,带回实验室,保存于-80 ℃待用。
1.3 总DNA和RNA提取总DNA和RNA的提取按照说明书分别使用E.Z.N.A.® Tissue DNA Kit (Omega)和Trizol(Invitrogen)进行操作。提取出的总DNA溶解于50 μL TE缓冲液中,并于-20 ℃保存备用。RNA提取后,使用RNeasy柱(Qiagen)进行纯化,-80 ℃保存备用。
1.4 mtDNA拷贝数检测以基因组DNA为模板,使用qRT-PCR法测定线粒体DNA拷贝数。通过分别检测单拷贝基因GCG和线粒体基因ATP6和ND1来衡量核DNA和mtDNA的含量。单个细胞mtDNA拷贝数的计算:mtDNA特异基因拷贝数/GCG基因拷贝数。使用Primer 5.0软件设计基因扩增的特异性引物,引物序列见表 1。利用SYBR® Premix EX TaqTM Ⅱ kit (TaKaRa)在CFX96TM Real-Time PCR检测系统对基因进行定量。
|
|
表 1 基因定量引物序列 Table 1 Primer sequences of genes used in qRT-PCR |
参照Prime-ScriptTM RT reagent kit (TaKaRa)方法,进行反转录合成cDNA。以cDNA为模板,使用qRT-PCR法测定基因的相对表达水平。ACTB、RPL4和TBP1作为内参基因,特异性引物序列见表 1。qRT-PCR定量检测见1.4。基因表达水平采用2-△△Ct法进行计算。
1.6 数据分析使用SPSS 22.0软件对数据进行One-way ANOVA、Student’s t-test和Pearson相关性分析。数据结果以“平均值±标准差”表示。*P < 0.05表示差异显著; **P < 0.01表示差异极显著;ns表示差异不显著(P>0.05)。
2 结果 2.1 猪体重增长率与环境温度变化的关系如图 1所示,试验场所1~12月的平均气温呈现先升高后降低的趋势,其中1月的平均气温最低(6.7 ℃),8月的平均气温最高(30.2 ℃)。猪体重1~12月的增长率与气温呈阶段性相关:1~4月,随气温升高而升高(r=0.96,P < 0.01);4~8月,随气温升高而降低(r=-0.94,P < 0.01);8~10月,随气温降低而升高(r=-0.77,P < 0.01);10~12月,随气温降低而降低(r=0.97,P < 0.01),说明4和10月是猪体重增长的较佳时期,即20 ℃左右的气温是猪体重增长的最佳温度范围[10],而温度过高或过低均会影响猪的体重增长,其中8月的猪体重增长率最低(12.24%),反映出猪体重增长率与气温的相关性。上述结果表明,极端气温环境对猪的生长发育存在明显的影响,为了解猪在极端气温条件下的代谢特点,而能量代谢则是反映猪对温度适应性的直接过程,故本试验进一步研究了低温(1月)和高温(8月)条件下荣昌猪能量代谢相关分子的表达情况。
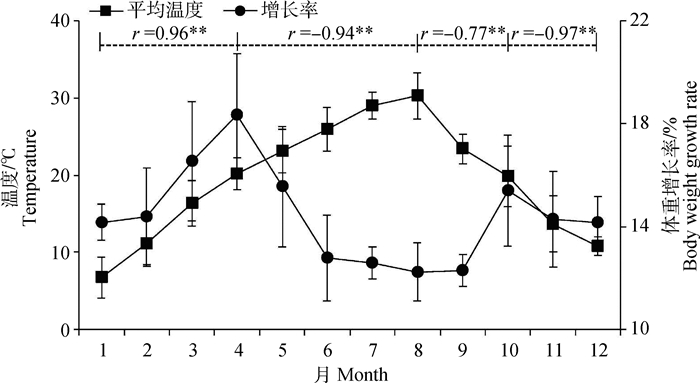
|
图 1 不同月份猪体重增长率与气温变化的关系 Figure 1 Correlation of body weight growth rate and temperature change in different months |
如图 2A所示,对低温和高温下的组织间mtDNA拷贝数进行单因素方差分析显示,组织间mtDNA拷贝数在低温组(P=3.14×10-10)和高温组(P=2.19×10-13)均差异极显著。其中,低温组mtDNA拷贝数相对较高的是眼肌和大脑,肾和肺相对较低;高温组mtDNA拷贝数最高和最低的分别是眼肌和肺,说明mtDNA拷贝数在低温组和高温组之间展现出非常高的一致性(r=0.92, P < 0.01;图 2B)。
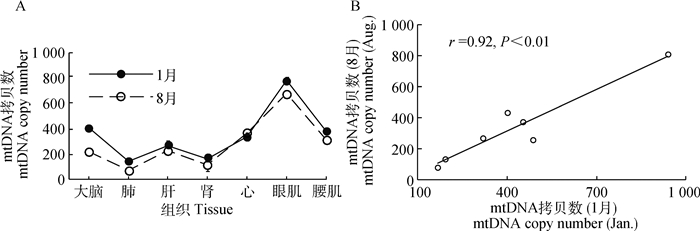
|
图 2 mtDNA拷贝数的变化(A)及季节间相关性分析(B) Figure 2 Characteristic of mtDNA copy number (A) and correlation analysis between seasons (B) |
如图 3A所示,除了心组织(P>0.05),其它组织的mtDNA拷贝数在低温组显著高于高温组(P < 0.05),尤其是大脑、肺和肾达到了极显著水平(P < 0.01),说明这3种组织比其他组织对环境温度的变化更敏感。TWINKLE基因是合成初始D-loop链和mtDNA完整复制所必需的,在维持mtDNA拷贝数上发挥着重要的作用[11]。因此,假设mtDNA拷贝数在高温组的降低可能受到TWINKLE基因表达的影响。如图 3B所示,所有组织TWINKLE基因表达在高温组均显著(P < 0.05)或极显著(P < 0.01)降低,这与假设一致,表明对TWINKLE基因表达的调节是mtDNA拷贝数适应环境温度变化机制中的重要一环。
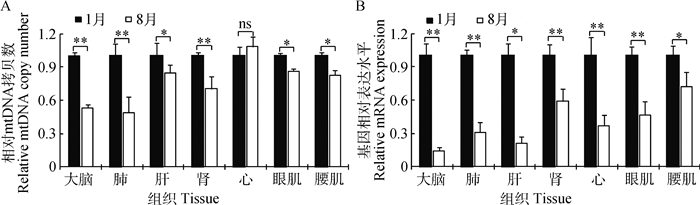
|
图 3 mtDNA拷贝数(A)和TWINKLE基因表达(B)在高低温度下的变化 Figure 3 Characteristic of mtDNA copy number (A) and TWINKLE gene expression (B) in high and low temperature |
如图 4所示,能量代谢相关基因(ND1、COX1、ATP6和CYTB)在组织中的表达水平基本上均是低温组显著(P < 0.05)或极显著(P < 0.01)高于高温组,而眼肌中ND1和COX1基因,肝和心中CYTB基因在低、高温组间无显著差异(P>0.05),大脑中CYTB基因在高温组显著高于低温组(P < 0.05),这可能与大脑的生理学功能相关。综上说明猪适应低温环境可通过补偿性调整特定基因转录水平来重塑代谢而实现。
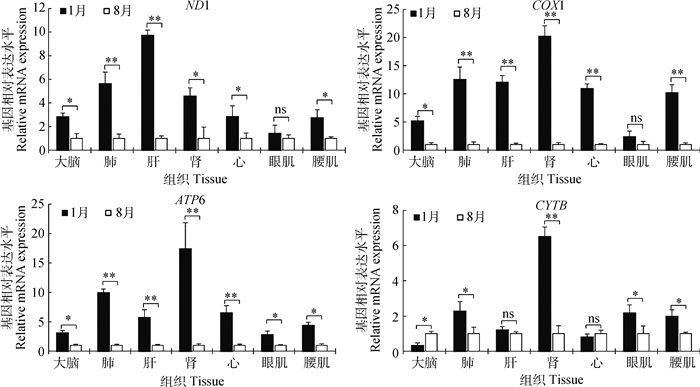
|
图 4 能量代谢相关基因在不同环境温度下表达水平 Figure 4 Energy metabolism-related genes expression in different ambient temperature |
对热应激基因HSP70的表达水平进行了测定(图 5),除了肺组织的HSP70基因表达水平在高温组极显著高于低温组(P < 0.01),其余组织的基因HSP70表达水平在两者之间无显著差异(P>0.05),说明极端环境温度对猪除了肺组织外,对其他器官无明显影响,猪已基本上适应了环境温度的变化,并产生了相应的分子适应性机制。
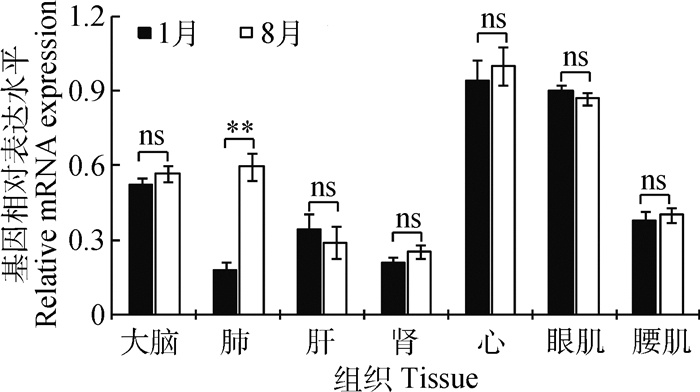
|
图 5 热应激基因HSP70在不同环境温度的表达水平 Figure 5 Expression of heat stress gene HSP70 in different ambient temperature |
猪的被毛和绝热层较少,并且缺乏汗腺功能,代谢产生的多余热量不能及时有效地散出,相较于其他动物对环境温度更敏感。研究表明,环境温度对育肥猪增重率的影响呈曲线关系[12]。在一定温度范围内,育肥猪的生长性能最佳,其产热与散热较少,仅靠物理调节就可维持体温平衡[13-14];低温环境下,育肥猪为了维持体温,不断地消耗饲料中的能量来加快分解代谢的速度,饲料并未完全用于生长,影响其体重的增加[15];高温环境下,育肥猪为了减少代谢产热,采食量大幅下降,导致其生长速度降低[16-17]。说明猪为了适应环境温度的变化,机体也在作出相应的变化,而环境温度的变化是季节性差异的主要因素之一,暗示了不同季节下猪的分子调控机制的差异,而本研究中的重庆地区季节性环境温度变化极大,育肥猪的增重率也表现出明显差异,故饲养在本地区的猪是研究环境温度适应性的理想材料。
不同组织细胞在生命活动中行使着不同的功能,具有不同的能量需求,故线粒体含量存在差异,而mtDNA拷贝数则是反映细胞线粒体含量的直接指标[18]。本试验中,环境温度变化对不同组织间mtDNA拷贝数的高低排序无影响,说明机体mtDNA拷贝数整体上不会轻易受外界影响,从而保证各组织生物学功能的实现,但同一组织在不同环境温度下mtDNA拷贝数存在差异。高温环境下,猪为了维持体热平衡,采食量减少,代谢产热减弱,能量需求减少,导致mtDNA拷贝数降低;低温环境下,猪为了维持体温恒定,代谢产热增强,能量需求增加,引起mtDNA拷贝数升高。长期生活在高原和平原的同一物种mtDNA拷贝数呈现极显著差异[19-20],说明机体为了适应外界环境变化对其组织mtDNA拷贝数作出相应的调整。基于mtDNA拷贝数对环境变化的敏感性,可考虑将mtDNA拷贝数作为反映环境变化的分子标记。
能量代谢过程除了受mtDNA拷贝数调节外,能量代谢相关基因的表达水平同样发挥重要作用,比如ND1、COX1、ATP6和CYTB基因。ND1基因可在呼吸链中直接参与电子传递并通过氧化磷酸化产生ATP而参与到能量代谢过程[21]。COX1基因能把氧化还原反应中的能量通过OXPHOS转变为ATP,在细胞有氧代谢过程中起着重要作用[22]。ATP6基因参与线粒体氧化磷酸化的调控,从而合成ATP[23]。CYTB基因可通过铁的化合价互变传递电子[24]。本试验中,低温环境下,ND1、COX1、ATP6和CYTB基因表达水平在绝大多数组织中显著高于高温环境,说明猪可通过调节特异基因表达和重塑能量代谢来适应环境温度的变化[25],高温环境下呼吸受到抑制,代谢产热减少,抑制能量代谢基因表达;低温环境下能量需求较高,代谢旺盛,促进能量代谢基因表达。
动物机体在受到高温刺激时,组织细胞会快速产生一类高度保守的保护性热休克蛋白来抵抗对细胞的损伤[26]。研究表明,HSP70是热休克蛋白家族中最保守,含量最丰富的一种非特异性的内源保护蛋白,具有保护细胞、减少炎症反应、抗细胞凋亡等功能,在高热环境刺激下,HSP70基因的表达量会显著升高[27-29]。本研究中,除了肺,其余组织HSP70基因的表达量在不同的季节差异不显著,可能是本研究的试验猪长期生活在该地区,已适应其自然环境温度的变化,说明猪具有极强的适应性。肺作为重要的呼吸系统组成部分,在高温环境下,为了散热控温,其呼吸频率升高,极易出现热喘行为,引起肺损伤[30],故HSP70基因表达量升高。
4 结论本研究通过检测季节性高低温环境下组织mtDNA拷贝数及能量代谢相关基因表达水平,初步探究了猪在分子水平对环境温度的适应性变化。结果提示,猪能量代谢相关基因转录水平及mtDNA拷贝数随着季节性环境温度的变化而发生了显著变化,但机体对环境的适应性变化是一个复杂性、系统性的过程,因此,还需进一步更为深入的研究来阐明具体的机制。
| [1] | BLOEMHOF S, MATHUR P K, KNOL E F, et al. Effect of daily environmental temperature on farrowing rate and total born in dam line sows[J]. J Anim Sci, 2013, 91(6): 2667–2679. DOI: 10.2527/jas.2012-5902 |
| [2] | VAN RENSBURG L J, SPENCER B T. The influence of environmental temperatures on farrowing rates and litter sizes in South African pig breeding units[J]. Onderstepoort J Vet Res, 2014, 81(1). DOI: 10.4102/ojvr.v81i1.824 |
| [3] | ⅡDAR, KOKETSUY. Climatic factors associated with peripartum pig deaths during hot and humid or cold seasons[J]. Prev Vet Med, 2014, 115(3-4): 166–172. |
| [4] | BODDICKER R L, SEIBERT J T, JOHNSON J S, et al. Gestational heat stress alters postnatal offspring body composition indices and metabolic parameters in pigs[J]. PLoS One, 2014, 9(11): e110859. DOI: 10.1371/journal.pone.0110859 |
| [5] | LARSON G, LIU R R, ZHAO X B, et al. Patterns of East Asian pig domestication, migration, and turnover revealed by modern and ancient DNA[J]. Proc Natl Acad Sci U S A, 2010, 107(17): 7686–7691. DOI: 10.1073/pnas.0912264107 |
| [6] |
国家畜禽遗传资源委员会.
中国畜禽遗传资源志:猪志[M]. 北京: 中国农业出版社, 2011: 2-16.
China National Commission of Animal Genetic Resources. Animal genetic resources in China: pigs[M]. Beijing: China Agricultural Press, 2011: 2-16. (in Chinese) |
| [7] | LI M Z, TIAN S L, JIN L, et al. Genomic analyses identify distinct patterns of selection in domesticated pigs and Tibetan wild boars[J]. Nat Genet, 2013, 45(12): 1431–1438. DOI: 10.1038/ng.2811 |
| [8] | AI H, FANG X, YANG B, et al. Adaptation and possible ancient interspecies introgression in pigs identified by whole-genome sequencing[J]. Nat Genet, 2015, 47(3): 217–225. DOI: 10.1038/ng.3199 |
| [9] | NRC. Nutrient requirements of swine[M]. Washington, DC: National Academy Press, 1998. |
| [10] |
李延森, 沈祥星, 李春梅. 母猪发情和产仔性能与环境温度变化相关性分析[J]. 畜牧兽医学报, 2016, 47(6): 1133–1139.
LI Y S, SHEN X X, LI C M. Correlation analysis between the ambient temperatures and reproductive performance of sows[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(6): 1133–1139. (in Chinese) |
| [11] | SHUTT T E, GRAY M W. Twinkle, the mitochondrial replicative DNA helicase, is widespread in the eukaryotic radiation and may also be the mitochondrial DNA primase in most eukaryotes[J]. J Mol Evol, 2006, 62(5): 588–599. DOI: 10.1007/s00239-005-0162-8 |
| [12] |
郭春华, 王康宁. 环境温度对生长猪生产性能的影响[J]. 动物营养学报, 2006, 18(4): 287–293.
GUO C H, WANG K N. Effects of environmental temperature on the performance of growing pigs[J]. Journal of Animal Nutrition, 2006, 18(4): 287–293. (in Chinese) |
| [13] | CAMERLINK I, BOLHUIS J E, DUIJVESTEIJN N, et al. Growth performance and carcass traits in pigs selected for indirect genetic effects on growth rate in two environments[J]. J Anim Sci, 2014, 92(6): 2612–2619. DOI: 10.2527/jas.2013-7220 |
| [14] | RENAUDEAU D, GOURDINE J L, ST-PIERRE N R. A meta-analysis of the effects of high ambient temperature on growth performance of growing-finishing pigs[J]. J Anim Sci, 2011, 89(7): 2220–2230. DOI: 10.2527/jas.2010-3329 |
| [15] | LE BELLEGO L, VAN MILGEN J, NOBLET J. Effect of high temperature and low-protein diets on the performance of growing-finishing pigs[J]. J Anim Sci, 2002, 80(3): 691–701. DOI: 10.2527/2002.803691x |
| [16] | HEITMAN Jr H, MORRISON S R. Ambient temperature and protein level in the ration of growing pigs[J]. Int J Biometeorol, 1988, 32(1): 44–46. DOI: 10.1007/BF01623993 |
| [17] | MORALES A, GRAGEOLA F, GARCÍA H, et al. Performance, serum amino acid concentrations and expression of selected genes in pair-fed growing pigs exposed to high ambient temperatures[J]. J Anim Physiol Anim Nutr, 2014, 98(5): 928–935. DOI: 10.1111/jpn.2014.98.issue-5 |
| [18] | ZHANG J, MA J D, LONG K, et al. Dynamic gene expression profiles during postnatal development of porcine subcutaneous adipose[J]. Peer J, 2016, 4(2): e1768. |
| [19] | LUO Y J, YANG X H, GAO Y Q. Mitochondrial DNA response to high altitude: A new perspective on high-altitude adaptation[J]. Mitochondr DNA, 2013, 24(4): 313–319. DOI: 10.3109/19401736.2012.760558 |
| [20] | LI Y, HUANG W, YU Q, et al. Lower mitochondrial DNA content relates to high-altitude adaptation in Tibetans[J]. Mitochondr DNA Part A, 2016, 27(1): 753–757. DOI: 10.3109/19401736.2014.915526 |
| [21] | SOLAINI G, SGARBI G, BARACCA A. Oxidative phosphorylation in cancer cells[J]. Biochim Biophys Acta, 2011, 1807(6): 534–542. DOI: 10.1016/j.bbabio.2010.09.003 |
| [22] | GENNIS R, FERGUSON-MILLER S. Structure of cytochrome c oxidase, energy generator of aerobic life[J]. Science, 1995, 269(5227): 1063–1064. DOI: 10.1126/science.7652553 |
| [23] | TAN G F, WANG F, ZHANG X Y, et al. Different lengths, copies and expression levels of the mitochondrial atp6 gene in male sterile and fertile lines of carrot (Daucus carota L.)[J]. Mitochondr DNA Part A, 2017: 1–9. |
| [24] | TAN A S, BATY J W, DONG L F, et al. Mitochondrial genome acquisition restores respiratory function and tumorigenic potential of cancer cells without mitochondrial DNA[J]. Cell Metab, 2015, 21(1): 81–94. DOI: 10.1016/j.cmet.2014.12.003 |
| [25] | ZHANG J, ZHOU C W, MA J D, et al. Breed, sex and anatomical location-specific gene expression profiling of the porcine skeletal muscles[J]. BMC Genet, 2013, 14: 53. |
| [26] | BAKTHISARAN R, TANGIRALA R, RAO C M. Small heat shock proteins: Role in cellular functions and pathology[J]. Biochim Biophys Acta, 2015, 1854(4): 291–319. DOI: 10.1016/j.bbapap.2014.12.019 |
| [27] | NAGAYACH R, GUPTA U D, PRAKASH A. Expression profiling of HSP70 gene during different seasons in goats (Capra hircus) under sub-tropical humid climatic conditions[J]. Small Rumin Res, 2017, 147: 41–47. DOI: 10.1016/j.smallrumres.2016.11.016 |
| [28] | BANERJEE D, UPADHYAY R C, CHAUDHARY U B, et al. Seasonal variation in expression pattern of genes under HSP70[J]. Cell Stress Chaperon, 2014, 19(3): 401–408. DOI: 10.1007/s12192-013-0469-0 |
| [29] | PARMAR M S, MADAN A K, HUOZHA R, et al. Heat shock protein70 (HSP70) gene expression pattern in peripheral blood mononuclear cells (PBMCs) during different seasons in Sahiwal cows (Bos indicus)[J]. J Anim Res, 2015, 5(1): 109–113. DOI: 10.5958/2277-940X.2015.00018.2 |
| [30] | LIU Z F, LI B L, TONG H S, et al. Pathological changes in the lung and brain of mice during heat stress and cooling treatment[J]. World J Emerg Med, 2011, 2(1): 50–53. DOI: 10.5847/wjem.j.1920-8642.2011.01.009 |



