2. 内蒙古农业大学动物科学学院, 呼和浩特 010018;
3. 内蒙古自治区综合疾病预防控制中心, 呼和浩特 010018;
4. 内蒙古农业大学生命科学学院, 呼和浩特 010018
2. College of Animal Science, Inner Mongolia Agricultural University, Hohhot 010018, China;
3. Inner Mongolia Center for Disease Control and Prevention, Hohhot 010018, China;
4. College of Life Science, Inner Mongolia Agricultural University, Hohhot 010018, China
骆驼斯氏副柔线虫(Parabronema skrjabini)属于旋尾目,副柔属[1-2],是一种寄生在骆驼、牛、羊等反刍动物真胃引起疾病的寄生虫,其中骆驼是其最适宜的终末宿主[3-4]。当骆驼大量感染斯氏副柔线虫时会导致腹泻、生长迟缓,真胃黏膜溃疡、出血,淋巴细胞浸润等现象,严重者会导致贫血死亡。国内自杨晓野[5]最早发现斯氏副柔线虫病以来,经流行病学调查发现斯氏副柔线虫已广泛存在,并且在不同地区的感染率为72%~100%。据报道,内蒙古地区双峰驼斯氏副柔线虫感染率高达100%,感染强度最高可达7 611条[6]。目前,斯氏副柔线虫病仍然严重危害我国牧区骆驼的健康,制约着当地养驼业的发展,给农牧民造成巨大的经济损失。
由于骆驼主要分布在经济欠发达国家或地区,导致斯氏副柔线虫病的相关研究资料较少,尤其是骆驼斯氏副柔线虫病致病作用的资料极度缺乏。目前,国内外对于骆驼斯氏副柔线虫病的研究主要在于形态学鉴定、流行病学调查等方面[7],尚无有效疫苗用于该病的预防,也未见报道该病有效的生前诊断方法。目前,在生产实践中,往往是在没有诊断依据的情况下盲目给药驱虫,不仅造成经济损失,而且也会导致药物残留超标和耐药虫株的产生。因此,对该病的研究应致力于在探索斯氏副柔线虫病生物学特征的基础上,寻找有效的生前诊断方法和制定新的防治措施。
本试验选取骆驼斯氏副柔线虫为研究对象,参考已报道的线虫免疫学诊断抗原的特点,在斯氏副柔线虫转录组测序分析的基础上[8-9],筛选出与斯氏副柔线虫免疫相关的基因,通过Blastx比对确定骆驼斯氏副柔线虫免疫相关基因CTPK (CAMK/TSSK protein kinase)。通过克隆斯氏副柔线虫CTPK基因的CDS区,利用生物信息学方法对该基因结构、理化性质及抗原表位等进行分析和预测,以期找到优势抗原表位,为骆驼斯氏副柔线虫病生前诊断方法iELISA的建立和DNA疫苗的研究奠定了理论基础。
1 材料与方法 1.1 试验材料 1.1.1 虫体和质粒骆驼斯氏副柔线虫样本采自于巴彦淖尔市乌拉特后旗双峰驼真胃。在体视镜下根据虫体不同形态特征鉴定雌雄后分装到2 mL冻存管中,标记后放入液氮中由本实验室保存,用于RNA的提取。
试验所用菌株:E. coli DH5α,北京全式金(TransGen Biotech)生物技术有限公司;质粒载体:克隆载体pMD19-T simple vector,大连宝生物工程有限公司。
1.1.2 主要试剂与仪器Trizol通用型RNA快速提取试剂盒、高保真酶Prime STAR® GXL DNA Polymerase、标准相对分子质量DNA Marker购自TaKaRa公司;DNA凝胶回收试剂盒,购自AXYGEN公司,DEPC处理水。NanoDROP核酸浓度测定仪,ND2000美国Thermo公司;凝胶影像仪,北京赛智创业科技有限公司等。
1.2 CTPK的扩增 1.2.1 总RNA提取与反转录按照Trizol[10]抽提总RNA说明书,提取斯氏副柔线虫总RNA。将获得的总RNA进行浓度、纯度检测后,利用反转录试剂盒合成cDNA。
1.2.2 目的基因的扩增CTPK序列来自本实验室骆驼斯氏副柔线虫高通量转录组测序序列,应用Primer5.0和Oligo6.24软件设计引物,同时在引物序列中加入酶切位点EcoRⅠ和XhoⅠ,并由华大基因有限公司合成,预期扩增的DNA片段约为519 bp。引物序列,上游引物:5′-GCAGAATTCATGTCCACAAAGACCAC-3′(下划线为EcoRⅠ酶切位点);下游引物:5′-CCACTCGAGACCGTTTTGTTGCACG-3′(下划线为XhoⅠ酶切位点)。以合成的cDNA为模板进行PCR扩增,PCR反应体系50 μL:cDNA 4 μL;Up-Primer 1 μL;Down-Primer 1 μL;Prime Star GXL DNA Polymerase 2 μL;5×Prime Star GXL Buffer(Mg2+ plus) 10 μL;dNTP Mixture 4 μL;ddH2O补齐至50 μL。PCR扩增条件:95 ℃预变性5 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min,共31个循环;72 ℃延伸10 min,4 ℃保存。将PCR产物用1%琼脂糖凝胶电泳进行检测。
1.3 CTPK基因的克隆和测序将PCR产物目的片段按照Axygen DNA胶回收试剂盒说明书进行胶回收,利用宝生物公司的DNA A-Tailing Kit对CTPK基因PCR产物进行平末端DNA片段的3′末端加一个“A-尾”,从而提高了平末端DNA片段的TA克隆效率。具体步骤:10×A-Tailing Buffer 5 μL;dNTP Mixture 4 μL;A-Tailing Enzyme 0.5 μL;平末端DNA片段0.5~5 μg;补ddH2O至50 μL;缓慢混匀后,72 ℃反应20 min;在冰浴中静置2 min。然后按照pMD19-T说明书,将加尾后的PCR产物与克隆载体pMD19-T 16 ℃过夜连接,将连接产物转化至Trans1-T1感受态细胞中:将100 μL感受态细胞放入冰盒中融化,加入过夜连接产物10 μL,混匀后冰浴30 min,42 ℃水浴45 s,冰浴2 min,然后加入无抗性LB培养液500 μL,37 ℃ 190 r·min-1摇菌2~3 h,最后取100 μL涂布在氨苄青霉素抗性的LB固体培养基上,37 ℃倒置过夜培养。挑取单克隆菌落放入含有氨苄抗性的LB液体培养基中,37 ℃摇菌一段时间后,观察菌液浑浊程度,如有浑浊可进行菌液PCR鉴定,阳性菌株送华大基因公司进行双向序列测定。将测序结果正确的质粒命名为pMD19T-CTPK。
1.4 CTPK基因生物信息学分析使用DNAStar中的EditSeq推导斯氏副柔线虫CTPK基因的氨基酸序列,然后利用Prot-Param在线软件分析CTPK蛋白的氨基酸序列组成和理化性质;利用ProtScale软件分析氨基酸残基的亲疏水性;利用TMHMM Server和SignaIP 4.1 Server[11]分析工具预测该蛋白质的跨膜区及信号肽;利用SOPMA在线软件分析蛋白质的二级结构;根据在线软件Phyre2.0[12]预测CTPK蛋白的三级结构;应用NetPhos3.0 Server软件预测磷酸化位点;运用SecretomeP软件预测非典型分泌蛋白;利用DNAStar分析CTPK蛋白质表面可行性,B细胞、T细胞抗原表位等[13]。
2 结果 2.1 目的基因的PCR扩增以cDNA为模板扩增CTPK基因,目的片段为519 bp,用1%琼脂糖凝胶电泳检测结果与预期目的基因片段大小一致(图 1)。
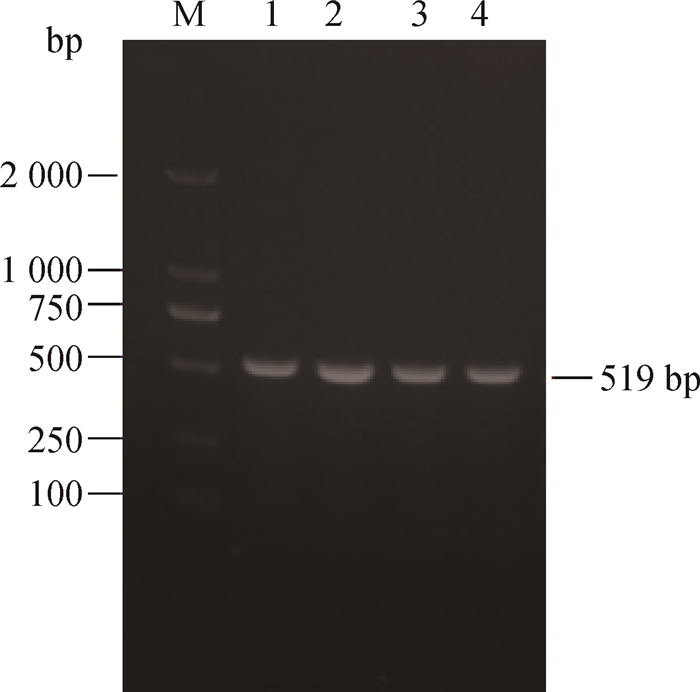
|
M. DNA相对分子质量标准DL2000;1~4. CTPK基因扩增产物 M. DNA marker DL2000;1-4. Products of CTPK gene 图 1 CTPK基因PCR扩增结果 Figure 1 PCR amplification of CTPK gene |
利用NCBI中的ORF Finder工具,对CTPK基因cDNA全长序列进行分析,结果表明,该基因cDNA序列全长共519 bp,含有一个编码173个氨基酸的完整ORF。重组基因测序后序列与转录组测序序列相似性为98.46%,发现8个突变位点:C139T、G165A、C189T、G199A、A213G、G321A、A348T、G408A。利用在线软件Rare Codon Caltor对斯氏副柔线虫重组基因测序序列和转录组序列进行稀有密码子分析,发现是同义突变,且突变前后稀有密码子种类、数量没有改变。
2.2.2 CTPK蛋白理化性质分析使用Port-Param在线软件分析CTPK蛋白的理化性质,结果表明CTPK蛋白相对分子质量为20.264 ku,理论等电点(theoretical PI)为9.53,原子总数共2 831个,分子式(Formula)为C895H1403N263O264S6,包含20种氨基酸,其中含量最高的是Lys(占8.7%),含量最低的是Trp占0.6%;带正电荷数(Arg+Lys)为27个,带负电荷数(Asp+Glu)为16个;蛋白质不稳定指数(instability index)为53.22,说明该蛋白质是不稳定蛋白(注:不稳定系数小于40,说明是稳定蛋白,反之则是不稳定蛋白);脂肪系数(Aliphatic index)为64.74;总平均亲水指数(Grand average of hydropathicity)为-0.925,说明该蛋白质是亲水性蛋白;假定所有的半胱氨酸都形成二硫键,CTPK在280 nm处的摩尔消光系数为23 630 (mol·cm),在0.1%(=1 g·L-1)溶液中的摩尔消光系数为1.154;而当所有的二硫键打开时280 nm处摩尔消光系数为23 380 (mol·cm),在0.1%(=1 g·L-1)溶液中摩尔消光系数1.517。当成熟肽N端的1个氨基酸为Met时,CTPK蛋白在体外哺乳动物网状红细胞中的半衰期为30 h,在酵母体内的半衰期大于20 h,在大肠埃希菌体内的半衰期大于10 h。
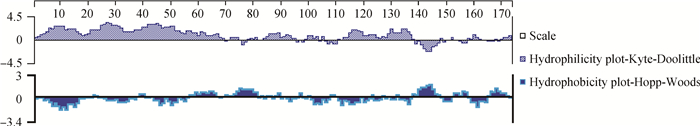
2.2.3 CTPK蛋白疏水性/亲水性预测分析氨基酸残基根据其亲疏水性可分为亲水性氨基酸和疏水性氨基酸。亲水性氨基酸位于蛋白质的表面,连续在一起形成抗原表位,同时也是判断B细胞抗原表位的主要参数[14]。而疏水性氨基酸位于蛋白内部。应用ProtScale在线软件对斯氏副柔线虫CTPK基因编码蛋白亲水性/疏水性进行分析(图 2)。由输出的文本可知其疏水性在143~144位点有最大值2.222,在27位点有最小值-3.267。根据氨基酸分值越高疏水性越强或氨基酸分值越低亲水性越强的规律分析得出,CTPK基因编码蛋白多肽链大部分区域分值为负值,说明该蛋白质是亲水性蛋白,与Port-Param预测结果相一致。
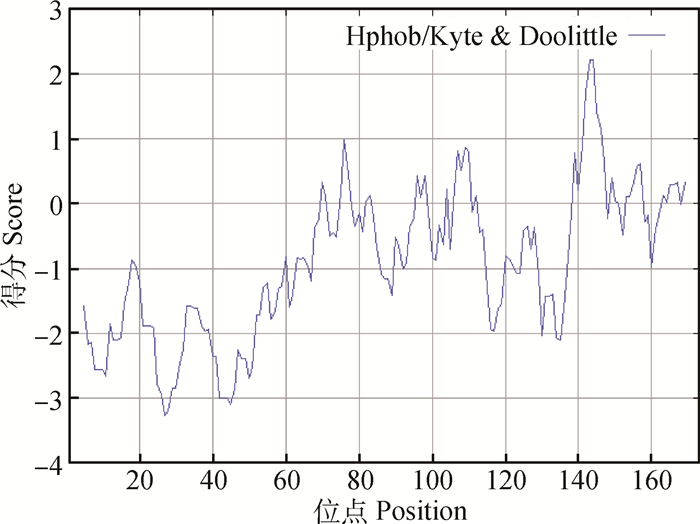
|
图 2 CTPK蛋白疏水性预测 Figure 2 Prediction of hydrophobicity of CTPK protein |
膜蛋白是镶嵌在生物膜中的蛋白质,在细胞中起着至关重要的作用,它们构成了各种激素、受体和神经信号,同时也是离子跨膜的通道。应用TMHMM预测CTPK蛋白无跨膜区,不是跨膜蛋白,且整个蛋白全部位于膜外(图 3),跨膜区中的氨基酸期望值约为0.176(该数值大于18时表明有跨膜区)。
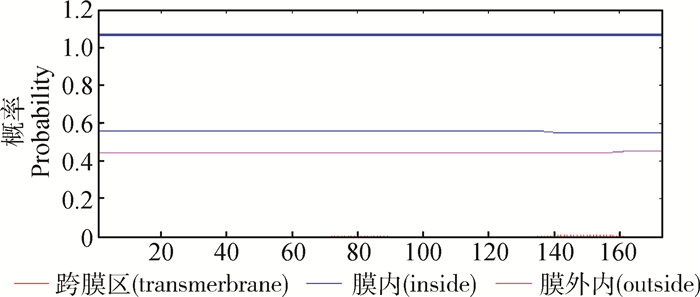
|
图 3 CTPK蛋白跨膜结构预测 Figure 3 Prediction of CTPK protein transmembarne domain |
信号肽是由蛋白质N端15~30个氨基酸残基组成的一段疏水肽段,决定蛋白质在细胞中的最终定位。信号肽不但决定蛋白质是否为分泌蛋白,而且与新生肽链在细胞内全方位定位有关。应用在线软件SignalP对CTPK蛋白的信号肽进行预测,综合图形和输出文本分析发现CTPK不存在信号肽(图 4),但是在运用SecretomeP软件预测非典型分泌蛋白发现,NN-Score=0.912(若NN-Score>0.5,判定为非典型分泌蛋白),所以该蛋白质属于分泌蛋白。
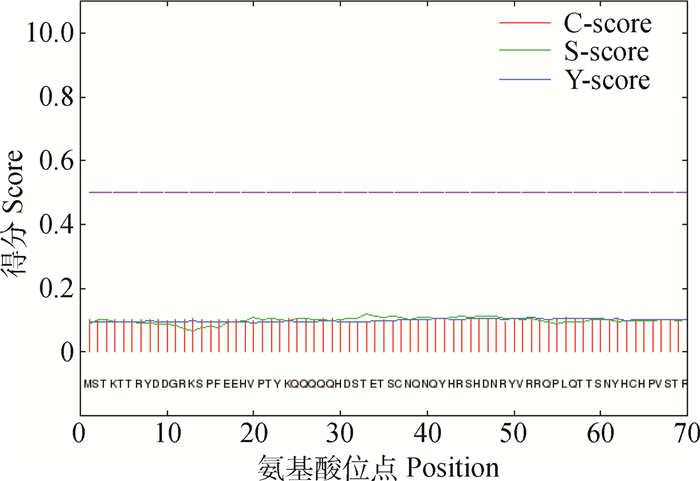
|
图 4 CTPK蛋白信号肽序列预测 Figure 4 Prediction of signal peptide sequence of CTPK protein |
磷酸化是最重要的蛋白质翻译后修饰之一,与蛋白质的功能密切相关,蛋白质的磷酸化与去磷酸化为真核细胞提供了调节机制。随着生物信息学的发展,对磷酸化位点的预测和发现已成为生物信息学焦点问题之一。通过在线软件NetPhos2.0 Server预测CTPK蛋白磷酸化位点发现(图 5),当潜在磷酸化位点的阈值为0.5时,CTPK蛋白存在24个潜在的磷酸化位点,其中包括10个丝氨酸位点,11个苏氨酸位点,3个酪氨酸位点。
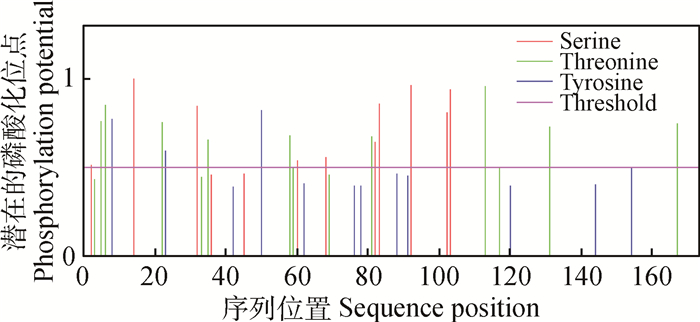
|
图 5 CTPK蛋白磷酸化位点预测 Figure 5 Prediction of CTPK protein phosphorylation sites |
通过在线软件SOPMA对斯氏副柔线虫CTPK基因编码蛋白的二级结构进行预测,可见CTPK蛋白中参与α螺旋形成的氨基酸有34个,占19.65%;参与β转角形成的氨基酸有25个,占14.45%;参与延伸链形成的氨基酸有32个,占18.50%;参与无规则卷曲形成的氨基酸为82个,占47.40%。
2.2.7 CTPK蛋白三级结构预测应用在线软件Phyre2.0对CTPK蛋白的三级结构预测,结果表明:CTPK蛋白由α螺旋、β转角、延伸链和无规卷曲组合形成超二级结构,经折叠、弯曲等一系列复杂的过程形成了CTPK蛋白的三级结构(图 6)。
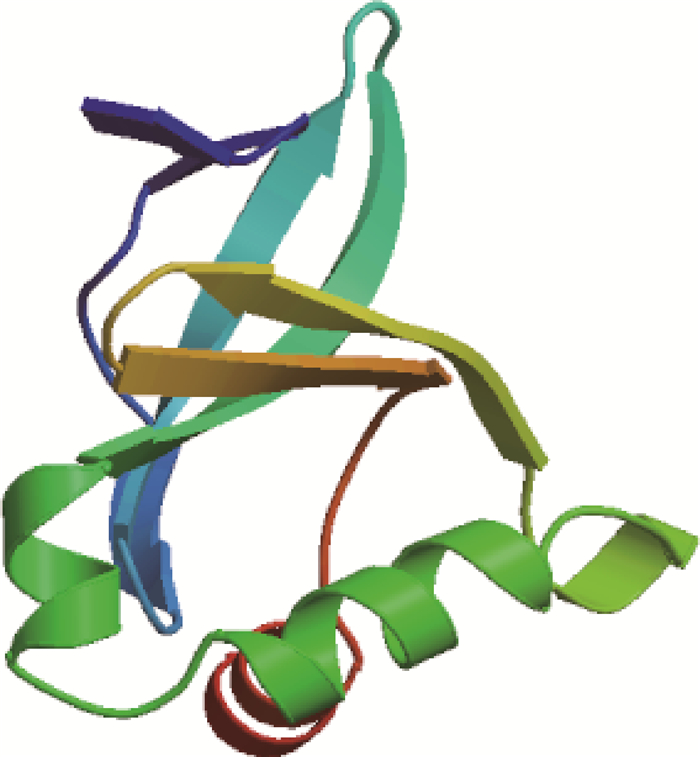
|
图 6 CTPK三级结构的预测 Figure 6 Prediction of tertiary structure of CTPK |
利用DNAStar软件对CTPK蛋白抗原表位进行分析,由图 7可知其亲水性位点主要在1—69、72—74、78—81、84—95、99—103、113—138、158—162、170—173位氨基酸处,与之前ProtScale预测的结果相符。根据Plot-Emini方法预测其表面可行性(图 8),在1—16、19—33、36—60、67—68、82—85、89—91、101—104、111—121、128—137、159—162、171—173位的氨基酸处有较高的可行性。根据Jameson-Wolf方法预测CTPK蛋白的抗原性系数,该蛋白质含有较多潜在的B细胞抗原表位(图 9),主要位于1—63、67—73、82—96、98—99、101—107、111—126、133—140、148—151、158—164、171—173位氨基酸残基或其附近,提示这些区段含有潜在优势抗原表位。然后再结合蛋白质二级结构、亲疏水性和表面可能性,综合分析CTPK蛋白潜在的蛋白抗原表位,在1—60、82—95、111—126、133—137、158—162、171—173等位氨基酸残基区域β转角和无规则卷曲结构较多、亲水性和表面可能性指数较高,暴露于表面的概率较大,提示作为B细胞抗原表位的可能性较大。T细胞表位是可以被TCR识别的抗原表位。应用Rothbard-Tayloy方法预测含有特定基序(motif)的潜在T淋巴细胞抗原表位,利用AMPHI方法预测免疫优势辅助性T淋巴细胞抗原位点,综合两种方法预测T细胞抗原表位(图 10),CTPK蛋白的T细胞抗原表位可能有9个,主要位于4—10、42—52、75—77、90—98、105—113、115—118、123—128、130—140、158—161位氨基酸残基或其附近,且分布较均匀。
|
图 7 CTPK蛋白亲水性预测 Figure 7 Prediction of hydrophilicity of CTPK protein |
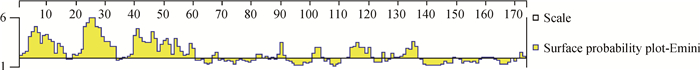
|
图 8 CTPK蛋白表面可及性预测结果 Figure 8 Prediction of surface probability of CTPK protein |

|
图 9 CTPK蛋白B细胞抗原表位预测结果 Figure 9 Prediction of B-cell antigenic epitopes of CTPK protein |

|
图 10 CTPK蛋白T细胞抗原表位预测结果 Figure 10 Prediction for T-cell antigenic epitopes of CTPK protein |
近年来,斯氏副柔线虫病在骆驼养殖中仍处于较高的感染水平,由于骆驼主要分布在经济欠发达国家或地区,导致斯氏副柔线虫病的相关研究资料较少,到目前为止,其确切的生活史仍未完全研究清楚。在斯氏副柔线虫病的防治上,尚缺乏有效的生前诊断方法和防治新措施。本研究通过克隆CTPK的编码区序列,利用生物信息学技术对分泌蛋白CTPK进行分析,采用不同方法和多种参数联合预测,以提高预测的准确性,为骆驼斯氏副柔线虫病的生前诊断建立和疫苗研制提供参考。
生物信息学是近年来发展起来的一种高效的研究手段,促进了生物学研究领域的快速发展[15],在寄生虫的研究上,也得到了广泛应用[16]。生物信息学技术已被广泛应用于预测和筛选有效的疫苗候选基因[17]。寄生虫的一些分泌性蛋白是虫体的表皮、肠上皮或分泌排泄器官不断分泌的抗原物质。这些抗原暴露于宿主免疫系统,是刺激宿主产生特异性免疫应答的重要靶抗原,被认为是与寄生虫免疫相关的抗原蛋白,在寄生虫建立寄生生活,免疫调节和免疫逃避中起关键作用[18-19]。这些功能性抗原可以为筛选疫苗抗原、发掘药物靶点和确定诊断抗原提供依据[20-21]。
本试验克隆了CTPK的编码区序列,采用生物信息学方法对分泌蛋白CTPK的理化性质、二级结构、B/T细胞抗原表位进行预测分析,结果表明:蛋白质理论相对分子质量为20.264 ku,理论等电点为9.53,在组成CTPK蛋白的20种氨基酸中,含量最高的是Lys占8.7%,含量最低的是Trp占0.6%;蛋白质不稳定指数为53.22,说明该蛋白质是不稳定蛋白;总平均亲水指数为-0.925,说明该蛋白质是亲水性蛋白,亲水区的存在有利于蛋白的高效表达;CTPK蛋白是一种不具有信号肽序列的非典型分泌蛋白(NN-Score=0.912),蛋白跨膜区预测分析表明该蛋白质不是跨膜蛋白,主要位于胞外区。利用DNAStar对斯氏副柔线虫CTPK蛋白的T/B细胞抗原表位进行预测,从该蛋白质的亲水性与表面可行性等方面分析,该蛋白质亲水性多肽较多,其合成的多肽很可能易溶于水;表面可行性较高区域可能位于CTPK蛋白分子表面。综合分析发现在1—60、82—95、111—126、133—137、158—162、171—173位氨基酸残基存在潜在的B细胞抗原表位;在4—10、42—52、75—77、90—98、105—113、115—118、123—128、130—140、158—161位氨基酸残基存在潜在的T细胞抗原表位,具有较好的抗原性,符合寄生虫免疫学诊断和免疫防治候选基因的特点。本试验为骆驼斯氏副柔线虫病早期快速诊断试剂盒的建立奠定基础,同时也为其DNA疫苗的研制提供理论依据。
4 结论扩增斯氏副柔线虫免疫相关基因CTPK的CDS区,其cDNA序列全长共519 bp,编码173个氨基酸,推导CTPK的相对分子质量为20.26 ku,理论等电点为9.53。结构分析发现,蛋白质无跨膜区,无规则卷曲构成二级结构的主要组分,亲水性氨基酸比例超过60%;抗原表位预测表明,CTPK蛋白是一种抗原性较高的亲水性蛋白。推测CTPK有望用作斯氏副柔线虫的免疫诊断抗原和疫苗候选抗原。
| [1] |
卢俊杰, 靳家声.
人和动物寄生线虫图谱[M]. 北京: 中国农业科学技术出版社, 2002: 405-459.
LU J J, JIN J S. The map of human and animal parasitic nematodes[M]. Beijing: China Agricultural Science and Technology Press, 2002: 405-459. (in Chinese) |
| [2] |
沈杰, 黄兵.
中国家畜家禽寄生虫名录[M]. 北京: 中国农业科学技术出版社, 2004: 105-111.
SHEN J, HUANG B. A list of parasites for livestock and poultry in China[M]. Beijing: China Agricultural Science and Technology Press, 2004: 105-111. (in Chinese) |
| [3] | HASHEMINASAB S S. Molecular characterization of the first internal transcribed spacer of rDNA of Parabronema skrjabini for the first time in sheep[J]. Ann Parasitol, 2015, 61(4): 241–246. |
| [4] | HASHEMINASAB S S, JALOUSIAN F, MESHGI B. Molecular and morphological characterization of Parabronema skrjabini of sheep and goats at three different geographical zones in Iran[J]. Ann Parasitol, 2016, 62(1): 55–61. |
| [5] |
杨晓野. 内蒙古自治区骆驼寄生虫种类调查[D]. 呼和浩特: 内蒙古农牧学院, 1985: 40-41.
YANG X Y. Camel parasitic species investigation in Inner Mongolia Autonomous Region[D]. Hohhot: Inner Mongolia Agricultural University, 1985: 40-41. (in Chinese) http://kns.cnki.net/KCMS/detail/detail.aspx?filename=nmgm198605016&dbname=CJFD&dbcode=CJFQ |
| [6] |
杨莲茹, 杨晓野, 刘珍莲, 等. 内蒙古地区骆驼斯氏副柔线虫(Parabronema skrjabini)病调查[J]. 内蒙古农业大学学报, 2004, 25(1): 43–45.
YANG L R, YANG X Y, LIU Z L, et al. Investigation of Parabronema Skrjabini disease of camels in Inner Mongolia Region[J]. Journal of Inner Mongolia Agricultural University, 2004, 25(1): 43–45. (in Chinese) |
| [7] |
赵治国. 我国骆驼斯氏副柔线虫病传播媒介的研究[D]. 呼和浩特: 内蒙古农业大学, 2010.
ZHAO Z G. Study on the vector of camel Parabronemosis in China[D]. Hohhot: Inner Mongolia Agricultural University, 2010. (in Chinese) http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=D198051 |
| [8] |
张晓东, 杨晓野, 杨莲茹, 等. 斯氏副柔线虫rDNA-ITS片段的克隆及序列分析[J]. 畜牧兽医学报, 2009, 40(5): 775–779.
ZHANG X D, YANG X Y, YANG L R, et al. Cloning and sequence analysis of the rDNA-ITS of Parabronema skrjabini[J]. Acta Veterinaria et Zootechnica Sinica, 2009, 40(5): 775–779. (in Chinese) |
| [9] |
冯陈晨, 王文龙, 呼和巴特尔. 骆驼斯氏副柔线虫转录组的高通量测序及分析[J]. 中国兽医学报, 2017, 37(4): 671–675.
FENG C C, WANG W L, HUHR B T E. Sequencing and analysis of the transcriptome of Parabronema skrjabini in camle[J]. Chinese Journal of Veterinary Science, 2017, 37(4): 671–675. (in Chinese) |
| [10] | SIMÕES A E S, PEREIRA D M, AMARAL J D, et al. Efficient recovery of proteins from multiple source samples after trizol® or trizol®·LS RNA extraction and long-term storage[J]. BMC Genomics, 2013, 14: 181. DOI: 10.1186/1471-2164-14-181 |
| [11] | PETERSEN T N, BRUNAK S, VON HEIJNE G, et al. SignalP 4.0:discriminating signal peptides from transmembrane regions[J]. Nat Methods, 2011, 8(10): 785–786. DOI: 10.1038/nmeth.1701 |
| [12] | KELLEY L A, MEZULIS S, YATES C M, et al. The Phyre2 web portal for protein modeling, prediction and analysis[J]. Nat Protoc, 2015, 10(6): 845–858. DOI: 10.1038/nprot.2015.053 |
| [13] |
李玲玲, 朱珊丽, 李文姝, 等. EB病毒核蛋白-1 B细胞表位的预测及其同源性分析[J]. 生物医学工程学杂志, 2011, 28(2): 371–375.
LI L L, ZHU S L, LI W S, et al. Prediction and research on homology of B-cell epitopes of Epstein-Barr virus nuclear antigen-1[J]. Journal of Biomedical Engineering, 2011, 28(2): 371–375. (in Chinese) |
| [14] | SIVALINGAM G N, SHEPHERD A J. An analysis of B-cell epitope discontinuity[J]. Mol Immunol, 2012, 51(3-4): 304–309. DOI: 10.1016/j.molimm.2012.03.030 |
| [15] | BLAZIE S M, BABB C, WILKY H, et al. Comparative RNA-Seq analysis reveals pervasive tissue-specific alternative polyadenylation in Caenorhabditis elegans intestine and muscles[J]. BMC Biol, 2015, 13: 4. DOI: 10.1186/s12915-015-0116-6 |
| [16] | LIU G H, XU M J, CHANG Q C, et al. De novo transcriptomic analysis of the female and male adults of the blood fluke Schistosoma turkestanicum[J]. Parasit Vectors, 2016, 9: 143. DOI: 10.1186/s13071-016-1436-2 |
| [17] |
成文敏, 潘伟荣, 尹俊芳, 等. 版纳微型猪近交系DLDH基因编码区序列扩增及生物信息学分析[J]. 畜牧兽医学报, 2015, 46(10): 1706–1712.
CHENG W M, PAN W R, YIN J F, et al. Amplification and bioinformatics analysis of coding region of the DLDH gene in Banna Mini-pig inbred line (BMI)[J]. Acta Veterinaria et Zootechnica Sinica, 2015, 46(10): 1706–1712. (in Chinese) |
| [18] | TRITTEN L, CLARKE D, TIMMINS S, et al. Dirofilaria immitis exhibits sex-and stage-specific differences in excretory/secretory miRNA and protein profiles[J]. Vet Parasitol, 2016, 232: 1–7. |
| [19] | EBERLE R, BRATTIG N W, TRUSCH M, et al. Isolation, identification and functional profile of excretory-secretory peptides from Onchocerca ochengi[J]. Acta Trop, 2015, 142: 156–166. DOI: 10.1016/j.actatropica.2014.11.015 |
| [20] | CAO X D, FU Z Q, ZHANG M, et al. iTRAQ-based comparative proteomic analysis of excretory-secretory proteins of schistosomula and adult worms of Schistosoma japonicum[J]. J Proteomics, 2016, 138: 30–39. DOI: 10.1016/j.jprot.2016.02.015 |
| [21] | CAO X D, FU Z Q, ZHANG M, et al. Excretory/secretory proteome of 14-day schistosomula, Schistosoma japonicum[J]. J Proteomics, 2016, 130: 221–230. |



