2. 中国动物卫生与流行病学中心, 青岛 266032;
3. 河北出入境检验检疫局技术中心, 石家庄 050051;
4. 江苏省农业科学院兽医研究所, 南京 210014;
5. 河北农业大学动物医学院, 保定 071001
2. China Animal Health and Epidemiology Center, Qingdao 266032, China;
3. Inspection and Quarantine Technical Center, Hebei Entry-Exit Inspection and Quarantine Bureau, Shijiazhuang 050051, China;
4. Institute of Veterinary Medicine, Jiangsu Academy of Agricultural Sciences, Nanjing 210014, China;
5. College of Veterinary Medicine, Hebei Agricultural University, Baoding 071001, China
猪病毒性腹泻是猪场常见疾病之一,主要病原是猪流行性腹泻病毒(porcine epidemic diarrhea virus, PEDV)、猪传染性胃肠炎病毒(porcine transmissible gastroenteritisvirus, TGEV)和猪轮状病毒(porcine rotavirus, PoRV)。这三种腹泻病毒以单独或混合感染方式引起猪群呕吐、水样腹泻、脱水、逐渐消瘦等临床症状,对仔猪危害较大,可导致仔猪高死亡率,造成养猪业严重的经济损失[1-2]。自2010年以来,我国多省大范围暴发以严重腹泻和仔猪高死亡率为症状的猪腹泻病,在2011年和2012年最为严重,PEDV变异株的流行是导致此次疫情暴发的主要原因[3-4]。在河北地区,2010年开始暴发腹泻疫情,腹泻病高发时期在2011年至2012年间,与其他省份的报道基本一致,而在2013年河北省腹泻病发生率明显回落[5-7]。2013年后,河北省邻近地区腹泻病发生情况略有不同,山东省部分地区腹泻病呈现缓和态势[8-9],而在山西、辽宁等地腹泻病仍呈现高发生率和高死亡率[10-11]。为摸清当前河北地区猪病毒性腹泻的流行状况和遗传变异规律,2016年4月至2017年2月,本研究采集河北省11个地市无害化处理厂、门诊部和屠宰场发病猪样品,进行PEDV、TGEV和PRoV的流行病学调查和PEDV S基因的遗传变异分析,为河北省猪病毒性腹泻的综合防控提供理论依据。
1 材料与方法 1.1 流行病学调查和样品采集2016年4月至2017年2月期间,通过对河北省11个地市的无害化处理厂和门诊部送检的1 855例患猪病例进行背景分析,腹泻发病猪180例。对腹泻发病猪采集小肠组织或小肠内容物用于后续检测。
1.2 主要试剂与设备Taq DNA聚合酶、dNTPs购自上海生工科技有限公司;磁珠法病毒DNA/RNA提取试剂盒购自天根生化科技(北京)有限公司;猪传染性胃肠炎病毒/流行性腹泻病毒/轮状病毒三重核酸检测试剂盒(PCR-荧光探针法)购自中山大学达安基因股份有限公司。
组织研磨仪购自德国Qiagen公司;核酸提取仪购自赛默飞世尔生物科技公司;四通道荧光PCR仪购自西安天隆科技有限公司。
1.3 样品处理和检测方法取腹泻病料小肠组织样品或肠内容物加1 mL PBS制成组织匀浆,反复冻融3次;5 000 r·min-1离心5 min,取200 μL上清液进行核酸提取。按荧光PCR检测试剂盒说明书配制反应体系并加入模板,利用荧光PCR仪,参照试剂盒说明书设定扩增程序进行荧光RT-PCR,待扩增结束后按说明书进行结果判定。
1.4 PEDV S基因引物的设计与目的片段扩增以PEDV流行株AJ1102株为参考模板,利用Oligo 6.0软件设计三对用于扩增PEDV S基因的引物(表 1),利用RT-PCR对9份来源不同的PEDV阳性病料扩增S基因,经测序拼接后获得完整的S基因序列。
|
|
表 1 PEDV S全长基因扩增序列 Table 1 The primers used for amplification of PEDV S gene |
PCR产物胶回收试剂盒购自天根生化科技(北京)有限公司;将胶回收产物送上海生工科技有限公司进行测序。
1.6 测序结果分析通过BioEdit将所克隆获得的S基因与GenBank上发表的33条国内外PEDV S基因推导氨基酸序列进行比对分析(表 2)。利用MEGA 7.0程序软件的邻位相接法(Neighbor-joining法)进行系统进化树的构建,并以Bootstrap值验证进化树的可信度。利用NetNGlyc 1.0 Server对S蛋白糖基化位点进行预测。
|
|
表 2 PEDV参考毒株信息 Table 2 Representative PEDV strains used in this study |
对全年送检的1 855例患猪病例进行统计分析,其中腹泻病例共180例,占全年总送检病例的9.70%。对腹泻病样品检测结果进行统计,发现25份病料有PEDV检出,检出率为13.89%;9份病料有PoRV检出,检出率为5.00%;TGEV仅在3份病料中检出,检出率为1.67%(表 3)。对河北地区11地市检出结果进行分别统计,结果显示,各地市PEDV和TGEV检出情况与河北省基本一致,但少数地市PoRV的检出率偏高(图 1)。综合以上结果表明,当前PEDV仍是引起河北地区猪群腹泻病的主要病毒性病原。
|
|
表 3 河北地区腹泻相关病毒检测情况 Table 3 The positive rate of PEDV, TGEV and PoRV |
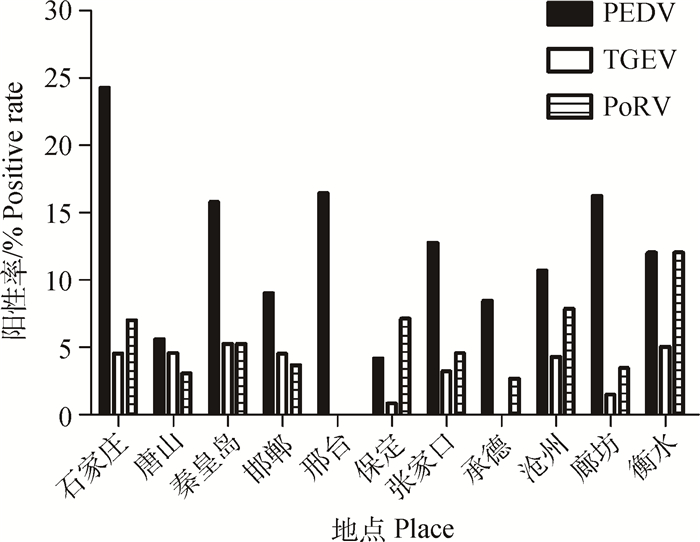
|
图 1 河北省11地市腹泻相关病原调查结果 Figure 1 Investigation of diarrhea related pathogen from Hebei province |
对腹泻病样品按猪年龄分类进行结果统计,结果显示,PEDV在仔猪和育肥猪中均有较高检出率,在仔猪中检出率最高,可达19.28%;TGEV和PoRV则在育肥猪中检出率最高,分别达6.90%和13.79%(表 3)。
对腹泻病样品按采样点来源进行结果统计,结果显示,PEDV在无害化处理厂检出率高达30.00%,而TGEV和PoRV没有检出;另外,PEDV和PoRV在门诊部均有一定检出,检出率分别为17.60%和6.40%,TGEV检出率仅为1.60%(表 3)。
在全年所检样品中,PEDV和TGEV发生率均在寒冷的冬春季节最高,在盛夏季节发生率最低;而PoRV发生率在春夏、夏秋季节变换时最高,在盛夏和深冬发生率较低(图 2),表明在河北地区,春冬季节仍是病毒性腹泻病原多发季节,而夏季较少发生。
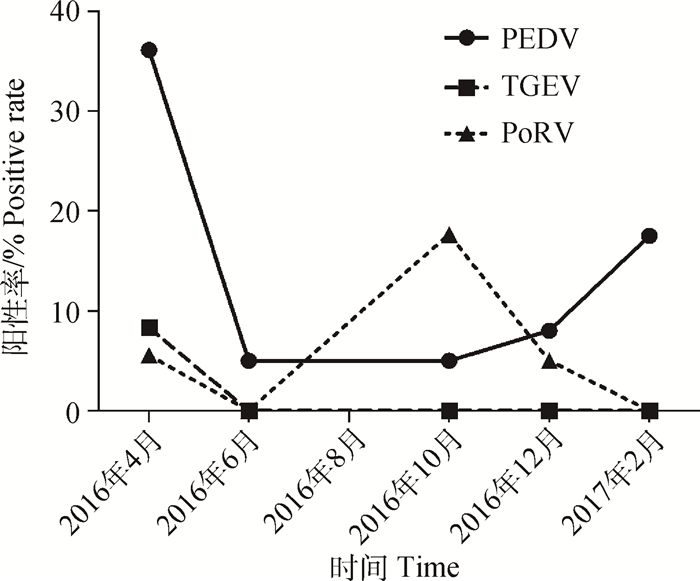
|
图 2 不同月份腹泻相关病原调查结果 Figure 2 Investigation of diarrhea related pathogen collected from different months |
在检出腹泻相关病原的样品中,PEDV单独感染率最高,达54.70%,PoRV单独感染率为22.11%,TGEV单独感染率为10.54%。在所检测具有腹泻相关病原的样品中,PEDV和TGEV共感染率为5.91%,PEDV和PoRV共感染率为6.17%,TGEV和PoRV共感染较少发生,共感染率仅为0.51%(图 3)。所检样品中未发现3种腹泻相关病原共感染情况。以上结果表明,PEDV引起的腹泻疾病主要以单独感染为主。
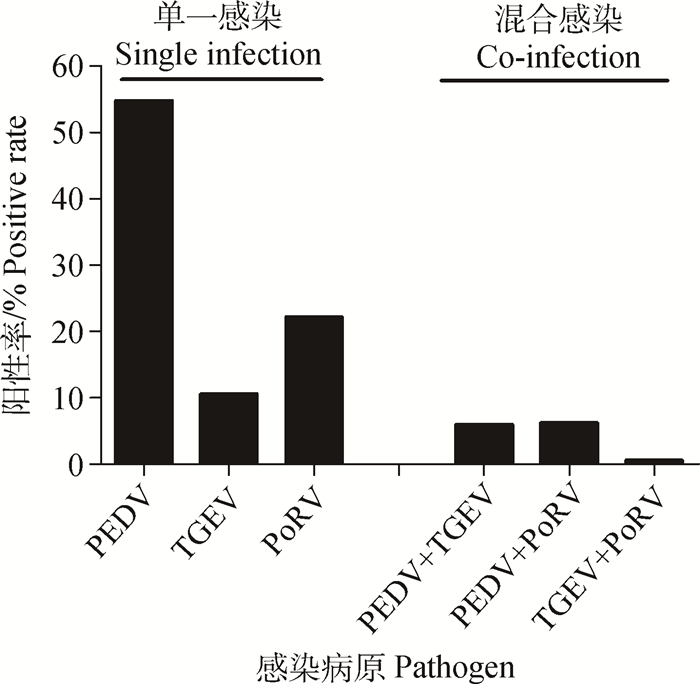
|
图 3 腹泻相关病原混合感染情况 Figure 3 Investigation of diarrhea related pathogen of single infection and co-infection |
S蛋白被认为是PEDV关键毒力蛋白,其遗传变异往往造成毒株毒力改变[12-13]。本研究选取来源不同的9份PEDV病料对S基因进行扩增并进行测序,得到9株PEDV S基因序列,并上传至GenBank(登录号:MG132631~MG132639)。经相似性比较分析,发现本研究所获得的9条S基因核苷酸序列相似性为96.8%~100.0%,推导氨基酸序列相似性为96.4%~100.0%。与GenBank上发表的33条国内外PEDV S基因推导氨基酸序列构建遗传进化树(图 4),发现进化树主要分为G1、G2和INDELs三支,本研究所获得的9条S基因全部位于G2上。G2这一大支又可以分为G2a和G2b小支,本研究所获得的9条S基因序列中,HB1601(MG132631)和HB1603(MG132632)株位于G2b分支,与美国流行毒株遗传关系较近;HB16123(MG132637)、HB16134(MG132638)和HB16135(MG132639)株位于G2b分支,与中国流行毒株遗传关系较近;HB1604(MG132633)和HB1605(MG132634)株以及HB1619(MG132635)和HB1622(MG132636)株位于G2a分支,与当前国内外流行毒株遗传关系较远。
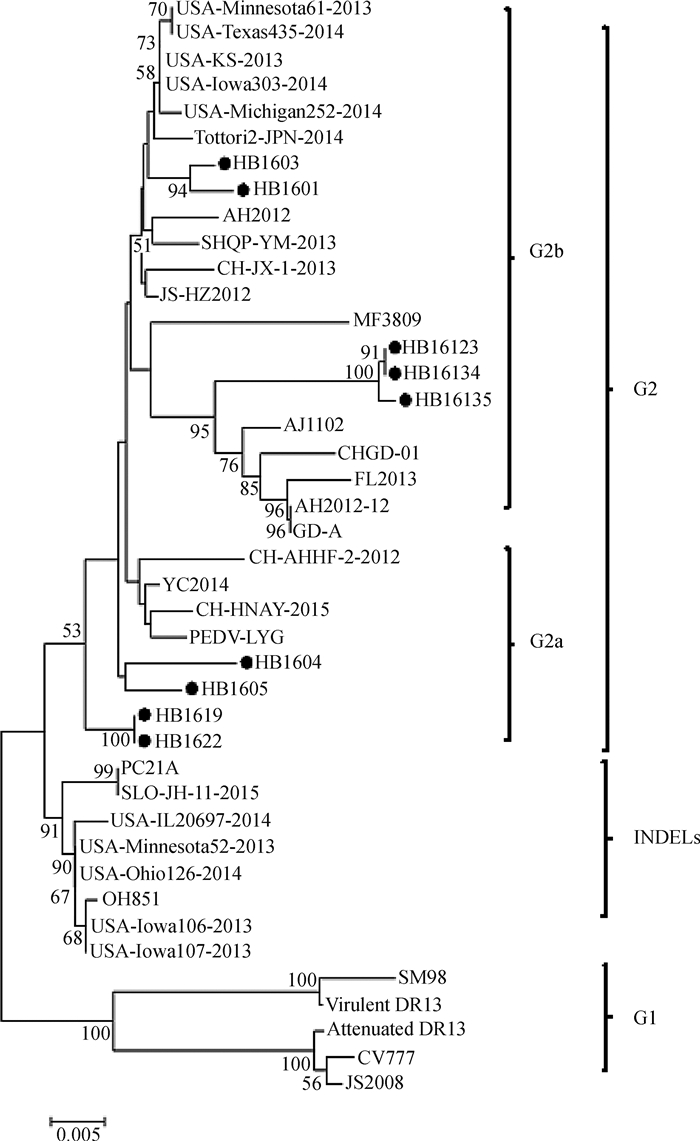
|
图 4 河北地区PEDV毒株S基因氨基酸序列遗传进化树 Figure 4 Phylogenetic analyses of PEDV strains based on the amino acid sequences of S gene |
将所得到的S基因与PEDV变异毒株AH2012进行氨基酸序列比对,发现所测9条S基因氨基酸序列均有多处突变。其中,处于G2b分支上的毒株突变位点较多,达38~39个;处于G2a分支上的毒株与CV777相似突变位点较多,达14个(表 4)。此外,HB1601和HB1603在382—383位氨基酸(TY)发生独特缺失,HB1619和HB1622株在234—236位有三个独特氨基酸插入(SAW)(表 4)。经NetNGlyc 1.0 Server软件预测AH2012株S蛋白有67处潜在糖基化位点,其中29处为NXT/S糖基化位点。对所获得的9条S基因氨基酸序列进行糖基化位点预测,发现多条S基因序列因氨基酸突变导致缺失或引入预测糖基化位点,其中HB1601株缺失一处预测NXT/S糖基化位点,HB16123、HB16134和HB16135株增加两处预测糖基化位点,HB1604和HB1605株分别增加一处预测糖基化位点(表 4)。与33株参考序列相比,所获得9条S基因氨基酸序列均发生多处独特氨基酸突变,其中HB1601、HB1603、HB1605、HB16123、HB16134和HB16135株在S蛋白核心抗原展示区域均有独特突变发生(表 4)。
|
|
表 4 河北省PEDV S基因突变位点分析 Table 4 Sequence analysis of mutant amino acid sites of PEDV S gene from Hebei province |
PEDV、TGEV和PoRV是引起猪腹泻病的主要病毒性病原,均以腹泻症状为主要临床特征,难以区分。河北地区自2010年开始暴发猪群腹泻病,在2011年和2012年疫情最为严重,2013年疫情趋于稳定[7, 14]。本研究对2016年2月至2017年4月河北地区送检临床样品进行发病情况统计和PEDV、TGEV和PoRV病原学检测。调查结果显示,当前河北省猪群腹泻病发生率较2010—2013年有明显缓解。不同于国内南方地区全年腹泻病均呈高发态势[6, 15-18],春冬季节为河北地区病毒性腹泻病多发季节。通过对腹泻病料调查,发现PEDV仍是腹泻病的主要病毒性病原,同时还发现少数地区正在流行PoRV,甚至个别地区PoRV是腹泻病的主要病原,这提示PoRV在河北省大面积流行的风险正在提高。
2011—2015年间,全国各地PEDV流行毒株以引起仔猪高发病率和高死亡率为主要特征,在保育猪和育肥猪中检出率较低[19-20]。本研究中,PEDV引起猪群腹泻病以单独感染为主,其在腹泻病样品中检出率较之2010—2013年均显著降低,但在育肥猪腹泻病料中有较高的检出率。母源抗体是保护猪群尤其仔猪抵抗PEDV感染的重要途径,然而其抗体水平往往受病毒感染滴度影响[21-22]。本研究中,河北地区育肥猪的高感染率提示成年猪分泌抗体水平较之2010—2013年可能显著提高,进而增强对猪群免疫保护能力,降低河北省猪群整体PEDV感染率。
PEDV S蛋白是病毒发生环境适应的关键蛋白,其遗传变异通常与病毒毒力相关,在分析流行毒株的流行趋势和遗传变异中具有重要的进化意义[23]。2010年来,全国多地报道PEDV流行毒株S蛋白与经典毒株变异较大,且分别进化形成多种不同分支[24-27]。Y. Y. Gao等通过对S蛋白遗传进化分析发现2010年后华北猪场中流行的PEDV毒株与韩国来源毒株进化关系较近[23]。本研究中,所获得9条PEDV S蛋白氨基酸序列同源性为96.4%~100.0%。遗传进化分析表明,河北省PEDV S蛋白分处于G2亚群中的2个不同进化分支,均与疫苗株CV777遗传关系较远。其中HB1601和HB1603株与美国分离株亲缘性较近,HB16123、HB16134和HB16135与国内其他地区分离株亲缘性较近,HB1604、HB1605株和HB1619、HB1622株分处的G2a分支则与当前国内外流行毒株遗传关系较远,表明,河北地区流行的PEDV优势毒株可能发生了改变,多种毒株并存。
PEDV S蛋白是Ⅰ型糖蛋白,有27~30个潜在的糖基化位点,这些糖基化位点往往与病毒毒力相关[13, 27]。其在S1区N端包含1个信号肽(l~18 aa),在S1和S2膜外域编码4个抗原表位(499—638、748—755、764—771和1 368—1 374 aa),其中499—638 aa为诱导中和抗体产生的核心区域,亦称为COE抗原区域[28-29]。T. Sato等将PEDV在细胞上连续传代致弱,发现S蛋白信号肽、S1和S2膜外区的氨基酸突变是造成毒株毒力减弱的关键因素[30]。本研究对9株PEDV S蛋白氨基酸序列分析,结果显示与国内流行毒株AH2012相比,9株S蛋白氨基酸序列均发生多处突变,部分突变位点与CV777疫苗株相同。同时,与33株参考序列相比,9株S蛋白氨基酸序列在S蛋白膜外区均发生多处独特性突变,表明PEDV S蛋白在河北地区的进化方向具有多样性,其是否影响S蛋白毒力和抗原性尚不得而知。其中,HB1601和HB1603在382—383 aa缺失两位氨基酸(TY),HB1619和HB1622在234—236 aa插入三位氨基酸突变位点(SAW),这一插入缺失特征目前尚未有报道。通过生物信息学软件分析发现,在HB1601、HB16123、HB16134、HB16135、HB1604和HB1605株中一些位点突变的发生可能会引起S蛋白糖基化位点失活或引入,推测这些位点突变有可能导致S蛋白毒力改变。此外,与33株参考序列相比,G2a分支和G2b分支S蛋白在COE区分别发生两处和三处独特性突变,这些突变可能会影响COE诱导中和抗体能力,改变毒株抗原性。综上,河北地区PEDV S基因进化分支较多,S蛋白的多种独特突变可能导致PEDV流行毒株毒力和抗原性改变,这一推测有待进一步研究证实。
4 结论2016年河北地区腹泻发生率较以往有所下降,PEDV对仔猪的感染情况有所缓解,而对育肥猪感染率较高。PEDV S基因氨基酸序列分析表明,河北省PEDV流行毒株分布在G2群中的2个进化分支,部分毒株发生独特氨基酸突变。
| [1] |
杨汉春. 2010年猪病流行情况与2011年流行趋势及防控对策[J]. 猪业科学, 2011, 28(1): 47–48.
YANG H C. The pig disease epidemic in 2010 and epidemic trend and prevention and control countermeasures in 2011[J]. Swine Industry Science, 2011, 28(1): 47–48. (in Chinese) |
| [2] | ZHANG B, TANG C, YUE H, et al. Viral metagenomics analysis demonstrates the diversity of viral flora in piglet diarrhoeic faeces in China[J]. J Gen Virol, 2014, 95(Pt 7): 1603–1611. |
| [3] | SUN R Q, CAI R J, CHEN Y Q, et al. Outbreak of porcine epidemic diarrhea in suckling piglets, China[J]. Emerg Infec Dis, 2012, 18(1): 161–163. DOI: 10.3201/eid1801.111259 |
| [4] |
王隆柏, 林裕胜, 车勇良, 等. 猪流行性腹泻病毒S、N和ORF3基因的遗传变异分析[J]. 畜牧兽医学报, 2014, 45(11): 1830–1836.
WANG L B, LIN Y S, CHE Y L, et al. Genetic variation analysis of S, N and ORF3 genes of porcine epidemic diarrhea virus[J]. Acta Veterinaria et Zootechnica Sinica, 2014, 45(11): 1830–1836. (in Chinese) |
| [5] |
于清磊. 2006-2015年甘肃省猪流行性腹泻流行病学分析[J]. 中国动物检疫, 2017, 34(4): 10–12.
YU Q L. Epidemiological analysis on Porcine Epidemic Diarrhea (PED) in Gansu province during 2006 to 2015[J]. China Animal Health Inspection, 2017, 34(4): 10–12. (in Chinese) |
| [6] |
赵振鹏, 杨振, 林伟东, 等. 江苏省猪流行性腹泻病毒的流行病学调查[J]. 安徽农业科学, 2015, 43(19): 125–127, 130.
ZHAO Z P, YANG Z, LIN W D, et al. Epidemiological investigation of porcine epidemic diarrhea disease in Jiangsu Province[J]. Journal of Anhui Agricultural Sciences, 2015, 43(19): 125–127, 130. DOI: 10.3969/j.issn.0517-6611.2015.19.047 (in Chinese) |
| [7] |
韩庆安, 张志, 李同山, 等. 2010-2013年河北省猪场腹泻病流行病学调查[J]. 中国动物检疫, 2015, 32(5): 5–8.
HAN Q A, ZHANG Z, LI T S, et al. Epidemiological survey of pig diarrhea in farms in Hebei Province during 2010-2013[J]. China Animal Health Inspection, 2015, 32(5): 5–8. (in Chinese) |
| [8] |
薛瑞雪, 田野, 田夫林, 等. 山东省部分地区仔猪病毒性腹泻流行病学调查[J]. 中国预防兽医学报, 2015, 37(4): 254–257.
XUE R X, TIAN Y, TIAN F L, et al. Epidemiological investigation of piglet diarrhea virus disease in Shandong province[J]. Chinese Journal of Preventive Veterinary Medicine, 2015, 37(4): 254–257. (in Chinese) |
| [9] |
赵梦姣, 陈书民, 成岩, 等. 山东省部分地区猪流行性腹泻流行病学调查及其M基因遗传变异分析[J]. 中国兽医学报, 2013, 33(10): 1504–1508.
ZHAO M J, CHEN S M, CHENG Y, et al. Epidemiological investigation of porcine epidemic diarrhea disease and phylogenetic analysis of M gene of PEDV in Shandong province[J]. Chinese Journal of Veterinary Science, 2013, 33(10): 1504–1508. (in Chinese) |
| [10] |
朱子健, 闫丽辉, 鞠妍, 等. 2015~2016年我国东北地区猪病毒性腹泻流行病学调查[J]. 中国预防兽医学报, 2017, 39(5): 356–360.
ZHU Z J, YAN L H, JU Y, et al. The epidemiological investigation on the viral diarrhea in pigs in the Northeast region of China during 2015-2016[J]. Chinese Journal of Preventive Veterinary Medicine, 2017, 39(5): 356–360. (in Chinese) |
| [11] |
王娟萍, 刘文俊, 米瑞娟, 等. 山西省猪流行性腹泻流行病学调查[J]. 畜牧与兽医, 2016, 48(9): 116–118.
WANG J P, LIU W J, MI R J, et al. Epidemiological investigation of porcine epidemic diarrhea in Shanxi Province[J]. Animal Husbandry & Veterinary Medicine, 2016, 48(9): 116–118. (in Chinese) |
| [12] | LAWRENCE P K, BUMGARDNER E, BEY R F, et al. Genome sequences of porcine epidemic diarrhea virus:in vivo and in vitro phenotypes[J]. Genome Announc, 2014, 2(3): e00503-14. DOI: 10.1128/genomeA.00503-14 |
| [13] | DUARTE M, LAUDE H. Sequence of the spike protein of the porcine epidemic diarrhoea virus[J]. J Gen Virol, 1994, 75(Pt 5): 1195–1200. |
| [14] |
王子龙, 韩庆安, 王玉清, 等. 河北省猪病毒性腹泻流行病学调查[J]. 黑龙江畜牧兽医, 2014, 5(5): 59–60.
WANG Z L, HAN Q A, WANG Y Q, et al. Porcine viral diarrhea epidemiological investigation in Hebei province[J]. Heilongjiang Animal Science and Veterinary Medicine, 2014, 5(5): 59–60. (in Chinese) |
| [15] |
阳酉萍, 黄小波, 曹三杰, 等. 四川部分地区猪流行性腹泻的分子流行病学调查[J]. 中国兽医学报, 2015, 35(5): 704–710.
YANG Y P, HUANG X B, CAO S J, et al. Molecular epidemiological investigation porcine epidemic diarrhea in some areas of Sichuan[J]. Chinese Journal of Veterinary Science, 2015, 35(5): 704–710. (in Chinese) |
| [16] |
李智丽, 黄淑坚, 马静云, 等. 2011-2015年我国南方地区PEDV分子流行病学调查及S基因遗传变异分析[J]. 广东农业科学, 2016, 43(11): 127–135.
LI Z L, HUANG S J, MA J Y, et al. Molecular epidemiological investigation of PEDV and analysis of genetic variation of S gene in South China during 2011-2015[J]. Guangdong Agricultural Sciences, 2016, 43(11): 127–135. (in Chinese) |
| [17] |
胡兴义, 张双翔, 金志强, 等. 贵州地区仔猪病毒性腹泻流行病学调查[J]. 中国预防兽医学报, 2016, 38(7): 542–545.
HU X Y, ZHANG S X, JIN Z Q, et al. The epidemiological investigation on viral diarrhea of piglets in Guizhou region[J]. Chinese Journal of Preventive Veterinary Medicine, 2016, 38(7): 542–545. (in Chinese) |
| [18] |
白小磊, 崔进, 崔甜甜, 等. 广东部分地区猪流行性腹泻病毒流行病学调查[J]. 中国兽医学报, 2016, 36(11): 1823–1828, 1863.
BAI X L, CUI J, CUI T T, et al. Molecular characterization and epidemiological survey of porcine epidemic diarrhea virus in some areas of Guangdong[J]. Chinese Journal of Veterinary Science, 2016, 36(11): 1823–1828, 1863. (in Chinese) |
| [19] |
林耀民, 宋万杰, 秦云, 等. 猪流行性腹泻的流行现状及防控策略[J]. 中国动物检疫, 2016, 33(5): 62–64.
LIN Y M, SONG W J, QIN Y, et al. The prevalence and prevention strategies of PEDV[J]. China Animal Health Inspection, 2016, 33(5): 62–64. (in Chinese) |
| [20] |
刘孝珍, 陈建飞, 时洪艳, 等. 2011年猪流行性腹泻病毒的遗传变异分析[J]. 中国预防兽医学报, 2012, 34(3): 180–183.
LIU X Z, CHEN J F, SHI H Y, et al. Genetic variation analysis of porcine epidemic diarrhea virus isolated in 2011[J]. Chinese Journal of Preventive Veterinary Medicine, 2012, 34(3): 180–183. (in Chinese) |
| [21] | PENSAERT M B, MARTELLI P. Porcine epidemic diarrhea:a retrospect from Europe and matters of debate[J]. Virus Res, 2016, 226: 1–6. DOI: 10.1016/j.virusres.2016.05.030 |
| [22] | POONSUK K, GIMÉNEZ-LIROLA L G, ZHANG J Q, et al. Does circulating antibody play a role in the protection of piglets against porcine epidemic diarrhea virus?[J]. PLoS One, 2016, 11(4): e0153041. DOI: 10.1371/journal.pone.0153041 |
| [23] | GAO Y Y, KOU Q W, GE X N, et al. Phylogenetic analysis of porcine epidemic diarrhea virus field strains prevailing recently in China[J]. Arch Virol, 2013, 158(3): 711–715. DOI: 10.1007/s00705-012-1541-2 |
| [24] | CHEN F Z, KU X G, LI Z H, et al. Genetic characteristics of porcine epidemic diarrhea virus in Chinese mainland, revealing genetic markers of classical and variant virulent parental/attenuated strains[J]. Gene, 2016, 588(1): 95–102. DOI: 10.1016/j.gene.2016.05.011 |
| [25] | SU Y F, LIU Y C, CHEN Y M, et al. Detection and phylogenetic analysis of porcine epidemic diarrhea virus in central China based on the ORF3 gene and the S1 gene[J]. Virol J, 2016, 13: 192. DOI: 10.1186/s12985-016-0646-8 |
| [26] | WANG E Y, GUO D H, LI C Q, et al. Molecular characterization of the ORF3 and S1 genes of porcine epidemic diarrhea virus Non S-INDEL strains in seven regions of China, 2015[J]. PLoS One, 2016, 11(8): e0160561. DOI: 10.1371/journal.pone.0160561 |
| [27] | FAN B C, JIAO D, ZHAO X N, et al. Characterization of Chinese porcine epidemic diarrhea virus with novel insertions and deletions in genome[J]. Sci Rep, 2017, 7: 44209. DOI: 10.1038/srep44209 |
| [28] | PARK S J, MOON H J, YANG J S, et al. Sequence analysis of the partial spike glycoprotein gene of porcine epidemic diarrhea viruses isolated in Korea[J]. Virus Genes, 2007, 35(2): 321–332. DOI: 10.1007/s11262-007-0096-x |
| [29] | CHANG S H, BAE J L, KANG T J, et al. Identification of the epitope region capable of inducing neutralizing antibodies against the porcine epidemic diarrhea virus[J]. Mol Cells, 2002, 14(2): 295–299. |
| [30] | SATO T, TAKEYAMA N, KATSUMATA A, et al. Mutations in the spike gene of porcine epidemic diarrhea virus associated with growth adaptation in vitro and attenuation of virulence in vivo[J]. Virus Genes, 2011, 43(1): 72–78. DOI: 10.1007/s11262-011-0617-5 |



