研究表明,鹅可以利用饲料中的粗纤维,添加适宜的粗纤维不仅可提高鹅生产性能,而且可以促进鹅消化道的发育以及维持肠道的健康[1-2]。鹅利用粗纤维的特性与消化道及肠道菌群结构有密切关系[3]。盲肠是消化利用纤维的重要场所[4],鹅具有发达的盲肠,其内容物pH和排空时间为微生物的定植创造了较好的条件[5],与其他肠段相比,盲肠菌种种类最为丰富[6]。盲肠微生物在鹅营养消化吸收、黏膜代谢、免疫应答等生长发育过程中具有重要作用[4, 7]。赫忠睿[8]、战利[9]、刘蓓一[3]研究了不同日粮纤维水平对鹅肠道内菌群多样性和丰度的影响,结果表明,提高日粮纤维水平可影响盲肠微生物多样性,而且可改变部分细菌的丰度。但目前鹅肠道微生物的研究方法多为体外培养、PCR-DGGE等分子生物学技术,而自然界中超过99%的微生物是不能被培养的[10],目前对鹅肠道细菌的研究普遍只能检测到较少部分细菌[11]。随着高通量测序策略技术的发展与进步,无需对细菌进行培养即可实现细菌的鉴定与分类,而且测序深度不断提高、读长不断增长,大大提高了测序结果的准确性[12]。Illumina公司开发的MiSeq测序平台有效解决了通量低、操作复杂等问题,目前已广泛应用于微生物群落多样性研究[13-15]。
本研究利用MiSeq PE300测序平台,采用双末端测序方法,通过OTUs聚类以及物种注释和丰度分析,揭示鹅盲肠黏膜微生态的特点,全面深入比较饲喂不同纤维水平日粮鹅肠道黏膜菌群结构的差异,为揭示鹅消化利用日粮纤维的机制提供参考。
1 材料与方法 1.1 试验动物及试验设计选取体重相近的35日龄健康卡洛斯公鹅90只,随机分为2组,每组3个重复,每个重复15只,试验期为42 d。参考NRC(1994),以苜蓿为纤维源,配制成粗纤维5%和8%的2个水平,5%日粮纤维水平记为L组、8%日粮纤维水平记为H组,日粮组成及营养水平如表 1所示。
|
|
表 1 日粮组成及营养水平 Table 1 Compositions and nutrient levels of diets |
试验在吉林农业大学动物科学技术学院养鹅基地进行,采用半开放式鹅舍,地面圈养,自由采食与饮水。
1.3 样品采集试验结束后,在3个重复中分别随机选取1只卡洛斯鹅进行屠宰,迅速取出盲肠并进行结扎,经75%酒精擦拭消毒后转移至无菌超净台内,将盲肠剪开,用无菌生理盐水冲洗肠道内壁,冲洗干净后,用灭菌手术刀片轻轻刮取肠道黏膜,收集于冻存管中,-80 ℃保存备用。
1.4 鹅盲肠黏膜细菌DNA提取与文库测序鹅盲肠黏膜细菌总DNA提取按照QIAamp DNA Mini kit步骤进行。细菌DNA经琼脂糖凝胶电泳检测后,采用通用引物515F(5′-GTGCCAGCMGCCGCGGTAA-3′)和806R(5′-GGACTACHVGGGTWTCTAAT-3′)对16S rDNA的V4区进行扩增。PCR产物纯化回收后采用NEB Next® UltraTM DNA Library Prep Kit for Illumina进行文库的构建,使用MiSeq PE300上机测序。
1.5 测序结果分析测序平台得到的原始数据经过数据拆分、PE Reads拼接[16]、Tags过滤[17]和去嵌合体序列后得到有效Tags[18]。采用Uparse软件(Uparse v7.0.1001,http://drive5.com/uparse/)[19]将样本有效Tags聚类成不同的操作分类单元(OTUs),并进行物种注释和样品复杂度分析。
1.6 数据处理采用SPSS16.0软件以及Metastats方法对各处理组间差异性进行分析。
2 结果 2.1 鹅盲肠黏膜细菌测序数据质量评价及OTU分析本研究对盲肠黏膜细菌样本进行了测序,经过数据的拆分、拼接、过滤和去嵌合体等一系列处理后,共得到有效序列(Effective tags)数为297 023条,平均每个样本Effective tags数为49 504条,Effective tags的平均长度为253 nt,错误率小于1%的碱基百分比为98.59%,错误率小于0.1%的碱基百分比为97.25%,测序数据正确可靠。
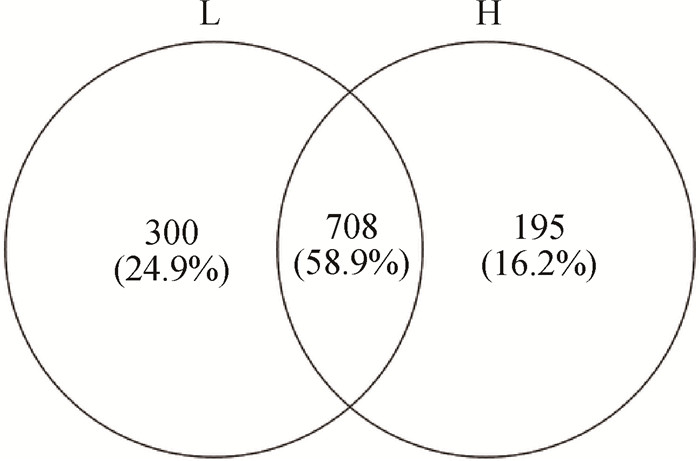
以97%相似度为聚类阈值,8%纤维水平组(H组)、5%纤维水平组(L组)盲肠样本OTUs分别为1 008和903个,两组共有OTUs为708个,如图 1所示。
|
图 1 不同处理组鹅盲肠细菌OTU韦恩图 Figure 1 Venn diagrams for OTUs of different groups in cecum of goose |
在97%相似度水平下,对两组处理的Chao1和Shannon进行显著性分析(表 2),均无显著性差异(P>0.05),表明两个处理组菌群多样性无显著性差异。
|
|
表 2 鹅盲肠样品细菌Alpha多样性 Table 2 Bacteria Alpha indexes in cecum samples of goose |
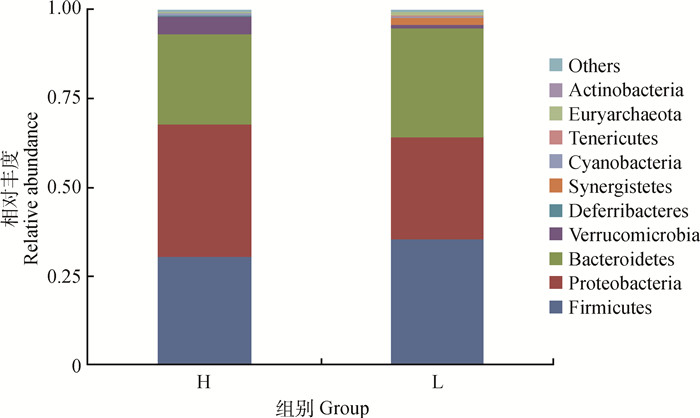
研究将相对丰度含量大于0.1%的菌门视为优势菌门,H和L优势菌门的数量分别为9和10。门水平上相对丰度排名前10的门丰度分布见图 2所示。相对丰度排名前10的门依次分别是Firmicutes(厚壁菌门)、Proteobacteria(变形菌门)、Bacteroidetes(拟杆菌门)、Verrucomicrobia(疣微菌门)、Deferribacteres(脱铁杆菌门)、Synergistetes(互养菌门)、Cyanobacteria(蓝细菌门)、Tenericutes(软壁菌门)、Euryarchaeota(广古菌门)、Actinobacteria(放线菌门),其中,H组相对丰度含量较高的前4菌门分别为Proteobacteria(37.15%)、Firmicutes(30.60%)、Bacteroidetes(25.58%)、Verrucomicrobia(4.86%),占总丰度的98%以上;L组中丰度含量较高的前4的菌门为Firmicutes(35.38%)、Bacteroidetes(30.97%)、Proteobacteria(28.57%)、Synergistetes(1.72%),占总丰度的96%以上。
|
图 2 各样本在门水平上的物种相对丰度 Figure 2 Relative abundance of species of samples at phylum level |
对H组和L组的优势菌门相对丰度进行差异显著性分析,其中有显著性差异的菌门如表 3所示。H组Verrucomicrobia、Deferribacteres相对丰度显著高于L(P<0.05),Synergistetes、Euryarchaeota则显著低于L组(P<0.05)。
|
|
表 3 在门水平上鹅盲肠菌群不同纤维组差异性分析 Table 3 Difference analysis of goose cecum microflora at phylum level between the two groups |
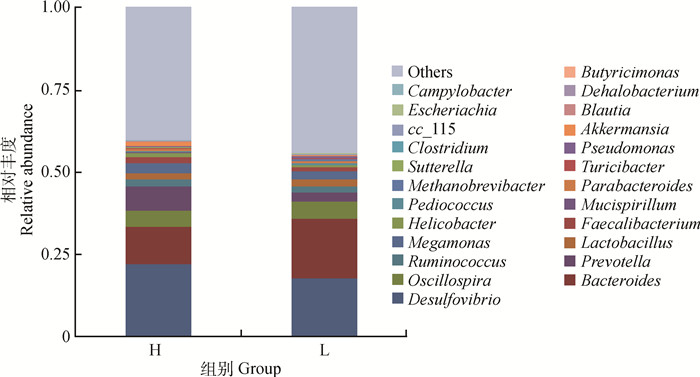
在属水平上,H组检测出的细菌属于17门类的199个属,相对丰度含量大于0.1%的属有22个,占细菌总丰度的59%;L组检测出的细菌属于19门类的226个属,相对丰度含量大于0.1%的属有24个,占细菌总丰度的55%。将相对丰度含量大于0.1%的菌属视为优势菌属,相对丰度柱形图见图 3。H组部分优势菌属的相对丰度:Desulfovibrio(22.02%)、Bacteroides(11.24%)、Prevotella(7.48%)、Oscillospira(4.87%)、Megamonas(3.18%)、Ruminococcus(2.11%)、Lactobacillus(1.78%)、Faecalibacterium(1.62%)、Akkermansia(1.29%)、Helicobacter(1.24%);L组部分优势菌属的相对丰度:Bacteroides(17.83%)、Desulfovibrio(17.80%)、Oscillospira(5.41%)、Prevotella(2.56%)、Megamonas(2.41%)、Lactobacillus(2.56%)、Ruminococcus(1.97%)、Faecalibacterium(1.07%)。
|
图 3 各样本在属水平上的物种相对丰度 Figure 3 Relative abundance of spieces of samples at genus level |
为了比较各处理组在属水平上菌群构成的差异,分别对各处理组优势菌属进行差异显著性分析,其中有显著性差异的菌属如表 4所示。H组Desulfovibrio、Prevotella、Helicobacter、Mucispirillum和Akkermansia菌属相对丰度显著高于L组(P<0.05);L组Bacteroides、Methanobrevibacter菌属相对丰度显著高于H组(P<0.05)。
|
|
表 4 属水平上盲肠菌群不同纤维组差异性分析 Table 4 Difference analysis of goose cecum microflora at genus level between the two groups |
鹅具有发达的盲肠[20, 5],与其他肠段相比,盲肠菌种种类最为丰富[6]。由于肠道内容物流动性等原因,内容物细菌多为过路菌,肠道黏膜细菌才是定植细菌的主体[21]。以细菌16S rRNA为基础的分子生物学技术由于其快速、准确、灵敏等优势,在细菌菌株的鉴定中得到了广泛的应用[22]。本研究采用Illumina MiSeq高通量测序技术,对盲肠黏膜细菌进行了分析,8%纤维水平组检测出的细菌属于17门类的199个属,5%纤维水平组检测出的细菌属于19门类的226个属,与以往鹅盲肠微生态研究结果相比,细菌的多样性明显提高。张名爱等[23]研究指出,鹅盲肠内优势菌群为双歧杆菌、梭菌和乳酸杆菌,而本研究中,Desulfovibrio(脱硫弧菌)、Bacteroides(拟杆菌)、Prevotella(普氏菌)、Oscillospira(颤螺旋菌)、Megamonas(巨单胞菌)、Ruminococcus(瘤胃球菌属)、Lactobacillus(乳酸菌)、Faecalibacterium、Akkermansia、Helicobacter(螺杆菌)均为相对丰度较高的优势菌属,与张名爱等[23]等研究结果不一致,一方面可能是由于鹅肠道黏膜和食糜中微生态存在一定的差异,另一方面可能是由于高通量技术具有无需分离培养菌群、客观还原菌群结构和测序深度高等优势。
动物对饲料中营养成分的消化吸收在一定程度上取决于肠道内微生物菌群的分布与数量,日粮组成对肠道微生物菌群结构具有一定的影响[5, 24]。A.Awati等[25]研究指出,动物肠道优势菌群受日粮组成的影响,且日粮纤维是影响优势菌群的重要因素。赫忠睿[8]、战利[9]研究指出,提高日粮纤维水平影响了鹅肠道内菌群的多样性和丰度。本试验发现,提高日粮纤维水平对细菌多样性无显著影响,但可显著影响部分细菌的丰度。提高日粮纤维水平可导致纤维分解菌以及在纤维降解中具有供能等作用的菌属丰度的增加。随着日粮纤维水平的提高,Verrucomicrobia、Deferribacteres菌门和Desulfovibrio、Prevotella、Helicobacter、Mucispirillum、Akkermansia菌属相对丰度显著增加;Synergistetes、Euryarchaeota菌门和Bacteroides、Methanobrevibacter菌属相对丰度显著降低。Desulfovibrio被证实在纤维降解中具有重要作用[26],Prevotella菌属含有可降解纤维的细菌[27]。Bacteroides是中温厌氧纤维素降解细菌,随日粮纤维水平升高而降低,机理需要进一步研究。本研究中,8%纤维水平组Akkermansia相对丰度显著高于5%纤维水平组,Akkermansia菌具有改善糖代谢和降低盲肠脂肪组织炎症的作用[10],可能与8%纤维日粮造成的盲肠炎症反应有关。
由于纤维水平不同导致鹅肠道菌群多样性和丰度差异的菌群并不一定直接参与肠道内纤维的分解。战利[9]研究发现,饲喂8.08%纤维水平日粮吉林白鹅肠道内容物埃希菌、粪肠球菌明显高于3.71%纤维水平组,但是这两组菌株均不能直接分解纤维素。单个菌株的纤维分解能力有限,甚至在体外试验中丧失纤维分解能力。另外,鹅肠道中除细菌外,还含有大量的真菌、病毒等微生物,其中部分具有纤维分解能力。综上,鹅肠道内纤维素的分解是一个复杂的过程,是多种微生物共同作用的结果,肠道内真菌、病毒等微生物对纤维的降解尚需深入研究。
4 结论5%和8%日粮纤维水平对卡洛斯鹅盲肠细菌Alpha多样性无显著影响,但可显著影响Verrucomicrobia、Deferribacteres、Synergistetes、Euryarchaeota菌门以及Desulfovibrio、Prevotella、Helicobacter、Mucispirillum、Akkermansia、Bacteroides、Methanobrevibacter菌属的相对丰度。
| [1] |
陈晓霞, 杨连玉. 日粮纤维构成对鹅营养物质利用的影响[J]. 黑龙江畜牧兽医, 2017(18): 59–60.
CHEN X X, YANG L Y. Effects of dietary fiber composition on the utilization of goose nutrition[J]. Heilongjiang Animal Science and Veterinary Medicine, 2017(18): 59–60. (in Chinese) |
| [2] |
杨艳红, 龙向华, 吕景智, 等. 饲粮纤维对单胃动物肠道的影响[J]. 中国畜牧杂志, 2015, 51(5): 81–85.
YANG Y H, LONG X H, LÜ J Z, et al. Effect of dietary fiber on intestines of monogastric animals[J]. Chinese Journal of Animal Science, 2015, 51(5): 81–85. (in Chinese) |
| [3] |
刘蓓一. 扬州鹅肠道微生物多样性及其受饲粮纤维水平的调控研究[D]. 扬州: 扬州大学, 2012.
LIU B Y. Molecular diversity of microoanisms in the gut of Yangzhou geese and its consequences of dietary fiber level variety[D]. Yangzhou: Yangzhou University, 2012. (in Chinese) |
| [4] |
黄萌, 刘国君, 郭镇华, 等. 鹅消化道内微生态的研究进展[J]. 黑龙江畜牧兽医, 2016(7): 78–80.
HUANG M, LIU G J, GUO Z H, et al. Research progress of goose gastrointestinal microbiota[J]. Heilongjiang Animal Science and Veterinary Medicine, 2016(7): 78–80. (in Chinese) |
| [5] | KATO S, HARUTA S, CUI Z J, et al. Stable coexistence of five bacterial strains as a cellulose-degrading community[J]. Appl Environ Microbiol, 2005, 71(11): 7099–7016. DOI: 10.1128/AEM.71.11.7099-7106.2005 |
| [6] |
胡平, 施寿荣, 王志跃, 等. 采用变性梯度凝胶电泳技术研究不同形态玉米日粮对鹅肠道微生物区系的影响[J]. 动物营养学报, 2010, 22(1): 169–175.
HU P, SHI S R, WANG Z Y, et al. Effects of different corn diet forms on intestinal microflora of geese by denaturing gradient gel electrophoresis[J]. Chinese Journal of Animal Nutrition, 2010, 22(1): 169–175. (in Chinese) |
| [7] |
马凤莲. 饲喂不同纤维分解菌对鹅肠道菌群多样性的影响[D]. 长春: 吉林农业大学, 2012.
MA F L. Feeding different cellulolytic bacteria impact on intestinal microflora of geese[D]. Changchun: Jilin Agricultural University, 2012. (in Chinese) |
| [8] |
赫忠睿. 日粮纤维对吉林白鹅消化功能及微生物多样性的影响[D]. 长春: 吉林农业大学, 2015.
HE Z R. Effects of dietary fiber on digestive function and microbial diversity of Jilin goose[D]. Changchun: Jilin Agricultural University, 2015. (in Chinese) |
| [9] |
战利. 高水平日粮纤维对鹅肠道菌群多样性的影响及部分差异菌株的确定[D]. 长春: 吉林农业大学, 2012.
ZHAN L. Influence of high levels of dietary fiber on the goose road flora diversity and some difference strains were ditermined[D]. Changchun: Jilin Agricultural University, 2012. (in Chinese) |
| [10] | SHIN N R, LEE J C, LEE H Y, et al. An increase in the Akkermansia spp. population induced by metformin treatment improves glucose homeostasis in diet-induced obese mice[J]. Gut, 2014, 63(5): 727–735. DOI: 10.1136/gutjnl-2012-303839 |
| [11] | MUYZER G, SMALLA K. Application of denaturing gradient gel electrophoresis (DGGE) and temperature gradient gel electrophoresis (TGGE) in microbial ecology[J]. Antonie Van Leeuwenhoek, 1998, 73(1): 127–141. DOI: 10.1023/A:1000669317571 |
| [12] | LUO C W, TSEMENTZI D, KYRPIDES N, et al. Direct comparisons of Illumina vs. Roche 454 sequencing technologies on the same microbial community DNA sample[J]. PLoS One, 2012, 7(2): e30087. DOI: 10.1371/journal.pone.0030087 |
| [13] | DEGNAN P H, OCHMAN H. Illumina-based analysis of microbial community diversity[J]. ISME J, 2012, 6(1): 183–194. DOI: 10.1038/ismej.2011.74 |
| [14] | POŁKA J, REBECCHI A, PISACANE V, et al. Bacterial diversity in typical Italian salami at different ripening stages as revealed by high-throughput sequencing of 16S rRNA amplicons[J]. Food Microbiol, 2015, 46: 342–356. DOI: 10.1016/j.fm.2014.08.023 |
| [15] |
汪文强, 赵生国, 马利青, 等. 动物基因组学重测序的应用研究进展[J]. 畜牧兽医学报, 2016, 47(10): 1947–1953.
WANG W Q, ZHAO S G, MA L Q, et al. The research progress and application of resequencing based on animal genomics[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(10): 1947–1953. (in Chinese) |
| [16] | MAGOĈ T, SALZBERG S L. FLASH:fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics, 2011, 27(21): 2957–2963. DOI: 10.1093/bioinformatics/btr507 |
| [17] | CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al. QⅡME allows analysis of high-throughput community sequencing data[J]. Nat Methods, 2010, 7(5): 335–336. DOI: 10.1038/nmeth.f.303 |
| [18] |
李红梅, 白林, 姜冬梅, 等. 基于16SrDNA高通量测序方法检测猪舍空气微生物多样性[J]. 中国畜牧杂志, 2015, 51(3): 81–84.
LI H M, BAI L, JIANG D M, et al. Microbial diversity of piggery air detected by 16S rDNA high-throughput sequencing[J]. Chinese Journal of Animal Science, 2015, 51(3): 81–84. (in Chinese) |
| [19] | EDGAR R C. UPARSE:highly accurate OTU sequences from microbial amplicon reads[J]. Nat Methods, 2013, 10(10): 996–998. DOI: 10.1038/nmeth.2604 |
| [20] |
王晔, 于辉. 日粮粗纤维水平对鹅肠道发育和免疫器官指数的影响[J]. 黑龙江畜牧兽医, 2015(18): 83–84.
WANG Y, YU H. Effects of dietary crude fiber level on intestinal development and immune organ index in goose[J]. Heilongjiang Animal Science and Veterinary Medicine, 2015(18): 83–84. (in Chinese) |
| [21] |
周映华, 胡新旭, 卞巧, 等. 无抗发酵饲料对生长育肥猪生长性能、肠道菌群和养分表观消化率的影响[J]. 动物营养学报, 2015, 27(3): 870–877.
ZHOU Y H, HU X X, BIAN Q, et al. Effects of fermented feed without antibiotic on growth performance, intestinal flora and nutrient apparent digestibility of growing-finishing pigs[J]. Chinese Journal of Animal Nutrition, 2015, 27(3): 870–877. (in Chinese) |
| [22] |
孙翠丽, 张阁, 程汝佳, 等. 16S rRNA高通量测序方法检测羊圈空气微生物群落结构及多样性[J]. 畜牧兽医学报, 2017, 48(7): 1314–1322.
SUN C L, ZHANG G, CHENG R J, et al. Microbial community structure and diversity of sheepfold atmosphere by 16S rRNA high-throughput sequencing[J]. Acta Veterinaria et Zootechnica Sinica, 2017, 48(7): 1314–1322. (in Chinese) |
| [23] |
张名爱, 王宝维, 龙芳羽, 等. 日粮纤维水平对鹅肠道正常菌群的影响[J]. 福建农林大学学报:自然科学版, 2007, 36(2): 159–162.
ZHANG M A, WANG B W, LONG F Y, et al. Effect of different fibre level diets on normal microbiological floras in goose intestines[J]. Journal of Fujian Agriculture and Forestry University:Natural Science Edition, 2007, 36(2): 159–162. (in Chinese) |
| [24] | GUO P, MOCHIDZUKI K, CHENG W, et al. Effects of different pretreatment strategies on corn stalk acidogenic fermentation using a microbial consortium[J]. Bioresour Technol, 2011, 102(16): 7526–7531. DOI: 10.1016/j.biortech.2011.04.083 |
| [25] | AWATI A, KONSTANTINOV S R, WILLIAMS B A, et al. Effect of substrate adaptation on the microbial fermentation and microbial composition of faecal microbiota of weaning piglets studied in vitro[J]. J Sci Food Agr, 2005, 85(10): 1765–1772. DOI: 10.1002/(ISSN)1097-0010 |
| [26] |
王佳婧. 厌氧复合菌系的筛选及含纤维素原料预处理复合菌剂的研究[D]. 成都: 中国科学院成都生物研究所, 2009.
WANG J J. Studies on screening anerobic microbial consortia and pretreatment of cellulose raw material by microbial agent[D]. Chengdu: Chengdu Institute of Biology, Chinese Academy of Sciences, 2009. (in Chinese) |
| [27] |
李志鹏. 梅花鹿瘤胃微生物多样性与优势菌群分析[D]. 北京: 中国农业科学院, 2013.
LI Z P. The analysis of microorganism diversity and dominant bacterial communities in the rumen of sika deer (Cervus nippon)[D]. Beijing: Chinese Academy of Agricultural Sciences, 2013. (in Chinese) |



