沙门菌污染是一个全球关注的重要问题,对人类的威胁已超过数百年。据估计,全球每年发生的沙门菌感染病例超过9 000万例,导致死亡人数达15.5万人[1]。而中国发生的细菌性食物中毒中,有70%~80%致病菌是沙门菌[2]。在美国,沙门菌是导致食源性感染的第二大病原菌[3]。动物饲料作为沙门菌的重要传染源受到广泛关注[4]。动物饲料产业链中从原料生产开始各个阶段均有较高沙门菌污染的风险。为控制饲料源致病菌的传播,抗生素自1950年美国食品和药品管理局(FDA)首次被批准用作饲料添加剂,半个多世纪以来一直被大量添加于养殖动物饲料中[5]。不可否认,抗生素对畜牧业起到十分积极的作用。但是,近年来,抗生素出现越来越多的负面效应,首当其冲就是耐药菌株的不断出现[6-7],抗生素被逐步替代的呼声越来越高。在2014年农业部2045号公告中,国家将甲酸、乙酸和柠檬酸等39种酸认定为安全的饲料添加剂品种,可应用于所有养殖动物饲料中,因此酸已成为替代抗生素的重要饲料添加剂组成部分[8]。
随着酸的使用越来越广泛,酸是否会如抗生素一样出现越来越多的耐酸菌株,答案是肯定的。致病菌在应对酸性压力环境时,为增加生存能力需要诱导酸耐受反应(acid tolerance response, ATR)。ATR是细菌在应对低pH值时产生的耐受[9-10],研究表明沙门菌暴露在较高的酸性环境中时就会产生ATR[11]。同时,也有证据表明,ATR可能会增加细菌的毒力[12],而细菌在不同生长阶段的ATR具有不同特征[13-14],在对数期和稳定期产生不同反应,需要酸休克蛋白全程合成[15-16]。ATR还可以使细菌对热、盐、乙醇等压力产生交叉耐受[17-18]。研究表明,沙门菌ATR可分为对数期ATR和稳定期ATR,对数期ATR(LP ATR)可以进一步分为依赖于fur的短暂耐酸系统和仅依靠rpoS的更持久有效的ATR[19]。同样,稳定期的鼠伤寒沙门菌具有两个独立调节的稳定期酸耐受系统。与pH无关的rpoS负责的酸耐受,以及依赖酸诱导的稳定期ATR,与ompR相关[14]。本研究选取与对数期ATR相关的fur基因及与稳定期ATR相关的ompR基因研究沙门菌的耐酸性。同时,N. Botteldoorn等[20-21]对比沙门菌rpoD、16S rRNA和gmK 3个管家基因表达的稳定性,结果表明,gmK是表达最稳定的管家基因。因此,本研究采用实时荧光定量PCR技术,以gmK作为管家基因,选取与对数期ATR相关的fur基因及与稳定期ATR相关的ompR基因研究沙门菌的耐酸性。并且,目前大部分对于ATR的研究是使用无机酸(如盐酸)做酸化剂在30或37 ℃条件下进行[15, 22],而在实际环境中,食源性致病菌更常暴露于20 ℃或者更低的温度环境下以及弱有机酸(如乙酸)环境下。同样,饲料的储存条件一般也更常暴露于20 ℃或者更低的温度环境下,并且随着酸的使用越来越广泛,饲料所带的致病菌在酸性环境产生ATR在预测模型和风险评估方面,对微生物安全具有重要意义。
响应面分析法(response surface methodology, RSM)是设计试验、建立模型和优化条件的最有效的方法之一,受到几个独立变量的影响[23-25]。与传统方法相比,响应面分析法不仅可以明确独立变量对试验的影响,确定最优的试验操作条件,而且可以有效的评估各变量之间的相互作用,以实现最佳的试验性能[26-28]。因此,本研究旨在单因素试验基础上,采用响应面分析法创造最优条件评估沙门菌在乙酸创造的酸性压力条件下的行为,评价它们存活率的变化和参与酸耐受基因的转录变化。同时,将优化后的模型应用于其他几种常用有机酸上,以此验证该模型对于其他几种常用酸所创造的诱导模型效果。
1 材料与方法 1.1 材料 1.1.1 菌种本试验所用细菌为肠炎沙门菌ATCC13076(福建省出入境检验检疫局检验检疫技术中心提供)。
1.1.2 试剂及溶液脑心浸液肉汤(BHI)(广东环凯生物科技有限公司)、冰乙酸(国药集团化学试剂有限公司)、TE缓冲液(北京索莱宝科技有限公司)、琼脂糖凝胶(Biowest Agarose)、SUPER Green I核酸染料(PCR级)(北京泛博生物化学有限公司)、Premix TaqTM (TaKaRa TaqTM Version 2.0)、PrimeScript RT reagent Kit with gDNA Eras、Easy Dilutio、SYBR® Premix Ex TaqTM (Tli RNaseH Plus)、DL1000 Marker、6×LoadingBuffer、RNAiso Plus(大连宝生物工程公司)、氯仿、无水乙醇、异丙醇(国药集团化学试剂有限公司)、DEPC-treated water(上海生工生物工程公司)、5×TAE缓冲液(北京索莱宝科技有限公司)。
1.1.3 设备单人双面超净工作台(苏州净化设备有限公司)、离心机(Allegra X-22,Beckman Co μlter)、恒温培养箱(上海精宏实验设备有限公司)、超纯水系统(MLli-Q)、电子天平(德国科恩ABT 320-4M)、PCR仪(美国BIO-RAD Thermal Cycler型号:C1000)、电泳仪(北京六一仪器厂)、Real-time PCR系统(美国BIO-RAD CFX96TMReal-time System)、凝胶成像系统(美国BIO-RAD Gel Doc XR+molecular imager)。
1.2 方法 1.2.1 沙门菌的培养取-80 ℃冻存沙门菌复活,培养24 h至稳定期,取1 mL接种于100 mL BHI肉汤中培养得到菌液浓度约为104 CFU·mL-1的菌悬液。
1.2.2 诱导产生酸耐受将100 mL无菌BHI(pH=7)肉汤转移到250 mL无菌锥形瓶中,加入菌液浓度约为104 CFU·mL-1的活菌做两个平行,用橡胶塞塞住后置于摇床中在细菌诱导温度条件下、190 r·min-1适应20 h,此时细菌处于稳定期阶段。预先用乙酸将无菌BHI肉汤的pH值调整至4.0,再用孔径为0.22 μm的微孔滤膜过滤,分装到无菌试管中,每管装3 mL肉汤,备用。将适应20 h后的细菌加入pH=4.0的BHI肉汤中,观察细菌的生长状况,应用MTT法[29]结合平板计数法检测沙门菌活菌数。
1.2.3 单因素试验分别以细菌培养温度(15、20、25、30、35、40 ℃)、母菌液添加量(1%、5%、10%、15%、20%、25%、30%)、培养时间(3、4、5、6、7、8 h)作为影响因素,研究各单因素对细菌酸耐受情况的影响。
1.2.4 模型优化前后酸性环境下细菌生存能力的变化相关文献报道的大部分酸耐受试验都是在30或37 ℃下诱导酸耐受2 h,因此在其他培养条件相同的情况下,本试验将肠炎沙门菌暴露于乙酸处理的pH4.0环境下,测定细菌在30、37 ℃以及模型优化后的温度环境下细菌在2 h内生存能力的变化。
1.2.5 其他酸性压力条件下模型优化效果验证其他培养条件相同的情况下,在30、37 ℃条件下诱导酸耐受2 h,利用实时荧光定量PCR方法检测模型优化前后耐酸肠炎沙门菌耐酸基因转录情况,验证模型优化对酸耐受的影响。同时将此模型应用于甲酸、丙酸、正丁酸3种有机酸及无机酸磷酸环境下验证此模型对其他几种酸性压力是否有效。
1.2.5.1RNA提取及反转录:采用传统法提取细菌RNA,并使用NANODROP超微量紫外分光光度计检测RNA的浓度及纯度。使用前用去核酸水对超微量紫外分光光度计进行清洗,用DEPC水进行校正调零。取1 μL RNA样品点样于超微量紫外分光光度计,直接读数即可。选取OD260 nm/OD280 nm 比值在1.8~2.0范围内样品进行下一步试验。
在逆转录之前,先去除gDNA,以消除总RNA中gDNA的影响。分别加入5×gDNA Eraser Buffer,2.0 μL;gDNA Eraser,1.0 μL;RNA,5.0 μL;RNase Free ddH2O加至10 μL。反应条件:42 ℃金属浴2 min或者室温下孵育30 min。取上述反应液10 μL;加入PrimeScript RT Enzyme Mix I 1.0 μL;RT Primer Mix 1.0 μL;5×PrimeScript Buffer 2(for Real Time) 4.0 μL,RNase Free ddH2O 4.0 μL,总共20 μL,反应条件:37 ℃,15 min;85 ℃,5 s;4 ℃保存,反应完成后,-20 ℃保存,用于下一步的试验。
1.2.5.2标准曲线的建立:荧光定量PCR的引物由上海生工生物工程股份有限公司合成,引物见表 1。
|
|
表 1 实时荧光定量PCR引物 Table 1 Real-time fluorescence quantitative PCR primers |
以未做处理的标准菌cDNA作为标准曲线建立的标准品,对目的基因进行普通PCR扩增,反应结束后取5 μL PCR产物,点样于10 g·L-1琼脂糖凝胶孔中,120 V电泳20 min后,置于凝胶成像仪中观察,并做好记录。
用Easy Dilution对上述普通PCR的产物进行稀释。取10个离心管,分别加入45 μL Easy Dilution,取5 μL上述普通PCR产物加入第一个离心管,吹打混匀以后再取5 μL混合液加入第二个离心管中,以此类推,直至第10个离心管。取后8个离心管作为标准曲线的模板,然后对目的基因进行荧光定量PCR扩增,从而优化荧光定量PCR反应的条件、获得标准曲线。
1.2.5.3耐酸基因相对转录水平检测:采用实时荧光定量PCR技术,对耐酸基因ompR、fur进行检测。根据SYBR® Premix Ex TaqTM试剂盒说明书配制反应体系。以gmK基因作为内参基因,每个样品设定3个生物重复,每个生物重复做两个技术重复。采用ΔΔCt法分析确定目的基因的相对转录水平。目的基因的相对转录水平= 2-ΔΔCt(式中ΔCt=Ct目的基因-Ct内参基因,ΔΔCt=ΔCt测试样品-ΔCt校准样品)。数据采用SPSS 19.0统计软件进行分析。
2 结果 2.1 响应面试验 2.1.1 单因素预试验分别以细菌培养温度(15、20、25、30、35、40 ℃)、母菌液添加量(1%、5%、10%、15%、20%、25%、30%)、培养时间(3、4、5、6、7、8 h)作为影响因素,研究各单因素对细菌酸耐受情况的影响,结果如图 1。试验结果表明,本试验所研究的所有变量均对细菌酸耐受产生影响,并且当培养温度为20 ℃、母菌液添加量为10%、培养时间为6 h时,肠炎沙门菌对pH 4.0最容易产生耐受。
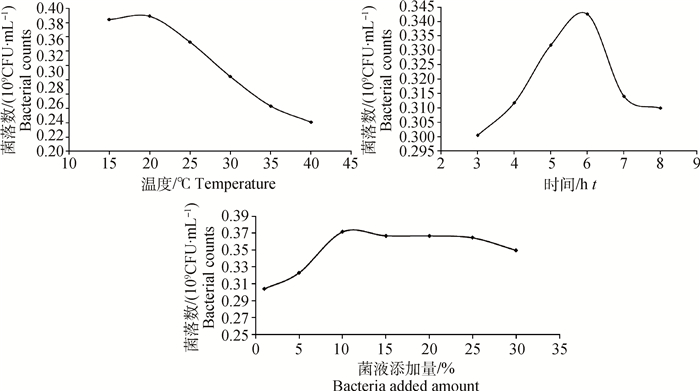
|
图 1 培养温度、时间、母菌液添加量对沙门菌耐酸性的影响 Figure 1 The influence of incubation temperature, incubation time and microbial amount on acid resistance of Salmonella |
在单因素预试验结果的基础上,确定响应面法的Box-Behnken设计来优化和分析选定的因素:细菌培养温度、培养时间、菌液添加量,分别记为A、B、C,在3个水平上进行优化研究,以菌落数为响应值,记为Y,建立数学模型。响应面设计的因素与水平见表 2,Box-Behnken设计的独立变量和响应值见表 3。
|
|
表 2 响应面设计的因素与水平 Table 2 Variables and levels in response surface design |
|
|
表 3 Box-Behnken设计的独立变量和响应值 Table 3 Box-Behnken design with independent variables and response values |
为了优化3个独立变量(细菌培养温度、培养时间、菌液添加量)对酸性压力条件下细菌菌落数的影响,采用3个因素和3个水平进行Box-Behnken设计,通过Design Expert 8.0软件采用多元回归分析对表 3的数据进行分析,得出以菌落数(Y)为响应值,细菌培养温度、培养时间、菌液添加量为变量真实值的二次多项式方程来描述酸性压力条件下菌落数与变量之间关系:Y=0.39+0.018A+0.011B+0.013C-0.015AB+0.017AC-3.1×10-3BC-0.035A2-0.049B2-5.925×10-4C2,R2=0.953 7。模型的方差分析及失拟项见表 4。
|
|
表 4 模型的方差分析及失拟项 Table 4 Analysis of variance (ANOVA) of the model and lack of fit |
方差分析(ANOVA)用于分析模型方程的意义,其结果如表 4所示。方差分析的两个重要指标是F值和P值,模型F值为16.04,说明模型显著,P<0.001同样说明该模型是显著,同时失拟项不显著(P>0.05)。因此,培养温度、菌液添加量、培养时间是酸性压力条件下菌落数的重要影响因素。
2.1.4 酸性模型优化应用Design Expert 8.0进行二次多项式模型拟合作图,通过3D响应面图和二维轮廓图(图 2~4)反应3个变量及其交互作用对酸性压力条件下菌落数的影响。
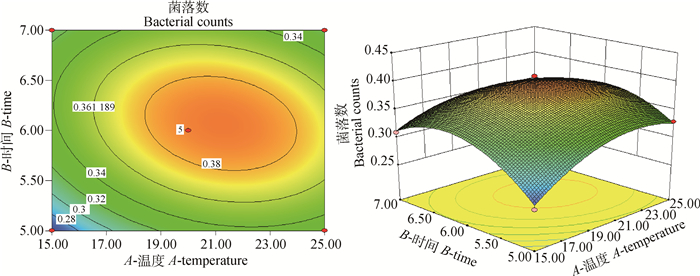
|
图 2 细菌培养温度与培养时间对沙门菌耐酸性的影响 Figure 2 The influence of cultivate bacteria temperature and time on the acid resistance of Salmonella |
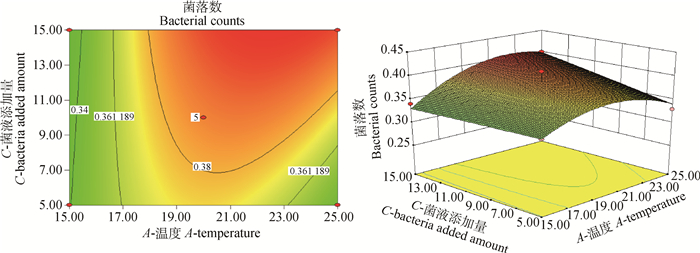
|
图 3 细菌培养温度与母菌液添加量对沙门菌耐酸性的影响 Figure 3 The influence of cultivate bacteria temperature and microbial amount on the acid resistance of Salmonella |
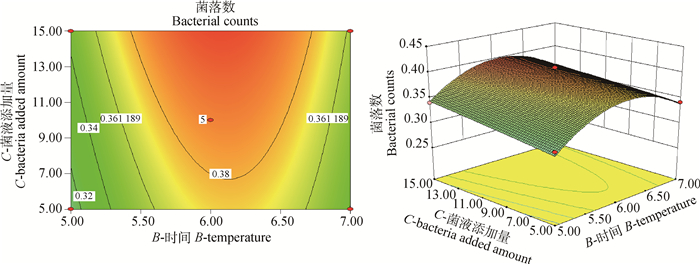
|
图 4 细菌培养时间与母菌液添加量对沙门菌耐酸性的影响 Figure 4 The influence of cultivate bacteria time and microbial amount on the acid resistance of Salmonella |
通过软件分析,诱导肠炎沙门菌耐酸性的最佳条件:培养温度22.49 ℃、培养时间6 h、菌液添加量15%。在此条件下,活菌菌落数达4.088×108 CFU·mL-1。从二维图可知,不同因素对诱导肠炎沙门菌耐酸性的影响大小顺序为培养温度>菌液添加量>培养时间。说明酸性压力条件下,菌落数最依赖于培养温度,其次是菌液添加量和培养时间。结合表 4模型的方差分析以及图 2~4的三维图可知,培养温度和菌液添加量交互作用对酸性压力条件下菌落数影响最大,其次是培养温度与培养时间的交互作用,培养时间与菌液添加量交互作用对酸性压力条件下菌落数影响最小,试验结果表明细菌培养温度和菌液添加量二者交互作用对诱导肠炎沙门菌耐酸性的影响最大。
2.1.5 验证试验采用“2.1.4”试验中得到的最佳酸耐受诱导条件培养温度22.49 ℃、培养时间6 h、菌液添加量15%进行诱导酸耐受重复试验,考虑到实际操作的方便性,将最适培养温度修正为22.5 ℃。按照修正后模型进行验证试验,设置3个平行试验,优化模型条件下耐酸细菌菌落数平均为0.396 6×109 CFU·mL-1,符合度为97%。试验结果与应用Design Expert 8.0进行预测的结果接近,说明优化模型适用于诱导肠炎沙门菌的耐酸性。
2.2 模型优化对肠炎沙门菌生存能力的影响由图 5可知,3种模型条件均对肠炎沙门菌的生存能力影响较大,首先,在pH4.0条件下,2 h的时间内,肠炎沙门菌的生存能力不断下降,结合图 1可知,优化温度条件下,肠炎沙门菌生存能力下降最慢,并在适应酸之后,生存能力再缓慢回升。对比3种生长模型,可以推测,酸性压力条件下,温度越低,细菌生长能力越强,并在优化温度条件下达到最强,试验结果表明,在温度较低条件下,肠炎沙门菌适应酸的能力越强,37 ℃环境下,细菌适应酸能力最弱。
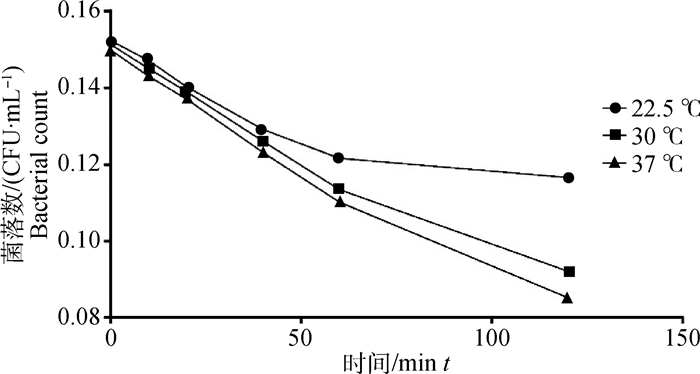
|
图 5 不同模型条件下肠炎沙门菌的生存能力 Figure 5 Survival rates of Salmonella enteritidis under different model conditions |
3对引物经过PCR扩增以后,其产物凝胶电泳所获得的条带如图 6所示。ompR基因与fur基因扩增条带大小与设计大小一致,将管家基因gmK的PCR扩增产物测序并与GenBank的序列(登录号:AF140283)进行对比,保守区序列相似性为98.01%。
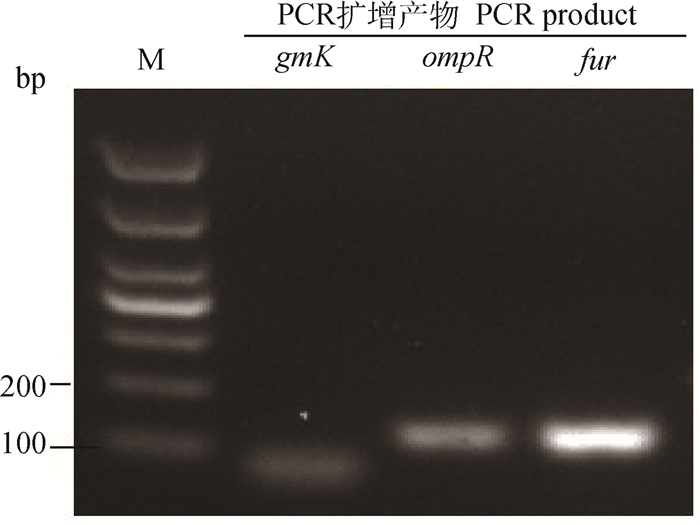
|
M. DL1000 DNA相对分子质量标准 M. DL1000 DNA marker 图 6 3对引物扩增基因的凝胶电泳结果 Figure 6 Electrophoresis results of three genes |
标准曲线分析:荧光定量PCR扩增获得的标准曲线显示,3个目的基因的标准曲线拟合度(R2)均在0.99~1,扩增效率(E)均在98%~105%,其中两个基因的标准曲线图如图 7所示。
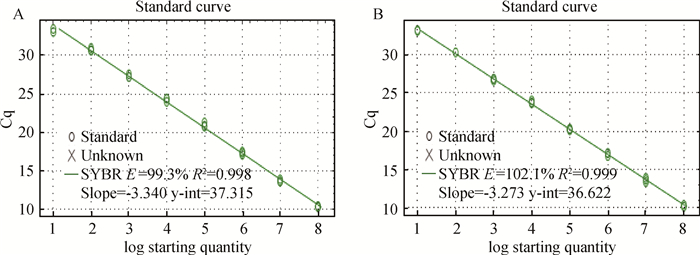
|
图 7 gmK(A)、ompR(B)的标准曲线 Figure 7 The standard curve of gmK(A), ompR(B) |
耐酸基因相对转录水平检测结果:以未经过酸处理的沙门菌标准菌(加入等量PBS)作为空白组对照,3种处理方式对沙门菌耐酸基因的相对转录水平的影响如图 8所示。
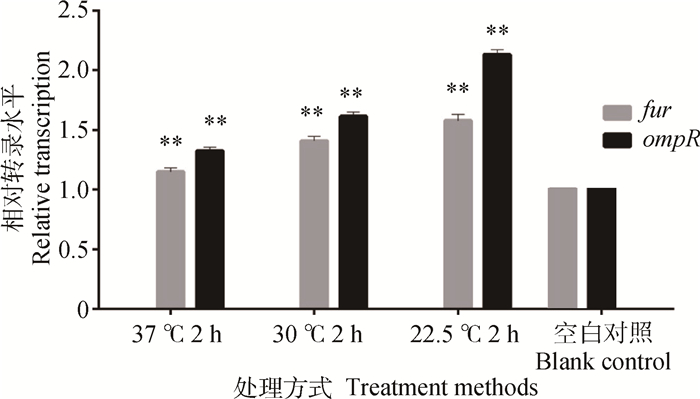
|
*.P < 0.05; **.P < 0.01 图 8 3种处理方式对沙门菌标准菌耐酸基因相对转录水平的影响 Figure 8 Effect of three kinds of treatment methods on the Salmonella standard acid tolerance related genes |
由图 8可知,优化前的两种方式对两种酸耐受基因的相对转录量虽然产生影响,但是影响很小,模型优化后耐酸基因fur的相对转录量是模型优化前的1.4、1.12倍,ompR的相对转录量是模型优化前的1.40、1.13倍,并且相互之间具有极显著差异(P < 0.01),因此可以验证模型优化结果对乙酸耐受性产生最大影响。
2.3.2.3优化模型对其他酸性压力环境下耐酸基因相对转录水平检测结果:由图 9可知,用甲酸、丙酸、正丁酸、磷酸调节培养基pH至4.0,菌液添加量一样的条件下,相比优化前大部分研究酸耐受文献常用的在37 ℃环境下恒温诱导酸耐受2 h,优化后在22.5 ℃诱导6 h耐酸基因fur的相对转录都有极显著提高(P < 0.01)。转录量升高最为明显的为丙酸压力环境下,fur相对转录升高2.04倍。
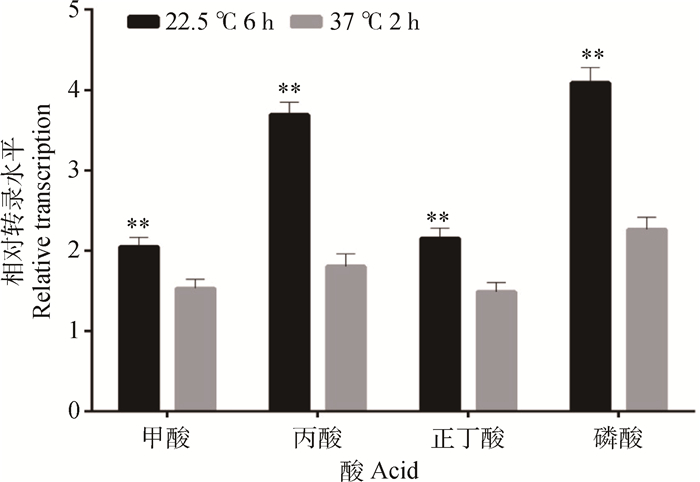
|
*.P < 0.05; **.P < 0.01 图 9 两种酸诱导模型对fur基因相对转录水平的影响 Figure 9 Effects of two acid-induced models on the relative expression of fur gene |
由图 10可知,用甲酸、丙酸、正丁酸、磷酸调节培养基pH至4.0,在菌液添加量一样的条件下,相比优化前在37 ℃环境下恒温诱导酸耐受2 h,优化后在22.5 ℃诱导6 h耐酸基因ompR的相对转录同样都有极显著提高(P < 0.01)。转录量升高最为明显的同样是丙酸压力环境下,ompR相对转录升高2.42倍。
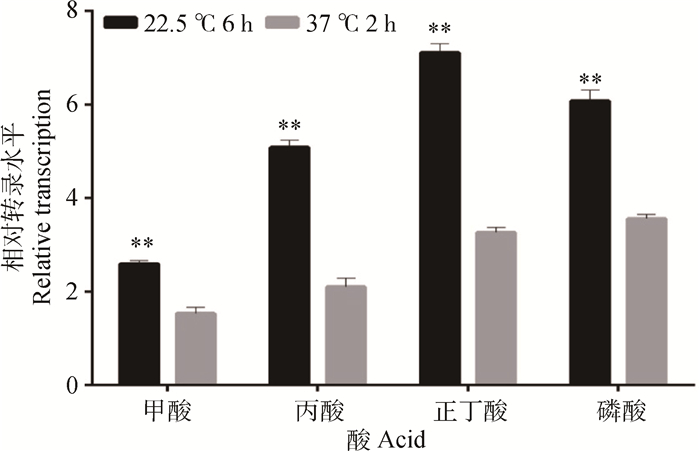
|
*.P < 0.05; **.P < 0.01 图 10 两种酸诱导模型对ompR基因相对转录水平的影响 Figure 10 Effects of two acid-induced models on the relative expression of ompR gene |
抗生素在为畜牧业做出巨大贡献的同时,也因被滥用产生许多负面作用,因此,畜禽养殖各阶段逐步应用酸替代抗生素,以期能控制致病菌的传播[32]。而酸作为防腐剂的成分之一在食品工业中应用已有近百年历史。研究表明,酸的使用可以有效减少一些食源性致病菌的生长,从而延长食品的保质期[33-34]。S. Bearson等[35]研究认为,酸性环境中的微生物可以感受到环境的恶化,合成特殊的应激蛋白。换而言之,在酸性环境中,不同的病原菌,以及一些进化后的新病原菌,都会逐渐适应酸性环境,并发展出在不同酸性环境中的不同生存方式[36],这种适应酸应激的方式称为酸耐受反应。A. Lianou等使用威布尔模型分析在葡萄糖存在条件下使用30种人或牛来源的不同血清型沙门菌研究沙门菌菌株诱导性酸耐受反应表型的变异性,结果表明这种酸耐受反应在不同菌株中存在较大的变异性[9]。P. S. Malheiros等也报道了在肠杆菌的适应性酸耐受反应中的菌株差异[37]。而目前对于沙门菌耐酸性的研究几乎没有选取肠炎沙门菌作为研究对象,据报道,在欧盟(EU)沙门菌是引起食源性疾病的首要原因,截至2013年,肠炎沙门菌与鼠伤寒沙门菌是从人类身上分离到的最常见的两个血清型(分离率分别为39.5%和20.2%,N=73 627)[38-39]。肠炎沙门菌在临床上具有重要地位,有必要对其酸耐受反应进行研究。本研究结果表明,经过乙酸处理,肠炎沙门菌酸耐受相关基因fur、ompR转录均极显著升高,因此可知肠炎沙门菌能在酸性环境下适应酸并产生酸耐受反应。本研究将响应面法应用于肠炎沙门菌酸耐受条件的优化,获得较好的结果。通过条件优化得出的最佳条件:培养温度22.5 ℃、培养时间6 h、菌液添加量15%,细菌菌落数平均为3.966×108 CFU·mL-1,与模型预测的提取率误差较小,说明该条件参数是合理可靠的。
对比3种模型条件对肠炎沙门菌的生存能力影响,首先,在pH4.0条件下,2 h的时间内,肠炎沙门菌的生存能力不断下降,同时,由试验结果可知,温度越低,肠炎沙门菌的生存能力下降越慢,优化后的模型条件下,肠炎沙门菌的生存能力下降最慢,说明在温度较低条件下,肠炎沙门菌适应酸的能力越强,37 ℃环境下,细菌适应酸能力最弱。经过酸处理后耐酸菌的fur和ompR相对转录均升高,相比已报道的常用于研究诱导酸耐受模型[10, 13, 17],应用优化后的诱导酸耐受模型进行诱导后酸耐受相关基因fur和ompR的相对转录极显著升高(P < 0.01)。换而言之,在优化后的诱导酸耐受模型条件下肠炎沙门菌更易产生耐酸性。同时,本研究还将此模型应用于其他常用酸甲酸、丙酸、正丁酸和磷酸诱导的酸耐受模型上,对比37 ℃环境下诱导酸耐受对fur和ompR基因相对转录的影响,结果同样验证了该模型更易诱导肠炎沙门菌产生耐酸性,提高肠炎沙门菌耐酸基因的相对转录。
4 结论成功建立肠炎沙门菌耐酸模型:培养温度22.5 ℃、培养时间6 h、菌液添加量15%,在此模型下,与已报道常用于进行诱导酸耐受的2种模型进行对比,优化模型条件下,肠炎沙门菌的生存能力下降最慢,说明在温度较低条件下,肠炎沙门菌适应酸的能力越强。同时应用实时荧光定量PCR方法测定耐酸基因fur和ompR相对转录的变化验证模型优化结果,验证了该方法能合理地优化细菌酸耐受诱导条件,并且,该方法对其他酸性压力条件下诱导的酸耐受同样有效。
| [1] | MAJOWICZ S E, MUSTO J, SCALLAN E, et al. The global burden of nontyphoidal Salmonella gastroenteritis[J]. Clin Infect Dis, 2010, 50(6): 882–889. DOI: 10.1086/649513 |
| [2] |
王军, 郑增忍, 王晶钰. 动物源性食品中沙门氏菌的风险评估[J]. 中国动物检疫, 2007, 24(4): 23–25.
WANG J, ZHENG Z R, WANG J Y. Risk assessment of Salmonella in animal derived food[J]. Chinese Journal of Animal Quarantine, 2007, 24(4): 23–25. (in Chinese) |
| [3] | SINGH P, MUSTAPHA A. Multiplex TaqMan® detection of pathogenic and multi-drug resistant Salmonella[J]. Int J Food Microbiol, 2013, 166(2): 213–218. DOI: 10.1016/j.ijfoodmicro.2013.07.023 |
| [4] | ANDREOLETTI O, BUDKA H, BUNCIC S, et al. Microbiological risk assessment in feedingstuffs for food-producing animals-scientific opinion of the panel on biological hazards[J/OL]. EFSA J, 2008, 6(7): 1-84. [2017-11-15]. http://onlinelibrary.wiley.com/doi/10.2903/j.efsa.2008.720/pdf. |
| [5] |
于文蕴, 杜柏林. 抗生素人类现代医学的灾难——在畜牧业应用的历史现状及未来展望[C]//第四届中国兽药大会——动物药品学分会论文集. 北京: 中国畜牧兽医学会, 2012.
YU W Y, DU B L. Antibiotics, the disaster of modern human medicine-historical present situation and future prospect of animal husbandry application[C]//2012 China Veterinary Drug Conference. Beijing: Chinese Society of Animal Husbandry and Veterinary Medicine, 2012. (in Chinese) |
| [6] |
李显彬, 李春香, 孟令玉, 等. 某院2011-2015年医院感染病原菌分布及耐药性[J]. 中国感染控制杂志, 2017, 16(1): 66–69.
LI X B, LI C X, MENG L Y, et al. Distribution and antimicrobial resistance of pathogens causing healthcare-associated infection in a hospital in 2011-2015[J]. Chinese Journal of Infection Control, 2017, 16(1): 66–69. (in Chinese) |
| [7] |
张秀芹. 饲料中沙门菌快速检测方法的建立及其分离株的耐药性与致病性分析[D]. 长春: 吉林大学, 2014.
ZHANG X Q. The establishment of the rapid detection method of Salmonella in feed and the drug resistance and pathogenicity analysis of isolates[D]. Changchun: Jilin University, 2014. (in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10183-1015513563.htm |
| [8] |
王彤, 刘显军, 陈静, 等. 对饲料酸化剂发展的思考[J]. 粮食与饲料工业, 2014(5): 54–56.
WANG T, LIU X J, CHEN J, et al. The development of feed acidifier thinking[J]. Cereal & Feed Industry, 2014(5): 54–56. (in Chinese) |
| [9] | LIANOU A, NYCHAS G J E, KOUTSOUMANIS K P. Variability in the adaptive acid tolerance response phenotype of Salmonella enterica strains[J]. Food Microbiol, 2017, 62: 99–105. DOI: 10.1016/j.fm.2016.10.011 |
| [10] | ABDUL-AZIZ A, ABDULLAH M F F, HUSSAIN N H. Inducible acid tolerance response in Shigella sonnei and Shigella flexneri[J]. Res J Microbiol, 2015, 10(7): 320–328. DOI: 10.3923/jm.2015.320.328 |
| [11] | FOSTER J W, HALL H K. Inducible pH homeostasis and the acid tolerance response of Salmonella typhimurium[J]. J Bacteriol, 1991, 173(16): 5129–5135. DOI: 10.1128/jb.173.16.5129-5135.1991 |
| [12] | BEAUBRUN J J G, TALL B D, FLAMER M L, et al. Increased secretion of exopolysaccharide and virulence potential of a mucoid variant of Salmonella enterica serovar Montevideo under environmental stress[J]. Microb Pathog, 2017, 103: 107–113. DOI: 10.1016/j.micpath.2016.12.005 |
| [13] | DAVIS M J, COOTE P J, O'BYRNE C P. Acid tolerance in Listeria monocytogenes:the adaptive acid tolerance response (ATR) and growth-phase-dependent acid resistance[J]. Microbiology, 1996, 142(10): 2975–2982. DOI: 10.1099/13500872-142-10-2975 |
| [14] | LEE I S, SLONCZEWSKI J L, FOSTER J W. A low-pH-inducible, stationary-phase acid tolerance response in Salmonella typhimurium[J]. J Bacteriol, 1994, 176(5): 1422–1426. DOI: 10.1128/jb.176.5.1422-1426.1994 |
| [15] | FOSTER J W. Salmonella acid shock proteins are required for the adaptive acid tolerance response[J]. J Bacteriol, 1991, 173(21): 6896–6902. DOI: 10.1128/jb.173.21.6896-6902.1991 |
| [16] | FOSTER J W. The acid tolerance response of Salmonella typhimurium involves transient synthesis of key acid shock proteins[J]. J Bacteriol, 1993, 175(7): 1981–1987. DOI: 10.1128/jb.175.7.1981-1987.1993 |
| [17] | KANG J H, WIEDMANN M, BOOR K J, et al. VirR-mediated resistance of Listeria monocytogenes against food antimicrobials and cross-protection induced by exposure to organic acid salts[J]. Appl Environ Microbiol, 2015, 81(13): 4553–4562. DOI: 10.1128/AEM.00648-15 |
| [18] | LOU Y, YOUSEF A E. Adaptation to sublethal environmental stresses protects Listeria monocytogenes against lethal preservation factors[J]. Appl Environ Microbiol, 1997, 63(4): 1252–1255. |
| [19] | LEE I S, LIN J, HALL H K, et al. The stationary-phase sigma factor σS (RpoS) is required for a sustained acid tolerance response in virulent Salmonella typhimurium[J]. Mol Microbiol, 1995, 17(1): 155–167. DOI: 10.1111/mmi.1995.17.issue-1 |
| [20] | BURIN R C K, SILVA A Jr, NERO L A. Influence of lactic acid and acetic acid on Salmonella spp. growth and expression of acid tolerance-related genes[J]. Food Res Int, 2014, 64: 726–732. DOI: 10.1016/j.foodres.2014.08.019 |
| [21] | BOTTELDOORN N, VAN COILLIE E, GRIJSPEERDT K, et al. Real-time reverse transcription PCR for the quantification of the mntH expression of Salmonella enterica as a function of growth phase and phagosome-like conditions[J]. J Microbiol Methods, 2006, 66(1): 125–135. DOI: 10.1016/j.mimet.2005.11.003 |
| [22] | KROLL R G, PATCHETT R A. Induced acid tolerance in Listeria monocytogenes[J]. Lett Appl Microbiol, 1992, 14(5): 224–227. DOI: 10.1111/j.1472-765X.1992.tb00691.x |
| [23] | BEZERRA M A, SANTELLI R E, OLIVEIRA E P, et al. Response surface methodology (RSM) as a tool for optimization in analytical chemistry[J]. Talanta, 2008, 76(5): 965–977. DOI: 10.1016/j.talanta.2008.05.019 |
| [24] | JIMÉNEZ J, GUARDIA-PUEBLA Y, ROMERO-ROMERO O, et al. Methanogenic activity optimization using the response surface methodology, during the anaerobic co-digestion of agriculture and industrial wastes. Microbial community diversity[J]. Biomass Bioenergy, 2014, 71: 84–97. DOI: 10.1016/j.biombioe.2014.10.023 |
| [25] | KANG J H, KIM S, MOON B K. Optimization by response surface methodology of lutein recovery from paprika leaves using accelerated solvent extraction[J]. Food Chem, 2016, 205: 140–145. DOI: 10.1016/j.foodchem.2016.03.013 |
| [26] | BELWAL T, DHYANI P, BHATT I D, et al. Optimization extraction conditions for improving phenolic content and antioxidant activity in Berberis asiatica fruits using response surface methodology (RSM)[J]. Food Chem, 2016, 207: 115–124. DOI: 10.1016/j.foodchem.2016.03.081 |
| [27] | ZAROUAL Z, CHAAIR H, ESSADKI A H, et al. Optimizing the removal of trivalent chromium by electrocoagulation using experimental design[J]. Chem Eng J, 2009, 148(2-3): 488–495. DOI: 10.1016/j.cej.2008.09.040 |
| [28] | FENG J, ZHANG J, ZHANG J, et al. Enhanced methane production of vinegar residue by response surface methodology (RSM)[J]. AMB Express, 2017, 7(1): 89. DOI: 10.1186/s13568-017-0392-3 |
| [29] |
汪燕秋, 吴丽云, 张洁, 等. 检测沙门菌活菌数的MTT方法建立[J]. 畜牧与兽医, 2017, 49(6): 130–137.
WANG Y Q, WU L Y, ZHANG J, et al. Establishment of MTT method in counting viable Salmonella[J]. Anim Husband Vet Med, 2017, 49(6): 130–137. (in Chinese) |
| [30] | BANG I S, KIM B H, FOSTER J W, et al. OmpR regulates the stationary-phase acid tolerance response of Salmonella enterica serovar typhimurium[J]. J Bacteriol, 2000, 182(8): 2245–2252. DOI: 10.1128/JB.182.8.2245-2252.2000 |
| [31] | HALL H K, FOSTER J W. The role of fur in the acid tolerance response of Salmonella typhimurium is physiologically and genetically separable from its role in iron acquisition[J]. J Bacteriol, 1996, 178(19): 5683–5691. DOI: 10.1128/jb.178.19.5683-5691.1996 |
| [32] |
URBAITYTE R. 饲料中添加有机酸是否值得考虑[J]. 毛慧, 译. 国外畜牧学-猪与禽, 2014, 34(11): 74-75.
URBAITYTE R. Organic acids: are they worth considering?[J]. MAO H, translated. Animal Science Abroad-Pigs and Poultry, 2014, 34(11): 74-75. (in Chinese) |
| [33] | THERON M M, LUES J F R. Organic acids and meat preservation:a review[J]. Food Rev Int, 2007, 23(2): 141–158. DOI: 10.1080/87559120701224964 |
| [34] | LACOMBE A, WU V C H, TYLER S, et al. Antimicrobial action of the American cranberry constituents; phenolics, anthocyanins, and organic acids, against Escherichia coli O157:H7[J]. Int J Food Microbiol, 2010, 139(1-2): 102–107. DOI: 10.1016/j.ijfoodmicro.2010.01.035 |
| [35] | BEARSON S, BEARSON B, FOSTER J W. Acid stress responses in enterobacteria[J]. FEMS Microbiol Lett, 1997, 147(2): 173–180. DOI: 10.1111/j.1574-6968.1997.tb10238.x |
| [36] | LIN J, LEE I S, FREY J, et al. Comparative analysis of extreme acid survival in Salmonella Typhimurium, Shigella flexneri, and Escherichia coli[J]. J Bacteriol, 1995, 177(14): 4097–4104. DOI: 10.1128/jb.177.14.4097-4104.1995 |
| [37] | MALHEIROS P S, BRANDELLI A, NOREÑA C P Z, et al. Acid and thermal resistance of a Salmonella enteritidis strain involved in several foodborne outbreaks[J]. J Food Saf, 2009, 29(2): 302–317. DOI: 10.1111/jfs.2009.29.issue-2 |
| [38] | European Food Safety Authority, European Centre for Disease Prevention and Control. The European Union summary report on trends and sources of zoonoses, zoonotic agents and food-borne outbreaks in 2013[J]. EFSA J, 2015, 13(1): 3991. DOI: 10.2903/j.efsa.2015.3991 |
| [39] |
李金鑫, 杨树青, 王莉莉, 等. 肠炎沙门菌SDBL-1分离株的生物学特性及传播途径探究[J]. 畜牧兽医学报, 2017, 48(7): 1323–1331.
LI J X, YANG S Q, WANG L L, et al. Study of Salmonella enteritidis SDBL-1 transmission and biological characteristics[J]. Acta Veterinaria et Zootechnica Sinica, 2017, 48(7): 1323–1331. (in Chinese) |



