当前耐药性细菌广泛存在,细菌病的防治现状形势严峻[1]。噬菌体是一种专性裂解细菌的病毒,作为细菌的天敌在自然界中广泛存在,由于其生产成本低、特异性强、不受耐药性的影响等优点,有望作为抗生素替代品用于养殖过程中细菌病的防治[2-3]。
噬菌体裂解细菌的过程依次为吸附、注入、增殖、装配和释放五个阶段,噬菌体吸附宿主菌表面受体是启动噬菌体侵染过程的关键步骤[4]。噬菌体识别的受体种类很多,如脂多糖、膜蛋白、鞭毛等,细菌表面受体的种类决定了噬菌体能否裂解细菌[5-6]。革兰阴性菌的脂多糖(LPS)是最常见的表面受体,作为细胞外膜的主要成分,根据其结构从内向外依次分为类脂A、核心多糖和O抗原,受多个脂多糖合成基因控制[7]。类脂A是LPS的疏水基团,决定LPS分子的内毒素活性;核心多糖与类脂A相连,可以进一步分为内核心多糖和外核心多糖区域,外核心多糖主要是己糖,根据其糖基构成可以分为五种类型,而内核心多糖主要由两个Kdo和3个Hep(HepⅠ、HepⅡ和HepⅢ)糖基组成,保守性强,维持细胞外膜的稳定性和完整性;O抗原与外核心多糖相连,变异性强,决定细菌的抗原性及血清型[8-10]。在长期进化过程中,细菌能够通过脂多糖合成相关基因的突变改变细胞表面脂多糖的构成,使噬菌体不能识别突变的LPS受体,进而抵御噬菌体的侵染。
笔者实验室前期分离到一株大肠杆菌噬菌体Bp7,属于肌尾噬菌体科T4类噬菌体,能够裂解大肠杆菌E. coli K12[11]。在培育过程中,得到E. coli K12的突变株K12-R,能够耐受噬菌体Bp7的裂解。为了明确K12-R的突变位点,本试验对其基因组序列进行测序分析,并对其生物学性能进行测定,以期为明确细菌耐受噬菌体侵染的机制提供理论基础。
1 材料与方法 1.1 材料大肠杆菌野生株E. coli K12(MG1655)、突变株K12-R均由本实验室保存。细菌基因组DNA小量纯化试剂盒购自宝生物工程有限公司;胰蛋白胨、酵母粉和琼脂粉购自Sigma公司。结晶紫染色液购自生工生物工程(上海)股份有限公司。
1.2 方法 1.2.1 菌株的培养及基因组序列分析将冻存的E. coli K12和K12-R菌液分别在普通营养琼脂板上划线,37 ℃条件下过夜培养,挑取单菌落接种到LB液体培养基,37 ℃振荡培养至对数期。增殖后的菌液用细菌基因组DNA小量纯化试剂盒提取基因组DNA,送安诺优达基因科技有限公司进行全基因组测序,根据测序结果分析比较突变株K12-R与野生株E. coli K12在基因组上的差异。
1.2.2 菌体电镜形态观察取增殖好的E. coli K12和K12-R菌液各500 μL,4 000 r·min-1离心1 min,去上清,菌体沉淀用1 mL PBS重悬,4 000 r·min-1离心1 min,去上清,重复3次。洗涤后的菌体沉淀用200 μL PBS重悬,取20 μL菌液滴加于铜网上,孵育10 min,滤纸吸去多余菌液,2%磷钨酸染色2 min,滤纸吸去多余染液,干燥后在日立透射电子显微镜HT7700下观察。
1.2.3 生长曲线的测定取增殖好E. coli K12和K12-R菌液,用LB调整初始菌液的浓度OD600 nm值为0.15,各取200 μL加到96孔培养板中,各设3个重复。在37 ℃的温箱中静止培养,每隔20 min测一次OD600 nm值,共测定5 h,根据测定结果绘制菌株的生长曲线。
1.2.4 自凝能力的测定取E. coli K12和K12-R菌液各10 mL,10 000 r·min-1离心2 min,去上清,PBS重悬,并调整菌液的初始浓度OD600 nm为1.4,将菌液静置于4 ℃冰箱中,每隔6 h从菌液最上层取样200 μL,测定OD600 nm值,共检测至54 h。
1.2.5 生物膜形成能力的测定取E. coli K12和K12-R菌液各1 mL,用LB调整菌液初始浓度OD600 nm值为0.2,各取200 μL菌液于96孔培养板中,各设3个重复,于37 ℃温箱中静置培养48 h,至其形成生物膜。弃去培养板各孔中上清,加入200 μL H2O洗去残余上清,重复3次。培养板各孔中滴加3滴结晶紫染液,静置20 min,使生物膜着色。弃去多余染液,加入200 μL H2O重复冲洗3次,晾干。各孔加入95%的乙醇200 μL,吹打混匀后,测定OD600 nm值。
1.2.6 数据分析试验数据用Prism 6.0进行处理,作图,应用ANOVA程序进行单因素方差分析。图中的试验数据均以“x±s”表示。
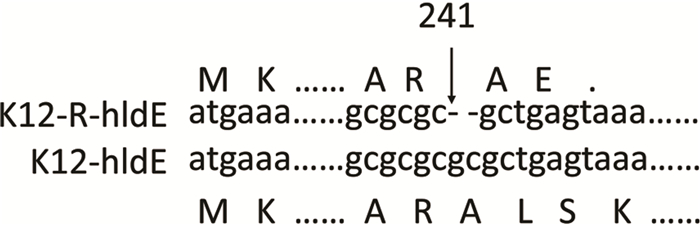
2 结果 2.1 E. coli K12及突变株K12-R基因组序列分析比较提取E. coli K12及其突变株K12-R的DNA,进行基因组测序,获得全基因组序列。与GenBank中的E. coli K12(MG1655)基因组序列(GenBank收录号NC_000913)进行比对,证实实验室保存的E. coli K12野生株核苷酸序列未见有义突变。对测序得到的E. coli K12野生株基因组序列与突变株K12-R基因组序列进行比对,检测K12-R的突变位点。比对结果显示E. coli K12及突变株K12-R基因组序列基本一致,仅K12-R的hldE基因发生有义突变,在241—242位碱基发生缺失,导致该基因转录提前终止(图 1)。
|
图 1 K12-R突变位点分析 Figure 1 Mutation sites analysis of K12-R |
E. coli K12和K12-R经磷钨酸染色处理后,透射电镜下观察其形态特征。E. coli K12菌体呈杆状,细胞膜轮廓清晰可见,菌体内两端负染。与E. coli K12比较,K12-R菌体形态没有明显变化,但是细胞膜轮廓不清晰,菌体内全部负染(图 2)。

|
图 2 E. coli K12和K12-R透射电镜形态观察(左10 000×,右15 000×) Figure 2 Morphology of K12 and K12-R in TEM(left 10 000×, right 15 000×) |
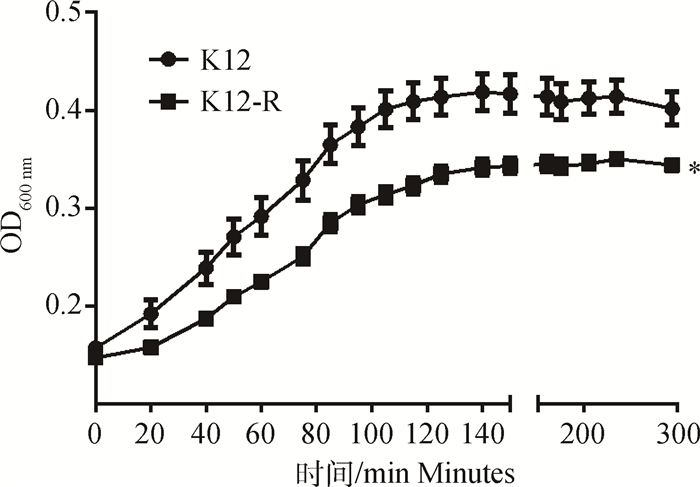
为了检测突变对K12-R菌体生长性能的影响,以E. coli K12为对照,进行菌液浊度测定,根据测定的OD600 nm值绘制生长曲线(图 3)。由图可知,在最开始的40 min内细菌生长缓慢,40~100 min大肠杆菌进入快速生长繁殖的对数期,140 min以后菌液浓度基本没有明显变化,细菌进入生长繁殖的平台期。与E. coli K12比较,K12-R的生长性能明显降低(P<0.05)。
|
*.P < 0.05 图 3 E. coli K12和K12-R的生长曲线 Figure 3 The growth curve of K12 and K12-R |
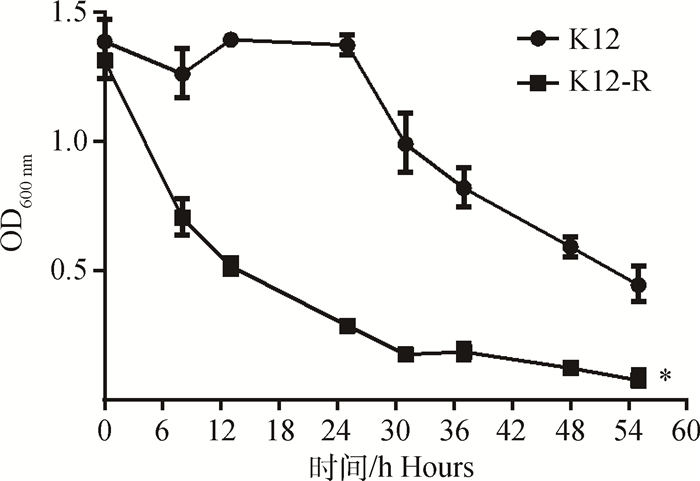
在4 ℃静置的条件下,测定不同时间点K12-R菌液的浊度,与E. coli K12比较,检测hldE基因突变对K12-R自凝能力的影响,结果见图 4。由图可知,随着时间的推移,菌液的浊度逐渐降低,说明菌液发生自凝现象。E. coli K12菌液在24 h内浊度基本没有变化,24 h后菌液出现自凝现象,到54 h菌液浊度维持在0.5左右,而K12-R菌液在6 h浊度即出现明显下降,至54 h菌体基本全部形成沉淀,说明K12-R的自凝能力明显增强(P < 0.05)。
|
*.P < 0.05 图 4 E. coli K12和K12-R的自凝能力(以浊度评价) Figure 4 The autoaggregation ability (evaluated by turbidity) of K12 and K12-R |
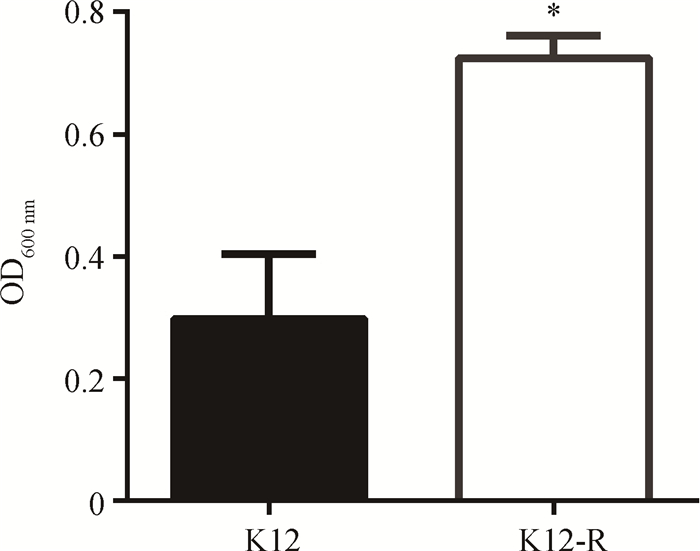
生物膜通常作为细菌应对环境变化的防御结构,对细菌的生存有积极作用。本试验对E. coli K12和突变株K12-R生物膜形成能力进行测定比较,结果见图 5。与野生株E. coli K12比较,K12-R生物膜形成的能力明显增强(P < 0.05)。
|
*.P < 0.05 图 5 E. coli K12和K12-R生物膜形成能力 Figure 5 The biofilm formation of K12 and K12-R |
在宿主菌E. coli K12培育过程中,获得了噬菌体Bp7的耐受株K12-R,为了明确其耐受机制,对K12-R的突变位点及其突变引起的生物学性能的改变进行了分析和测定。基因组测序比对结果表明,K12-R的hldE基因发生了2个碱基缺失,导致该基因转录提前终止,因此初步证实E. coli K12脂多糖是噬菌体Bp7的识别受体。
革兰阴性菌细胞外膜是细菌抵御多种抗生素及不良环境的第一道防线,在细菌的营养吸收和废物代谢过程中也具有重要的作用。脂多糖作为外膜的主要成分,决定细胞膜的稳定性。脂多糖成分的变化,能够引起细菌表型的变化,如细胞膜的通透性、完整性、生物膜形成能力等[12-13]。大肠杆菌的内核心多糖由Kdo和Hep两种糖基组成,抑制Kdo糖基合成通常导致细菌死亡,而抑制Hep糖基的合成会引起细菌表型的改变[14]。控制核心多糖生物合成的是一系列waa基因,其中hldE基因(旧称waaE或者rfaE)编码HepⅠ转移酶,该基因的缺失能够导致脂多糖内核心多糖主链上HepⅠ和HepⅡ之间的连接键发生断裂,使细胞外膜表面的亲水基团缺失,暴露出细胞膜内部的疏水基团,导致细菌表面疏水性增强[15]。本试验中K12-R突变株的hldE基因转录提前终止,引起细胞生物学性能一系列的变化。由于细胞外膜疏水性增强,导致K12-R突变株的自凝能力明显增强,这与之前报道一致[10, 12]。电镜观察突变株K12-R形态发现其细胞内全部负染,说明磷钨酸染液能够顺利进入细胞内部,证明hldE基因缺失使细胞膜通透性增加,甚至影响细胞膜的完整性。R. Nakao等[10]报道了hldE基因缺失并不影响菌体的生长性能,Z. Wang等[12]报道了敲除核心多糖各合成基因后,各突变株生长性能普遍增强,认为其生长状态的改变与外核心多糖的半乳糖糖基相关。但是本试验中生长曲线测定结果显示K12-R的生长性能明显降低,推测其原因是由于hldE基因突变影响了菌体细胞膜的完整性,进而影响了细菌生长状态。细菌的黏附能力是影响生物膜形成的重要因素,据报道,LPS能够促使细菌黏附在适宜的介质表面,形成生物膜[16]。大肠杆菌核心多糖的构成与大肠杆菌生物膜的形成密切相关[10]。前期研究表明,在多种革兰阴性菌中,核心多糖的缺失均能够促进生物膜的形成[10, 17-18]。本试验中hldE基因突变也同样导致K12-R的生物膜形成能力显著增强,这说明在革兰阴性菌中,其脂多糖成分的缺失能够增强细菌生物膜形成能力可能是一个普遍现象,因为脂多糖的缺失暴露了内部的疏水性基团,提高了细胞膜表面的疏水性,促进了生物膜的生成[19]。
细菌在长期进化过程中,通过多种耐受机制抵御噬菌体的侵染。本研究证实K12-R通过hldE基因突变,耐受了噬菌体Bp7的侵染,初步证实噬菌体Bp7通过识别宿主菌E. coli K12表面的脂多糖受体侵染细菌,后续将进一步验证E.coli K12的内核心多糖与噬菌体Bp7的相互作用,这将为进一步阐明细菌耐受噬菌体侵染机制提供理论依据。
4 结论E.coli K12的脂多糖合成基因hldE突变能够改变K12-R脂多糖的结构,从而抑制噬菌体Bp7的吸附。突变株K12-R细胞膜通透性增加,生长繁殖能力降低,自凝能力增强,生物膜形成能力增强。
| [1] | WANG Y, YI L, WANG Y X, et al. Isolation, phylogenetic group, drug resistance, biofilm formation, and adherence genes of Escherichia coli from poultry in central China[J]. Poult Sci, 2016, 95(12): 2895–2901. DOI: 10.3382/ps/pew252 |
| [2] | CRISCUOLO E, SPADINI S, LAMANNA J, et al. Bacteriophages and their immunological applications against infectious threats[J]. J Immunol Res, 2017, 2017: 3780697. |
| [3] | CISEK A A, DABROWSKA I, GREGORCZYK K P, et al. Phage therapy in bacterial infections treatment:one hundred years after the discovery of bacteriophages[J]. Curr Microbiol, 2017, 74(2): 277–283. DOI: 10.1007/s00284-016-1166-x |
| [4] | YOUNG R. Phage lysis:three steps, three choices, one outcome[J]. J Microbiol, 2014, 52(3): 243–258. DOI: 10.1007/s12275-014-4087-z |
| [5] | BERTOZZI SILVA J, STORMS Z, SAUVAGEAU D. Host receptors for bacteriophage adsorption[J]. FEMS Microbiol Lett, 2016, 363(4): fnw002. DOI: 10.1093/femsle/fnw002 |
| [6] | RAKHUBA D V, KOLOMIETS E I, DEY E S, et al. Bacteriophage receptors, mechanisms of phage adsorption and penetration into host cell[J]. Pol J Microbiol, 2010, 59(3): 145–155. |
| [7] | HEINRICHS D E, YETHON J A, WHITFIELD C. Molecular basis for structural diversity in the core regions of the lipopolysaccharides of Escherichia coli and Salmonella enterica[J]. Mol Microbiol, 1998, 30(2): 221–232. DOI: 10.1046/j.1365-2958.1998.01063.x |
| [8] | GONZÁLEZ-GARCÍA V A, PULIDO-CID M, GARCIA-DOVAL C, et al. Conformational changes leading to T7 DNA delivery upon interaction with the bacterial receptor[J]. J Biol Chem, 2015, 290(16): 10038–10044. DOI: 10.1074/jbc.M114.614222 |
| [9] | ROJAS-MACIAS M A, STÅHLE J, LVTTEKE T, et al. Development of the ECODAB into a relational database for Escherichia coli O-antigens and other bacterial polysaccharides[J]. Glycobiology, 2015, 25(3): 341–347. DOI: 10.1093/glycob/cwu116 |
| [10] | NAKAO R, RAMSTEDT M, WAI S N, et al. Enhanced biofilm formation by Escherichia coli LPS mutants defective in Hep biosynthesis[J]. PLoS One, 2012, 7(12): e51241. DOI: 10.1371/journal.pone.0051241 |
| [11] | ZHANG C, LI W L, LIU W H, et al. T4-like phage Bp7, a potential antimicrobial agent for controlling drug-resistant Escherichia coli in chickens[J]. Appl Environ Microbiol, 2013, 79(18): 5559–5565. DOI: 10.1128/AEM.01505-13 |
| [12] | WANG Z, WANG J L, REN G, et al. Influence of core oligosaccharide of lipopolysaccharide to outer membrane behavior of Escherichia coli[J]. Mar Drugs, 2015, 13(6): 3325–3339. |
| [13] | BOGDANOV M, MILEYKOVSKAYA E, DOWHAN W. Lipids in the assembly of membrane proteins and organization of protein supercomplexes:implications for lipid-linked disorders[J]. Subcell Biochem, 2008, 49: 197–239. DOI: 10.1007/978-1-4020-8831-5 |
| [14] | RAETZ C R H, WHITFIELD C. Lipopolysaccharide endotoxins[J]. Annu Rev Biochem, 2002, 71: 635–700. DOI: 10.1146/annurev.biochem.71.110601.135414 |
| [15] | DESROY N, MOREAU F, BRIET S, et al. Towards Gram-negative antivirulence drugs:new inhibitors of HldE kinase[J]. Bioorg Med Chem, 2009, 17(3): 1276–1289. DOI: 10.1016/j.bmc.2008.12.021 |
| [16] | KARUNAKARAN E, MUKHERJEE J, RAMALINGAM B, et al. "Biofilmology":a multidisciplinary review of the study of microbial biofilms[J]. Appl Microbiol Biotechnol, 2011, 90(6): 1869–1881. DOI: 10.1007/s00253-011-3293-4 |
| [17] | NAKAO R, SENPUKU H, WATANABE H. Porphyromonas gingivalis galE is involved in lipopolysaccharide O-Antigen synthesis and biofilm formation[J]. Infect Immun, 2006, 74(11): 6145–6153. DOI: 10.1128/IAI.00261-06 |
| [18] | XU D, ZHANG W, ZHANG B, et al. Characterization of a biofilm-forming Shigella flexneri phenotype due to deficiency in Hep biosynthesis[J]. Peer J, 2016, 4(e2178). |
| [19] | MITZEL M R, SAND S, WHALEN J K, et al. Hydrophobicity of biofilm coatings influences the transport dynamics of polystyrene nanoparticles in biofilm-coated sand[J]. Water Res, 2016, 92: 113–120. DOI: 10.1016/j.watres.2016.01.026 |



