梅花鹿(Cervus nippon)属于哺乳纲、偶蹄目,雌性无角,雄性有角,是重要的经济动物,具有极高的观赏价值、药用价值和食用价值。中国有着丰富的梅花鹿资源和悠久的梅花鹿驯鹿历史,已经育成品种有双阳梅花鹿、敖东梅花鹿、四平梅花鹿、东丰梅花鹿、兴凯湖梅花鹿、西丰梅花鹿,培育的品系有长白山梅花鹿。梅花鹿种公鹿则是养殖企业为培育优质的鹿群,依据生产性能、年龄、体质外貌、遗传性能等综合因素选育出来的种用资源。
动物的起源进化研究主要围绕线粒体DNA和Y染色体。线粒体DNA遵循母系遗传,进化速率快,具有多态性,被广泛的用于母系起源的研究,研究热点主要集中在Cytb基因、D-loop区、12S rRNA和16S rRNA基因以及线粒体DNA全序列。Y染色体遵循父系遗传,具有非重组区,有效群体小,是研究遗传多样性[1-2]、父系起源[3-4]及迁徙路线[5]的重要工具,研究热点主要集中在Y-SNP、Y-STR及Y-CNV,其中Y-SNP研究主要围绕AMELY、DBY、SRY、USP9Y、ZFY、UTY等基因,在牛[6-7]、羊[8-10]、马[11-13]、猪[14-16]等物种中研究的较多。目前,关于中国家养梅花鹿的起源进化和遗传多样性的研究主要集中在线粒体DNA,吴华等[17]对来自9个东北家养群体的45只梅花鹿的D-loop区进行研究,定义了14种单倍型,分成了3个单系,并指出东北家养梅花鹿的遗传多样性较高。李欢霞[18]测定了来自4个家养群体的194只梅花鹿的Cytb基因,发现23个变异位点,定义了8种单倍型,并分成了两个单系。而目前中国家养梅花鹿Y染色体的研究却不多见,仅见周盼伊[19]对来自8个家养群体的690只生产鹿群梅花鹿的Y染色体进行研究,定义了10种单倍型,有两个父系类型。虽然线粒体DNA可为家养梅花鹿的遗传多样性和母系起源的研究提供较为明确的分子生物学证据,但单一的线粒体DNA的研究结果并不能全面反映家养梅花鹿的遗传多样性和起源进化,仍需利用Y染色体从父系遗传多样性和父系起源进化的角度进行补充。梅花鹿种公鹿是家养梅花鹿群体的核心,决定着整个梅花鹿群体的生产性能。
本研究利用Y染色体的AMELY、DBY、USP9Y、SRY基因作为分子标记对我国梅花鹿种公鹿的遗传多样性和父系起源进行探讨,摸清梅花鹿种公鹿的父系类型,为我国梅花鹿保种和育种工作提供数据支持与理论指导。
1 材料与方法 1.1 试验材料分别从吉林省和黑龙江省采集梅花鹿种公鹿抗凝血样171份(表 1)。
|
|
表 1 样品信息 Table 1 Sample information |
全血基因组DNA提取试剂盒(DP1101)购自北京百泰克公司;Taq DNA聚合酶购自于大连宝生物工程有限公司;50×TAE、琼脂糖、异丙醇、无水乙醇、Loading Buffer、DNA Marker(DL 2000和DL15000)均购自上海生工生物工程技术服务有限公司。
PCR扩增仪(Eppendorf);电泳仪(Bio-Rad);凝胶成像系统(Bio-Rad);离心机(Sigma公司);电子天平(Mettler Toledo公司)。
1.3 基因组DNA提取和引物设计按照百泰克全血基因组DNA提取试剂盒的说明书对梅花鹿种公鹿的基因组DNA进行提取,参考文献[19]对梅花鹿Y染色体上的AMELY、DBY、USP9Y、SRY基因片段设计引物,并在此基础上进行修改,引物由上海生工生物工程技术服务有限公司合成,引物序列见表 2。
|
|
表 2 引物信息 Table 2 Primer information |
50 μL扩增体系:ddH2O 35 μL,dNTP和10×buffer各5 μL,Taq酶0.5 μL,上、下游引物(10 μmol·L-1)各1 μL,模板基因组DNA 2.5 μL。反应条件:94 ℃预变性5 min;94 ℃变性30 s,最佳Tm 30 s,72 ℃延伸1 min,共30个循环;72 ℃延伸5 min,最后4 ℃保存。使用1%的凝胶电泳对PCR产物进行检验,将合格的样品送往上海生工生物工程技术服务有限公司进行测序。
1.5 序列的生物信息学分析利用BioEdit7.2.5软件进行人工校正,删除不准确的序列;DNAMAN V6软件进行序列的拼接;Mega6.0软件[20]确定核苷酸突变位点、碱基组成、转换与颠换比(Ti/Tv);DNASP5.1软件[21]定义单倍型,计算单倍型多样性(Hd)、种群内核苷酸多样性(π)和种群间的核苷酸歧义度(Dxy);使用Mega6.0软件的邻接法(Neighbor-Joining,NJ)构建系统进化树,马鹿(Cervus elaphus)相应的同源序列作为外源,将Bootstrap设置1 000次重复抽样来检验NJ系统进化树各个分支的置信度[22];利用Mega6.0和iTOLS3.5.3构建171只梅花鹿种公鹿个体的分子系统进化树;Structure2.3.4软件计算群体遗传结构,设定群体数(K)的预测值范围为2~9,MCM(Markov chain monte carlo)开始时的不作数迭代(Length of burn-in period)设为20 000次,不作数迭代后的MCMC为120 000次,重复运行6次。使用Structure Harvester Web v0.6.94在线工具及J.K.Pritchard等[23]描述的方法确定最优群体数。
2 结果 2.1 梅花鹿种公鹿Y染色体上5个基因片段的扩增结果采用普通PCR方法进行扩增,取3 μL PCR扩增产物进行1%的琼脂凝胶电泳检测,AMELY1、AMELY2、DBY、USP9Y、SRY基因片段均符合目的片段的大小,无杂带且条带清晰,特异性好,可用于后续的PCR产物测序(图 1)。
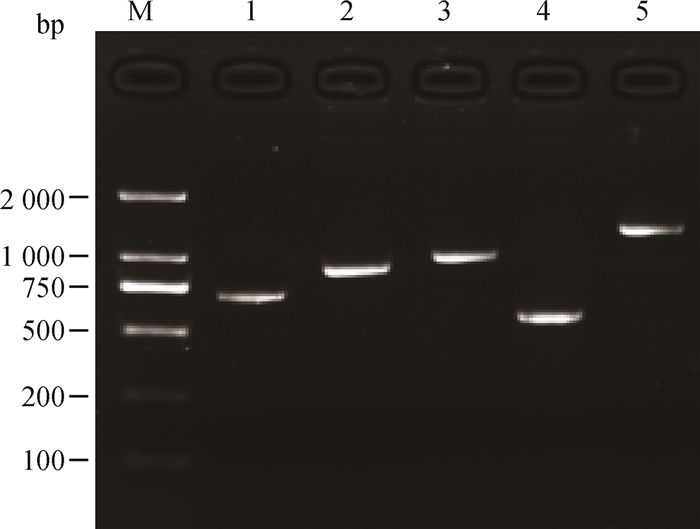
|
M.DNA相对分子质量标准DL2000;1.AMELY1;2.AMELY2;3.DBY;4.USP9Y;5.SRY M.DL2000 DNA marker; 1.AMELY1; 2.AMELY2; 3.DBY; 4.USP9Y; 5.SRY 图 1 AMELY1、AMELY2、DBY、USP9Y、SRY扩增结果 Figure 1 Result for PCR amplification of AMELY1, AMELY2, DBY, USP9Y, SRY |
利用BioEdit7.2.5软件对比峰图对测序结果进行人工校正,删除不准确的序列,可得到AMELY1、AMELY2、DBY、USP9Y、SRY基因片段的长度分别为708、877、1 086、581、1 523 bp,种公鹿间的各片段无序列长度差异。利用Mega6.0计算5个片段总碱基组成,东丰梅花鹿种公鹿A+T的含量为63.8%、C+G含量为36.2%。兴凯湖梅花鹿种公鹿A+T含量为63.6%、C+G含量为36.4%。其余梅花鹿种公鹿群体的A+T和C+G含量均为63.7%和36.3%。可知A+T含量明显大于C+G含量,说明Y染色体的这5个基因片段存在AT碱基偏好性。由表 3可知,东丰梅花鹿种公鹿和四平梅花鹿种公鹿间具有最大的核苷酸歧义度(0.000 50),敖东梅花鹿种公鹿和和双阳梅花鹿种公鹿具有最低的核苷酸歧义度(0.000 14),东丰梅花鹿种公鹿具有最高的核苷酸多样性(0.000 42),敖东梅花鹿种公鹿具有最低的核苷酸多样性(0.000 12)。
|
|
表 3 种群间核苷酸歧义度(Dxy)、种群内核苷酸多样性(π)和单倍型多样性(Hd) Table 3 The Dxy between populations, π and Hd within the population |
利用DNASP5.1软件共定义10种单倍型,分别为H1、H2、H3、H4、H5、H6、H7、H8、H9、H10(表 4)。如表 5所示,兴凯湖梅花鹿种公鹿有4种单倍型(H1、H3、H4、H6),东大梅花鹿种公鹿有6种单倍型(H1、H2、H3、H7、H8、H9),四平梅花鹿种公鹿有3种单倍型(H1、H2、H7),双阳梅花鹿种公鹿有4种单倍型(H1、H4、H6、H8),敖东梅花鹿种公鹿有3种单倍型(H1、H2、H9),长白山梅花鹿种公鹿有3种单倍型(H1、H2、H3),东丰梅花鹿种公鹿有5种单倍型(H1、H2、H3、H5、H10),四平梅花鹿种公鹿和敖东梅花鹿种公鹿分别具有最高和最低的单倍型多样性(表 3)。H5和H10为东丰梅花鹿种公鹿独有。H1为所有梅花鹿种公鹿群体共有,所占频率为49.1%,是优势单倍型,而H5、H6、H7、H8、H9、H10所占频率较低,分别为0.59%、3.5%、2.9%、1.75%、1.17%、0.59%,为稀有单倍型(表 5)。
|
|
表 4 变异位点和单倍型 Table 4 Mutation sites and haplotypes |
|
|
表 5 单倍型频率及其在群体中的分布 Table 5 Distribution and frequency of haplotypes |
利用Mega6.0软件比对分析后,未发现缺失和插入,全为碱基替换,共统计出8个突变位点(表 4),其中在AMELY1的66位出现C→T转换、136位T→G颠换、624位T→C转换,AMELY2的128位T→A颠换、590位A→G转换,DBY的404位G→A转换,USP9Y的362位置出现C→T转换,SRY的38位T→A颠换,转换与颠换比(Ti/Tv)为2.223,表明AMELY、DBY、USP9Y、SRY基因的所有突变以转换为主。对AMELY、DBY、USP9Y、SRY基因进行中性检验,得到的Tajima’S D值[24]分别为-0.445 03、0.351 58、1.243 27、1.341 26,P>0.1,为不显著差异,表明4个基因的所有的突变均符合中性进化。梅花鹿种公鹿Y染色体的5个基因片段Hd=0.696 80,π=0.000 36。
2.5 梅花鹿种公鹿的单倍型进化树分析对171只梅花鹿种公鹿的10种单倍型进行系统进化分析(图 2),发现H9是最原始的单倍型,与其它单倍型的亲缘关系较远,并独自形成单系Ⅰ(Y1)。H3、H5和H10形成单系Ⅱ(Y2)。H2、H4、H6、H7和H8是由H1进化而来,并和H1一起形成单系Ⅲ(Y3)。结合表 5,敖东梅花鹿种公鹿和东大梅花鹿种公鹿的一部分个体是最原始种群,另外一部分个体则由最原始种群进化而来。除双阳梅花鹿种公鹿和四平梅花鹿种公鹿均来自单系Ⅲ(Y3),其余梅花鹿种公鹿群体来自不同单系。
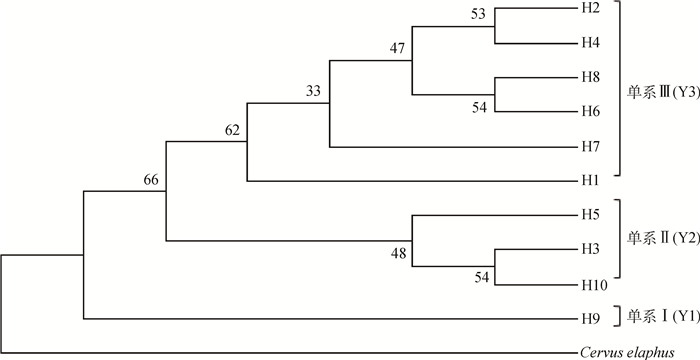
|
图 2 10种单倍型的NJ系统进化树 Figure 2 NJ phylogenetic tree of 10 haplotypes |
利用Mega6.0软件和iTOL3.5.3构建171只梅花鹿种公鹿的圆形分子系统进化树,由图 3可知,各群体间分界不明显,存在基因交流和基因渗入现象,这可能是养殖企业在配种时引种杂交引起的。
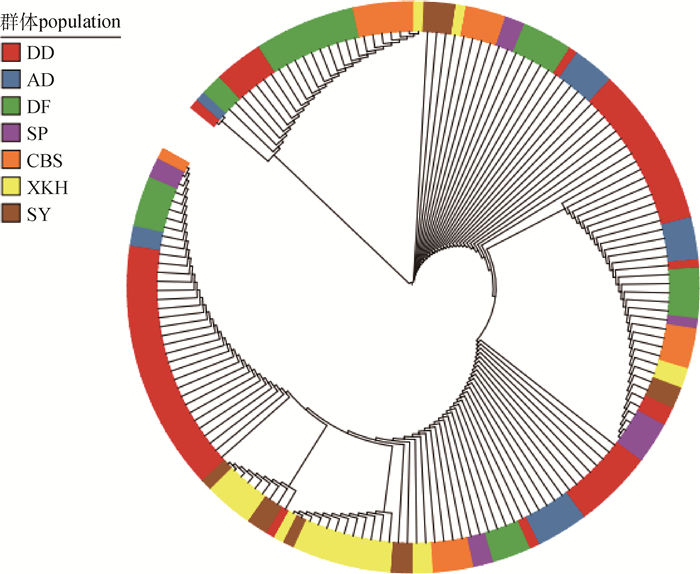
|
图 3 171只梅花鹿种公鹿的圆形分子系统进化树 Figure 3 Circular phylogenetic tree of 171 breeder sika deer |
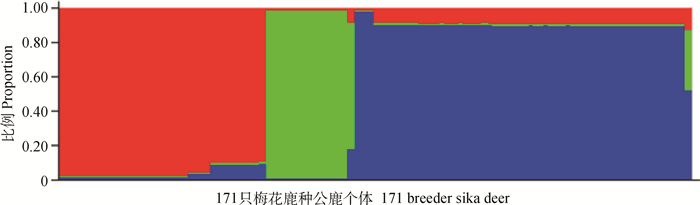
利用Structure2.3.4软件分析171只梅花鹿种公鹿的群体结构,如图 4所示,根据J.K.Pritchard等[23]描述的方法,ΔK值在K=3时达到最大值,即K的最优值为3。绘制K=3的群体遗传结构图(图 5),结果表明171只梅花鹿种公鹿来自于3个祖先基因型。
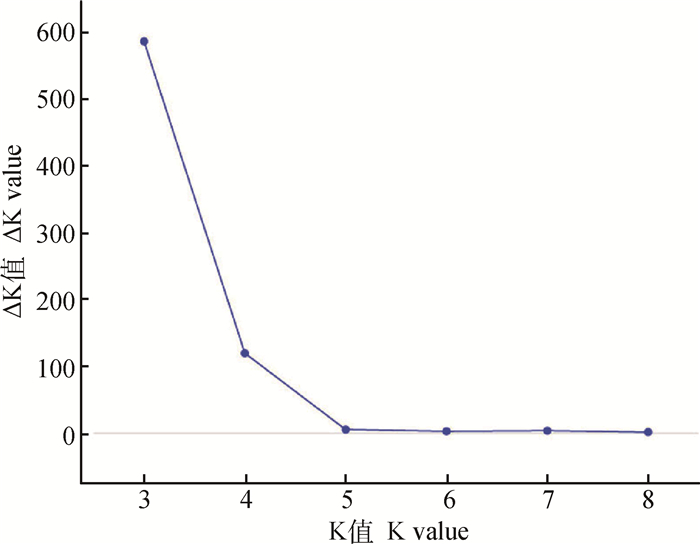
|
图 4 171只梅花鹿种公鹿的最优K值 Figure 4 The optimal K value of 171 breeder sika deer |
|
3种颜色分别代表 3种父系类型 Three colours represent three paternal types 图 5 171只梅花鹿种公鹿的群体遗传结构 Figure 5 Population genetic structure of 171 breeder sika deer |
本试验对171只梅花鹿种公鹿Y染色体的AMELY1、AMELY2、DBY、USP9Y、SRY基因进行PCR扩增测序,得到长度为708、877、1 086、581、1 523 bp的基因片段,并分别检测到3、2、1、1、1个突变位点。AMELY、DBY、USP9Y、SRY基因的Hd值分别为0.368、0.234、0.384、0.401,π值分别为0.001 90、0.001 46、0.001 06、0.000 96。AMELY基因共有5个突变位点,且具有较高的π值,而DBY、USP9Y、SRY基因均只有1个突变位点,且具有较低的π值,这可能是由基因功能的不同所导致的,AMELY基因是牙釉质蛋白基因[25],DBY和USP9Y基因与精子生成有关[26-27],SRY基因则是性别决定基因[28-29]。从整体来看,本研究中梅花鹿种公鹿Y染色体的5个基因片段的Hd和π低于吴华等[17]对我国东北区驯养梅花鹿线粒体的Hd=0.889,π=0.031 73,一方面因为生产实践中梅花鹿的配种方式为单公群母,另一方面因为种公鹿的选择会参考重要产茸性能,人工选择强度远远大于种母鹿。
10种单倍型中,H1所占频率为49.1%,是优势单倍型,为所有梅花鹿种公鹿群体共有。H5、H6、H7、H8、H9、H10所占的频率分别为0.59%、3.5%、2.9%、1.75%、1.17%、0.59%,为稀有单倍型,其中H5和H10为东丰梅花鹿种公鹿独有,H6存在于兴凯湖梅花鹿种公鹿和双阳梅花鹿种公鹿,H7存在于东大梅花鹿种公鹿和四平梅花鹿种公鹿,H8存在于东大梅花鹿种公鹿和双阳梅花鹿种公鹿,H9存在于东大梅花鹿种公鹿和敖东梅花鹿种公鹿。各单倍型频率相差悬殊的现象很可能是养殖企业只关注种公鹿的重要产茸性能并长期对其定向选择造成的,而单公群母的配种方式更会导致稀有单倍型慢慢消失。某群体种公鹿的遗传结构代表了该群体未来几年的遗传结构,为防止稀有单倍型慢慢消失而造成的遗传多样性降低,养殖企业应在保证生产需求的情况下,根据该群体种公鹿的单倍型分布情况进行针对性的配种,从而缩小各单倍型之间的频率差距,扩大稀有单倍型在梅花鹿群体中的比例,以达到保护我国梅花鹿遗传多样性与种质资源的目的。
单倍型系统进化树和群体结构分析表明,中国梅花鹿种公鹿存在3种父系类型,分别为Y1、Y2和Y3,其中Y1由H9组成,是最原始的父系类型。而H9仅存在于东大梅花鹿种公鹿和敖东梅花鹿种公鹿,且在两种公鹿群体所占频率仅为1.7%和6.3%,为防止该原始单倍型在梅花鹿群体中消失,应针对性的选用该单倍型的种公鹿进行配种,以增加该单倍型的后代,达到保护原始单倍型的目的。另外,除双阳梅花鹿种公鹿和四平梅花鹿种公鹿均来自Y3,其余梅花鹿种公鹿均为多父系类型,而某个群体的生产鹿绝大部分是该群体种公鹿的后代,所以本试验对梅花鹿种公鹿父系类型的研究结果,也可代表梅花鹿群体的父系类型,为防止我国梅花鹿种质资源因近交而引起的衰退,同一父系类型内部的梅花鹿应尽量减少基因交流,而相同单倍型的梅花鹿更应避免近交。
由图 3可知,家养梅花鹿群体间存在着基因交流和基因渗入现象,这很可能是养殖企业引种杂交所引起的,为了避免引种杂交造成的种质资源退化,养殖企业应将科学研究与生产实践结合起来,建立梅花鹿群体内部种公鹿的表型性状、生产性状和遗传性状体系,并制定长远的选种选配方案,逐步提高该群体的遗传多样性,提高梅花鹿群体的环境适应能力与进化潜力,发挥我国家养梅花鹿的种质资源优势,培育出更优秀的梅花鹿新品种。
4 结论本研究结果表明,171只梅花鹿种公鹿的单倍型多样性为0.696 80,核苷酸多样性为0.000 36,表明我国梅花鹿种公鹿Y染色体具有较高的单倍型多样性和极低的核苷酸多样性。NJ系统进化树和Structure群体结构分析的结果表明,梅花鹿种公鹿有3个父系类型,分别为Y1、Y2和Y3,除双阳梅花鹿种公鹿和四平梅花鹿种公鹿均来自Y3,其余梅花鹿种公鹿群体均为多父系类型。
| [1] | IMMEL U D, KLEIBER M, KLINTSCHAR M. Y chromosome polymorphisms and haplotypes in South Saxony-Anhalt (Germany)[J]. Forensic Sci Int, 2005, 155(2-3): 211–215. DOI: 10.1016/j.forsciint.2005.01.004 |
| [2] | SEO Y, TAKAMI Y, NAKAYAMA T, et al. Y chromosome DNA polymorphisms and their haplotypes in a Japanese population[J]. Leg Med, 1999, 1(3): 145–149. DOI: 10.1016/S1344-6223(99)80027-3 |
| [3] | UNDERHILL P A, PASSARINO G, LIN A A, et al. Maori origins, Y-chromosome haplotypes and implications for human history in the Pacific[J]. Hum Mutat, 2001, 17(4): 271–280. DOI: 10.1002/(ISSN)1098-1004 |
| [4] |
常振华, 卫利选, 张润锋, 等. 中国黄牛Y-SNPs遗传多样性与起源研究[J]. 畜牧兽医学报, 2011, 42(11): 1537–1542.
CHANG Z H, WEI L X, ZHANG R F, et al. Genetic diversity and origin based on Y-SNPs in Chinese cattle[J]. Acta Veterinaria et Zootechnica Sinica, 2011, 42(11): 1537–1542. (in Chinese) |
| [5] | SU B, XIAO C J, DEKA R, et al. Y chromosome haplotypes reveal prehistorical migrations to the Himalayas[J]. Hum Genet, 2000, 107(6): 582–590. DOI: 10.1007/s004390000406 |
| [6] | LI R, XIE W M, CHANG Z H, et al. Y chromosome diversity and paternal origin of Chinese cattle[J]. Mol Biol Rep, 2013, 40(12): 6633–6636. DOI: 10.1007/s11033-013-2777-y |
| [7] | LI R, WANG S Q, XU S Y, et al. Novel Y-chromosome polymorphisms in Chinese domestic yak[J]. Anim Genet, 2014, 45(3): 449–452. DOI: 10.1111/age.2014.45.issue-3 |
| [8] | ZHANG G, VAHIDI S M F, MA Y H, et al. Limited polymorphisms of two Y-chromosomal SNPs in Chinese and Iranian sheep[J]. Anim Genet, 2012, 43(4): 479–480. DOI: 10.1111/age.2012.43.issue-4 |
| [9] | NIEMI M, BLÄUER A, ISO-TOURU T, et al. Mitochondrial DNA and Y-chromosomal diversity in ancient populations of domestic sheep (Ovis aries) in Finland:comparison with contemporary sheep breeds[J]. Genet Sel Evol, 2013, 45: 2. DOI: 10.1186/1297-9686-45-2 |
| [10] | ZHANG M, PENG W F, YANG G L, et al. Y chromosome haplotype diversity of domestic sheep (Ovis aries) in northern Eurasia[J]. Anim Genet, 2014, 45(6): 903–907. DOI: 10.1111/age.2014.45.issue-6 |
| [11] | WALLNER B, VOGL C, SHUKLA P, et al. Identification of genetic variation on the horse Y chromosome and the tracing of male founder lineages in modern breeds[J]. PLoS One, 2013, 8(4): e60015. DOI: 10.1371/journal.pone.0060015 |
| [12] | BRANDARIZ-FONTES C, LEONARD J A, VEGA-PLA J L, et al. Y-chromosome analysis in retuertas horses[J]. PLoS One, 2013, 8(5): e64985. DOI: 10.1371/journal.pone.0064985 |
| [13] | HAN H Y, ZHANG Q, GAO K X, et al. Y-single nucleotide polymorphisms diversity in Chinese indigenous horse[J]. Asian-Australas J Anim Sci, 2015, 28(8): 1066–1074. DOI: 10.5713/ajas.14.0784 |
| [14] | RAMÍREZ O, OJEDA A, TOMÀS A, et al. Integrating Y-chromosome, mitochondrial, and autosomal data to analyze the origin of pig breeds[J]. Mol Biol Evol, 2009, 26(9): 2061–2072. DOI: 10.1093/molbev/msp118 |
| [15] | CLIFFE K M, DAY A E, BAGGA M, et al. Analysis of the non-recombining Y chromosome defines polymorphisms in domestic pig breeds:ancestral bases identified by comparative sequencing[J]. Anim Genet, 2010, 41(6): 619–629. DOI: 10.1111/age.2010.41.issue-6 |
| [16] |
王兆龙. 实验动物贵州白香猪母系及父系遗传检测[D]. 贵州: 贵州大学, 2009.
WANG Z L. Matrilineal and patrilineal genetic detection on laboratory animalization of Guizhou white Xiang pig[D]. Guizhou: Guizhou University, 2009. (in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10657-2010070816.htm |
| [17] |
吴华, 胡杰, 方盛国, 等. 中国圈养梅花鹿的遗传多样性和遗传结构[J]. 动物学杂志, 2006, 41(4): 41–47.
WU H, HU J, FANG S G, et al. Genetic diversity and genetic structure of domestic Sika deer in China[J]. Chinese Journal of Zoology, 2006, 41(4): 41–47. (in Chinese) |
| [18] |
李欢霞. 梅花鹿线粒体基因组全序列测定及种群遗传结构分析[D]. 镇江: 江苏科技大学, 2012.
LI H X. The sequencing of the mitochondrial complete genome of Sika deer (Cervus nippon) and genetic structure among populations[D]. Zhenjiang: Jiangsu University of Science and Technology, 2012. (in Chinese) http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=D222507 |
| [19] |
周盼伊. 利用Y染色体基因分析家养梅花鹿的父系类型[D]. 北京: 中国农业科学院, 2016.
ZHOU P Y. Using the Y chromosome gene analysis the paternal type of Cervus nippon[D]. Beijing: Chinese Academy of Agricultural Sciences, 2016. (in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-82101-1016171335.htm |
| [20] | TAMURA K, STECHER G, PETERSON D, et al. MEGA6:molecular evolutionary genetics analysis version 6.0[J]. Mol Biol Evol, 2013, 30(12): 2725–2729. DOI: 10.1093/molbev/mst197 |
| [21] | LIBRADO P, ROZAS J. DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 2009, 25(11): 1451–1452. DOI: 10.1093/bioinformatics/btp187 |
| [22] | FELSENSTEIN J. Confidence limits on phylogenies:an approach using the bootstrap[J]. Evolution, 1985, 39(4): 783–791. DOI: 10.1111/j.1558-5646.1985.tb00420.x |
| [23] | PRITCHARD J K, STEPHENS M, DONNELLY P. Inference of population structure using multilocus genotype data[J]. Genetics, 2000, 155(2): 945–959. |
| [24] | TAJIMA F. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism[J]. Genetics, 1989, 123(3): 585–595. |
| [25] | OIDA S, NAGANO T, YAMAKOSHI Y, et al. Amelogenin gene expression in porcine odontoblasts[J]. J Dent Res, 2002, 81(2): 103–108. DOI: 10.1177/0810103 |
| [26] | SESSION D R, LEE G S, WOLGEMUTH D J. Characterization of D1Pas1, a mouse autosomal homologue of the human AZFa region DBY, as a nuclear protein in spermatogenic cells[J]. Fertil Steril, 2001, 76(4): 804–811. DOI: 10.1016/S0015-0282(01)01996-3 |
| [27] | LEE K H, SONG G J, KANG I S, et al. Ubiquitin-specific protease activity of USP9Y, a male infertility gene on the Y chromosome[J]. Reprod Fertil Dev, 2003, 15(1-2): 129–133. |
| [28] | WHITFIELD L S, LOVELL-BADGE R, GOODFELLOW P N. Rapid sequence evolution of the mammalian sex-determining gene SRY[J]. Nature, 1993, 364(6439): 713–715. DOI: 10.1038/364713a0 |
| [29] |
李讨讨, 马友记, 赵兴绪. SOX9基因调控雄性哺乳动物生殖活动的研究进展[J]. 畜牧兽医学报, 2017, 48(6): 971–978.
LI T T, MA Y J, ZHAO X X. Research progress on reproduction regulation of SOX9 gene in male mammals[J]. Acta Veterinaria et Zootechnica Sinica, 2017, 48(6): 971–978. (in Chinese) |



