2. 中国农业科学院北京畜牧兽医研究所, 北京 100193;
3. 信阳师范学院大别山农业生物资源保护与利用研究院, 信阳 464000
2. Institute of Animal Science, Chinese Academy of Agricultural Sciences, Beijing 100193, China;
3. Institute for Conservation and Utilization of Agro-bioresources in Dabie Mountains, Xinyang Normal University, Xinyang 464000, China
c-Src作为非受体型酪氨酸蛋白激酶的一员,能专一性磷酸化蛋白底物中的Tyr残基,几乎存在于所有多细胞动物体内。c-Src蛋白的结构在不同物种间高度保守,主要包含N末端的肉豆蔻酰基的膜结合序列、2个介导蛋白-蛋白相互作用的结构域(SH3和SH2)、1个保守的酪氨酸激酶结构域(PTK)以及负责调控c-Src激酶活性的C-末端[1]。SH3可以与富含脯氨酸序列的靶蛋白互作,影响靶蛋白的结合与定位,同时对c-Src自身的催化能力的调节有重要作用;SH2能识别并结合含磷酸化酪氨酸的短序列,是c-Src蛋白调控磷酸化酪氨酸信号通路的重要元件[2-3]。SH2和SH3结构域以及自身的Tyr416/527磷酸化一起调节c-Src的催化活性。在正常细胞和肿瘤细胞中,c-Src直接与多种生长因子、细胞因子、激素受体以及涉及细胞黏附和迁移的信号转导因子和激活剂发生作用,参与激活或抑制p38 MAPK、Grb2-Ras-Raf-MEK-ERK、PI3K/AKT、FAX-paxillin-p130-Cas以及STAT等信号通路,调节细胞生长、骨架改变、分化、生存、黏附以及迁移等[4-7]。
hnRNPK (Heterogeneous nuclear ribonucleoprotein K)是一个多功能蛋白,参与调控转录、特定mRNA的翻译,或作为支架蛋白整合不同信号传导通路的信号级联,在骨骼肌发育过程中亦起着重要作用[8-10]。hnRNPK蛋白主要包含3个可以结合RNA或DNA的KH (K-homology domain)结构域和1个与蛋白质结合的KI (K protein interactive region)结构域,而在N端和C端分别有1个NLS(Nuclear localization sequence)和KNS(Nuclear shuttling domain)结构域[11]。在这些结构域的介导下,hnRNPK可以在核与胞浆间穿梭,通过与RNA、DNA及蛋白质的相互作用而参与多个细胞过程,在生长发育中发挥重要作用[12-14]。其中,hnRNPK蛋白KI结构域中的脯氨酸富集区(RXXPXXP和PXXPXR)可以特异地与SH3结构域结合,尤其是酪氨酸激酶Src家族蛋白的SH3结构域,从而影响激酶活性[15];而c-Src能磷酸化hnRNPK位于KH3结构域上的Tyr458位点,磷酸化后将阻碍KH3结构域与RNA‘UCCC’Motif的结合,从而影响其对mRNA翻译水平的调控[16]。因此,hnRNPK蛋白和c-Src蛋白间的相互作用对二者生物学功能有着重要影响。
目前,关于猪hnRNPK和c-Src蛋白的研究还很少,本研究分离克隆猪hnRNPK和c-Src基因,利用酵母双杂交和GST-Pull down技术从体内外研究它们的互作关系,为进一步揭示hnRNPK和c-Src在猪中的分子功能提供了研究基础。
1 材料与方法 1.1 试验材料组织样品:淮南猪6月龄背最长肌组织,样品放入RNase Free的1.5 mL离心管中后迅速投于液氮中,而后转入-80 ℃保存备用。
菌株:E. coli DH5α、E. coli BL21和酵母AH109菌株由本实验室保存。
载体:酵母双杂交载体(pGBKT7 BD和pGADT7 AD)、酵母双杂交阳性对照载体(pGBKT7 BD-p53和pGADT7 AD-SV40)、酵母双杂交阴性对照载体(pGBKT7 BD-lam和pGADT7 AD-SV40)和GST pull-down载体(pGEX-6p1和pET28a)为本实验室保存。
试剂:构建载体所需限制性内切酶、T4连接酶、RNAiso Plus、反转录试剂盒购自TaKaRa公司;Glutathione MagBeads、Ni-Chraged Magbeads购自南京金斯瑞生物科技有限公司;鲑鱼精DNA、聚乙二醇3350购自Sigma公司;一抗(hnRNPK、c-Src和GST)购自Santa Cruz公司;HPR标记的二抗、BeyoECL Plus购自碧云天公司。
1.2 总RNA提取及cDNA合成采用Trizol法(RNAiso Plus,TaKaRa,No. 9109)从肌肉提取RNA,用TaKaRa公司的反转录试剂盒(No. 6110A)对提取的RNA进行反转录,具体操作步骤参照说明书进行。
1.3 利用PCR和酶切连接法构建酵母双杂交质粒通过对GenBank公布的猪c-Src基因(XM_021077973.1)和hnRNPK基因(XM_021064018.1)的mRNA序列进行分析,用Premier 5.0软件设计1对引物(pGBKT7-hnRNPK,表 1)用于扩增猪hnRNPK基因的完整编码区,设计5对引物(表 1)用于扩增猪c-Src基因的完整编码区及不同结构域缺失片段。根据酵母质粒载体pGPBKT7与pGADT7上的限制性酶切位点,扩增hnRNPK基因,引物上下游分别加入EcoRⅠ和BamHⅠ的酶切位点;而扩增c-Src基因引物上下游分别加入EcoR I和Xho I酶切位点。PCR产物经胶回收、双酶切后分别克隆至pGBKT7及pGADT7中,转化E. coli DH5α。菌液PCR筛选阳性克隆并测序验证,获得序列正确的载体质粒即为重组载体pGBKT7-hnRNPK、pGADT7-c-Src、pGADT7-ΔSH3、pGADT7-ΔSH2、pGADT7-ΔPTKc和pGADT7-SH3。
|
|
表 1 载体构建引物信息 Table 1 Primers used for vector construction |
对c-Src和hnRNPK基因序列进行分析,与酵母双杂交相同,均采用编码区作为插入目的片段,用Premier 5.0软件设计1对引物(pET28a-hnRNPK,表 1)用于扩增猪hnRNPK基因的完整编码区,设计5对引物(表 1)用于扩增猪c-Src基因的完整编码区及不同结构域缺失片段(引物同酵母双杂交pGADT7质粒构建)。根据GST pull-down所需质粒pET28a和pGEX-6p1的限制性酶切位点,扩增hnRNPK基因,引物上下游分别加入BamH I和Xho I酶切位点,而扩增c-Src基因引物上下游分别加入EcoR I和Xho I酶切位点。PCR产物经胶回收后双酶切并克隆至pET28a和pGEX-6p1中,转化E. coli DH5α中,菌液PCR筛选阳性克隆并测序验证,获得序列正确的载体质粒即为重组载体pET28a-hnRNPK与pGEX-6p1-c-Src、pGEX-6p1-ΔSH3、pGEX-6p1-ΔSH2、pGEX-6p1-ΔPTKc和pGEX-6p1-SH3。
1.5 酵母双杂交检测酵母双杂交系统(Yeast Two-hybrid System,Y2H)是将待研究的两种蛋白质分别融合到酵母表达质粒的转录激活因子(如GAL4等)的DNA结合结构域(DNA-binding domain,BD)和转录激活域(Transcription-activating domain,AD)上,构建成融合表达载体,从表达产物分析两种蛋白质相互作用的系统,是一种在活细胞体内研究蛋白质与蛋白质之间相互作用的有效方法[17-19]。采用醋酸锂法制备酵母AH109感受态细胞,首先从冻存的酵母菌种刮取接种于YPDA(全营养培养基)平板培养,挑单个菌落接种于3 mL YPDA培养基中,30 ℃,200~220 r·min-1培养8 h;然后接5 μL培养物于50 mL YPDA培养基中,30 ℃,200~220 r·min-1培养16~20 h;OD600 nm达到0.2~0.3后,离心收集细胞并重悬浮于100 mL YPDA,然后30 ℃,200~220 r·min-1培养3~5 h;OD600 nm达到0.4~0.6,离心收集细胞,用60 mL灭菌水重悬,然后再次离心收集并重悬于3 mL 1.1×TE-LiAc中,转入离心管,高速短暂离心15 s,重新悬浮于600 μL 1.1×TE-LiAc中,即为酵母感受态细胞。
利用PEG-LiAc法转化AH109细胞,在1.5 mL离心管中加入0.1 μg质粒DNA、0.1 mg蛙鱼精DNA、0.1 mL酵母感受态细胞和0.6 mL PEG-LiAc溶液,混合均匀;30 ℃水浴30 min,然后加入70 μL DMSO,轻轻混匀,42 ℃水浴热激15 min后,12 000 r·min-1离心15 s,去上清,加入1 mL YPDA培养基,30 ℃培养60 min;再次12 000 r·min-1离心15 s,去上清,加入1 mL无菌水悬浮细胞,取100 μL细胞悬液平铺于相应的SD选择性培养基上,30 ℃培养3~4 d;挑取单克隆并接种于5 mL液体双缺培养基中,30 ℃,220 r·min-1培养至OD600 nm=1.0,3 000 r·min-1离心5 min收集酵母菌体,加入1 mL无菌水重悬,测定OD600 nm值,并根据具体浓度将其稀释到OD600 nm=1.0;最后将酵母菌液按10-1、10-2和10-3浓度梯度稀释,并将不同浓度的酵母菌液滴在双缺和四缺SD培养基上,30 ℃生长4~5 d。根据酵母生长情况判断其自激活活性及蛋白之间的相互作用,而后进一步通过检测β-半乳糖苷酶活性来检测相互作用的强弱。
1.6 GST pull-down检测GST pull-down(GST融合蛋白沉降技术)是靶蛋白与GST融合表达,利用GST对谷胱甘肽(GSH)偶联磁珠的亲和性,将GST融合蛋白固化在谷胱甘肽亲和树脂上,经层析从目的蛋白溶液中捕获与之相互作用的蛋白,从而证实两种蛋白间的相互作用或筛选相应的目的蛋白,是体外研究蛋白质互作的主要手段[20]。融合蛋白的提取,将在16或37 ℃用100 mmol·L-1 IPTG诱导表达8 h的细菌培养物于5 000 r·min-1,4 ℃离心10 min,收集细胞,弃上清,用预冷的PBS重新悬浮细胞,然后5 000 r·min-1,4 ℃离心10 min,收集细胞;把细胞放于-80 ℃冻1 h,再解冻细胞,用预冷的PBS重悬,然后超声波破碎,直到细胞不黏稠;12 000 r·min-1,4 ℃离心10 min,将上清移到一个新的预冷的离心管,用预冷的PBS重悬沉淀物;最后将等量的10 μL样品(可溶的和难溶的),加等量的2×SDS上样缓冲液,煮沸5 min,用于SDS-PAGE分析。
融合蛋白的纯化(亲和层析法),用前先把磁珠(Glutathione MagBeads或Ni-Chraged Magbeads)溶液混匀,取100 μL磁珠加入1.5 mL离心管,再加入1 mL PBS,上下颠倒混匀,用磁性分离架分离磁珠,弃上清液,重复此步骤3次以上;然后向离心管加入100 μL PBS重悬磁珠,再加入50~1 000 μL提取的蛋白样品溶液,室温振荡孵育1 h后用磁性分离架分离磁珠;再加入1 mL PBS,充分混匀,磁性分离架分离磁珠,重复此步骤3次;最后向离心管中加入100 μL谷胱甘肽洗脱缓冲液,充分混匀,室温孵育5 min,磁性分离架分离磁珠,将上清移入一新的预冷的离心管,保存备用。
取GST-c-Src、GST-ΔSH3、GST-ΔSH2、GST-ΔPTKc、GST-SH3和GST标签蛋白各1 mg,在4 ℃条件下分别与5 mg His-hnRNPK融合蛋白在100 μL结合缓冲液中孵育1 h;然后向反应混合物中加入20 μL Glutathione MagBeads,4 ℃冷室摇床上孵育1 h;用50倍体系预冷的PBS清洗Glutathione MagBeads 3次,再将磁珠用100 μL SDS-PAGE上样缓冲液重悬,并在95 ℃处理10 min;最后取20 μL处理好的样品用于10% SDS-PAGE分析和免疫印迹检测。
1.7 数据分析β-半乳糖苷酶活性测定时每个样品重复3次,活性值用“平均值±标准差”表示。试验数据通过SPSS 18.0软件进行显著性分析,显著性分析方法采用LSD法(最小显著性差异法),分别用“*”和“**”表示与对照相比差异显著(P<0.05)和极显著(P<0.01)。
2 结果 2.1 酵母双杂交pGBKT7-hnRNPK和c-Src不同缺失体pGADT7质粒构建以猪肌肉总RNA为模板反转录cDNA,通过PCR扩增,获得目的基因片段hnRNPK的完整编码区1 392 bp,包含两个酶切位点;c-Src基因的完整编码区及不同缺失体分别为1 629 bp(全长)、1 182 bp(SH3结构域缺失)、870 bp(SH3和SH2结构域缺失)、804 bp(PTKc结构域缺失)和168 bp(SH3结构域),包含两个酶切位点(图略)。然后将这些片段分别克隆到pGBKT7和pGADT7酵母双杂交载体中(图 1A和1B)。经测序验证插入序列的正确性后,利用质粒试剂盒抽提质粒。以上结果表明,酵母双杂交pGBKT7-hnRNPK和c-Src不同缺失体pGADT7载体构建成功,可以用于酵母双杂交试验。
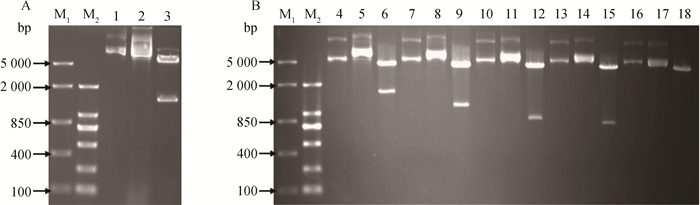
|
A.琼脂糖凝胶电泳检测猪hnRNPK基因的BD融合重组质粒载体;B.琼脂糖凝胶电泳检测猪c-Src及缺失体的AD融合重组质粒载体:M1. DL5000 DNA相对分子质量标准;M2. DL2000 DNA相对分子质量标准(2 000、1 000、750、500、250、100 bp);1. pGBKT7质粒;2. pGBKT7-hnRNPK质粒;3. pGBKT7-hnRNPK质粒双酶切;4、7、10、13和16. pGADT7质粒;5. pGADT7-c-Src质粒;6. pGADT7-c-Src质粒双酶切;8. pGADT7-ΔSH3质粒;9. pGADT7-ΔSH3质粒双酶切;11. pGADT7-ΔSH2质粒;12. pGADT7-ΔSH2质粒双酶切;14. pGADT7-ΔPTKc质粒;15. pGADT7-ΔPTKc质粒双酶切;17. pGADT7-SH3质粒;18. pGADT7-SH3质粒双酶切 A. Electrophoresis results of porcine hnRNPK BD recombinant plasmid on agarose gel; B. Electrophoresis results of porcine c-Src AD recombinant plasmid on agarose gel; M1. DL5000 DNA marker; M2. DL2000 DNA marker(2 000, 1 000, 750, 500, 250, 100 bp); 1. pGBKT7 plasmid; 2. pGBKT7-hnRNPK plasmid; 3. The double enzyme digestion of pGBKT7-hnRNPK plasmid; 4, 7, 10, 13, 16. pGADT7 plasmid; 5. pGADT7-c-Src plasmid; 6. The double enzyme digestion of pGADT7-c-Src plasmid; 8. pGADT7-ΔSH3 plasmid; 9. The double enzyme digestion of pGADT7-ΔSH3 plasmid; 11. pGADT7-ΔSH2 plasmid; 12. The double enzyme digestion of pGADT7-ΔSH2 plasmid; 14. pGADT7-ΔPTKc plasmid; 15. The double enzyme digestion of pGADT7-ΔPTKc plasmid; 17. pGADT7-SH3 plasmid; 18. The double enzyme digestion of pGADT7-SH3 plasmid 图 1 猪hnRNPK基因和c-Src基因及缺失体的酵母双杂交质粒构建 Figure 1 The construction of yeast two-hybrid plasmids of porcine hnRNPK and different mutants of c-Src |
为了检验体内条件下,hnRNPK和c-Src相互作用情况,我们进行了酵母双杂交试验,将转化pGBKT7-hnRNPK质粒的酵母AH109细胞和转化c-Src不同缺失体pGADT7质粒的酵母AH109细胞分别涂布在SD-Trp-Leu和SD-His-Ade平板上,结果不能生长,His、Trp、Ade、Leu是酵母体内含有的特殊报告基因,说明pGBKT7-hnRNPK和c-Src不同缺失体pGADT7质粒不具有自激活宿主菌的作用,表明构建的pGBKT7-hnRNPK和c-Src不同缺失体pGADT7质粒能够适用于酵母双杂交系统进行蛋白相互作用的验证。
将hnRNPK分别和c-Src的不同缺失体pGADT7质粒同时转入酵母AH109细胞中,再分别涂布在SD-Trp-Leu和SD-His-Trp-Leu-Ade平板上。在SD-Trp-Leu平板上均有酵母单菌落生长,表明AD和BD载体已成功转入酵母中(图 2);在SD-Trp-Leu-His-Ade平板上,转入阴性对照的酵母不能在平板上生长,转入阳性对照的酵母生长良好,表明酵母双杂系统可以正常工作。同时,转入pGBKT7-hnRNPK与pGADT7-ΔSH3、pGBKT7-hnRNPK与pGADT7-ΔSH2的酵母也不能在平板上生长,表明c-Src蛋白的SH3结构域缺失以及SH3和SH2结构域共同缺失都不能激活酵母双杂交系统;转入pGBKT7-hnRNPK与pGADT7-c-Src、pGBKT7-hnRNPK与pGADT7-ΔPTKc、pGBKT7-hnRNPK与pGADT7-SH3的酵母能正常生长,表明hnRNPK和c-Src之间存在蛋白互作,其中位于c-Src激酶N端的SH3结构域在互作中起着关键作用,SH2结构域与hnRNPK互作较弱或不存在互作(图 2)。为了确定hnRNPK和c-Src蛋白互作强度,进一步测定了β-半乳糖苷酶活性,结果表明,阴性对照在本底水平上存在一定的β-半乳糖苷酶活性((0.80±0.09) Miller units),阳性对照β-半乳糖苷酶活性最高为(10.84±1.01) Miller units,c-Src、SH3和ΔPTKc的活性较高分别为(3.96±0.43)、(2.51±0.26)和(1.96±0.15) Miller units,均极显著高于阴性对照(P<0.01);ΔSH3和ΔSH2的β-半乳糖苷酶活性均较低,表现为阴性对照相似的本底水平活性(图 2)。这些结果进一步表明,hnRNPK和c-Src之间存在直接的相互作用,互作区域主要标记在c-Src激酶N端的SH3结构域。
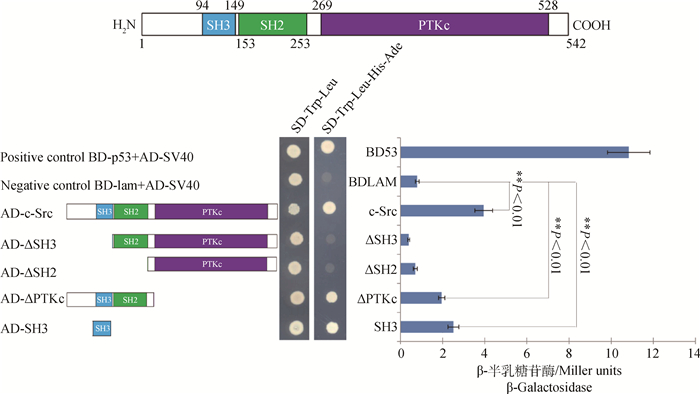
|
图 2 hnRNPK与c-Src在酵母细胞中的相互作用 Figure 2 Interaction between hnRNPK and c-Src in the yeast cells |
为了验证hnRNPK与c-Src蛋白质的体外互作,构建了hnRNPK和c-Src基因及缺失体的原核表达载体,并进行诱导表达及纯化。首先利用PCR扩增hnRNPK基因CDS和c-Src基因CDS及缺失体(c-Src、PTKc缺失、SH2缺失、SH3缺失和SH3结构域),分别克隆到pGEX-6p-1和PET28a原核表达载体中(图 3A和3B)。经测序验证插入序列的正确性后,将载体转化到BL21大肠杆菌,利用IPTG诱导表达重组目的蛋白(图 3D)。经SDS-PAGE检测,发现各重组蛋白实际分子量和理论分子量大小一致,初步证明了目的蛋白的成功表达;进一步利用Western blotting验证了诱导后产生的蛋白,用hnRNPK和c-Src特异抗体检测到His-hnRNPK、GST-c-Src、GST-ΔSH3和GST-ΔSH2的表达,但没有检测到GST-ΔPTKc和GST-SH3(c-Src抗体的抗原表位在c-Src的C端,由于它们的C端缺失,因此检测不到);而用GST标签抗体检测c-Src及其缺失体的表达,GST、GST-c-Src、GST-ΔSH3、GST-ΔSH2、GST-c-Src-ΔPTKc和GST-SH3均能检测到,表明这些融合蛋白均成功表达(图 3D)。
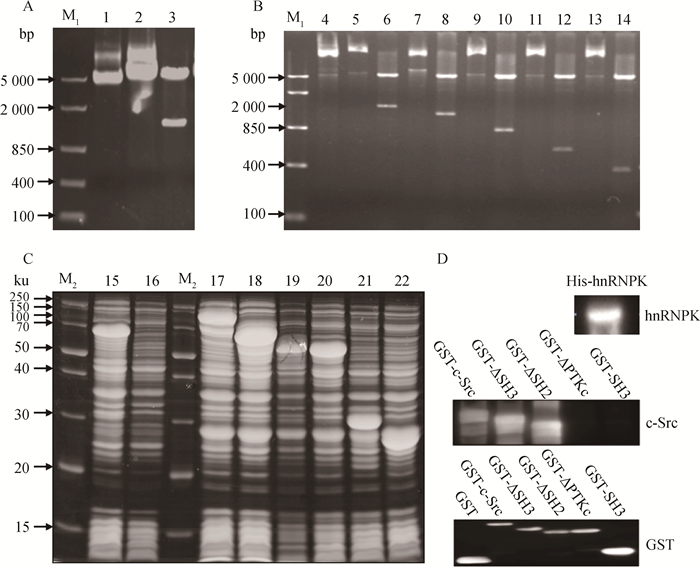
|
A.琼脂糖凝胶电泳检测猪hnRNPK重组载体;B.琼脂糖凝胶电泳检测猪c-Src及缺失体重组载体;C.融合蛋白SDS-PAGE电泳检测;D.蛋白质印迹:M1. DL5000 DNA相对分子质量标准;M2.蛋白质相对分子质量标准;1. pET28-a质粒;2. pET28-a-hnRNPK质粒;3. pET28-a-hnRNPK质粒双酶切;4. pGEX-6p1质粒;5. pGEX-6p1-c-Src质粒;6. pGEX-6p1-c-Src质粒双酶切;7. pGEX-6p1-ΔSH3质粒;8. pGEX-6p1-ΔSH3质粒双酶切;9. pGEX-6p1-ΔSH2质粒;10. pGEX-6p1-ΔSH2质粒双酶切;11. pGEX-6p1-ΔPTKc质粒;12. pGEX-6p1-ΔPTKc质粒双酶切;13. pGEX-6p1-SH3质粒;14. pGEX-6p1-SH3质粒双酶切;15. His-hnRNPK;16. His;17. GST-c-Src;18. GST-ΔSH3;19. GST-ΔSH2;20. GST-ΔPTKc;21. GST-SH3;22. GST A. Electrophoresis results of porcine hnRNPK prokaryotic expression recombinant plasmid on agarose gel; B. Electrophoresis results of porcine c-Src and mutants prokaryotic expression recombinant plasmid on agarose gel; C. Detection of fusion protein by SDS-PAGE; D. Western bloting: M1. DL5000 DNA marker; M2. Protein marker; 1. pET28-a plasmid; 2. pET28-a-hnRNPK plasmid; 3. The double enzyme digestion of pET28-a-hnRNPK plasmid; 4. pGEX-6p1 plasmid; 5. pGEX-6p1-c-Src plasmid; 6. The double enzyme digestion of pGEX-6p1-c-Src plasmid; 7. pGEX-6p1-ΔSH3 plasmid; 8. The double enzyme digestion of pGEX-6p1-ΔSH3 plasmid; 9. pGEX-6p1-ΔSH2 plasmid; 10. The double enzyme digestion of pGEX-6p1-ΔSH2 plasmid; 11. pGEX-6p1-ΔPTKc plasmid; 12. The double enzyme digestion of pGEX-6p1-ΔPTKc plasmid; 13. pGEX-6p1-SH3 plasmid; 14. The double enzyme digestion of pGEX-6p1-SH3 plasmid; 15. His-hnRNPK; 16. His; 17. GST-c-Src; 18. GST-ΔSH3; 19. GST-ΔSH2; 20. GST-ΔPTKc; 21. GST-SH3; 22. GST 图 3 猪hnRNPK和c-Src基因及缺失体原核表达载体的构建和重组蛋白的表达纯化 Figure 3 The construction of prokaryotic expression vector and the expression and purification of recombinant protein of porcine hnRNPK and different mutants of c-Src |
然后再利用GST融合蛋白纯化介质和Ni-IDA亲和层析介质分别纯化带GST和His标签重组蛋白,检测发现,His-hnRNPK和GST-SH3有较少的杂带,其他都较纯,说明纯化效果较好,可以用于后续的GST pull-down分析。
2.4 GST pull-down验证hnRNPK与c-Src蛋白的相互作用c-Src含有SH3、SH2和PTKc等3个重要的功能性结构域,通过构建不同截短的缺失体,诱导全长及截短蛋白的表达,将纯化好的带His-hnRNPK融合蛋白与结合在GST亲和树脂(beads)上的c-Src及其缺失体GST融合蛋白一起孵育,然后用清洗液充分清洗,再经SDS-PAGE胶分离结合在树脂上的蛋白,通过考马斯亮蓝染色(CBB-R250)与免疫印迹检测分析发现,GST-c-Src和GST-ΔPTKc在体外与His-hnRNPK较强相互作用,GST-ΔSH3、GST-ΔSH在体外与His-hnRNPK的相互作用则较弱,但作为阴性对照的GST标签蛋白则不能与His-hnRNPK相互作用(图 4)。以上结果表明,猪hnRNPK蛋白与c-Src的相互作用是特异的,互作的区域主要位于c-Src激酶的N端,这与酵母双杂交结果是一致的。
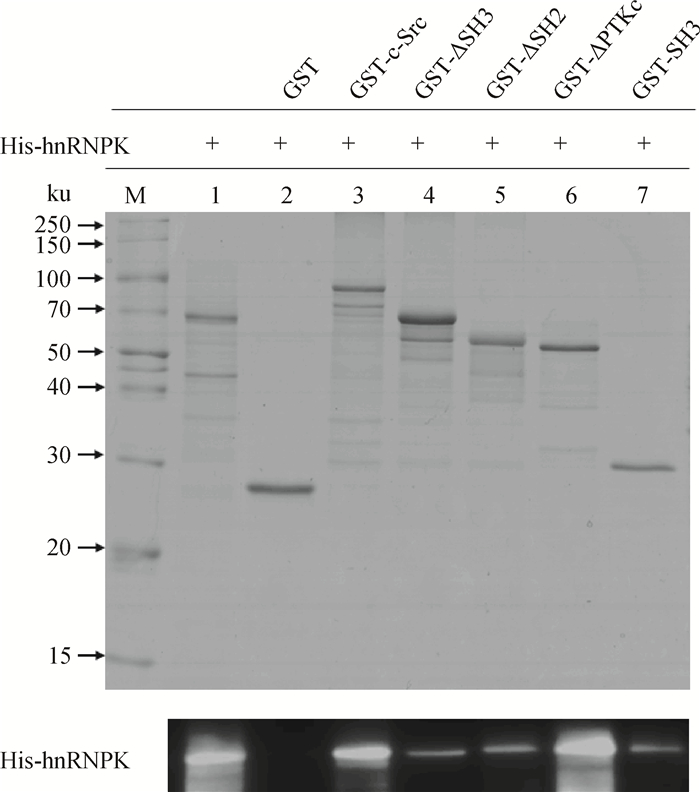
|
1. His-hnRNPK纯化产物;2~7. GST pull-down试验中SDS-PAGE上样缓冲液洗脱物 1. The purified product of His-hnRNPK; 2-7. The SDS-PAGE loading buffer elution in GST pull-down assay 图 4 GST pull-down验证猪hnRNPK与c-Src体外蛋白相互作用 Figure 4 Interaction between porcine hnRNPK and c-Src in vitro by GST pull-down analysis |
c-Src可以通过胰岛素和生长因子信号通路对细胞增殖分化、葡萄糖运输、糖原合成、蛋白质合成等生物学过程起着重要调节作用[21]。hnRNPK作为一个多功能蛋白,参与染色质重塑、转录、翻译的调控以及多种信号转导过程[22]。ERK介导的hnRNPK蛋白的丝氨酸磷酸化修饰可以促进其在细胞质的集聚表达[23]。而在细胞质中hnRNPK可以与c-Src特意结合并激活c-Src激酶活性,同时也可作为酪氨酸磷酸化底物,其中hnRNPK位于KH3结构域上的Tyr458位点磷酸化后将阻碍KH3结构域与RNA‘UCCC’模体的结合,从而影响其对mRNA的翻译水平的调控[13, 15]。hnRNPK与c-Src激酶均在动物肌肉发育中起着重要调控作用,例如,M. J. Lim等[24-25]研究发现,抑制c-Src的活性可以抑制Raf/MEK/ERK通路,却激活了p38 MAPK通路,从而抑制了成肌细胞增殖并促进分化,而这种抑制随着p38 MAPK通路的失活而解除;hnRNPK Ser284/353位点的磷酸化能促进hnRNPK在分化C2C12成肌细胞中特异集聚表达,并调节成肌分化[8]。因此,对hnRNPK蛋白与c-Src激酶的相互作用的分析有助于揭示hnRNPK与c-Src在骨骼肌成肌分化中相互调控的分子机制及其介导的复杂信号通路。D. Adolph等[26]运用体外BiFC(双分子荧光互补技术)和GST-pull down技术分析了人hnRNPK蛋白及不同缺失体与c-Src的在体内外的互作情况,发现hnRNPK蛋白主要通过KI结构域与c-Src相互作用,而位于KI结构域中的富含脯氨酸模体(218-242位氨基酸)对互作及激活c-Src激酶活性至关重要。但c-Src激酶具有多个功能域,其中哪一段或哪一结构域在与hnRNPK蛋白互作中发挥作用尚不清楚。
酵母双杂交技术是一种在活细胞内检测蛋白质-蛋白质相互作用的经典方法,本研究运用该技术证实了多功能蛋白hnRNPK与c-Src激酶的相互作用,并分析了c-Src激酶主要功能域的不同缺失体与互作情况,结果表明,位于c-Src蛋白N-端的SH3结构域是互作的关键,SH3结构域和PTKc结构域的互作效应则比较弱。为了排除酵母双杂交技术中的假阳性,进一步采用了GST pull-down的方法在体外对两者之间的相互作用进行鉴定。通过分别将hnRNPK蛋白与c-Src蛋白及不同功能域缺失体用标签蛋白树脂洗脱纯化下来,在体外研究hnRNPK蛋白与c-Src蛋白不同缺失体的相互作用关系,结果与酵母双杂交是一致的。因此,利用酵母双杂交技术和GST pull-down技术从体内外条件下证实了猪hnRNPK与c-Src互作,并将c-Src蛋白的互作区定位在c-Src蛋白N端的SH3结构域,这些验证了前期在小鼠和人中hnRNPK与c-Src蛋白互作的结果[15, 26-28],并加深了对二者相互作用的认识,也为进一步研究两者协同表达具体的作用机制以及如何调控猪骨骼肌生长发育奠定了基础。
4 结论通过酵母双杂交和GST pull-down技术,从体内外对猪hnRNPK蛋白与c-Src激酶之间的相互作用作了分析,不仅加深了蛋白质之间相互作用方法的认识,而且将c-Src蛋白的互作区定位在c-Src蛋白N端的SH3结构域,两种技术互为补充,互相印证,更加确定了结果的真实可靠。但是两者相互调控的具体作用机制以及如何调节骨骼肌成肌分化的问题还有待更深入一步的研究。
| [1] | MARTIN G S. The hunting of the Src[J]. Nat Rev Mol Cell Biol, 2001, 2(6): 467–475. DOI: 10.1038/35073094 |
| [2] | YOUNG M A, GONFLONI S, SUPERTI-FURGA G, et al. Dynamic coupling between the SH2 and SH3 domains of c-Src and Hck underlies their inactivation by C-terminal tyrosine phosphorylation[J]. Cell, 2001, 105(1): 115–126. DOI: 10.1016/S0092-8674(01)00301-4 |
| [3] | ZHANG J, WANG S L, JIANG B, et al. c-Src phosphorylation and activation of hexokinase promotes tumorigenesis and metastasis[J]. Nat Commun, 2017, 8: 13732. DOI: 10.1038/ncomms13732 |
| [4] | JIN W, YUN C, JEONG J, et al. c-Src is required for tropomyosin receptor kinase C (TrkC)-induced activation of the phosphatidylinositol 3-kinase (PI3K)-AKT pathway[J]. J Biol Chem, 2008, 283(3): 1391–1400. DOI: 10.1074/jbc.M705052200 |
| [5] | HAMZEH M T, SRIDHARA R, ALEXANDER L D. Cyclic stretch-induced TGF-β1 and fibronectin expression is mediated by β1-integrin through c-Src-and STAT3-dependent pathways in renal epithelial cells[J]. Am J Physiol Renal Physiol, 2015, 308(5): F425–F436. DOI: 10.1152/ajprenal.00589.2014 |
| [6] | TRUFFI M, DUBREUIL V, LIANG X, et al. RPTPα controls epithelial adherens junctions, linking E-cadherin engagement to c-Src-mediated phosphorylation of cortactin[J]. J Cell Sci, 2014, 127(Pt 11): 2420–2432. |
| [7] | YANG Y, DING L, GUO Z K, et al. The epigenetically-regulated miR-34a targeting c-SRC suppresses RAF/MEK/ERK signaling pathway in K-562 cells[J]. Leuk Res, 2017, 55: 91–96. DOI: 10.1016/j.leukres.2017.01.020 |
| [8] | PUENTE L G, VOISIN S, LEE R E C, et al. Reconstructing the regulatory kinase pathways of myogenesis from phosphopeptide data[J]. Mol Cell Proteomics, 2006, 5(12): 2244–2251. DOI: 10.1074/mcp.M600134-MCP200 |
| [9] | GONNET F, BOUAZZA B, MILLOT G A, et al. Proteome analysis of differentiating human myoblasts by dialysis-assisted two-dimensional gel electrophoresis (DAGE)[J]. Proteomics, 2008, 8(2): 264–278. DOI: 10.1002/(ISSN)1615-9861 |
| [10] | XU Y J, QIAN H, FENG X T, et al. Differential proteome and transcriptome analysis of porcine skeletal muscle during development[J]. J Proteomics, 2012, 75(7): 2093–2108. DOI: 10.1016/j.jprot.2012.01.013 |
| [11] | BARBORO P, FERRARI N, BALBI C. Emerging roles of heterogeneous nuclear ribonucleoprotein K (hnRNP K) in cancer progression[J]. Cancer Lett, 2014, 352(2): 152–159. DOI: 10.1016/j.canlet.2014.06.019 |
| [12] | LIU Y Y, GERVASI C, SZARO B G. A crucial role for hnRNP K in axon development in Xenopus laevis[J]. Development, 2008, 135(18): 3125–3135. DOI: 10.1242/dev.022236 |
| [13] | OSTARECK-LEDERER A, OSTARECK D H. Precision mechanics with multifunctional tools:how hnRNP K and hnRNPs E1/E2 contribute to post-transcriptional control of gene expression in hematopoiesis[J]. Curr Protein Pept Sci, 2012, 13(4): 391–400. DOI: 10.2174/138920312801619484 |
| [14] | THOMPSON P J, DULBERG V, MOON K M, et al. hnRNP K coordinates transcriptional silencing by SETDB1 in embryonic stem cells[J]. PLoS Genet, 2015, 11(1): e1004933. DOI: 10.1371/journal.pgen.1004933 |
| [15] | OSTARECK-LEDERER A, OSTARECK D H, RUCKNAGEL K P, et al. Asymmetric arginine dimethylation of heterogeneous nuclear ribonucleoprotein K by protein-arginine methyltransferase 1 inhibits its interaction with c-Src[J]. J Biol Chem, 2006, 281(16): 11115–11125. DOI: 10.1074/jbc.M513053200 |
| [16] | LIEPELT A, MOSSANEN J C, DENECKE B, et al. Translation control of TAK1 mRNA by hnRNP K modulates LPS-induced macrophage activation[J]. RNA, 2014, 20(6): 899–911. DOI: 10.1261/rna.042788.113 |
| [17] | BAKER M. Proteomics:The interaction map[J]. Nature, 2012, 484(7393): 271–275. DOI: 10.1038/484271a |
| [18] |
袁维峰, 吴保明, 张鑫宇, 等. 应用酵母双杂交系统初步筛选IBDV VP2结合蛋白[J]. 畜牧兽医学报, 2009, 40(6): 958–962.
YUAN W F, WU B M, ZHANG X Y, et al. Preliminary screening candidate IBDV VP2-binding proteins by yeast two hybrid system[J]. Acta Veterinaria et Zootechnica Sinica, 2009, 40(6): 958–962. (in Chinese) |
| [19] |
陈大雷, 朱文娟, 杨述林, 等. 酵母双杂交筛选猪CALSARCIN-3基因相互作用蛋白的研究[J]. 畜牧兽医学报, 2009, 40(11): 1594–1599.
CHEN D L, ZHU W J, YANG S L, et al. Yeast two hybrid study of pig Calsarcin-3 interaction proteins[J]. Acta Veterinaria et Zootechnica Sinica, 2009, 40(11): 1594–1599. DOI: 10.3321/j.issn:0366-6964.2009.11.004 (in Chinese) |
| [20] | WISSMUELLER S, FONT J, LIEW C W, et al. Protein-protein interactions:Analysis of a false positive GST pulldown result[J]. Proteins, 2011, 79(8): 2365–2371. DOI: 10.1002/prot.23068 |
| [21] | MOLLER L, DALMAN L, NORRELUND H, et al. Impact of fasting on growth hormone signaling and action in muscle and fat[J]. J Clin Endocr Metab, 2009, 94(3): 965–972. DOI: 10.1210/jc.2008-1385 |
| [22] | GALLARDO M, HORNBAKER M J, ZHANG X R, et al. Aberrant hnRNP K expression:All roads lead to cancer[J]. Cell Cycle, 2016, 15(12): 1552–1557. DOI: 10.1080/15384101.2016.1164372 |
| [23] | HABELHAH H, SHAH K, HUANG L, et al. ERK phosphorylation drives cytoplasmic accumulation of hnRNP-K and inhibition of mRNA translation[J]. Nat Cell Biol, 2001, 3(3): 325–330. DOI: 10.1038/35060131 |
| [24] | LIM M J, SEO Y H, CHOI K J, et al. Suppression of c-Src activity stimulates muscle differentiation via p38 MAPK activation[J]. Arch Biochem Biophys, 2007, 465(1): 197–208. DOI: 10.1016/j.abb.2007.06.004 |
| [25] | ROSOFF W J, SWOPE S L. Role for cellular Src kinase in myoblast proliferation[J]. J Cell Physiol, 2002, 193(3): 328–339. DOI: 10.1002/(ISSN)1097-4652 |
| [26] | ADOLPH D, FLACH N, MUELLER K, et al. Deciphering the cross talk between hnRNP K and c-Src:the c-Src activation domain in hnRNP K is distinct from a second interaction site[J]. Mol Cell Biol, 2007, 27(5): 1758–1770. DOI: 10.1128/MCB.02014-06 |
| [27] | MORITZ B, LILIE H, NAARMANN-DE VRIES I S, et al. Biophysical and biochemical analysis of hnRNP K:arginine methylation, reversible aggregation and combinatorial binding to nucleic acids[J]. Biol Chem, 2014, 395(7-8): 837–853. |
| [28] | OSTARECK-LEDERER A, OSTARECK D H, CANS C, et al. C-Src-mediated phosphorylation of hnRNP K drives translational activation of specifically silenced mRNAs[J]. Mol Cell Biol, 2002, 22(13): 4535–4543. DOI: 10.1128/MCB.22.13.4535-4543.2002 |



