噬菌体是一种专性寄生于细菌的病毒,作为细菌的天敌,在自然界中广泛存在,其数量达1031个[1]。在19世纪初,噬菌体曾广泛用于细菌病的防治,随后抗生素的发现及其广谱抗菌特性导致各国噬菌体的研究工作停滞。然而近年来,细菌耐药情况日益严重,新型抗生素发现和开发进程进展缓慢,人们注意到使用抗生素存在着不可忽视的缺陷[2-3]。因此,噬菌体作为抗生素替代品又重新引起关注[4-5]。噬菌体裂解细菌特异性强,且不受细菌耐药性限制,已成为科学研究的热点,广泛应用于病原体检测、食品和环境消毒、细菌性疾病防控等领域[6-7]。
噬菌体种类繁多,其中T-even类(T2、T4、T6等)噬菌体能够裂解肠杆菌科(Enterobacteriaceae)细菌。目前GenBank数据库中已经登录了300多种能够裂解大肠杆菌的噬菌体,其中37种属于T4类噬菌体[8]。T4类噬菌体属于有尾噬菌体科,其尾部结构复杂,包括基板、6个短尾丝和6个长尾丝[9]。在感染宿主菌过程中,T4类噬菌体长尾丝末端的受体识别蛋白能够识别并吸附宿主菌表面受体,启动噬菌体侵染过程[10-11]。T4类噬菌体长尾丝的受体识别蛋白主要有两种:在T4噬菌体中,gp37蛋白位于长尾丝末端,在伴侣分子gp57和gp38的作用下形成三聚体结构,是识别受体的关键蛋白[12-13];在T2和T6噬菌体中,gp38蛋白作为结构蛋白与gp37相邻,位于长尾丝末端,是识别受体的关键蛋白[14]。
笔者在前期研究工作中分离到一株鸡源大肠杆菌噬菌体Bp7,基因组测序结果显示其属肌尾噬菌体科T4类噬菌体[15-16]。为了检测噬菌体Bp7的gp38蛋白在噬菌体吸附宿主菌过程中的作用,本试验在原核系统表达gp38蛋白,分析其序列特征,测定其生物学活性,并对其进行噬菌体表面定位,以期为明确噬菌体Bp7的吸附机制提供理论基础。
1 材料与方法 1.1 材料 1.1.1 噬菌体及其宿主菌噬菌体Bp7为鸡源大肠杆菌噬菌体,属肌尾噬菌体科,T4类噬菌体,已完成基因组测序(GenBank登录号:NC_019500.1),由青岛农业大学微生物实验室保存。宿主菌为E.coli K12,用于噬菌体Bp7的增殖,由青岛农业大学微生物实验室保存。
1.1.2 引物及主要试剂限制性内切酶、T4 DNA连接酶和PCR产物回收试剂盒购自TaKaRa公司,质粒提取试剂盒、胶回收试剂盒和His-Ni蛋白纯化柱购自康为世纪有限公司。根据噬菌体Bp7基因组注释的gp38基因序列,设计gp38上、下游引物,序列分别如下,gp38F:5'-CAGGAGCTCATGGCAGTAGTAGGAAT-3', gp38R:5'-GGGCTCGAGTTAA-TACTTTCGTATTAT-3'(下划线分别表示限制性酶切位点SacⅠ和XhoⅠ), 由上海生工生物工程技术服务有限公司合成。
1.2 方法 1.2.1 噬菌体Bp7蛋白gp38序列分析根据噬菌体Bp7全基因组注释信息,获得预测的gp38氨基酸序列,与NCBI网站GenBank中已登录的其他噬菌体尾丝蛋白gp38序列进行同源性比对,分析噬菌体Bp7蛋白gp38与其他噬菌体的亲缘关系;选取相似性较高的7个序列及有代表性的2个序列,利用DNAman软件对尾丝蛋白gp38的氨基酸序列进行保守区域分析,阐明其序列特征。选取的序列及其GenBank登录号分别为Shf125875(YP_009100789.1)、vB_EcoM_VR5(YP_009205945.1)、RB51(YP_002854204.1)、RB69(NP_861945.1)、PHAPEC2(YP_009056827.1)、JS10(YP_002922591.1)、JS98(YP_001595373.1)、T6(AAC61977.1)、T2(CAA28935.1)。
1.2.2 gp38基因的克隆及重组表达质粒的构建以噬菌体Bp7基因组DNA为模板,PCR扩增gp38基因,预期目的基因大小为780 bp。PCR反应条件:95 ℃预变性5 min;94 ℃变性1 min,55 ℃退火1 min,72 ℃延伸1 min,35个循环;72 ℃延伸10 min。1%琼脂糖凝胶电泳检测PCR产物,送上海生工生物工程技术服务有限公司测序。将鉴定正确的基因片段插入原核表达载体pColdTF中,构建原核表达质粒pColdTF-gp38,分别进行PCR和双酶切鉴定,测序。
1.2.3 重组蛋白gp38的表达及抗体制备鉴定正确的重组质粒pColdTF-gp38转化大肠杆菌感受态细胞BL21,制备可表达gp38蛋白的工程菌pColdTF-gp38/BL21。挑取单菌落,接入LB液体培养基中(含100 μg·mL-1氨苄青霉素),16 ℃条件下振荡培养至对数生长期,加入0.6 mmol·L-1的IPTG诱导表达12 h,4 000 r·min-1离心10 min收集菌体沉淀,PBS洗涤3次,重悬菌体。超声裂解菌体,分离上清和沉淀,SDS-PAGE电泳检测重组蛋白的表达形态,亲和层析法纯化蛋白,进行Western blot鉴定[17]。鉴定正确的纯化蛋白gp38免疫家兔,4免后收集血清,琼扩试验检测蛋白gp38抗体效价。
1.2.4 重组蛋白gp38在噬菌体表面的定位分析10 μL噬菌体Bp7增殖液与10 μL gp38抗血清混匀,4 ℃条件下孵育4 h。取10 μL孵育液滴加于铜网上,吸附10 min,PBS洗涤3次。磷钨酸染色3 min, 自然干燥后,电镜观察gp38蛋白在噬菌体Bp7表面的定位。
1.2.5 重组蛋白gp38的受体识别活性分析将200 μL过夜培养的宿主菌E. coli K12与200 μL纯化蛋白gp38(0.2 mg·mL-1)混匀,在37 ℃条件下孵育10 min,同时以等量PBS代替纯化蛋白作为对照。加入100 μL适宜浓度的噬菌体Bp7增殖液,37 ℃条件下作用5 min,双层平板法检测噬菌斑生长情况。对照组和试验组各设3个重复。根据噬菌斑数量计算噬菌体Bp7相对吸附率,检测尾丝蛋白gp38竞争抑制噬菌体Bp7吸附宿主菌E. coli K12作用。
取100 μL适宜浓度的噬菌体Bp7增殖液,加入100 μL gp38抗血清混匀,同时设阴性血清为对照,在37 ℃条件下孵育10 min。加入200 μL新鲜增殖的E. coli K12菌液混匀,37 ℃条件下作用5 min,双层平板法检测噬菌斑生长情况。对照组和试验组各设3个重复。根据噬菌斑数量计算噬菌体Bp7相对吸附率,检测gp38蛋白抗体阻断噬菌体Bp7吸附宿主菌E. coli K12作用。
噬菌体相对吸附率=试验组噬菌斑数/对照组噬菌斑数×100%。
2 结果 2.1 噬菌体Bp7蛋白gp38序列分析噬菌体Bp7蛋白gp38氨基酸序列与GenBank数据库中其他噬菌体的gp38序列进行相似性比对,结果显示噬菌体Bp7尾丝蛋白gp38序列与T4类噬菌体尾丝蛋白序列相似性高,其中与噬菌体Shf125875的尾丝蛋白氨基酸序列相似性最高(97%),与噬菌体T6、T2的相似性较低(59%、33.9%),与T4噬菌体无同源性。根据相似性比对结果,选取相似性较高的序列,对噬菌体Bp7尾丝蛋白gp38进行进化分析,结果见图 1。噬菌体Bp7尾丝蛋白gp38与其他7株噬菌体亲缘关系较近,同属一个进化分支,与另外4个分支的噬菌体遗传距离较远。
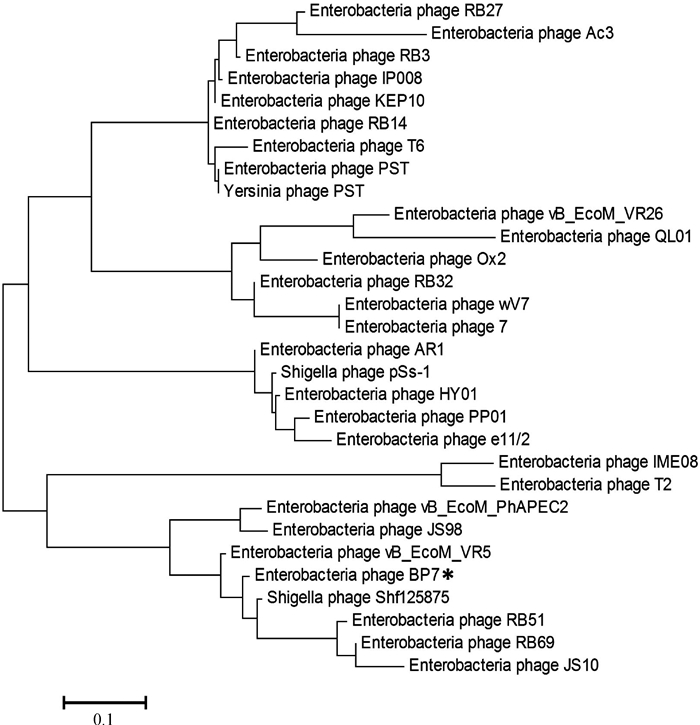
|
图 1 尾丝蛋白gp38序列同源性分析 Figure 1 Homology analysis of gp38 amino acid sequence |
用DNAman软件对噬菌体Bp7尾丝蛋白gp38及其亲缘关系较近的7株噬菌体gp38蛋白序列以及有代表性的T2、T6噬菌体gp38蛋白序列进行比对,结果见图 2。由图 2可知,gp38蛋白N端序列保守性相对较强,C端序列高度变异。在C端存在4个高度保守的富甘氨酸保守区(glycine-rich motifs, GRM),将C端序列分割成五个高变区(hypervariable segments, HVS),呈现出马赛克特征。

|
图 2 尾丝蛋白gp38氨基酸序列特征分析 Figure 2 Character analysis of gp38 amino acid sequence |
以噬菌体Bp7的基因组序列为模板,设计引物,PCR扩增目的基因gp38,测序结果表明获得了正确的gp38基因产物。将鉴定正确的gp38基因片段插入原核表达载体pColdTF中构建原核表达质粒pColdTF-gp38,进行PCR和双酶切鉴定,结果见图 3。由图可知,在大约780 bp处出现目的条带,与预期片段大小相同。

|
M.DNA相对分子质量标准(DL2000); 1. pColdTF-gp38 PCR扩增; 2. pColdTF-gp38双酶切鉴定 M. DNA marker (DL2000); 1. PCR product of pColdTF-gp38; 2. Double enzyme digestion product of pColdTF-gp38 图 3 pColdTF-gp38的PCR与双酶切鉴定 Figure 3 PCR and double enzyme digestion products of recombined plasmid pColdTF-gp38 |
重组质粒pColdTF-gp38转入大肠杆菌BL21中,IPTG低温诱导,进行SDS-PAGE电泳检测和Western blot鉴定,结果见图 4。由图 4可知,在85 ku左右出现预期目的条带,蛋白在上清中表达量较高,为可溶性表达。亲和层析法纯化蛋白后,免疫家兔,4免后采血,检测血清中抗体效价达1: 16。
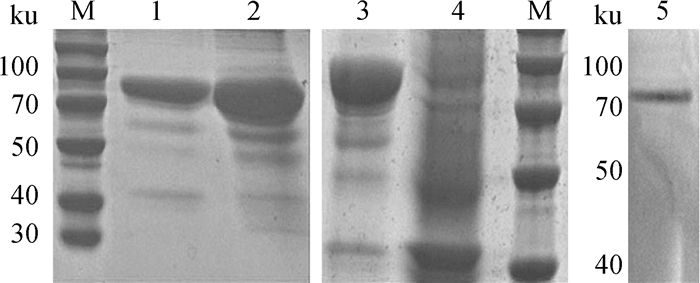
|
M.蛋白质相对分子质量标准; 1. pColdTF-gp38/BL21菌体裂解沉淀; 2. pColdTF-gp38/BL21菌体裂解上清; 3.重组质粒pColdTF-gp38的诱导表达; 4.空载体质粒pColdTF的诱导表达; 5. gp38蛋白Western blot检测 M. Protein marker; 1. The precipitation of pColdTF-gp38/BL21; 2. The supernatant of pColdTF-gp38/BL21; 3. The expression of recombinant plasmid pColdTF-gp38; 4. The expression of plasmid pColdTF; 5. Western blot of gp38 protein 图 4 重组蛋白gp38的SDS-PAGE及Western blot鉴定 Figure 4 SDS-PAGE and Western blot analysis of recombined protein gp38 |
利用尾丝蛋白gp38制备的抗血清与噬菌体Bp7相互作用后,进行免疫电镜观察,结果见图 5。由图 5可知,加入gp38蛋白的抗血清后,噬菌体Bp7的长尾丝相对,聚集在一起,说明gp38蛋白的抗血清能够与噬菌体Bp7的长尾丝结合,引起噬菌体的聚集,表明蛋白gp38位于噬菌体Bp7的尾丝末端,是尾丝蛋白的组成部分。
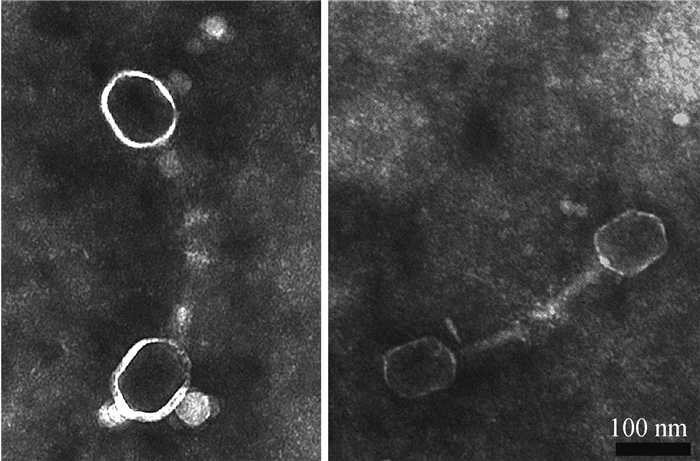
|
图 5 重组蛋白gp38免疫电镜观察 Figure 5 Immuno-electron microscope observation of recombined protein gp38 |
gp38蛋白与E. coli K12孵育后,加入噬菌体Bp7,检测噬菌体Bp7吸附宿主菌E. coli K12的效果,结果见图 6。由图 6可知,gp38蛋白组噬菌体Bp7相对吸附率为0,说明gp38蛋白能够与E. coli K12表面受体结合,使噬菌体Bp7不能结合到宿主菌表面受体,完全竞争抑制噬菌体Bp7对宿主菌E. coli K12的吸附。
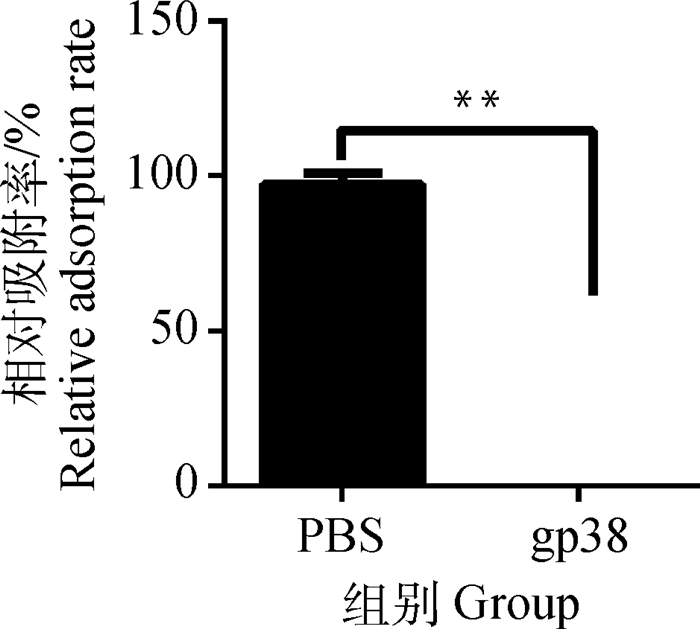
|
图 6 gp38蛋白抑制噬菌体Bp7吸附作用 Figure 6 gp38 protein inhibits the adsorption of phage Bp7 |
gp38抗血清与噬菌体Bp7孵育后,加入宿主菌E. coli K12,检测噬菌体Bp7吸附效果,结果见图 7。由图 7可知,gp38抗血清组噬菌体Bp7相对吸附率为0,说明gp38抗血清能够与噬菌体Bp7的蛋白gp38特异性结合,从而对噬菌体起到封闭作用,使噬菌体尾丝蛋白无法识别宿主菌的受体,完全阻断噬菌体Bp7对宿主菌E. coli K12的吸附。
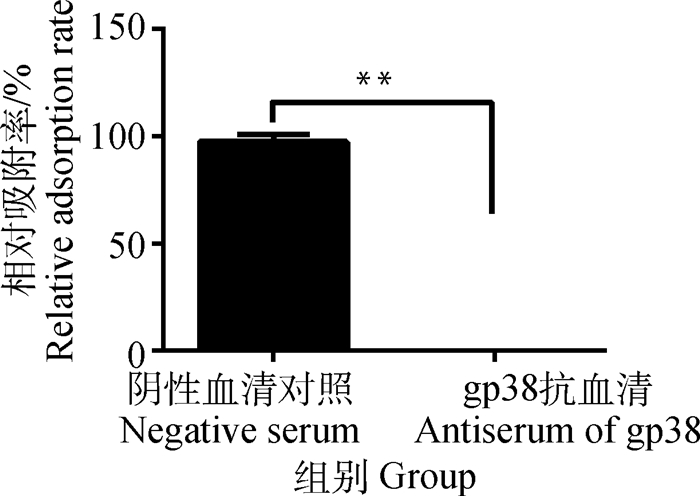
|
图 7 gp38抗血清阻断噬菌体Bp7吸附作用 Figure 7 Antiserum of gp38 protein blocks the adsorption of phage Bp7 |
噬菌体作为细菌的天敌,具有潜在的开发价值和广阔的应用前景,近年来备受关注[18-19]。有尾噬菌体gp38蛋白在不同的噬菌体中具有不同的作用。在T4噬菌体中,gp38作为伴侣分子,保证受体识别蛋白gp37形成正确的三聚体空间构象,而在T2、T6噬菌体中,gp38是关键的受体识别蛋白[20]。本试验对噬菌体Bp7的gp38蛋白开展研究,明确其在噬菌体Bp7吸附过程中的作用。
噬菌体Bp7是一株从鸡场分离的大肠杆菌噬菌体,具有宽宿主谱的特征,属于肌尾噬菌体科、T4类噬菌体,与T4噬菌体全基因组序列相似性达76.56%[15]。但是,本试验噬菌体Bp7蛋白gp38氨基酸序列比对分析发现其与T4噬菌体gp38蛋白几乎没有同源性,而与T2、T6噬菌体的亲缘关系较近。对其序列特征进一步分析发现,gp38蛋白序列C端变异程度高,4个GRM区与5个HVS区间隔排列,呈现出马赛克特征,这与之前对gp38序列报道结果一致[20]。因此,可以推测噬菌体Bp7蛋白gp38与T2、T6噬菌体的gp38蛋白相似,可能作为受体识别蛋白在噬菌体吸附宿主菌过程中具有关键作用。本试验在原核系统中可溶性表达了噬菌体Bp7蛋白gp38,并制备了相应多克隆抗体。gp38蛋白竞争抑制试验及抗体阻断试验证实,两者均能完全阻止噬菌体Bp7的吸附过程,这表明gp38蛋白在噬菌体Bp7吸附过程中具有关键作用。同时,通过免疫电镜观察,gp38抗体能够与噬菌体Bp7长尾丝结合,引发噬菌体聚集,说明gp38蛋白位于噬菌体Bp7的长尾丝末端,这表明噬菌体Bp7蛋白gp38与T2噬菌体尾丝蛋白gp38功能相似,是噬菌体Bp7关键的受体识别蛋白。
本试验证实尾丝蛋白gp38在噬菌体Bp7识别宿主菌过程中具有重要作用,gp38蛋白特征决定了其能够吸附宿主菌受体的种类,进而决定了噬菌体Bp7的宿主谱。Drexler等[21]推测GRM构成gp38蛋白的结构核心,HVS形成Ω环,识别宿主菌表面受体。对不同噬菌体gp38的序列比对发现与HVS序列相似的噬菌体其宿主范围相同,利用同源重组替换HVS序列可以改变噬菌体宿主谱,这进一步证实了HVS序列是gp38的受体结合区域[20]。因此,尾丝蛋白gp38的HVS结构特征决定了噬菌体Bp7识别受体的种类,进而决定了噬菌体Bp7宽宿主谱的特征,但是gp38蛋白的HVS识别受体的作用机制尚不明确,将在后续工作中进行深入研究。
4 结论噬菌体Bp7蛋白gp38位于长尾丝末端,是噬菌体的受体识别蛋白,在噬菌体识别与吸附宿主菌E.coli K12表面受体过程中具有关键作用,这为后期阐明宽宿主谱噬菌体Bp7识别受体的作用机制提供了理论基础。
| [1] | HATFULL G F, HENDRIX R W. Bacteriophages and their genomes[J]. Current Opin Virol, 2011, 1(4): 298–303. DOI: 10.1016/j.coviro.2011.06.009 |
| [2] | FISCHBACH M A, WALSH C T. Antibiotics for emerging pathogens[J]. Science, 2009, 325(5944): 1089–1093. DOI: 10.1126/science.1176667 |
| [3] | HARDS K, COOK G M. Targeting bacterial energetics to produce new antimicrobials[J]. Drug Resist Updat, 2018, 36: 1–12. DOI: 10.1016/j.drup.2017.11.001 |
| [4] | REARDON S. Phage therapy gets revitalized:The rise of antibiotic resistance rekindles interest in a century-old virus treatment[J]. Nature, 2014, 510(7503): 15–16. DOI: 10.1038/510015a |
| [5] | CRISCUOLO E, SPADINI S, LAMANNA J, et al. Bacteriophages and their immunological applications against infectious threats[J]. J Immunol Res, 2017, 2017: 3780697. |
| [6] | XU Y Q, LIU Y, LIU Y, et al. Bacteriophage therapy against Enterobacteriaceae[J]. Virol Sin, 2015, 30(1): 11–18. |
| [7] | OFIR G, SOREK R. Contemporary phage biology:From classic models to new insights[J]. Cell, 2018, 172(6): 1260–1270. DOI: 10.1016/j.cell.2017.10.045 |
| [8] | BARTUAL S G, OTERO J M, GARCIA-DOVAL C, et al. Structure of the bacteriophage T4 long tail fiber receptor-binding tip[J]. Proc Natl Acad Sci U S A, 2010, 107(47): 20287–20292. DOI: 10.1073/pnas.1011218107 |
| [9] | LEIMAN P G, ARISAKA F, VAN RAAIJ M J, et al. Morphogenesis of the T4 tail and tail fibers[J]. Virol J, 2010, 7: 355. DOI: 10.1186/1743-422X-7-355 |
| [10] | STORMS Z J, SAUVAGEAU D. Modeling tailed bacteriophage adsorption:Insight into mechanisms[J]. Virology, 2015, 485: 355–362. DOI: 10.1016/j.virol.2015.08.007 |
| [11] | HU B, MARGOLIN W, MOLINEUX I J, et al. The bacteriophage T7 virion undergoes extensive structural remodeling during infection[J]. Science, 2013, 339(6119): 576–579. DOI: 10.1126/science.1231887 |
| [12] | MONTAG D, HASHEMOLHOSSEINI S, HENNING U. Receptor-recognizing proteins of T-even type bacteriophages. The receptor-recognizing area of proteins 37 of phages T4 TuIa and TuIb[J]. J Mol Biol, 1990, 216(2): 327–334. DOI: 10.1016/S0022-2836(05)80324-9 |
| [13] | YAP M L, ROSSMANN M G. Structure and function of bacteriophage T4[J]. Future Microbiol, 2014, 9(12): 1319–1327. DOI: 10.2217/fmb.14.91 |
| [14] | RIEDE I, DREXLER K, SCHWARZ H, et al. Teven-type bacteriophages use an adhesin for recognition of cellular receptors[J]. J Mol Biol, 1987, 194(1): 23–30. DOI: 10.1016/0022-2836(87)90712-1 |
| [15] | ZHANG C, LI W L, LIU W H, et al. T4-like phage Bp7, a potential antimicrobial agent for controlling drug-resistant Escherichia coli in chickens[J]. Appl Environ Microbiol, 2013, 79(18): 5559–5565. DOI: 10.1128/AEM.01505-13 |
| [16] | ZHANG C, LI W H, REN H Y. Complete genome sequence of Bp7, an Escherichia coli bacteriophage with a wide host range[J]. J Virol, 2012, 86(24): 13832. DOI: 10.1128/JVI.02631-12 |
| [17] |
齐心, 陈培培, 邹玲, 等. 噬菌体Bp7穿孔素holin的克隆、表达及其活性分析[J]. 畜牧兽医学报, 2017, 48(12): 2392–2398.
QI X, CHEN P P, ZOU L, et al. Cloning, expression and activity analysis of phage Bp7 holin[J]. Acta Veterinaria et Zootechnica Sinica, 2017, 48(12): 2392–2398. (in Chinese) |
| [18] | PIRES D P, MELO L, VILAS BOAS D, et al. Phage therapy as an alternative or complementary strategy to prevent and control biofilm-related infections[J]. Curr Opin Microbiol, 2017, 39: 48–56. DOI: 10.1016/j.mib.2017.09.004 |
| [19] | LIN D M, KOSKELLA B, LIN H C. Phage therapy:An alternative to antibiotics in the age of multi-drug resistance[J]. World J Gastrointest Pharmacol Ther, 2017, 8(3): 162–173. DOI: 10.4292/wjgpt.v8.i3.162 |
| [20] | TROJET S N, CAUMONT-SARCOS A, PERRODY E, et al. The gp38 adhesins of the T4 Superfamily:A complex modular determinant of the phage's host specificity[J]. Genome Biol Evol, 2011, 3: 674–686. DOI: 10.1093/gbe/evr059 |
| [21] | DREXLER K, RIEDE I, MONTAG D, et al. Receptor specificity of the Escherichia coli T-even type phage Ox2:Mutational alterations in host range mutants[J]. J Mol Biol, 1989, 207(4): 797–803. DOI: 10.1016/0022-2836(89)90245-3 |



