副猪嗜血杆菌(Haemophilus parasuis,HPS)为革兰阴性菌,属于巴氏杆菌科(Pasteurellaceae),无运动性,多形态[1-2],主要引起猪的多发性浆膜炎和关节炎[3]。随着猪场的规模化、集约化发展,副猪嗜血杆菌病给养猪业带来巨大的经济损失[4],近年来国内外对于HPS的研究愈来愈热。在对HPS毒力因子、信号通路及致病机制等的相关研究中,常采用自然转化的方法构建基因缺失株和回补株以用于研究相关基因编码的蛋白功能[5]。
细菌将遗传物质从亲代传递给子代的过程称作基因的纵向转移(vertical transfer)。此外,细菌通过另一种基因传递方向——平行基因转移(horizontal gene transfer,HGT)来实现遗传物质水平方向的交流。HGT被认为促进了细菌基因组的可塑性,使细菌可以有效地适应环境中的改变而获得一个更广泛的生存空间,在细菌进化过程中扮演了重要角色[6]。接合转移(conjugation)、转导(transduction)和自然转化(natural transformation)作为细菌平行基因转移的三种模式[7],在耐药性的传播、编码毒素噬菌体的分布以及致病岛(pathogenicity islands)的转移等过程中都发挥着重要作用[8-9]。在自然条件下,细菌主动摄取外源DNA并将其稳定遗传的过程称作自然转化[10]。自1928年Griffith首次发现肺炎链球菌(Streptococcus pneumoniae)的自然转化过程后,陆续有许多细菌被发现具有自然转化能力,这些细菌被称作自然感受态细菌(naturally competent bacteria)[11-12]。
HPS同样展现出了自然转化的能力[5]。自2004年Bigas等[13]以该菌自然转化系统为基础,创建了一套在HPS上的基因操作技术,借此构建该菌的基因缺失株来研究相关基因功能,HPS自然转化的应用便推广开来。与接合转移、转导相比,自然转化的发生很少受到外源DNA的影响及转化方法的局限,如感受态细菌的制备。自然转化只与受体菌的诱导相关, 且操作相对简单[14]。研究表明,自然转化的发生是很多基因精密调控的结果,包括作为核心调控因子的tfox(Sxy)、crp、cyaA,以及作为DNA摄取和同源重组的组件结构的dprA、rec2、四型菌毛系统(pilA、pilB、pilC、pilD、pilF)及comABCD操纵子等[15]。不同细菌,尤其是革兰阴性菌的自然转化能力的系统性调控存在着很大差异,使自然转化机制的研究变得更加困难[14]。近年来,许多研究者采用自然转化的方法构建基因缺失株对相关基因功能进行研究,例如周鹏[16]构建了HAPS_RS00465基因缺失株探究其在HPS生物被膜形成、生长速度方面的功能。然而关于HPS自然转化机制的研究鲜有报道,对于HPS自然转化的调控机制目前尚不清楚。因此,本研究通过对实验室鉴定和保存的HPS田间分离株及15株标准株进行筛选,发现一株高自然转化效率的菌株(SC1401),并对该菌株进行了全基因组测序,分析其基因组特征,以亲缘关系相对较近,且自然转化机制研究相对全面的流感嗜血杆菌Rd KW20菌株[17]的自然感受态相关调控基因为参考,初步挖掘SC1401中与自然转化机制相关的基因,为后续进一步研究HPS自然转化机制提供生物材料,并奠定一定的分子背景基础。
1 材料与方法 1.1 菌株、质粒及试剂HPS田间分离株由四川农业大学动物医学院猪病研究中心分离、鉴定并保存;HPS 15个血清型的标准株由澳大利亚Yeerongpilly动物研究中心细菌学实验室惠赠,并由四川农业大学动物医学院猪病研究中心保存;质粒cheY LKR-pK18由四川农业大学动物医学院猪病研究中心构建和保存;分型血清由四川农业大学猪病研究中心制备、鉴定并保存;胰蛋白胨大豆琼脂(TSA)、胰蛋白胨大豆肉汤(TSB)购自美国BD公司;LA培养基、LB培养基、小牛血清购自Gibco公司;NAD、cAMP、卡那霉素、质粒提取试剂盒、细菌总DNA提取试剂盒购自天根公司。
1.2 引物设计与合成参照何绿琴等[18]构建cheY基因缺失株所用引物,及文献[19]中用于HPS分子分型引物,合成本研究涉及的所有引物(表 1)。HPS、Kan、cheY引物用于亲本株、缺失株的鉴定;其余引物用于HPS血清型的鉴定。
|
|
表 1 本研究所用的引物序列 Table 1 Primers used in this study |
使用cheY LKR-pK18质粒对实验室保存的75株HPS田间分离株和15株标准株进行自然感受态细菌的初步筛选,具体操作参考谨瑾等[20]确定的HPS自然转化最优条件:将受体菌挑单至3 mL TSB液体培养基(添加5%小牛血清和0.01%NAD, TSB++)中,37 ℃摇床培养14 h;取出后将菌液点在TSA固体培养基中(添加5%小牛血清和0.01%NAD, TSA++)平板上,使其分散成直径约为10 mm的圆形斑点,37 ℃培养13 h。用30 μL TSB++重悬菌苔,以调整菌液浓度到5×1010CFU·mL-1;取20 μL重悬液,加入1 μg的外源质粒cheY LKR-pK18(使用等体积的TE buffer做阴性对照[13]),混匀后37 ℃孵育10 min;将混合液(约30 μL)点在TSA++平板上,使其形成10 mm左右的圆斑,37 ℃培养箱中孵育5 h;收集菌体,用100 μL TSB++重悬,涂于含50 μg·mL-1卡那霉素的TSA++平板,37 ℃孵育36~48 h后,对抗性平板上生长的菌落进行PCR鉴定以确定自然转化的发生。
1.3.2 自然转化效率的测定测定初步筛选为自然感受态的田间分离株的转化效率,具体操作如下:前期操作同上,得到用100 μL TSB++重悬的菌苔后,37 ℃孵育5 h,再使用900 μL生理盐水将重悬液进行10倍等比稀释。取100 μL 10-4稀释的菌液涂布于含50 μg·mL-1卡那霉素的TSA++平板,取100 μL 10-7稀释的菌液涂布于不含任何抗生素的TSA++平板,37 ℃孵育36~48 h后进行菌落计数,参考文献中的计数方法[21-22],计算各菌株的自然转化效率(转化效率=阳性转化子的数量/质粒的量),使用与外源质粒等体积的TE buffer作为阴性对照。
1.4 各自然感受态菌株血清型的测定根据已报道的HPS PCR分型方法[19]操作,引物如表 1所示,PCR扩增程序:94 ℃预变性5 min;94 ℃变性30 s,54 ℃复性30 s,68 ℃延伸60 s,30个循环;68 ℃延伸5 min;4 ℃保存。扩增产物在1.5%琼脂糖凝胶中电泳鉴定。
1.5 全基因组测序 1.5.1 DNA的提取挑取“1.3”中所得自然转化效率最高的菌株单菌落于TSB++培养基中,37 ℃,220 r·min-1培养16 h。使用细菌基因组DNA提取试剂盒提取基因组,操作步骤按照试剂盒说明书进行。
1.5.2 基因组测序、组装及注释基因组DNA提取后,进行质量鉴定,在纯度和浓度均达到测序要求后进行全基因组测序。基因组的测序采用PacBio三代测序技术,构建10 kb SMRTbell DNA文库,测序深度为180×。并基于Linux系统平台,利用C语言、C++语言、perl语言以及相应程序、脚本等,开发适用于Haemophilus parasuis基因组分析的软件BacCompMap-Haemophilus parasuis-WYP,在评估其有效性后,对SC1401基因组进行de novo 组装和分析。使用GeneMarks对拼接好的SC1401基因组进行基因功能的预测。为了预测编码蛋白的基因,将全基因组与包括KEGG(Kyoto encyclopedia of genes and genomes)、COG(Clusters of orthologous groups)、NR(非冗余蛋白数据库)、Swiss-Prot、GO(Gene ontology)和TrEMBL在内的六个数据库进行比对。使用RNAmmer和tRNAscan-SE软件分别进行rRNA和tRNA的预测。
1.5.3 全基因组序列的上传与比较基因组学分析将所得菌株的全基因组序列上传GenBank、EMBL、DDBJ数据库中。将该菌株与其他四株完成全基因组测序的副猪嗜血杆菌基因组序列进行比较分析,包括SH0165株(GenBank:CP001321)、SH03株(GenBank:CP009158)、KL0318株(GenBank:CP009237)和ZJ0906株(GenBank:CP005384)。四个菌株的全基因组序列及注释信息均来自GenBank数据库。比较五株菌株基因组的基本特征,并使用Mauve软件进行基因组共线性分析。
1.5.4 自然转化相关基因分析目前自然转化机制在流感嗜血杆菌中研究的较为透彻,且流感嗜血杆菌与副猪嗜血杆菌为亲缘关系相近,同属于巴斯德菌属。将试验所得菌株的测序结果与流感嗜血杆菌Rd KW20菌株进行比对,分析HPS中存在的与自然转化可能相关的基因,并与“1.5.3”中提到的四株完成全基因组测序的HPS进行比对。
2 结果 2.1 高自然转化效力菌株的筛选使用HPS-F/R检测,亲本株与缺失株均产生目的条带,大小为822 bp;使用引物cheY-F/R检测,亲本株有条带,大小为672 bp,缺失株无条带;使用引物Kan-F/R检测,亲本株无条带,缺失株产生条带,大小为935 bp(图 1),证明该菌株为自然感受态细菌。
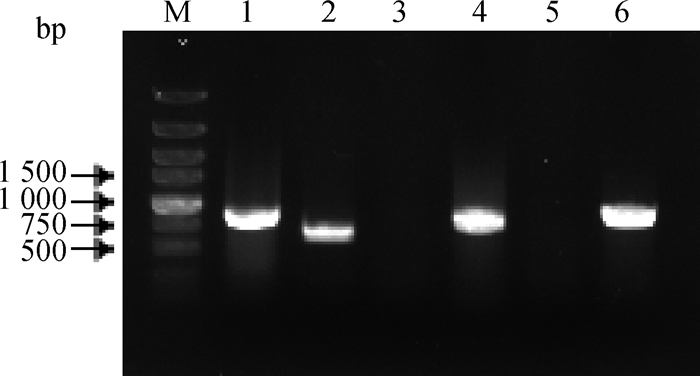
|
M.DNA相对分子质量标准;1、4.引物HPS-F/R鉴定;2、5.引物cheY-F/R鉴定;3、6.引物Kan-F/R鉴定;1~3.亲本菌;4~6.突变株 M. DNA marker; 1, 4. Identification with primers HPS-F/R; 2, 5. Identification with primers cheY-F/R; 3, 6. Identification with primers Kan-F/R; 1-3. SC1401; 4-6. SC1401 ΔcheY::kan 图 1 cheY缺失株的PCR鉴定 Figure 1 Identification of the mutant strain SC1401 ΔcheY::kan by PCR |
对本实验室75株HPS田间分离株及15株标准株进行筛选,共得到11株自然感受态细菌,并测得各菌株的自然转化效率。菌株名称及自然转化效率如表 2所示。其中自然转化效率最高的菌株为SC1401。
|
|
表 2 自然感受态菌株的血清型及其转化效率 Table 2 Serotype and transformation efficiency of natural competence |
使用表 1中的分型引物对所得自然感受态细菌进行血清分型,根据产生特异性条带的引物及目的条带大小判断菌株的血清型,结果如表 2所示。
2.3 基因组组装与注释本研究获得完整的副猪嗜血杆菌SC1401基因组序列,序列全长2 277 540 bp,其GC含量为40.03%,共编码2 220个基因,其占整个基因组序列的87.75%。将其基因组基本信息与其他四株完成全基因组测序的HPS菌株——SH0165株、SH03株、KL0318株和ZJ0906株进行比较,结果如表 3所示。
|
|
表 3 5株副猪嗜血杆菌全基因组比较 Table 3 Genome comparison of 5 HPS strains |
5株副猪嗜血杆菌基因组的基本特征如表 3所示,各菌株在基因组大小、GC含量方面非常相似,在编码蛋白质的数量、RNA数量以及基因总数方面存在一些差异,表明各菌株在进化阶段存在一定的差异,在进化过程中丢失或新获取了一些基因。将SC1401菌株编码的所有基因与其余四株进行比对(图 2),发现1 412个基因为5个菌株共有,385个基因为SC1401菌株特有,SH0165、SH03、KL0318及ZJ0906特有的基因数量分别为190、47、71、201个。
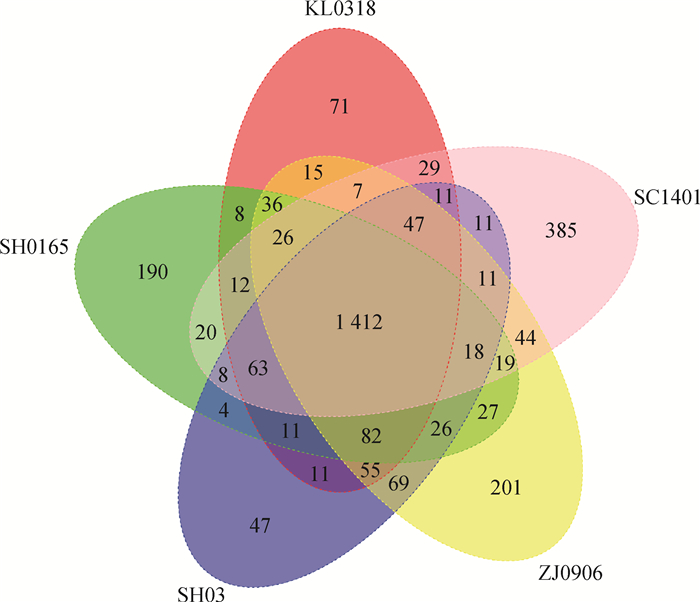
|
图 2 core-pan基因分析 Figure 2 Analysis of core-pan gene |
采用Mauve软件将SC1401全基因组与其余四株菌株的基因组进行比对分析,比对结果见图 3。从图中可以看到SC1401菌株与其余四株菌株相似性较高,有着非常保守的基因组结构和共线性关系。
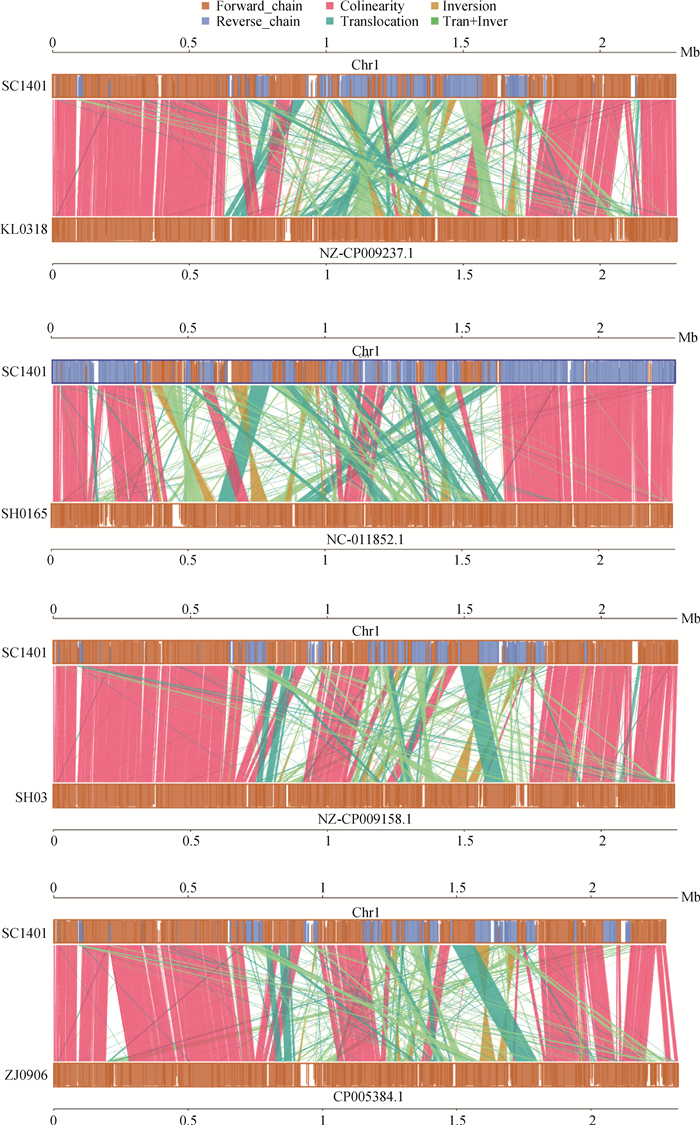
|
图 3 SC1401与其余4株菌株的共线性分析 Figure 3 Syntenic analysis between SC1401 and the other 4 strains |
将SC1401菌株序列与流感嗜血杆菌Rd KW20菌株序列比对后,得到多个与自然转化相关的基因(图 4)。图 4中各个基因同样均存在于SH0165、SH03、KL0318和ZJ0906菌株中。在SC1401中,comA B C D E共用一个操纵子,pilA B C D共用一个操纵子。

|
图 4 5株副猪嗜血杆菌菌株与自然转化相关的基因 Figure 4 Genes related to natural transformation of 5 HPS strains |
比起人工制作的感受态细菌(电化学方法),自然转化是细菌在天然状态下的转化情形,人们根据HPS的这种特性,创建了相应的基因缺失的方法,用以研究该菌的基因功能[13]。比起电转化和接合转移,该方法无需对亲本株进行特殊处理,操作更简单,试验周期短,更易获得阳性转化子。因此,该方法很快便得到推广与使用,尤其在研究基因功能和毒力因子方面。然而并非所有副猪嗜血杆菌都是自然感受态细菌,因此筛选HPS自然感受态菌株也为HPS基因功能研究提供了生物材料,且在实际操作中,自然转化效率往往很低,获得正确的阳性克隆子往往需要反复试验,延长了试验周期。
CheY/QseC属于HPS的一对二元信号转导系统,CheY负责调控特定基因的表达水平,属于细胞质应答调节蛋白,cheY对于HPS至关重要,且在HPS中高度保守,此前何绿琴等[18]成功使用自然转化方法构建出HPS cheY基因缺失株,因此本研究通过cheY LKR-pK18来筛选自然感受态细菌,其原理和方法大致如下:通过overlap-PCR将cheY基因上游同源臂L、下游同源臂R和卡那霉素抗性盒Kan融合成cheY LKR片段;将此片段连接至pK18mobsacB自杀质粒得到cheY LKR-pK18质粒;利用同源重组原理及自然转化法将该质粒上的Kan转化到HPS中置换cheY原位基因以获得具有卡那霉素抗性的cheY突变菌株。
本研究在75株田间分离株中共筛出11株自然感受态细菌,包括高自然转化效率的SC1401菌株,说明自然感受态细菌在田间分离株中占有一定的比例,而15株标准株均未检测到自然转化的发生,可能是由于其自身本就不具备自然转化能力,或者目前未能摸索到有效诱导其自然转化发生的条件。
通过全基因组测序,我们获得了高自然转化效率菌株SC1401的基因组序列,并上传到GenBank、EMBL、DDBJ数据库中,登录号为CP015099。将SC1401菌株与其他四株完成全基因组测序的副猪嗜血杆菌基因组序列进行比较分析,包括SH0165株、SH03株、KL0318株和ZJ0906株。分析表明SC1401菌株基因组的基本特征与其他HPS属的菌株基因组的共线性关系较好,但core-pan基因分析表明各菌株亦存在一定的差异,其中SC1401菌株特有的基因高达385个,而SH0165、SH03、KL0318及ZJ0906特有的基因数量则分别为190、47、71、201个,远低于SC1401所含有的特有基因数量。该现象可能是由于SC1401具备高自然转化效率,在进化的历程中更容易获得外源基因所导致的。
在之前自然感受态细菌筛选的过程中,各个菌株的自然转化能力存在明显的差别。通过验证尚未发现副猪嗜血杆菌参考菌株SH0165具备自然转化能力,SH03、KL0318和ZJ0906菌株尚无关于自然转化的报道,自然转化能力未知。然而序列比对发现,SH0165、SH03、KL0318和ZJ0906菌株与SC1401菌株基因组均存在与自然转化相关的基因(图 3):负责自然转化过程中DNA摄取的com系统和T4P四型菌毛系统,负责细菌自然转化调控的crp、cyaA、icc和tfox基因,与重组或DNA程序化进程(生理生化进程)有关的comM、dprA、radc、recA、ssb基因,以及在自然转化过程中功能未知的ligA、murE、luxS和rbsB基因。
通过比对发现,大多数基因在二者间的一致性都高达90%以上,而tfox基因一致性只为74%(该基因在多种细菌中已被证实是自然转化上游核心调控因子),这提示二者自然转换能力的不同可能是由于各基因转录水平/表达差异以及/或tfox基因的分子差异造成的,亦有可能副猪嗜血杆菌中还存在其他未知的与自然转化相关的重要基因/分子基础。研究表明流感嗜血杆菌的感受态建立受到转录调节因子tfox和cAMP受体蛋白CRP共同调控(cAMP-CRP复合物),tfox缺失株失去了自然转化的能力,而tfox表达上调的突变株能够进行持续性的自然转化[21]。由于tfox基因在副猪嗜血杆菌高、低自然感受态细菌中具有明显的分子差异,而在流感嗜血杆菌、大肠杆菌等中高度保守(相似度达95%以上),且该基因作为自然感受态系统的核心调控元件,其在HPS中的后续研究则很有必要。同时SH0165与SC1401菌株自然转化能力的差异及基于此差异背后的分子背景的挖掘,为笔者后续开展HPS自然转化机制的研究奠定了基础。
Bossé等[23]在筛选传染性胸膜肺炎放线杆菌(Actinobacillus pleuropneumoniae, APP)的自然感受态菌株时,所检测的血清3型菌株均具有自然转化能力,而血清7型菌株均未检测到自然转化能力。本研究筛选出的11株自然感受态细菌的分型结果显示:血清13型4株,血清4型、5型、11型各2株,血清1型1株。在HPS中,自然转化能力与血清型之间的关系目前未见报道,二者之间是否像在APP中一样存在一定关联,有待进一步研究。
4 结论筛选出11株HPS自然感受态菌株,均为野生型,其中SC1401菌株自然转化效率最高;SC1401菌株基因组大小为2 277 540 bp,GC含量为40.03%,共编码2 220个基因;基因组共线性分析表明SC1401与其他4株(SH0165、SH03、KL0318和ZJ0906株)全基因组测序菌株的共线性良好;比较基因组分析表明,SC1401菌株含特有基因385个,多于其他4株;5株HPS含有一系列相同的与自然转化相关的基因,且除tfox基因外,各基因氨基酸序列一致性均达90%以上。
| [1] | BLACKALL P J, TURNI C. Understanding the virulence of Haemophilus parasuis[J]. Vet J, 2013, 198(3): 549–550. DOI: 10.1016/j.tvjl.2013.09.070 |
| [2] | BROCKMEIER S L, REGISTER K B, KUEHN J S, et al. Virulence and draft genome sequence overview of multiple strains of the swine pathogen Haemophilus parasuis[J]. PLoS One, 2014, 9(8): e103787. DOI: 10.1371/journal.pone.0103787 |
| [3] | ANGEN Ø, OLIVEIRA S, AHRENS P, et al. Development of an improved species specific PCR test for detection of Haemophilus parasuis[J]. Vet Microbiol, 2007, 119(2-4): 266–276. DOI: 10.1016/j.vetmic.2006.10.008 |
| [4] | ZHOU Q, FENG S X, ZHANG J M, et al. Two glycosyltransferase genes of Haemophilus parasuis SC096 implicated in lipooligosaccharide biosynthesis, serum resistance, adherence, and invasion[J]. Front Cell Infect Microbiol, 2016, 6: 100. |
| [5] | ZHANG L H, LI Y, DAI K, et al. Establishment of a successive markerless mutation system in Haemophilus parasuis through natural transformation[J]. PLoS One, 2015, 10(5): e0127393. DOI: 10.1371/journal.pone.0127393 |
| [6] | PERRON G G, LEE A E G, WANG Y, et al. Bacterial recombination promotes the evolution of multi-drug-resistance in functionally diverse populations[J]. Proc R Soc B Biol Sci, 2012, 279(1733): 1477–1484. DOI: 10.1098/rspb.2011.1933 |
| [7] | BERTOLLA F, SIMONET P. Horizontal gene transfers in the environment:natural transformation as a putative process for gene transfers between transgenic plants and microorganisms[J]. Res Microbiol, 1999, 150(6): 375–384. DOI: 10.1016/S0923-2508(99)80072-2 |
| [8] | CLAVERYS J P, PRUDHOMME M, MARTIN B. Induction of competence regulons as a general response to stress in gram-positive bacteria[J]. Ann Rev Microbiol, 2006, 60: 451–475. DOI: 10.1146/annurev.micro.60.080805.142139 |
| [9] | NIKAIDO H. Multidrug resistance in bacteria[J]. Annu Rev Biochem, 2009, 78: 119–146. DOI: 10.1146/annurev.biochem.78.082907.145923 |
| [10] | KRÜGER N J, STINGL K. Two steps away from novelty-principles of bacterial DNA uptake[J]. Mol Microbiol, 2011, 80(4): 860–867. DOI: 10.1111/mmi.2011.80.issue-4 |
| [11] |
孙东昌, 张衍梅, 施跃峰. 细菌自然转化的分子机制研究进展[J]. 微生物学报, 2012, 52(1): 6–11.
SUN D C, ZHANG Y M, SHI Y F. Advances in the molecular mechanism of natural bacterial transformation-a review[J]. Acta Microbiologica Sinica, 2012, 52(1): 6–11. (in Chinese) |
| [12] | BOSSÉ J T, NASH J H E, KROLL J S, et al. Harnessing natural transformation in Actinobacillus pleuropneumoniae:a simple method for allelic replacements[J]. FEMS Microbiol Lett, 2004, 233(2): 277–281. DOI: 10.1111/fml.2004.233.issue-2 |
| [13] | BIGAS A, GARRIDO M E, DE ROZAS A M P, et al. Development of a genetic manipulation system for Haemophilus parasuis[J]. Vet Microbiol, 2005, 105(3-4): 223–228. DOI: 10.1016/j.vetmic.2004.10.015 |
| [14] | SEITZ P, BLOKESCH M. Cues and regulatory pathways involved in natural competence and transformation in pathogenic and environmental Gram-negative bacteria[J]. FEMS Microbiol Rev, 2012, 37(3): 336–363. |
| [15] | DUBNAU D. DNA uptake in bacteria[J]. Annu Rev Microbiol, 1999, 53: 217–244. DOI: 10.1146/annurev.micro.53.1.217 |
| [16] |
周鹏.副猪嗜血杆菌HAPS_RS00465蛋白的免疫保护性及其基因缺失株生物学特性研究[D].雅安: 四川农业大学, 2016.
ZHOU P. Studies on the immunoprotection of H. Parasuis HAPS_RS00465 protein and the biological characteristics of the gene deletion strains[D]. Ya'an: Sichuan Agricultural University, 2016. (in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10626-1017013114.htm |
| [17] | MAUGHAN H, REDFIELD R J. Tracing the evolution of competence in Haemophilus influenzae[J]. PLoS One, 2009, 4(6): e5854. DOI: 10.1371/journal.pone.0005854 |
| [18] | HE L Q, WEN X T, YAN X F, et al. Effect of cheY deletion on growth and colonization in a Haemophilus parasuis serovar 13 clinical strain EP3[J]. Gene, 2016, 577(1): 96–100. DOI: 10.1016/j.gene.2015.11.046 |
| [19] | HOWELL K J, PETERS S E, WANG J H, et al. Development of a multiplex PCR assay for rapid molecular serotyping of Haemophilus parasuis[J]. J Clin Microbiol, 2015, 53(12): 3812–3821. DOI: 10.1128/JCM.01991-15 |
| [20] |
谨瑾, 代科, 文心田, 等. 副猪嗜血杆菌自然转化方法的建立及条件优化[J]. 农业生物技术学报, 2017, 25(12): 2058–2065.
IN J, DAI K, WEN X T, et al. Establishment and condition optimization for natural transformation method of Haemophilus parasuis[J]. Journal of Agricultural Biotechnology, 2017, 25(12): 2058–2065. (in Chinese) |
| [21] | KRISTENSEN B M, SINHA S, BOYCE J D, et al. Natural transformation of Gallibacterium anatis[J]. Appl Environ Microbiol, 2012, 78(14): 4914–4922. DOI: 10.1128/AEM.00412-12 |
| [22] | LIU M F, ZHANG L, HUANG L, et al. Use of natural transformation to establish an easy knockout method in Riemerella anatipestifer[J]. Appl Environ Microbiol, 2017, 83(9): e00127–17. |
| [23] | BOSSÉ J T, SINHA S, SCHIPPERS T, et al. Natural competence in strains of Actinobacillus pleuropneumoniae[J]. FEMS Microbiol Lett, 2009, 298(1): 124–130. DOI: 10.1111/fml.2009.298.issue-1 |



