转基因动物在定向育种、生物反应器、疾病模型、器官移植、功能基因组学等领域发挥着重要作用。如何提高转基因动物的生产效率,定点整合外源基因及维持外源基因高丰度表达[1]是转基因研究领域面临的主要问题。整合有外源基因的细胞其基因表达水平会伴随着传代次数的增加被抑制[2-4],表观遗传修饰的动态变化可能是影响基因表达的主要因素。
组蛋白修饰作为表观遗传中重要的调控机制之一, 在包括基因表达调控等多种生物学过程中起着重要作用。哺乳动物中,组蛋白甲基转移酶EHMT2被认为是常染色质区催化组蛋白H3K9二甲基化(H3K9me2),并调节该区域基因表达的主要酶[5]。H3K9me2是基因转录抑制及异染色质形成的主要原因[6],异染色质作为染色体的组成部分,在细胞周期中保持浓缩状态,具有重要的生物学功能。克隆胚胎中异常的组蛋白H3K9me2导致多能性基因Oct4无法正常表达[7],高丰度的组蛋白H3K9甲基化水平是阻碍细胞获得全能性的主要因素[8]。组蛋白赖氨酸甲基转移酶EHMT2特异性抑制剂BIX01294 [9]成功应用于多能干细胞的诱导及克隆胚胎发育率的提高[10-11],但由于较高的细胞毒性在绵羊与小鼠克隆胚胎的应用中并未获益[12-13]。Vedadi等[14]报道的小分子抑制剂UNC0638可高效特异性的结合组蛋白H3K9me2甲基化转移酶EHMT2与EHMT1,并具较低的细胞毒性。供体细胞经UNC0638处理后,可显著降低克隆胚胎H3K9me2水平,并影响克隆胚胎发育关键基因Nanog与 Oct4的表达[15]。
转基因细胞在体外培养、胚胎发育过程及转基因动物个体中普遍存在外源基因沉默现象[16]。UNC0638作为一种组蛋白修饰小分子药物能否通过调节H3K9me2甲基化水平,进而促进外源基因表达,仍未见相关的报道。因此,初步探讨UNC0638对转基因细胞外源基因表达的影响,有助于人们进一步了解外源基因整合后的表达机制,并为转基因水牛生产效率的提高提供理论依据。
1 材料与方法 1.1 材料 1.1.1 细胞水牛胎儿成纤维细胞(BFFs)与以绿色荧光蛋白(eGFP)基因为报告基因的转PRL基因水牛胎儿成纤维细胞系由广西亚热带农业生物资源保护与利用国家重点实验室培养保存。
1.1.2 试剂本试验所用试剂除特别注明外,其它试剂均来自美国Sigma公司;UNC0638购自Cayman公司,组蛋白H3K9me2甲基化抗体购自Abcam公司。胎牛血清(FBS)购自HyClone公司;无Ca2+、Mg2+的杜氏磷酸盐缓冲液(PBS),0.25%胰酶(trypsin),细胞培养液:DMEM+10% FBS;一抗:Anti-Histone H3 (di methyl K9)的鼠源单克隆抗体(1:100);二抗:FITC标记的兔抗鼠IgG (1:200);UNC0638液: DMSO预溶,以DMEM液配制50 μmol·L-1母液,根据试验要求用DMEM液稀释成不同浓度的工作液,在工作液中,DMSO的浓度要控制在0.1%以内。
1.2 方法 1.2.1 BFFs传代培养及生长曲线绘制BFFs复苏后传代培养,分别用添加有不同浓度(0.0、0.5、1.5、2.5、5.0 μmol·L-1)UNC0638的DMEM培养液重悬,调整细胞密度约为(2~3)×104·mL-1,接种于24孔细胞培养板中,每孔500 μL,置于38.5 ℃、5% CO2及饱和湿度的培养箱中培养,每3 d换液1次。每隔24 h消化细胞,每个培养浓度消化,分别收集3个孔的细胞,连续8 d,用细胞计数板计数,每孔记数3次,取其平均值进行生长曲线的绘制。
1.2.2 BFFs染色体核型分析UNC0638处理第7代BFFs 48 h后,使用浓度为0.1~0.2 μg·mL-1秋水仙素继续培养3 h,收集细胞。加入预热至37 ℃的0.075 mol·L-1 KCl低渗处理,加入2 mL新鲜配制且预冷的甲醇-冰乙酸(3:1)固定液,固定细胞1~2 min,离心弃上清。加5 mL新鲜配置的甲醇-冰乙酸(3:1)固定液,使用枪头吹出来的气泡轻轻吹打细胞,重悬细胞,室温条件下固定30 min。离心弃上清后,加入200 μL固定液,轻轻吹打混匀细胞。吸取细胞悬液,在距离20 cm以上的高度滴1~2滴细胞悬液于-20 ℃预冷的载玻片上,固定细胞。用Giemsa染液染色15~20 min,清水冲洗后晾干。在显微镜下观察细胞染色体数目,对典型的中期染色体拍照,随机抽取统计染色体数目。
1.2.3 免疫荧光检测BFFs组蛋白H3K9me2甲基化水平BFFs分别经不同浓度(0.0、0.5、1.5、2.5、5.0 μmol·L-1)UNC0638处理48 h后,取出爬片上的细胞玻片,放入12孔板内,PBS清洗两次后,室温下用4%多聚甲醛固定40 min。阻断液(含有100 mmol·L-1的甘氨酸和0.3% BSA的PBS缓冲液)冲洗3次,每次5 min,用1% Tritonx-100室温透化25 min,阻断液冲洗3次,每次5 min。1% BSA室温封闭1 h,Triton-BSA-PBS洗3次后加入100 mL一抗,4 ℃过夜孵育,次日37 ℃生化培养箱孵育30 min,Triton-BSA-PBS洗3次。二抗室温避光孵育1.5 h,Triton-BSA-PBS冲洗3次。10 μg·mL-1碘化丙啶(PI)室温避光染核10 min,Triton-BSA-PBS冲洗3次,凡士林压片,使用激光共聚焦扫描拍照。
1.2.4 BFFs细胞RNA提取使用不同浓度(0.0、0.5、1.5、2.5 μmol·L-1)UNC0638处理水牛转基因BFFs(24、48 h)后,Trizol法提取RNA,分光光度计及电泳检测其纯度和完整性,置于-80 ℃冰箱保存备用。按照一步快速反转录试剂盒(北京全式金生物公司)进行反转录反应,反转录体系20 μL:2 μg总RNA,5×TransScript All- in-One SuperMix for qPCR 4 μL,gDNA Remover 1 μL,RNase-free Water补足20 μL,轻轻混匀后,42 ℃孵育15 min,85 ℃灭活5 min,4 ℃终止。
1.2.5 qRT-PCR检测qRT-PCR的反应体系为20 μL:RT-PCR产物1 μL,2×FastStart Universal SYBR Green Master(ROX)10 μL,Primer F/R (10 μmol·L - 1) 0.6 μL,RNase Free water 8.4 μL。反应条件:95 ℃变性10 min;95 ℃ 15 s,60 ℃ (或者58 ℃) 1 min,40个循环,试验对每个样品进行3次重复。β-actin作内参对其进行标准化,运用2-△△Ct方法计算目的基因的相对表达量,组间差异利用SPSS软件进行统计分析。引物信息见表 1。
|
|
表 1 用于PCR反应各个基因引物序列和反应条件 Table 1 Details of primers used for PCR |
免疫荧光强度相对水平的分析和基因相对表达量的分析均采用SPSS17.0软件进行单因素方差分析(ANOVA)。每个试验重复至少3次,染色体核型分析采用卡方检验,P>0.05表示差异不显著;P < 0.05表示差异显著。
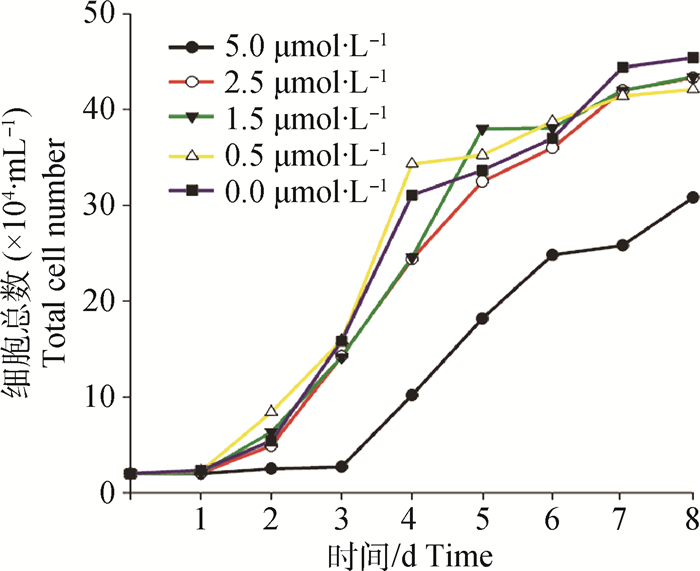
2 结果 2.1 不同浓度UNC0638对BFFs增殖的影响不同浓度(0.0(对照组)、0.5、1.5、2.5、5.0 μmol·L-1)UNC0638处理BFFs 8 d。结果如图 1所示,UNC0638处理组与对照组相比,生长曲线趋势大致相同,均呈“S”形分布。0.5、1.5、2.5 μmol·L-1处理组与对照组相比细胞增殖效率没有显著变化,5.0 μmol·L-1处理组细胞生长受到抑制,3 d后开始增殖,与其他各组相比细胞增殖效率下降。
|
图 1 不同UNC0638浓度对BFFs生长曲线的影响 Figure 1 Growth curves of BFFs cultured in different concentration of UNC0638 |
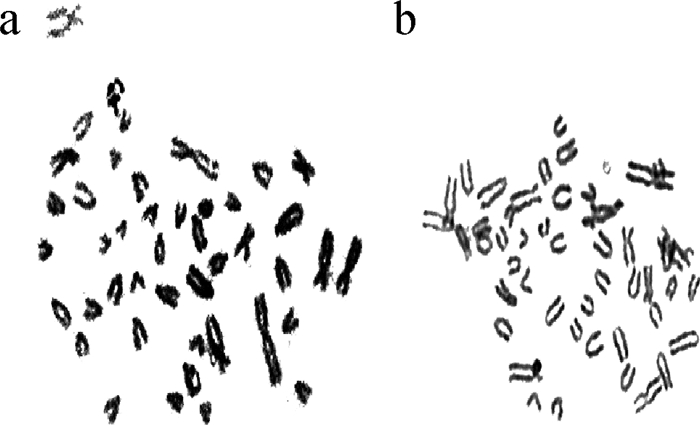
BFFs正常数目染色体如图 2 a所示,异常染色体数如图 2 b所示。结果显示BFFs经不同浓度(0.0、0.5、1.5、2.5 μmol·L-1)UNC0638处理48 h后对其细胞核型无显著影响(表 2)。
|
a.正常染色体数(46, XX);b.异常染色体数 a.Normal chromosome No.; b.Abnormal chromosome No. 图 2 BFFs染色体中期分裂相 Figure 2 The metaphase spreads of chromosome of BFFs |
|
|
表 2 不同浓度UNC0638处理BFFs后的核型分析 Table 2 Karyotype analysis of BFFs in different concentration of UNC0638 |
BFFs分别经不同浓度(0.0(对照组)、0.5、1.5、2.5、5.0 μmol·L-1)UNC0638处理48 h,检测各组细胞组蛋白H3K9me2甲基化水平(图 3a), 并统计荧光强度,结果如图 3b所示:各处理组与对照组相比组蛋白H3K9me2甲基化水平显著降低(P < 0.05),其中0.5、1.5、2.5 μmol·L-1组间不存在显著差异(P>0.05),5.0 μmol·L-1组显著低于0.5 μmol·L-1组(P < 0.05),但与1.5、2.5 μmol·L-1组间差异不显著(P>0.05)。

|
a.BFFs组蛋白H3K9me2的变化(200×);b.相对荧光强度。不同小写字母表示组间差异显著(P < 0.05),相同字母表示组间差异不显著(P>0.05)。下同 a.The H3K9me2 status of BFFs(200×); b.The relative fluorescence intensity of BFFs.Different small letters mean significant difference among the groups (P < 0.05), same small letters mean not significant difference among the groups (P>0.05).The same as below 图 3 UNC0638处理48 h后对BFFs组蛋白H3K9me2的影响 Figure 3 Effect of H3K9me2 on BFFs after 48 h UNC0638 treatment |
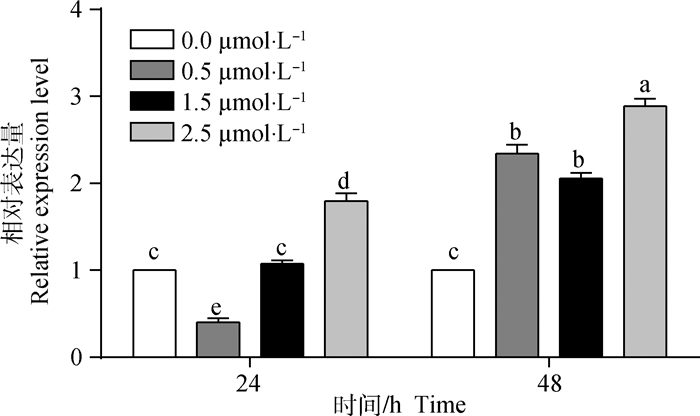
不同浓度(0.0(对照组)、0.5、1.5、2.5 μmol·L-1)UNC0638处理转基因BFFs(24、48 h)后,结果如图 4所示:处理24 h,仅2.5 μmol·L-1处理组eGFP的表达显著高于对照组(P < 0.05);处理48 h,各处理组与对照组相比eGFP的表达水平显著提高(P < 0.05)。
|
图 4 不同浓度UNC0638对转基因BFFs eGFP基因表达的影响 Figure 4 Effect of different concentration of UNC0638 on the expression of eGFP gene in transgenic BFFs |
转基因动物研究始于20世纪70年代末,是生物技术领域研究热点之一,该技术具有广阔的应用前景及重要的经济价值。然而转基因动物生产过程中外源基因导入仍面临着效率低及无法精确控制基因表达等问题。有报道指出一些与转录激活有关的表观遗传小分子抑制剂可提高外源基因在体内外的表达水平[17]。本研究探讨组蛋白甲基化小分子抑制剂UNC0638处理水牛转基因BFFs后对外源基因表达效率的影响。
组蛋白是染色质的核心,其尾部的乙酰化、磷酸化、甲基化等共同组成了组蛋白密码并控制基因表达。组蛋白H3与H4赖氨酸甲基化参与多种生物学过程,包括转录的控制、DNA甲基化、异染色质区域形成、X染色体的失活等[18-19],一些效应分子可特异性阅读不同的赖氨酸甲基化密码调节转录过程[20]。催化组蛋白赖氨酸甲基化的蛋白含有一个共同的结构域(SET结构域),主要有Suv39h1、Suv39h2、EHMT2、EHMT1,其中EHMT2与EHMT1是控制常染色质区域组蛋白甲基化的主要蛋白。组蛋白修饰在重编程过程中发挥重要的作用,H3K9me2修饰与基因的转录调节有关[21-22],并可调控Oct4在胚胎中的表达[23]。Chen等[8]报道,在诱导多能干细胞过程中组蛋白H3K9me2甲基化是抑制重编程的主要因素。此外,PGC7可以与含有H3K9me2的染色质结合,阻断TET3蛋白的主动去甲基化活性,以保护受精卵中的特异性的印记基因[13]。使用H3K9me2抑制剂BIX01294处理羊克隆胚胎可以显著降低胚胎组蛋白H3K9me2水平,但对囊胚发育率无显著影响[24]。Park等[25]指出BIX01294的高细胞毒性可能是影响胚胎发育的主要原因,阻碍了其在细胞重编程领域的研究与应用。UNC0638是一种新型的组蛋白甲基化抑制剂[14],具更高效的组蛋白甲基化抑制效率与细胞毒性,因而在提高体细胞克隆效率和外源基因表达上有极高的利用价值。
Vedadi等[14]报道,UNC0638处理浓度大于0.5 μmol·L-1时可显著降低人乳腺癌细胞株的H3K9me2水平。本研究基于此设置浓度梯度,在探讨UNC0638对BFFs生长特性及细胞毒性的影响时发现,BFFs经5.0 μmol·L-1浓度的UNC0638处理后细胞增殖减缓,处理48 h不影响细胞的核型正常率,结果与Vedadi等[14]报道的细胞毒性检测试验(MTT)结果相符,UNC0638药物安全指标(EC50)为(EC50=(11 000±710) nmol·L-1 (n=3)),BIX01294为(EC 50=(2 700±76) nmol·L-1 (n=3)),说明UNC0638较低的细胞毒性,一定浓度范围内不影响BFFs的生长特性与核型。对不同浓度UNC0638处理BFFs后,其组蛋白H3K9me2去甲基化能力分析,结果显示,0.5 μmol·L-1 UNC0638在处理BFFs 12 h,即可检测到组蛋白H3K9me2甲基化水平的显著降低,Fu等[26]报道使用0.1 μmol·L-1UNC0638处理牛供体细胞48 h,可检测到H3K9me2甲基化水平的显著降低。以上结果表明,低浓度UNC0638可显著抑制组蛋白H3K9me2的甲基化水平。经UNC0638处理后的转基因细胞外源基因表达分析发现,不同浓度UNC0638处理48 h,与对照组相比可显著提高细胞eGFP的表达量,黄波[27]使用组蛋白去乙酰化抑制剂TSA处理转基因细胞可增强eGFP基因的转录。该结果证实UNC0638可改变由表观遗传修饰导致的外源基因沉默,组蛋白H3K9me2可能是抑制转基因细胞外源基因表达的因素之一。
张一军[28]研究发现,山羊克隆胚胎经UNC0638处理后,H3K9me2水平显著下降与卵胞浆内单精子注射胚胎不存在显著差异,且克隆胚胎囊胚的Oct4和Nanog基因表达量与囊胚质量显著提高。UNC0638是否可以提高水牛转基因克隆胚胎发育率有待进一步的研究。
4 结论本研究结果表明,适宜浓度的UNC0638处理BFFs,不影响其细胞增殖与核型;UNC0638可显著降低BFFs组蛋白H3K9me2甲基化水平,并提高水牛转基因细胞外源基因的表达效率。
| [1] | HOUDEBINE L M. Transgenic animal bioreactors[J]. Transgenic Res, 2000, 9(4-5): 305–320. |
| [2] | MIGLIACCIO A R, BENGRA C, LING J H, et al. Stable and unstable transgene integration sites in the human genome:extinction of the Green Fluorescent Protein transgene in K562 cells[J]. Gene, 2000, 256(1-2): 197–214. DOI: 10.1016/S0378-1119(00)00353-X |
| [3] | YAMAUCHI Y, DOE B, AJDUK A, et al. Genomic DNA damage in mouse transgenesis[J]. Biol Reprod, 2007, 77(5): 803–812. DOI: 10.1095/biolreprod.107.063040 |
| [4] | KONG Q R, WU M L, HUAN Y J, et al. Transgene expression is associated with copy number and cytomegalovirus promoter methylation in transgenic pigs[J]. PLoS One, 2009, 4(8): e6679. DOI: 10.1371/journal.pone.0006679 |
| [5] | TACHIBANA M, SUGIMOTO K, FUKUSHIMA T, et al. Set domain-containing protein, G9a, is a novel lysine-preferring mammalian histone methyltransferase with hyperactivity and specific selectivity to lysines 9 and 27 of histone H3[J]. J Biol Chem, 2001, 276(27): 25309–25317. DOI: 10.1074/jbc.M101914200 |
| [6] | NAKAYAMA J I, RICE J C, STRAHL B D, et al. Role of histone H3 lysine 9 methylation in epigenetic control of heterochromatin assembly[J]. Science, 2001, 292(5514): 110–113. DOI: 10.1126/science.1060118 |
| [7] | BORTVIN A, EGGAN K, SKALETSKY H, et al. Incomplete reactivation of Oct4-related genes in mouse embryos cloned from somatic nuclei[J]. Development, 2003, 130(8): 1673–1680. DOI: 10.1242/dev.00366 |
| [8] | CHEN J K, LIU H, LIU J, et al. H3K9 methylation is a barrier during somatic cell reprogramming into iPSCs[J]. Nat Genet, 2013, 45(1): 34–42. DOI: 10.1038/ng.2491 |
| [9] | KUBICEK S, O'SULLIVAN R J, AUGUST E M, et al. Reversal of H3K9me2 by a small-molecule inhibitor for the G9a histone methyltransferase[J]. Mol Cell, 2007, 25(3): 473–481. DOI: 10.1016/j.molcel.2007.01.017 |
| [10] | SHI Y, DESPONTS C, DO J T, et al. Induction of pluripotent stem cells from mouse embryonic fibroblasts by Oct4 and Klf4 with small-molecule compounds[J]. Cell Stem Cell, 2008, 3(5): 568–574. DOI: 10.1016/j.stem.2008.10.004 |
| [11] | HUANG J J, ZHANG H Y, YAO J, et al. BIX-01294 increases pig cloning efficiency by improving epigenetic reprogramming of somatic cell nuclei[J]. Reproduction, 2016, 151(1): 39–49. DOI: 10.1530/REP-15-0460 |
| [12] | HUANG Y F, JIANG X H, YU M, et al. Beneficial effects of diazepin-quinazolin-amine derivative (BIX-01294) on preimplantation development and molecular characteristics of cloned mouse embryos[J]. Reprod Fertil Dev, 2017, 29(6): 1260–1269. DOI: 10.1071/RD15463 |
| [13] | FU L, ZHANG J, YAN F X, et al. Abnormal histone H3K9 dimethylation but normal dimethyltransferase EHMT2 expression in cloned sheep embryos[J]. Theriogenology, 2012, 78(9): 1929–1938. DOI: 10.1016/j.theriogenology.2012.07.017 |
| [14] | VEDADI M, BARSYTE-LOVEJOY D, LIU F, et al. A chemical probe selectively inhibits G9a and GLP methyltransferase activity in cells[J]. Nat Chem Biol, 2011, 7(8): 566–574. DOI: 10.1038/nchembio.599 |
| [15] | WANG Y S, ZHANG Y J, MAO T C, et al. Treatment donor cells with UNC0638 modify the abnormal histone H3K9 dimethylation and gene expression in cloned goat embryos[J]. Small Ruminant Res, 2017, 156: 27–32. DOI: 10.1016/j.smallrumres.2017.08.011 |
| [16] | GARRICK D, FIERING S, MARTIN D I K, et al. Repeat-induced gene silencing in mammals[J]. Nat Genet, 1998, 18(1): 56–59. DOI: 10.1038/ng0198-56 |
| [17] | GAETANO C, CATALANO A, PALUMBO R, et al. Transcriptionally active drugs improve adenovirus vector performance in vitro and in vivo[J]. Gene Ther, 2000, 7(19): 1624–1630. DOI: 10.1038/sj.gt.3301296 |
| [18] | LACHNER M, JENUWEIN T. The many faces of histone lysine methylation[J]. Curr Opin Cell Biol, 2002, 14(3): 286–298. DOI: 10.1016/S0955-0674(02)00335-6 |
| [19] | LACHNER M, O'SULLIVAN R J, JENUWEIN T. An epigenetic road map for histone lysine methylation[J]. J Cell Sci, 2003, 116(11): 2117–2124. DOI: 10.1242/jcs.00493 |
| [20] | BANNISTER A J, ZEGERMAN P, PARTRIDGE J F, et al. Selective recognition of methylated lysine 9 on histone H3 by the HP1 chromo domain[J]. Nature, 2001, 410(6824): 120–124. DOI: 10.1038/35065138 |
| [21] | NISHIOKA K, CHUIKOV S, SARMA K, et al. Set9, a novel histone H3 methyltransferase that facilitates transcription by precluding histone tail modifications required for heterochromatin formation[J]. Genes Dev, 2002, 16(4): 479–489. DOI: 10.1101/gad.967202 |
| [22] | FELDMAN N, GERSON A, FANG J, et al. G9a-mediated irreversible epigenetic inactivation of Oct-3/4 during early embryogenesis[J]. Nat Cell Biol, 2006, 8(2): 188–194. DOI: 10.1038/ncb1353 |
| [23] | EPSZTEJN-LITMAN S, FELDMAN N, ABU-REMAILEH M, et al. De novo DNA methylation promoted by G9a prevents reprogramming of embryonically silenced genes[J]. Nat Struct Mol Biol, 2008, 15(11): 1176–1183. DOI: 10.1038/nsmb.1476 |
| [24] | KANG J, KALANTRY S, RAO A. PGC7, H3K9me2 and Tet3:regulators of DNA methylation in zygotes[J]. Cell Res, 2013, 23(1): 6–9. DOI: 10.1038/cr.2012.117 |
| [25] | PARK K E, JOHNSON C M, CABOT R A. IVMBIX-01294, an inhibitor of the histone methyltransferase EHMT2, disrupts histone H3 lysine 9(H3K9) dimethylation in the cleavage-stage porcine embryo[J]. Reprod Fertil Dev, 2012, 24(6): 813–821. DOI: 10.1071/RD11205 |
| [26] | FU L, YAN F X, AN X R, et al. Effects of the histone methyltransferase inhibitor UNC0638 on histone H3K9 dimethylation of cultured ovine somatic cells and development of resulting early cloned embryos[J]. Reprod Domest Anim, 2014, 49(2): e21–e25. DOI: 10.1111/rda.2014.49.issue-2 |
| [27] |
黄波.TSA浓度及处理时间对猪体细胞核移植胚胎体外发育及转基因eGFP表达的影响[D].哈尔滨: 东北农业大学, 2010.
HUANG B.Effect of TSA concentration and treatment period on the in vitro development and transgene eGFP expression of pig somatic cell nuclear transfer embryos[D]. Harbin: Northeast Agricultural University, 2010.(in Chinese) http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y1787354 |
| [28] |
张一军.UNCO638和2-PCPA对山羊早期克隆胚胎组蛋白甲基化的影响[D].杨凌: 西北农林科技大学, 2014.
ZHANG Y J.Effect of UNC0638 and 2-PCPA on histone modifications in early goat cloned embryos[D]. Yangling: Northwest A & F University, 2014.(in Chinese) |



