环二鸟苷酸(c-di-GMP)、环二腺苷酸(c-di-AMP)和环腺苷酸-鸟苷酸(cGAMP)作为第二信使分子,日益成为研究的热点。c-di-GMP作为在细菌中合成的第二信使分子,参与细菌生理活动的许多方面,例如:生物被膜的形成、分化、运动性、毒力及胞外多糖的形成[1-5]。尽管在真核细胞中尚未发现天然存在的c-di-GMP,但是已有研究报道在小鼠和人细胞中转入c-di-GMP后,可以通过TBK-IRF3信号通路刺激Ⅰ型干扰素的生成[6-7],预示它可以作为潜在的免疫佐剂。cGAMP是一种新发现的哺乳动物第二信使,可以结合并且激活干扰素基因刺激因子(STING),诱导机体产生I型干扰素,进而刺激机体产生天然免疫应答反应[8-9]。并且已有研究报道利用原核表达系统纯化得到的环二鸟苷酸环化酶能够在体外酶促合成c-di-GMP[10-13]。
霍乱弧菌中的DncV(VC0179)是一种环二核苷酸环化酶,可以利用ATP和GTP作为底物催化合成c-di-GMP、cGAMP和c-di-AMP[14]。为了研究VC0179的催化功能以及通过体外酶促反应合成产生c-di-GMP和cGAMP,需要一种简单、快速的方法检测c-di-GMP和cGAMP。迄今为止已经开发了一些体外检测c-di-GMP和cGAMP的方法,包括高效液相色谱-质谱联用技术(HPLC-MS/MS)、高效液相色谱(HPLC)、荧光共振能量转移(FRET)和薄层色谱分析(TLC)[15-18]。这些方法虽然已被证明有效,但是它们也存在一些缺点。例如,HPLC虽然分析小分子比较灵敏,可以检测c-di-GMP和cGAMP,但是对于体外酶促反应体系来说,需要严谨的纯化操作去除酶蛋白等大分子,耗费时间和人力。TLC虽然能够快速检测c-di-GMP和cGAMP,但是待检测物通常需要放射性标记,对操作者健康产生潜在的不利影响。
最近,RNA适配体逐渐成为能够在体外和体内检测小分子的通用工具。Paige等[19]发现了一种基于RNA的适配体:spinach,其一旦与3, 5-二氟-4-羟基亚苄基咪唑啉酮(3, 5-difluoro-4-hydroxybenzylidene imidazolinone,DFHBI)结合,能够产生类似绿色荧光蛋白的激发荧光。Kellenberger等[20]将天然的GEMM-I核糖开关与spinach适配体结合,构建得到的适配体用于细胞内c-di-GMP和cGAMP检测[21-22]。为了检测体外酶促合成的c-di-GMP和cGAMP,本研究将VC2 GEMM-I核糖开关和它的突变体VC2/g20a连接到spinach2和tRNA支架上,开发出了一种基于RNA的核酸适配体,spinach2与spinach相比具有更高的热稳定性和折叠效率[23]
本研究以含有T7启动子、VC2 GEMM-1核糖开关序列或其突变序列VC2/g20a、spinach2和tRNA序列的双链DNA为模板,利用T7 RNA聚合酶体外转录体系制备VC2、VC2/g20a RNA适配体。通过检测绿色荧光强度分析RNA适配体对c-di-GMP和cGAMP的结合能力以及VC2和VC2/g20a适配体的检测下限,为相关研究提供参考。
1 材料与方法 1.1 主要试剂材料VC0179蛋白编码序列由宝生物工程(大连)有限公司合成;含有T7启动子、VC2或VC2/g20a适配体、spinach2和tRNA序列的单链DNA(表 1)由生工生物工程(上海)股份有限公司合成;限制性内切酶BamH I、EcoR I购自TaKaRa公司;T7 RNA聚合酶、NTP混合物、RNA酶抑制剂、脱氧核糖核酸酶I、RNA酶、无机焦磷酸酶和DEPC水购自赛默飞世尔科技公司;ATP、GTP标准品购自Aladdin公司;IPTG购自北京索莱宝科技有限公司;Ni-NTA Agarose购自QIAGEN公司;DFHBI购自Lucerna technologies(美国);质粒小量快速提取试剂盒、胶回收试剂盒、RNA纯化试剂盒、PCR纯化试剂盒均购自生工生物工程(上海)股份有限公司;E.coli DH5α、E.coli BL21(DE3)感受态细胞及含His标签的IPTG诱导型载体为本实验室保存。
|
|
表 1 本研究中使用的单链DNA序列 Table 1 The single strand DNA sequences used in this study |
以合成的单个DNA作为模板并采用以下引物:Sp-u:5′-CGATCCCGCGAAATTAATACG-3′, Sp-d:5′-TGGCGCCCGAACAGGGAC-3′,通过PCR得到用于体外转录的线性双链DNA(dsDNA),PCR产物dsDNA经PCR纯化试剂盒纯化。以dsDNA为模板体外转录得到RNA适配体:1 μg dsDNA,60 U T7聚合酶,NTPs(各6 mmol·L-1),80 U RNase抑制剂,0.3 U无机焦磷酸酶,加ddH2O至100 μL,混合均匀后37 ℃孵育4 h。孵育后加入2 U DNase I并孵育15 min以消化DNA模板,然后用RNA纯化试剂盒进行纯化回收。用酶标仪测定RNA的浓度后-80 ℃保存备用。
1.3 VC0179的表达和纯化将VC0179编码序列进行PCR扩增,PCR产物纯化后用BamH I和Xho I双酶切,回收后连接至IPTG诱导型载体的BamH I和Xho I位点并转化至E.coli DH5α中。转化菌液涂布于含氨苄青霉素的LB平板,37 ℃倒置培养过夜。挑选阳性克隆菌小提质粒,用BamH I和Xho I双酶切验证后送往生工生物工程(上海)股份有限公司测序。将测序正确的重组质粒转化至E.coli BL21(DE3)中,37 ℃培养。待其OD600 nm达到0.6~0.8时,加入IPTG使其终浓度为1 mmol·L-1,室温诱导12 h。诱导结束后4 ℃ 8 000 r·min-1离心10 min收集菌体,加入裂解缓冲液(50 mmol·L-1 NaH2PO4·H2O,300 mmol·L-1 NaCl,10 mmol·L-1咪唑)重悬,低温超声破碎,然后4 ℃ 13 000 r·min-1离心30 min除去细胞碎片。取上清液用QIAGEN公司的Ni-NTA填料按照操作说明书进行蛋白纯化,根据说明书用BCA蛋白质测定试剂盒测定蛋白质浓度。
1.4 VC0179体外酶促合成c-di-GMP和cGAMP用先前报道的方法由VC0179合成c-di-GMP和cGAMP[14]:1 μmol·L-1 VC0179与2 mmol·L-1的GTP合成c-di-GMP,1 μmol·L-1 VC0179与各2 mmol·L-1的GTP和ATP合成cGAMP;反应体系均为100 μL,缓冲液中包含5 mmol·L-1 MgCl2和10 mmol·L-1 HEPES(pH 7.5)。37 ℃孵育3 h后,将反应溶液在99 ℃煮沸5 min,12 000 r·min-1离心10 min,收集上清液进行c-di-GMP和cGAMP的检测。
1.5 RNA适配体检测c-di-GMP和cGAMP在100 μL反应体积中,将100 nmol·L-1纯化的RNA适配体与指定浓度的c-di-GMP或cGAMP在25 ℃下孵育结合,结合缓冲液中包含1 mmol·L-1 DFHBI、40 mmol·L-1 HEPES(pH 7.5)、125 mmol·L-1 KCl和30 mmol·L-1 MgCl2;将5 μL转录的RNA混合物与90 μL VC0179反应溶液在相同的结合缓冲液中25 ℃孵育3 h。然后将溶液转移到96孔黑色板中,在荧光和化学发光分析仪(激发:450 nm;发射:530 nm)中测量荧光强度。
1.6 数据分析本研究中所有测量经过3次重复。结果用“平均值±标准误差”表示。利用Graphpad Prism5软件的单因素方差分析进行差异显著性判断,P < 0.001表示差异极显著。
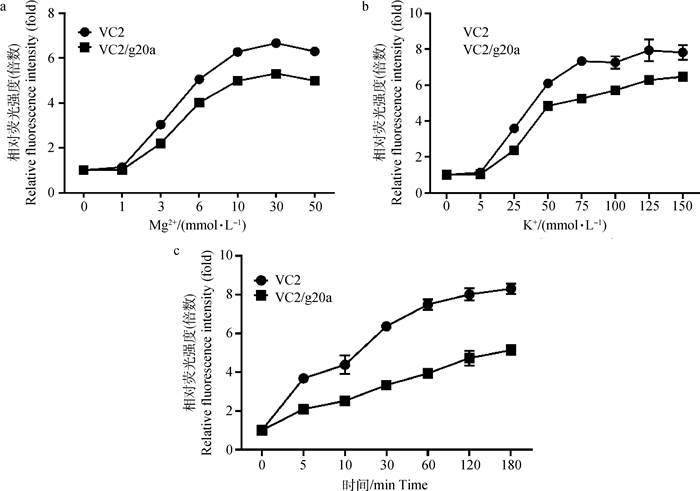
2 结果 2.1 RNA适配体与c-di-GMP和cGAMP结合条件的优化已有研究结果表明,一价和二价金属离子可以通过稳定RNA三级结构从而促进RNA折叠[24-27]。因此,本试验过改变K+和Mg2+浓度来优化RNA适配体与c-di-GMP和cGAMP的结合。该RNA适配体通过利用T7 RNA聚合酶体转录体系转录得到的RNA加热变性、缓慢退火得到。随着KCl从0 mmol·L-1增加到150 mmol·L-1,荧光强度逐渐增加。当MgCl2的浓度从0 mmol·L-1增加到50 mmol·L-1时,可以得到类似结果:在30 mmol·L-1 MgCl2下,VC2和VC2/g20a适配体分别与c-di-GMP和cGAMP结合后产生的荧光强度与对照相比分别增加大约5.6倍和4.3倍(图 1a)。在125 mmol·L-1 KCl溶液中,与未添加KCl的对照相比VC2和VC2/g20a适配体与c-di-GMP和cGAMP结合后产生的荧光强度分别增加约7倍和5.3倍(图 1b)。
|
a、b、c分别为MgCl2、KCl浓度和结合时间对荧光强度的影响。所有的测量经过3次重复,数据误差用标准差表示,下同 a, b, c. Effect of MgCl2, KCl concentrations and binding time on the fluorescence intensity produced by VC2 and VC2/g20a aptamers. All measurements were conducted with 3 duplicates and error bars represent the S.D, the same as below 图 1 RNA适配体与c-di-GMP和cGAMP结合的条件优化 Figure 1 Condition optimizing for c-di-GMP and cGAMP binding to RNA aptamers |
然后检测反应时间对荧光强度的影响。RNA适配体与c-di-GMP和cGAMP配体孵育的时间越长,产生的荧光越强(图 1c)。当在25 ℃孵育180 min时,VC2和VC2/g20a适配体的荧光强度分别增加到8.3倍和5.1倍。综合上述结果,优化的试验条件定为RNA适配体与c-di-GMP和cGAMP配体在125 mmol·L-1 KCl和30 mmol·L-1 MgCl2溶液中孵育180 min。
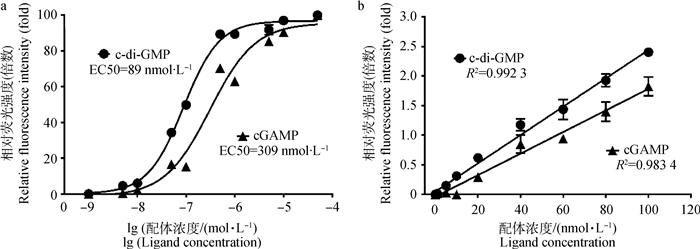
2.2 VC2和VC2/g20a适配体对c-di-GMP和cGAMP的检测灵敏度和检测极限为确定VC2和VC2/g20a适配体对c-di-GMP和cGAMP检测的灵敏度,发现当添加的c-di-GMP和cGAMP从10-9 mol·L-1增加到10-3 mol·L-1时,两个适配体产生的荧光强度逐渐增加。通过将荧光强度和相应的c-di-GMP、cGAMP浓度作图,得到两条较好的拟合曲线(图 2a)。根据结合曲线,可以得到c-di-GMP对VC2适配体的半数效应浓度(EC50)为(89±1.7) nmol·L-1,cGAMP对VC2/g20a适配体的半数效应浓度为(309±4.5) nmol·L-1。
|
a. VC2和VC2/g20a适配体对c-di-GMP和cGAMP的检测灵敏度;b. VC2和VC2/g20a适配体对c-di-GMP和cGAMP的检测限。未添加c-di-GMP和cGAMP配体的样品荧光强度作为背景扣除 a. The sensitivity of VC2 and VC2/g20a aptamers detecting c-di-GMP and cGAMP; b. The detection limit of VC2 and VC2/g20a aptamers towards c-di-GMP and cGAMP. Background without ligand was removed from every point 图 2 确定VC2和VC2/g20适配体分别对c-di-GMP和cGAMP的检测灵敏度及检测限 Figure 2 The datection sensitivity and limit of VC2 and VC2/g20 aptamers towards c-di-GMP and cGAMP, respectively |
为了确定两种适配体的检测限,适配体的浓度保持在100 nmol·L-1,c-di-GMP和cGAMP的浓度从0 nmol·L-1增加到100 nmol·L-1。通过对荧光强度与相应的配体浓度作图,二者呈现较好的线性关系(VC2适配体R2=0.992 3,线性范围:5~100 nmol·L-1;VC2/g20a适配体R2=0.983 4,线性范围:20 ~100 nmol·L-1)(图 2b)。另外从图上可以看到,VC2和VC2/g20a适配体对c-di-GMP和cGAMP的检测限分别为5 nmol·L-1和20 nmol·L-1。
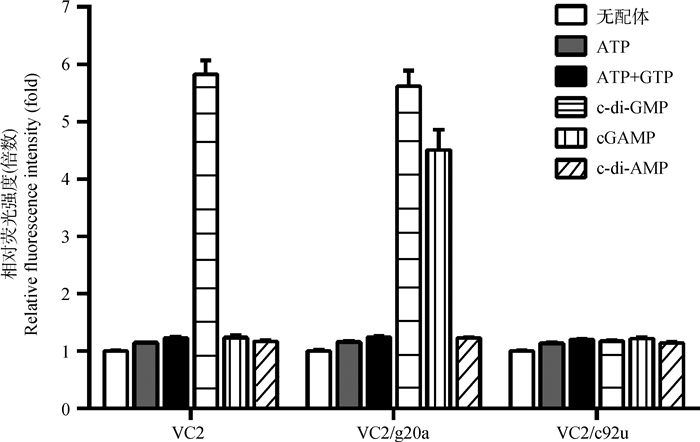
2.3 RNA适配体对c-di-GMP和cGAMP的选择性为了确定适配体对c-di-GMP和cGAMP的选择性,使用VC2和VC2/g20a适配体分别检测c-di-GMP、cGAMP、c-di-AMP、ATP和GTP;其中以VC2适配体的C92U突变体(VC2/c92u)作为阴性对照。在优化的结合条件下,与不加配体的对照相比,VC2适配体选择性结合c-di-GMP并产生5.9倍的荧光强度。VC2/g20a适配体与cGAMP特异性结合,相对于阴性对照样品产生接近4.3倍的荧光强度(图 3)。VC2/g20a适配体也能够与c-di-GMP结合并产生明显的激发荧光,这与Kellenberger等[20]报道的数据相似。
|
VC2和VC2/g20a适配体及其阴性对照(VC2/c92u)与配体(ATP、ATP+GTP、c-di-AMP、c-di-GMP和cGAMP)在优化的条件下结合,并测定相应的荧光强度。将不含配体的样品荧光值作为背景扣除 The VC2 and VC2/g20a aptamers and their negative control(VC2/c92u) bound to the ligands (ATP, ATP + GTP, c-di-AMP, c-di-GMP and cGAMP) under optimal binding conditions and the corresponding fluorescence intensities were detected. Fluorescence was normalized by sample without ligands 图 3 RNA适配体对不同配体的选择性 Figure 3 The selectivity of RNA aptamers toward different ligands |
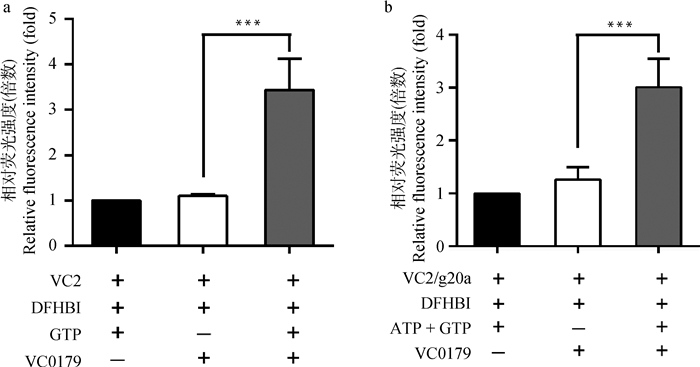
为了简单、快速的测定体外酶合成的c-di-GMP和cGAMP,本研究使用转录后未经纯化的RNA适配体混合物来检测VC0179合成的c-di-GMP和cGAMP。与未添加GTP的阴性对照相比,VC2适配体可以与VC0179合成的c-di-GMP结合并产生大约3.2倍的荧光强度(图 4a, P < 0.01)。与未添加ATP和GTP的阴性对照相比,VC2/g20a适配体结合VC0179合成的cGAMP后,可以产生接近2.5倍的荧光强度(图 4b, P < 0.01)。上述数据表明转录的RNA适配体混合物能够检测酶促反应产生的c-di-GMP和cGAMP。
|
a.转录的VC2适配体混合物检测VC0179合成的c-di-GMP;b.转录的VC2/g20a适配体混合物检测VC0179合成的cGAMP。***. P < 0.001 a. Transcribed VC2 aptamer mixture detected c-di-GMP by VC0179 synthesized; b. Transcribed VC2/g20a aptamer mixture detected cGAMP by VC0179 synthesized. ***. P < 0.001 图 4 RNA适配体混合物对VC0179合成的c-di-GMP和cGAMP的检测 Figure 4 Transcribed RNA mixture was used to detect c-di-GMP and cGAMP by VC0179 synthesized |
第二信使分子c-di-GMP和cGAMP能够刺激机体产生I型干扰素,具有潜在作为疫苗佐剂功能,因此,c-di-GMP和cGAMP对家畜重大疫病的治疗具有潜在应用价值。目前获得c-di-GMP和cGAMP的一个方法是通过化学合成,此方法虽然可行但也存在一些不足,比如:合成步骤复杂、成本高[10]。另一个主要方法是通过体外酶促反应获得,例如Korovashkina等[11]利用重组的环化酶在体外合成c-di-GMP,最近Launer-Felty和Strobel[28]报道利用环化酶体外合成c-di-GMP和cGAMP。与此同时,对于酶促合成的c-di-GMP和cGAMP的检测也是一个需要研究的重要问题。以往检测c-di-GMP和cGAMP的方法包括HPLC-MS/MS、HPLC、FRET、TLC等,这些方法虽然有效但也存在缺点。例如使用高效液相色谱-质谱联用技术检测c-di-GMP和cGAMP时仪器操作复杂,准备时间长,并且检测成本高。而高效液相色谱检测酶促合成的c-di-GMP和cGAMP时需要去除产物中的酶蛋白,耗费人力和物力等[15-18]。
因此为了研究体外酶促反应合成产生的c-di-GMP和cGAMP,需要一种方法能够简单、快速的对其进行检测。本研究通过将VC2核糖开关和其突变体VC2/g20a与spinach2和tRNA支架融合构建得到RNA适配体,通过结合DFHBI染料产生绿色激发荧光来检测c-di-GMP和cGAMP,进而开发一种基于RNA适配体的简单、快速检测c-di-GMP和cGAMP的方法。
本研究利用T7 RNA聚合酶体外转录体系转录得到的RNA经过加热变性和缓慢退火后,得到RNA适配体[29]。RNA适配体由3个模块构建(图 5):(1)与c-di-GMP和cGAMP结合区域; (2)spinach2区域;(3)tRNA支架,VC2 GEMM-1核糖开关或其突变体VC2/g20a用于结合c-di-GMP或cGAMP;Spinach 2用来结合DFHBI;tRNA支架增加适配体的热稳定性[30]。在没有c-di-GMP或cGAMP时,RNA适配体不能够产生激发荧光;在有c-di-GMP或cGAMP存在时,RNA适配体与c-di-GMP或cGAMP结合后,spinach2形成封闭结构,能够与DFHBI结合,形成的复合物在450 nm波长激发下产生类似绿色荧光蛋白的激发荧光。
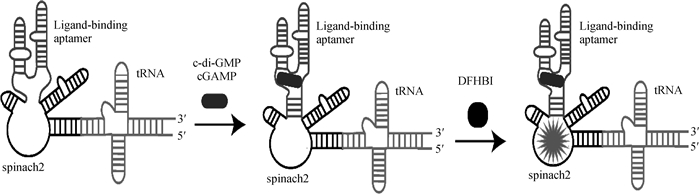
|
图 5 用于检测c-di-GMP和cGAMP的RNA适配体的示意图 Figure 5 Schematic illustration of designed RNA aptamers for detecting c-di-GMP and cGAMP |
已有文献报道一价和二价金属离子能够促进RNA折叠并且稳定形成三级结构[24-27],所以在本研究中,通过改变结合溶液中的K+和Mg2+浓度和孵育时间,对RNA适配体检测c-di-GMP和cGAMP的反应条件进行了优化。在含有30 mmol·L-1 MgCl2和125 mmol·L-1 KCl的结合溶液中孵育180 min的优化条件下,VC2适配体对c-di-GMP的检测限为5 nmol·L-1;VC2/g20a适配体对cGAMP的检测限为20 nmol·L-1。同时进一步研究发现,c-di-GMP对VC2适配体的半数效应浓度(EC50)为(89±1.7)nmol·L-1,cGAMP对VC2/g20a适配体的半数效应浓度为(309±4.5) nmol·L-1。这表明,本研究构建的两种RNA适配体对c-di-GMP和cGAMP的检测是灵敏的,并且能够在较大的范围内检测到c-di-GMP和cGAMP。
此外,本研究通过使用VC2和VC2/g20a适配体分别检测c-di-GMP、cGAMP、c-di-AMP、ATP和GTP,试验结果证明,VC2适配体能够特异性的检测c-di-GMP,VC2/g20a适配体能够特异性的检测cGAMP,这与先前报道的研究结果相似[20]。同时发现,与空白对照相比,VC2和VC2/g20a适配体对ATP和GTP没有明显的结合能力,这说明酶促反应产物中的底物ATP和GTP对RNA适配体检测c-di-GMP和cGAMP的结果没有影响,因此可以使用VC2和VC2/g20a适配体直接检测酶促反应产物中的c-di-GMP和cGAMP,并且该检测方法是灵敏的。
进一步的研究发现,转录未经纯化的RNA适配体混合物也能够显著检测体外VC0179酶促合成的c-di-GMP和cGAMP。这就进一步简化了使用RNA适配体检测c-di-GMP和cGAMP的步骤,使得使用VC2和VC2/g20a适配体分别检测c-di-GMP和cGAMP时更加简单、快速,同时也为在其他条件下检测c-di-GMP和cGAMP提供了可能。需要说明的是,如果要定量检测体外酶促合成的c-di-GMP和cGAMP,需要用c-di-GMP和cGAMP标准样品做标准曲线,并且给出相应的线性范围。
4 结论本研究通过将VC2核糖开关和其突变体VC2/g20a与spinach2和tRNA支架融合构建得到RNA适配体,开发了一种基于RNA适配体简单、快速的检测c-di-GMP和cGAMP的方法,进一步发现,转录未经纯化的RNA混合物也能够较灵敏地检测VC0179酶促合成的c-di-GMP和cGAMP。这种检测方法也为在其他条件下检测c-di-GMP和cGAMP提供了可能。
| [1] | RÖMLING U, GALPERIN M Y, GOMELSKY M. Cyclic di-GMP:The first 25 years of a universal bacterial second messenger[J]. Microbiol Mol Biol Rev, 2013, 77(1): 1–52. DOI: 10.1128/MMBR.00043-12 |
| [2] | LEONI L, PAWAR S V, RAMPIONI G.Genetic tools to study c-di-GMP-dependent signaling in Pseudomonas aeruginosa[M]//SAUER K.c-di-GMP Signaling: Methods and Protocols.New York, NY: Humana Press, 2017: 471-480. |
| [3] | BARAQUET C, HARWOOD C S.Use of nonradiochemical DNAse footprinting to analyze c-di-GMP modulation of DNA-binding proteins[M]//SAUER K.c-di-GMP Signaling: Methods and Protocols.New York, NY: Humana Press, 2017: 303-315. |
| [4] | AHMAD I, CIMDINS A, BESKE T, et al. Detailed analysis of c-di-GMP mediated regulation of csgD expression in Salmonella typhimurium[J]. BMC Microbiol, 2017, 17: 27. DOI: 10.1186/s12866-017-0934-5 |
| [5] | SLOUP R E, KONAL A E, SEVERIN G B, et al. Cyclic di-GMP and VpsR induce the expression of type Ⅱ secretion in Vibrio cholerae[J]. J Bacteriol, 2017, 199(19): e00106–17. |
| [6] | YIN Q, TIAN Y, KABALEESWARAN V, et al. Cyclic di-GMP sensing via the innate immune signaling protein STING[J]. Mol Cell, 2012, 46(6): 735–745. DOI: 10.1016/j.molcel.2012.05.029 |
| [7] | ALYAQOUB F S, ALDHAMEN Y A, KOESTLER B J, et al. In vivo synthesis of Cyclic-di-GMP using a recombinant adenovirus preferentially improves adaptive immune responses against extracellular antigens[J]. J Immunol, 2016, 196(4): 1741–1752. DOI: 10.4049/jimmunol.1501272 |
| [8] | TAO J L, ZHOU X, JIANG Z F. cGAS-cGAMP-STING:The three musketeers of cytosolic DNA sensing and signaling[J]. IUBMB Life, 2016, 68(11): 858–870. DOI: 10.1002/iub.v68.11 |
| [9] | WU J X, SUN L J, CHEN X, et al. Cyclic GMP-AMP is an endogenous second messenger in innate immune signaling by cytosolic DNA[J]. Science, 2013, 339(6121): 826–830. DOI: 10.1126/science.1229963 |
| [10] | SHCHOKOLOVA A S, RYMKO A N, KVACH S V, et al. Enzymatic synthesis of 2'-ara and 2'-deoxy analogues of c-di-GMP[J]. Nucleosides, Nucleotides Nucleic Acids, 2015, 34(6): 416–423. DOI: 10.1080/15257770.2015.1006775 |
| [11] | KOROVASHKINA A S, RYMKO A N, KVACH S V, et al. Enzymatic synthesis of c-di-GMP using inclusion bodies of Thermotoga maritima full-length diguanylate cyclase[J]. J Biotechnol, 2013, 164(2): 276–280. DOI: 10.1016/j.jbiotec.2012.12.006 |
| [12] | SPEHR V, WARRASS R, HÖCHERL K, et al. Large-scale production of the immunomodulator c-di-GMP from GMP and ATP by an enzymatic cascade[J]. Appl Biochem Biotechnol, 2011, 165(3-4): 761–775. DOI: 10.1007/s12010-011-9294-z |
| [13] | RAO F, PASUNOOTI S, NG Y, et al. Enzymatic synthesis of c-di-GMP using a thermophilic diguanylate cyclase[J]. Anal Biochem, 2009, 389(2): 138–142. DOI: 10.1016/j.ab.2009.03.031 |
| [14] | DAVIES B W, BOGARD R W, YOUNG T S, et al. Coordinated regulation of accessory genetic elements produces cyclic di-nucleotides for V. cholerae virulence[J]. Cell, 2012, 149(2): 358–370. DOI: 10.1016/j.cell.2012.01.053 |
| [15] | SPANGLER C, BÖHM A, JENAL U, et al. A liquid chromatography-coupled tandem mass spectrometry method for quantitation of cyclic di-guanosine monophosphate[J]. J Microbiol Methods, 2010, 81(3): 226–231. DOI: 10.1016/j.mimet.2010.03.020 |
| [16] | IRIE Y, PARSEK M R.LC/MS/MS-based quantitative assay for the secondary messenger molecule, c-di-GMP[M]//FILLOUX A, RAMOS J L.Pseudomonas Methods and Protocols.New York, NY: Humana Press, 2014: 271-279. |
| [17] | PAIJO J, KAEVER V, KALINKE U.cGAMP quantification in virus-infected human monocyte-derived cells by HPLC-coupled tandem mass spectrometry[M]//MOSSMAN K.Innate Antiviral Immunity: Methods and Protocols.New York, NY: Humana Press, 2017: 153-166. |
| [18] | BAIRD N J, INGLESE J, FERRÉ-D'AMARÉ A R. Rapid RNA-ligand interaction analysis through high-information content conformational and stability landscapes[J]. Nat Commun, 2015, 6: 8898. DOI: 10.1038/ncomms9898 |
| [19] | PAIGE J S, WU K Y, JAFFREY S R. RNA mimics of green fluorescent protein[J]. Science, 2011, 333(6042): 642–646. DOI: 10.1126/science.1207339 |
| [20] | KELLENBERGER C A, WILSON S C, SALES-LEE J, et al. RNA-based fluorescent biosensors for live cell imaging of second messengers cyclic di-GMP and cyclic AMP-GMP[J]. J Am Chem Soc, 2013, 135(13): 4906–4909. DOI: 10.1021/ja311960g |
| [21] | SUDARSAN N, LEE E R, WEINBERG Z, et al. Riboswitches in eubacteria sense the second messenger cyclic Di-GMP[J]. Science, 2008, 321(5887): 411–413. DOI: 10.1126/science.1159519 |
| [22] | SMITH K D, LIPCHOCK S V, AMES T D, et al. Structural basis of ligand binding by a c-di-GMP riboswitch[J]. Nat Struct Mol Biol, 2009, 16(12): 1218–1223. DOI: 10.1038/nsmb.1702 |
| [23] | STRACK R L, DISNEY M D, JAFFREY S R. A superfolding Spinach2 reveals the dynamic nature of trinucleotide repeat-containing RNA[J]. Nat Methods, 2013, 10(12): 1219–1224. DOI: 10.1038/nmeth.2701 |
| [24] | TRACHMAN Ⅲ R J, DRAPER D E. Comparison of interactions of diamine and Mg2+ with RNA tertiary structures:Similar versus differential effects on the stabilities of diverse RNA folds[J]. Biochemistry, 2013, 52(34): 5911–5919. DOI: 10.1021/bi400529q |
| [25] | LEIPPLY D, DRAPER D E. Dependence of RNA tertiary structural stability on Mg2+ concentration:Interpretation of the Hill equation and coefficient[J]. Biochemistry, 2010, 49(9): 1843–1853. DOI: 10.1021/bi902036j |
| [26] | TAN Z J, CHEN S J. RNA helix stability in mixed Na+/Mg2+ solution[J]. Biophys J, 2007, 92(10): 3615–3632. DOI: 10.1529/biophysj.106.100388 |
| [27] | KLEIN D J, MOORE P B, STEITZ T A. The contribution of metal ions to the structural stability of the large ribosomal subunit[J]. RNA, 2004, 10(9): 1366–1379. DOI: 10.1261/rna.7390804 |
| [28] | LAUNER-FELTY K D, STROBEL S A. Enzymatic synthesis of cyclic dinucleotide analogs by a promiscuous cyclic-AMP-GMP synthetase and analysis of cyclic dinucleotide responsive riboswitches[J]. Nucleic Acids Res, 2018, 46(6): 2765–2776. DOI: 10.1093/nar/gky137 |
| [29] | KELLENBERGER C A, HAMMOND M C. In vitro analysis of riboswitch-spinach aptamer fusions as metabolite-sensing fluorescent biosensors[J]. Methods Enzymol, 2015, 550: 147–172. DOI: 10.1016/bs.mie.2014.10.045 |
| [30] | PONCHON L, DARDEL F. Recombinant RNA technology:The tRNA scaffold[J]. Nat Methods, 2007, 4(7): 571–576. DOI: 10.1038/nmeth1058 |

 图 1(Figure 1)
图 1(Figure 1)


