2. 中国农业科学院北京畜牧兽医研究所, 北京 100193;
3. 福建省家畜传染病防治与生物技术重点实验室, 龙岩 364000
2. Institute of Animal Science, Chinese Academy of Agricultural Sciences, Beijing 100193, China;
3. Key Laboratory of Fujian Province for Livestock Epidemic Prevention and Control and Biological Technology, Longyan 364000, China
可变剪接(alternative splicing,AS)是mRNA通过不同的方式产生多种不同剪接RNA转录产物的过程[1],是高等真核生物蛋白质多样性的重要基因调解机制之一[2]。人基因组中有约95%的多外显子基因通过100 000多种不同的AS事件产生转录产物[3]。新一代RNA测序(RNA-seq)技术已经证实RNA转录产物的可变剪接是人基因组中编码蛋白质多样性的重要机制[4]。通过对基因组中AS事件的鉴定,已经发现一些基因的异常AS可导致人类多种疾病[5-7]。
畜禽基因和基因组中一些AS事件逐渐被发现和证实。研究人员通过设计跨越Lmbr 1基因外显子4的引物,发现与丝羽乌骨鸡趾型呈现显著相关的Lmbr 1基因中Lmbr 1-α为其主要转录产物,而Lmbr 1-β为次要转录产物[8]。经重叠延伸RT-PCR结合巢式PCR法扩增,在东北民猪中发现TLR4基因的3个可变剪接体[9],鸭TLR4基因中也发现了两种不同的剪接体[10]。研究人员通过PCR扩增和5′RACE方法获得白色和黑色山羊皮肤组织Agouti基因部分基因组序列及mRNA序列,经过拼接比对,在白色山羊中发现5种Agouti基因可变剪接体,而黑色山羊中发现2种可变剪接体[11]。运用RNA-seq技术,畜禽基因组的AS事件得到了全面的鉴定和分析。通过对绒山羊肌内脂肪细胞在成熟前后RNA-seq数据分析发现,绒山羊肌内脂肪细胞中存在A3SS、A5SS、SE和RI 4种主要可变剪接类型,其中A3SS最为普遍[12]。牛基因组的21 755个基因中4 567(21%)个基因发生了可变剪接[13]。猪睾丸基因组中存在92 738个AS事件,以TSS、TTS、SE和AE 4种类型为主;通过分析发现,与睾丸素代谢相关的基因中63个基因发生了可变剪接,以TSS和TTS为主[14]。这些AS事件的鉴定与分析有利于全面研究基因组中AS事件和蛋白质的多样性。由于AS事件大多数表现出组织特异性变异,其中10%~30%的AS事件易受细胞、组织和条件特异性调节的影响[3]。上述的研究并未对组织间差异剪接,特别是差异剪接基因(differential splicing gene,DSG)进行深入的分析。差异剪接分析描述了两个样本间AS位点使用情况和剪接异构体的差异,这对基因组中AS及其调控机制的研究至关重要,同时揭示了差异基因表达分析错过的功能多样性[15]。DSG指不同组织样品间因差异剪接而导致基因亚型的相对丰度有差异的基因。因此,DSG分析对于研究组织间AS及其调控机制具有重要的意义。
本研究以AA肉鸡RNA-seq测序数据为基础,通过分析鉴定肉鸡肌肉和脂肪组织基因组中DSG,分析这些DSG功能和其信号通路,为进一步解析肉鸡基因组中组织间AS事件及组织间基因表达的特异性奠定理论基础。
1 材料与方法 1.1 试验样品AA肉鸡母鸡苗由福建正大食品股份有限公司提供,于龙岩学院动物饲养房饲养至42日龄。肉鸡饲养管理和屠宰参考肉鸡生产技术规范(DB11/T 328-2005)与肉鸡屠宰质量管理规范(NY/T 1174-2006)。3只肉鸡禁食12 h后进行屠宰,取胸肌和腹脂组织样品转入2 mL RNase-free离心管中,标记后迅速置于液氮中保存备用。
1.2 组织总RNA提取采用TRIzol试剂(Invitrogen,Carlsbad,美国)提取组织样品总RNA,使用0.8%的琼脂糖凝胶电泳检测总RNA是否降解和受污染,并通过Agilent 2100生物分析仪使用RNA 6000 Nano试剂盒(Agilent Technologies,美国)检测总RNA的浓度、完整度(RIN)、28S/18S比值和片段大小。用于构建RNA-seq文库和测序的总RNA均满足以下条件:总RNA量≥6 μg,RIN≥8.0,28S/18S≥1.0。
1.3 肉鸡胸肌和腹部脂肪组织RNA-seq文库的构建和测序RNA-seq文库构建和测序均由深圳华大基因科技有限公司完成。分别取3只肉鸡的胸肌和腹脂组织RNA样品各3份,构建6个文库,使用Illumina Hiseq 2500测序平台进行测序,测序获得长度为150 bp的序列(reads)。去除带接头的reads、未知碱基N含量大于5%的reads、去除低质量的reads(质量值低于15的碱基占该reads总碱基数的比例大于20%的reads为低质量的reads),得到clean reads。
1.4 肉鸡胸肌和腹部脂肪组织参考基因组比对及AS事件和DSG检测得到clean reads之后,使用HISAT软件[16]将clean reads比对到鸡参考基因组序列。使用StringTie软件[17]对每个样品进行转录本重构,然后用Cuffmerge软件[18]将所有样品的重构信息整合在一起,再使用Cuffcompare软件将整合后的转录本与参考注释信息进行比较,计算转录本数量。
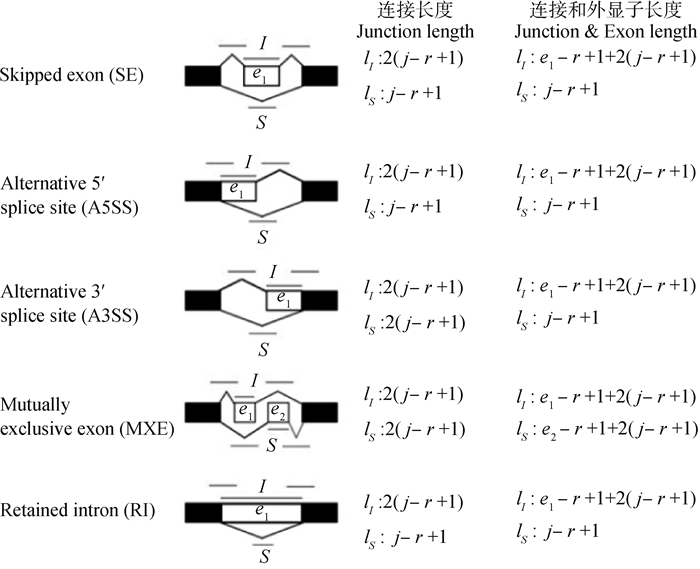
使用rMATS软件[19]检测样品中5种AS事件,分别为外显子跳跃(skipped exon,SE)、可变供体位点(alternative 5′ splicing site,A5SS)、可变受体位点(alternative 3′ splicing site,A3SS)、互斥外显子(mutually exclusive exons,MXE)和单内含子保留(retained intron,RI),并检测肌肉和脂肪组织样品间的DSG。rMATS软件是一款用于RNA-seq数据检测DSG的软件,它可以计算inclusion亚型和skipping亚型的相对丰度(图 1),同时计算相应的P-value和FDR值来衡量样品间剪接差异的显著性。本研究中使用FDR≤0.05为判断标准,小于该阈值的DSG定义为显著差异的DSG。
|
I.内含子异构体读长; S.跳跃异构体读长;j.连接长度;e1,e2.外显子长度;r.读长;lI.内含子有效长度;ls.外显子异构体有效长度 I. Reads of the inclusion isoform; S. Reads of the skipping isoform; j. Junction length; e1, e2. Exon length; r. Read length; lI. Effective length of the inclusion isoform; ls. Effective length of the skipping exon isoform 图 1 AS事件中各种剪接类型的相对丰度计算 Figure 1 Calculation of relative abundance of different splicing types in AS |
使用KOBAS 3.0软件[20-21]进行DSG的注释、GO功能富集分析、信号通路(pathway)分析,并对相关信号通路和基因进行进一步分析。进行GO功能富集分析时,以基因组为背景,统计每个GO分析中基因与背景基因之间的差异,对P值进行校正,定义校正后的P<0.05为这些基因的功能在该GO类别中显著富集。进行KEGG分析时,以基因组为背景,根据注释结果以及功能分类,将DSG进行生物通路分类,校正后的P<0.05时,表明DSG富集在一条通路中。同时对通路中DSG的表达量进行检测。具体方法为:与参考基因组比对之后,使用RSEM软件[22]计算基因和转录本的表达水平,采用校正的P≤0.05来筛选肌肉和脂肪组织基因的差异表达基因。
1.6 数据统计分析使用IBM SPSS Statistics 19.0软件(SPSS Inc., Chicago, IL, USA)对肌肉和脂肪组织基因组中的可变剪接数量进行独立样本T检验和多重比较(SLD法),数据结果以“平均数±标准差”表示。
2 结果 2.1 肉鸡肌肉和脂肪组织AS事件鉴定测序原始数据经过质量控制后,肉鸡胸肌3个组织样分别获得6.69、6.71和6.75 G,腹脂3个组织样分别获得6.68、6.69、6.73 G可用于注释分析的序列片段(clean reads)。得到clean reads之后,使用HISAT软件[16]将clean reads比对到鸡参考基因组序列。肌肉组织样品比对率分别为69.18%、69.49%和68.47%,脂肪组织样品对比率分别为74.60%、73.10%和73.75%。相同组织样品间均匀的比对率表明样品之间的数据具有可比性。
肉鸡肌肉和脂肪组织基因组中AS事件鉴定结果见表 1。由表 1可知,肉鸡肌肉和脂肪组织基因组中分别检测到(5 966.00±111.66)和(6 757.00±156.51)个AS,脂肪组织中AS数量比肌肉组织中高13.26%(P < 0.05)。共检测到的5种AS类型中,除MXE外,脂肪组织中4种类型剪接事件(SE、A5SS、A3SS和RI)的数量均显著高于肌肉组织(P < 0.05);且以SE为主,分别占肌肉和脂肪组织中总AS事件数量的54.92%和52.67%。
|
|
表 1 肉鸡肌肉和脂肪组织基因组中AS事件鉴定 Table 1 The result of alternative splicing (AS) events identified in muscle and fat tissues genome in broilers |
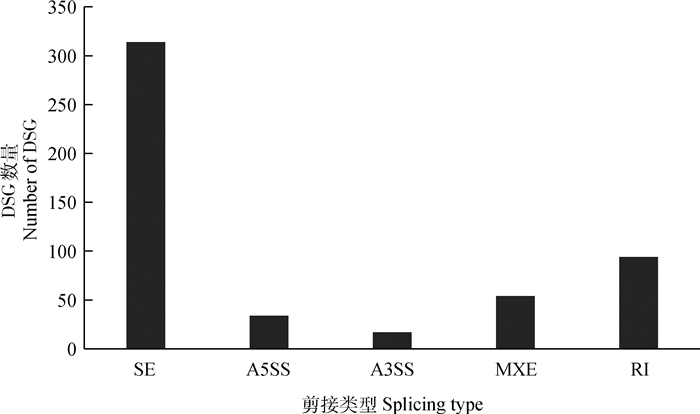
经rMATS软件[19]检测分析发现,AA肉鸡肌肉和脂肪组织中检测到513个显著的DSGs,其中SE、A5SS、A3SS、MXE和RI 5种类型的DSG数量分别为314、34、17、54和94个(图 2)。DSG中发生SE可变剪接的基因数量最多;其次为RI和MEX;发生这3种类型的DSG占总AS事件数量的90.06%。
|
图 2 肉鸡肌肉和脂肪组织差异可变剪接基因数量 Figure 2 The number of DSG between muscle and fat tissues in broilers |
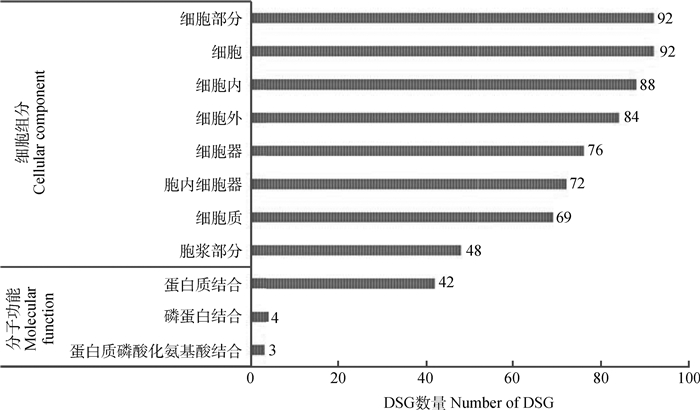
为了进一步了解肉鸡肌肉和脂肪组织DSG的功能,使用KOBAS 3.0在线软件[20-21]对所有DSG基因进行了基因注释、基因本体(gene ontology,GO)注释和功能分析。选取校正P ≤ 0.05的DSG作为GO富集分析结果(图 3)。通过GO富集分析发现,肉鸡肌肉和脂肪组织中513个DSGs中的93个DSGs显著富集在细胞组分(cellular component)和分子功能(molecular function)类别中的11个GO项内,同一基因多富集在多个GO项中。影响肉鸡肌肉和脂肪组织DSG涉及的细胞组分包括细胞(GO:0005623)、细胞部分(GO:0044464)、细胞内(GO:0005622)、细胞外(GO:0044424)、细胞器(GO:0043226)、胞内细胞器(GO:0043229)、细胞质(GO:0005737)、胞浆部分(GO:0044444)8个GO项;还有少量DSG分类到分子功能中,包括蛋白质结合(GO:0005515)、磷蛋白结合(GO:0051219)和蛋白质磷酸化氨基酸结合(GO:0045309)3个GO项。
|
图 3 肉鸡肌肉和脂肪组织基因组DSG的GO富集结果 Figure 3 The result of GO analysis for DSG in genome between muscle and fat tissues in broilers |
以鸡基因组为参照,使用KOBAS 3.0在线软件[20-21]对肉鸡肌肉和脂肪组织DSG进行了通路分析。分析发现,31个DSGs富集在肌动蛋白细胞骨架调节、焦点粘连、丙酮酸代谢和细胞内吞作用4条信号通路中。肌动蛋白细胞骨架调节与焦点粘连两条信号通路关系密切,其中10个基因均在两条信号通路之中(表 2)。肌动蛋白细胞骨架在维持细胞形状、调控细胞粘连等方面具有重要功能;焦点粘连则参与细胞膜受体和肌动蛋白骨架之间的结构连接;丙酮酸代谢作为三羧酸循环的重要组成部分,在调解物质代谢中具有重要作用;细胞内吞作用在分子运输转运方面具有重要作用。
|
|
表 2 肉鸡肌肉和脂肪组织基因组中DSGs的通路分析结果 Table 2 The result of pathway analysis for DSGs in genome between muscle and fat tissues in broilers |
为了验证DSG在肌肉和脂肪组织中的表达情况,以脂肪组织为参照,经RSEM软件[22]分析,信号通路中的31个DSGs有13个在肌肉组织中上调表达,4个下调表达;其中14个DSGs(占信号通路基因中48.39%)表达量在肌肉和脂肪组织中未发生显著的表达量变化(表 3)。
|
|
表 3 肉鸡肌肉和脂肪组织基因组中DSGs差异表达结果 Table 3 The result of differential expression levels for DSGs in genome between muscle and fat tissues in broilers |
自1978年Gilbert[23]发现AS现象以来,Ensembl数据库[24]已经收录了人、小鼠、斑马鱼、鸡、鸭、猪、牛、羊等80多种生物的可变剪接事件。基于高通量RNA-seq技术挖掘基因组AS的应用[25],使得人、小鼠、猪、牛、果蝇等多个物种基因组的可变剪接事件得到了全面的鉴定和分析[14]。冉茂良等[14]研究发现,AS事件具有发育时期特异性。徐铁山等[26]在北京鸭肌肉和皮脂中分别发现了5 958.00和7 528.67个AS。本研究中,在肉鸡肌肉和脂肪组织基因组中分别检测到5 966.00和6 757.00个AS,其中脂肪组织中AS数量显著高于肌肉组织。由此可见,可变剪接事情具有组织特异性,脂肪组织发生AS事件的频率远远高于肌肉组织。本研究发现,肌肉和脂肪组织中DSG发生SE可变剪接的基因数量最多。SE是选择性剪接最简单和最普遍存在的一种方式,这与Li等[27]对猪肌肉组织中差异剪接类型的研究相一致,说明SE可变剪接类型在肉鸡肌肉和脂肪组织中发生频率较高。
差异剪接分析对于理解细胞和器官的生理过程十分重要[28]。为了探索肌肉和脂肪组织生理过程的差异,本研究进行了肉鸡肌肉和脂肪组织间差异剪接分析。肉鸡肌肉和腹脂组织基本组成差异较大,前者由肌纤维构成,后者则属于白色脂肪,由脂肪细胞构成[29]。本研究肌肉和脂肪组织间的DSG的功能大部分与细胞基本结构相关。由此可见,差异剪接在调控肌肉和脂肪组织细胞的基本组成上具有重要作用。信号通路分析发现,DSG富集在肌动蛋白细胞骨架调节、焦点粘连、丙酮酸代谢和细胞内吞作用4条信号通路中,这些通路与肌肉组成、脂肪代谢等密切相关。由此可见,肌肉和脂肪组织细胞组成和行使功能的差异,部分源于基因组中差异可变剪接。可变剪接调控的基因通常不同于差异表达所调控的基因。因此,在基因组水平描述基因调控时,差异剪接与差异基因表达通常互相补充[15]。本研究发现,约50%的DSG在肌肉和脂肪组织中的表达水平未发生显著变化,说明肌肉和脂肪组织中DSG和差异表达基因发挥各自的功能,共同调控肌肉和脂肪组织的基本组成、生理功能等。
对于DSG可以通过剪接因子对整个基因组的可变剪接进行研究,也可以针对某一基因进行具体的结构和功能的研究。剪接因子在mRNA前体剪接过程中发挥重要作用。研究发现,可以通过剪接因子SRSF10对鸡基因组的剪接作用来研究整个基因组中的可变剪接[30]。在与本试验相关的研究中发现,对脂肪代谢具有重要调控功能的Apelin信号通路对肉鸡腹脂的沉积具有重要的调控作用(未发表)。其中Apelin前体由77个氨基酸组成,编码基因通过可变剪接可指导合成多种成熟的Apelin活性多肽片段:Apelin-36、Apelin-17、Pyr-apelin-13、Apelin-13和Apelin-12等[31]。因此,对Apelin基因的可变剪接、时空表达规律以及功能验证等研究,能够加深对肉鸡中该基因DSG、基因功能的深入理解。
4 结论 4.1本研究在肉鸡肌肉和脂肪组织中分别检测到(5 966.00±111.66)和(6 757.00±156.51)个可变剪接事件,脂肪组织发生可变剪接的频率显著高于肌肉组织(P=0.002)。
4.2本研究鉴定的5种可变剪接类型中以外显子跳跃为主,分别占肌肉和脂肪组织中可变剪接事件的54.92%和52.67%。
4.3本研究在肌肉和脂肪组织基因组中检测到513个显著的DSGs,其中93个DSGs显著富集于细胞组分和分子功能中;31个DSGs富集在肌动蛋白细胞骨架调节、焦点粘连、丙酮酸代谢和细胞内吞作用信号通路中,14个DSGs在肌肉和脂肪组织基因组中的表达水平未发生显著变化。
4.4本研究对肉鸡肌肉和脂肪组织基因组中DSG的鉴定与分析,为研究可变剪接及其调控的机制、组织间差异剪接的多样性提供理论依据。
注:DSGs的详细信息需要者可向作者索取。
| [1] | TRESS M L, ABASCAL F, VALENCIA A. Alternative splicing may not be the key to proteome complexity[J]. Trends Biochem Sci, 2017, 42(2): 98–110. DOI: 10.1016/j.tibs.2016.08.008 |
| [2] | KELEMEN O, CONVERTINI P, ZHANG Z Y, et al. Function of alternative splicing[J]. Gene, 2013, 514(1): 1–30. DOI: 10.1016/j.gene.2012.07.083 |
| [3] | BLENCOWE B J. The relationship between alternative splicing and proteomic complexity[J]. Trends Biochem Sci, 2017, 42(6): 407–408. DOI: 10.1016/j.tibs.2017.04.001 |
| [4] | LIU Y S, GONZÀLEZ-PORTA M, SANTOS S, et al. Impact of alternative splicing on the human proteome[J]. Cell Rep, 2017, 20(5): 1229–1241. DOI: 10.1016/j.celrep.2017.07.025 |
| [5] | DE MIGUEL F J, PAJARES M J, MARTÍNEZ-TERROBA E, et al. A large-scale analysis of alternative splicing reveals a key role of QKI in lung cancer[J]. Mol Oncol, 2016, 10(9): 1437–1449. DOI: 10.1016/j.molonc.2016.08.001 |
| [6] | LI Y, YUAN Y. Alternative RNA splicing and gastric cancer[J]. Mutat Res, 2017, 773: 263–273. DOI: 10.1016/j.mrrev.2016.07.011 |
| [7] | LI H, WANG Z Y, MA T Y, et al. Alternative splicing in aging and age-related diseases[J]. Transl Med Aging, 2017, 1: 32–40. DOI: 10.1016/j.tma.2017.09.005 |
| [8] |
黄艳群, 陈文, 李宁, 等. 鸡Lmbr1基因一种异常可变剪接的克隆和表达分析[J]. 畜牧兽医学报, 2010, 41(5): 518–523.
HUANG Y Q, CHEN W, LI N, et al. The cloning and expression analysis of one abnormal alternative splice in chicken Lmbr1[J]. Acta Veterinaria et Zootechnica Sinica, 2010, 41(5): 518–523. (in Chinese) |
| [9] |
杨秀芹, 翟春媛, 李海涛, 等. 猪TLR4基因可变剪接体的鉴定[J]. 畜牧兽医学报, 2011, 42(8): 1051–1056.
YANG X Q, ZHAI C Y, LI H T, et al. Identification of alternative splicing isoforms of pig TLR4 mRNA[J]. Acta Veterinaria et Zootechnica Sinica, 2011, 42(8): 1051–1056. (in Chinese) |
| [10] |
黄正洋, 陈阳, 李欣钰, 等. 鸭TLR4基因可变剪接体的克隆、鉴定及组织表达分析[J]. 畜牧兽医学报, 2013, 44(5): 697–702.
HUANG Z Y, CHEN Y, LI X Y, et al. Identification and expression analysis of alternative splicing of TLR4 in duck[J]. Acta Veterinaria et Zootechnica Sinica, 2013, 44(5): 697–702. (in Chinese) |
| [11] |
张天, 李祥龙, 周荣艳, 等. 不同毛色山羊皮肤组织Agouti基因剪接体类型研究[J]. 畜牧兽医学报, 2015, 46(11): 1934–1943.
ZHANG T, LI X L, ZHOU R Y, et al. Study on Agouti spliceosome types in goat skin with different coat color[J]. Acta Veterinaria et Zootechnica Sinica, 2015, 46(11): 1934–1943. (in Chinese) |
| [12] |
杜琛, 付绍印, 高鸿雁, 等. 绒山羊肌内脂肪细胞成熟前后比较转录组分析[J]. 畜牧兽医学报, 2014, 45(5): 714–721.
DU C, FU S Y, GAO H Y, et al. Transcriptome analysis of intramuscular preadipocytes and matureadipocyte in cashmere goats[J]. Acta Veterinaria et Zootechnica Sinica, 2014, 45(5): 714–721. (in Chinese) |
| [13] | CHACKO E, RANGANATHAN S. Genome-wide analysis of alternative splicing in cow:Implications in bovine as a model for human diseases[J]. BMC Genomics, 2009, 10(S3): S11. |
| [14] |
冉茂良, 陈斌, 李智, 等. 基于RNA-seq测序数据鉴定和分析猪基因组可变剪接事件[J]. 中国科学:生命科学, 2016, 46(3): 274–284.
RAN M L, CHEN B, LI Z, et al. Identification and analysis of alternative splicing events in Sus Scrofa using RNA-seq data[J]. Scientia Sinica Vitae, 2016, 46(3): 274–284. (in Chinese) |
| [15] | HOOPER J E. A survey of software for genome-wide discovery of differential splicing in RNA-Seq data[J]. Hum Genomics, 2014, 8: 3. DOI: 10.1186/1479-7364-8-3 |
| [16] | KIM D, LANGMEAD B, SALZBERG S L. HISAT:A fast spliced aligner with low memory requirements[J]. Nat Methods, 2015, 12(4): 357–360. DOI: 10.1038/nmeth.3317 |
| [17] | PERTEA M, PERTEA G M, ANTONESCU C M, et al. StringTie enables improved reconstruction of a transcriptome from RNA-seq reads[J]. Nat Biotechnol, 2015, 33(3): 290–295. DOI: 10.1038/nbt.3122 |
| [18] | TRAPNELL C, ROBERTS A, GOFF L, et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks[J]. Nat Protoc, 2012, 7(3): 562–578. DOI: 10.1038/nprot.2012.016 |
| [19] | SHEN S, PARK J W, LU Z X, et al. rMATS:Robust and flexible detection of differential alternative splicing from replicate RNA-Seq data[J]. Proc Natl Acad Sci U S A, 2014, 111(51): E5593–E5601. DOI: 10.1073/pnas.1419161111 |
| [20] | WU J M, MAO X Z, CAI T, et al. KOBAS server:A web-based platform for automated annotation and pathway identification[J]. Nucleic Acids Res, 2006, 34(S2): W720–W724. |
| [21] | XIE C, MAO X Z, HUANG J J, et al. KOBAS 2.0:A web server for annotation and identification of enriched pathways and diseases[J]. Nucleic Acids Res, 2011, 39(S2): W316–W322. |
| [22] | LI B, DEWEY C N. RSEM:Accurate transcript quantification from RNA-Seq data with or without a reference genome[J]. BMC Bioinformatics, 2011, 12: 323. DOI: 10.1186/1471-2105-12-323 |
| [23] | GILBERT W. Why genes in pieces?[J]. Nature, 1978, 271(5645): 501. DOI: 10.1038/271501a0 |
| [24] | STALKER J, GIBBINS B, MEIDL P, et al. The Ensembl Web site:Mechanics of a genome browser[J]. Genome Res, 2004, 14(5): 951–955. DOI: 10.1101/gr.1863004 |
| [25] | WANG Z, GERSTEIN M, SNYDER M. RNA-Seq:A revolutionary tool for transcriptomics[J]. Nat Rev Genet, 2009, 10(1): 57–63. DOI: 10.1038/nrg2484 |
| [26] |
徐铁山, 顾丽红, 侯水生, 等. 应用RNA-seq数据开展鸭基因组可变剪接的鉴定与分析[J]. 中国家禽, 2016, 38(17): 10–16.
XU T S, GU L H, HOU S S, et al. Identification and analysis of alternative splicing in duck genome using RNA-seq data[J]. China Poultry, 2016, 38(17): 10–16. (in Chinese) |
| [27] | LI B J, DONG C, LI P H, et al. Identification of candidate genes associated with porcine meat color traits by genome-wide transcriptome analysis[J]. Sci Rep, 2016, 6: 35224. DOI: 10.1038/srep35224 |
| [28] | ZIMMERMANN K, JENTSCH M, RASCHE A, et al. Algorithms for differential splicing detection using exon arrays:A comparative assessment[J]. BMC Genomics, 2015, 16(1): 136. DOI: 10.1186/s12864-015-1322-x |
| [29] | ZHANG S, MCMILLAN R P, HULVER M W, et al. Chickens from lines selected for high and low body weight show differences in fatty acid oxidation efficiency and metabolic flexibility in skeletal muscle and white adipose tissue[J]. Int J Obesity, 2014, 38(10): 1374–1382. DOI: 10.1038/ijo.2014.8 |
| [30] | ZHOU X X, WU W W, WEI N, et al. Genome-wide analysis of SRSF10-regulated alternative splicing by deep sequencing of chicken transcriptome[J]. Genom Data, 2014, 2: 20–23. DOI: 10.1016/j.gdata.2014.02.001 |
| [31] | BOUCHER J, MASRI B, DAVIAUD D, et al. Apelin, a newly identified adipokine up-regulated by insulin and obesity[J]. Endocrinology, 2005, 146(4): 1764–1771. DOI: 10.1210/en.2004-1427 |



