2. 黑龙江省农业科学院, 哈尔滨 150086
2. Heilongjiang Academy of Agricultural Sciences, Harbin 150086, China
真核生物基因多数是断裂的,由外显子和内含子组成,转录时首先形成初始转录本——pre-mRNA,再经过剪接去掉内含子并把外显子按照5′→3′的顺序连接起来,得到成熟mRNA。传统剪接发生于同一个pre-mRNA内部,成熟RNA的外显子都来源于同一个初始转录本,称为顺式剪接(cis-splicing, CS)。反式剪接(trans-splicing, TS)则发生于不同的初始转录本之间,剪接产物是含有不同来源外显子的嵌合RNAs。TS最早发现于低等真核生物,哺乳动物的相关研究起步较晚,近年来,第二代高通量测序技术的发展为TS鉴定提供了有力工具,TS及其产物在生命活动、疾病发生中的作用也逐渐得到重视。本文对TS概念、类型、产生机制及其在哺乳动物中的作用进行综述。
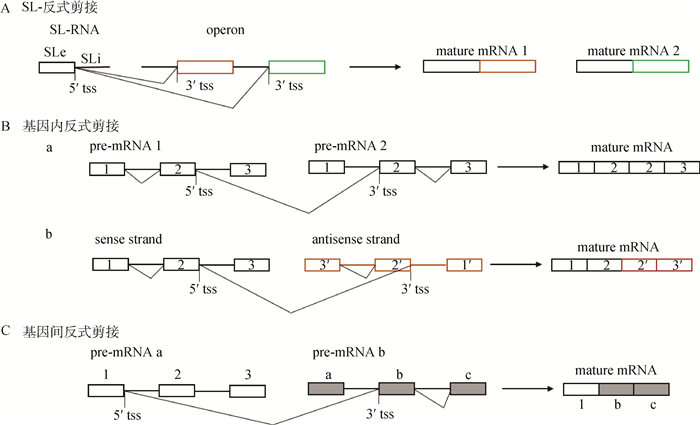
1 反式剪接类型TS可归纳为两种类型:SL(spliced-leader)-型和非SL-型,非SL-型又包括基因内TS和基因间TS。SL-型TS只存在于低等真核生物,在脊椎动物中非SL-型取代了SL型,且发生的频率显著降低。SL-型TS与操纵子密切相关,有研究认为,随着操纵子的丢失和基因组复杂性的增加,TS逐渐转化为CS,说明剪接在生物进化过程中具有重要意义[1]。
1.1 SL-型反式剪接SL-型TS是指来源于一个非编码RNA(SL RNA)5′末端的前导序列被剪接到pre-mRNA的5′端,在产生的成熟mRNA中前导序列不具有编码作用,故SL-型TS不影响原有mRNA的翻译结果,该过程类似于高等生物基因的5′加帽,能够提高mRNA稳定性,促进翻译。SL RNA的前导序列也被称为外显子(SLe),余下部分为内含子(SLi)。SLe的长度随着物种的不同而改变,介于16~51 nt之间。同一个SLe可以与许多、甚至是物种内全部pre-mRNA进行TS,由此形成的成熟mRNA含有共同的5′末端。各物种SL RNA序列相似性不高,但都具有高甲基化的5′帽子结构、较短的SLe序列和茎环二级结构,并且茎环结构的一个配对区含有5′剪接位点(splice site, SS)和Sm蛋白结合位点[2]。
SL-型TS存在两种类型:一种剪接发生于SLe和pre-mRNA的5′游离末端;此外,许多低等真核生物基因以操纵子形式存在,即多个基因共用一个启动子,所以也存在着SL RNA和操纵子内部基因间的TS,剪接发生于SLe和pre-mRNA的5′非游离序列(图 1A)。有些生物(如锥体虫)只有一种SL RNA参与所有的TS反应[3];而在秀丽线虫及其亲缘种中,上述两种TS反应分别由不同的SL RNA(SL1和SL2)负责,这可能是不同环境和发育阶段下转录本差异表达的原因[4]。SL-型TS广泛存在于低等真核生物中,锥体虫每个蛋白编码基因都会通过该方式形成SL帽状RNA[3]。
|
a.同一pre-mRNA的两个拷贝或等位基因间的剪接;b.同一基因座正反两条链间的剪接。方框表示外显子,不同颜色或背景的方框表示来自不同基因。tss.反式剪接位点 a. Trans-splicing between two copies of the same pre-mRNA or alleles; b. Trans-splicing between sense and antisense DNA at the same locus. Boxes indicate exon, those boxes with different colors or backgrounds indicate exons from different genes. Tss. Trans-splicing site 图 1 反式剪接类型 Figure 1 Types of trans-splicing |
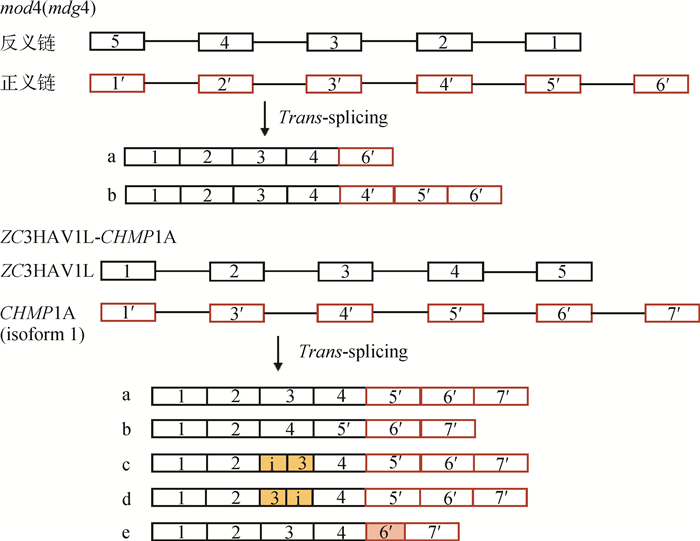
基因内TS的初始转录本来源于同一个基因座,可发生于同一pre-mRNA的两个拷贝、等位基因以及同一DNA分子两条链的转录产物间(图 1B),往往造成外显子重复和正义-反义链融合[4]。在果蝇[5]和家蚕[6]中研究得比较透彻的mod(mdg)基因座能形成一系列基因内TS产物,这些成熟mRNAs的5′外显子相同,均来自于同一个基因的5′端,家蚕是BGIBMGA006426基因的外显子1~4;3′外显子是可变的,分别来自于反向平行链的不同基因,家蚕中已鉴定了8个不同基因。家蚕的1个上游基因和8个下游基因产生了9种成熟mRNAs,其中两个mRNAs的3′端来源于同一个基因,但外显子组成不同(图 2),这说明TS也存在着可变剪接。目前可变反式剪接(alternative trans-splicing, ATS)还没有统一的定义,笔者认为应该为两个或多个特定的pre-RNAs分子经过TS形成多种嵌合RNAs的过程。mod(mdg)基因编码产物在两个物种间非常相似:5′外显子编码产生BTB(BR2C, ttk and bab)结构域;3′外显子尽管来源于不同的基因,但都编码产生保守的FLYWCH基序。家蚕9个成熟mRNAs均编码产生317 aa残基的蛋白质多肽链。大鼠COT(carnitine octanoyltransferase)基因cDNA的外显子重复现象及产生机制分析,首次证明了哺乳动物中存在着自发的TS反应[7];对绵羊T细胞受体β链(T cell receptor β-chain, TRB)基因座转录产物的克隆分析表明,在该区域存在着较高频率的基因内TS事件,这也是其编码蛋白功能复杂的原因之一[8]。
|
方框表示外显子,直线表示内含子,不同颜色的方框分别表示部分外显子和内含子(用i表示)。a、b、c、d、e表示可变转录本 Boxes indicate exon, lines indicate intron, those closed boxes with different colors indicate partial of an exon or retained intron (indicated with i). a, b, c, d, e indicate the alternative transcripts 图 2 家蚕mod4(mdg4)和人ZC3HAV1L-CHMP1A基因的可变反式剪接[6, 13] Figure 2 Alternative trans-splicing of silk worm mod4(mdg4) and human ZC3HAV1L-CHMP1A genes[6, 13] |
基因间TS是不同来源的初始转录本间发生剪接,在pre-mRNA之间以及pre-mRNA和非编码RNA之间都能发生;进行剪接的基因可位于同一条染色体上,也可位于不同染色体上(图 1C)。自二十世纪九十年代初期在胎鼠肝组织中发现基因间TS后[9],这方面的研究逐渐增多,人、小鼠和果蝇中都鉴定到该现象。虽然一度被认为是转录组中的稀有事件,深度测序技术的普及应用及高性能生物信息学软件的开发、完善,为在全转录组水平上进行TS的高通量鉴定提供了有效手段,大量基因间TS现象也随之被鉴定,其在生命活动中的重要性正逐渐被揭示[10-12]。
基因间TS也存在着复杂的可变剪接现象。嵌合子ZC3HAV1L-CHMP1A的两个亲本基因分别位于人7和16号染色体上,两个亲本通过TS产生的嵌合子有5种可变转录本(图 2),其中转录本a是主要的ATS产物,广泛表达于多种组织和细胞内,两个亲本基因为框内融合,预期可编码产生融合蛋白;转录本a~d使用经典的SSs进行TS;转录本e的嵌合点位于ZC3HAV1L外显子4末端和CHMP1A外显子6内部,不符合GT-AG规则,在交界处存在着一个5 bp的正向重复序列,笔者认为其可能是转录过程中模板转换导致的(图 2)[13]。
2 反式剪接反应机制Ⅰ型内含子[14]、Ⅱ型内含子[15]和tRNA[16]都存在TS现象,但他们都是不依赖于剪接体的TS,本文只对剪接体介导的RNA反式剪接(spliceosome-mediated RNA trans-splicing, SMaRT)产生机制进行介绍。
2.1 反式剪接具有和顺式剪接相同的反应机制一系列研究表明,SMaRT具有和CS相同的剪接机制。首先,具有相同的剪接信号。TS不存在特殊的反式剪接位点(trans-splice sites, TSSs)。绵羊TRB基因座含有3个D-J-C(多样区-连接区-恒定区)基因簇,该区域的TS都发生于D/J和J/C连接点,没有形成新的SSs[8];Ma等[17]通过高通量测序技术在猪上鉴定了251个嵌合RNAs,其中大多数分子的SSs符合GT-AG规则。
其次,具有相似的剪接体组分。U1、U2、U4/U6和U5等核内小核糖核蛋白(small nuclear ribonucleoproteins, snRNP)共同组装成顺式剪接体。早期在秀丽线虫和蛔虫中的研究表明,U2、U4/U6、U5等snRNPs都是SL-型TS的基本因子[18],后来发现,锥体虫中U1 snRNP在CS和TS中发挥着双重作用[19]。
另外,具有类似的剪接调控因子。SR蛋白、Sm蛋白、核内不均一核糖核蛋白和多聚嘧啶序列结合蛋白等顺式剪接调控因子均参与调控TS[20-23]。在目前鉴定的哺乳动物TS反应中,尚未发现特殊的剪接调控因子。
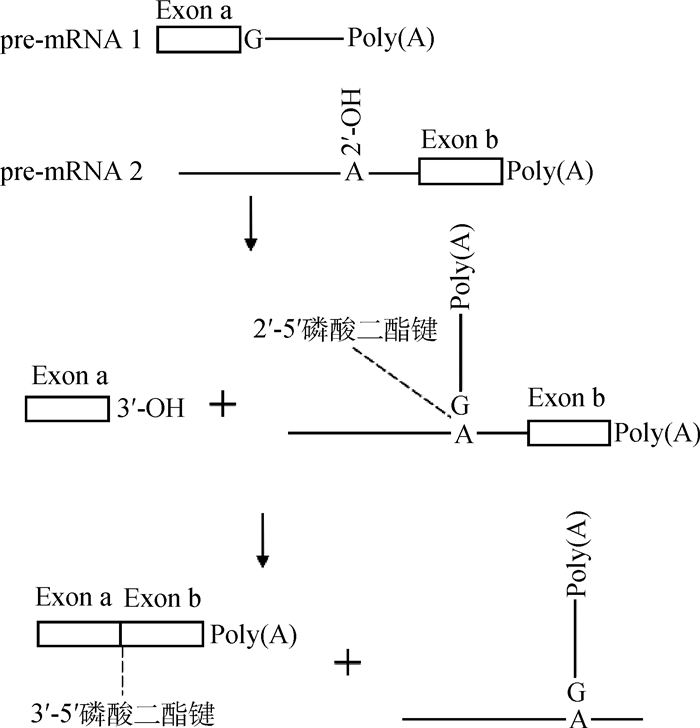
2.2 反式剪接的特点 2.2.1 形成Y-结构不同于CS的套马索结构,TS的中间产物是一个Y-结构。Y-结构最早是在研究锥体虫SL-型TS中发现的[24],全转录组水平上的研究进一步证明了Y-结构存在的普遍性[25]。在进行TS时,被剪接掉的区域不是一个连续的内含子,而是来自于两个不同的基因,剪接供体位点和分支点分别位于不同的基因上,当分支点游离羟基与5′ SS通过2′-5′磷酸二酯键结合后,反式内含子的上游和下游之间不能形成一个闭合的套马索结构,而是开放的Y-结构(图 3)。
|
图 3 反式剪接的Y结构 Figure 3 Y-structure in trans-splicing |
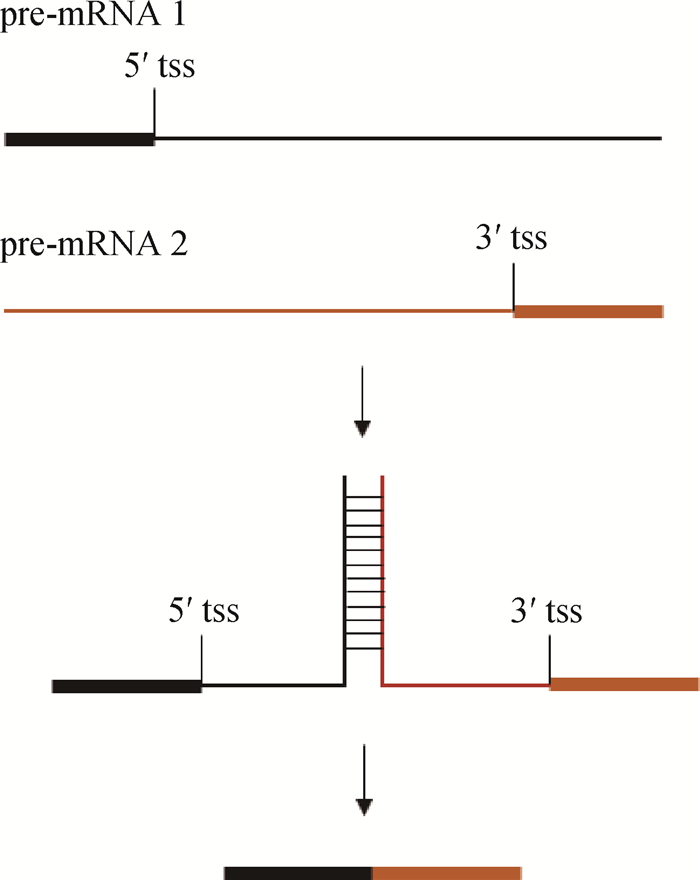
CS的外显子都位于一个基因上,彼此具有较近的物理距离。TS的反式内含子则含有互补配对序列,通过该序列形成二级结构,促进两个独立pre-mRNA彼此靠近,为剪接反应创造条件(图 4)。人和小鼠TS的上游内含子含有嘧啶富集区,下游内含子具有嘌呤富集区,二者几乎能够完全互补配对[26]。肠贾第虫HSP90和OADβ基因都是TS产物,其中HSP90反式内含子的上、下游区域含有26 bp互补配对区,OADβ基因在配对的碱基数和序列组成上存在着一定的差别,但也能形成二级结构[27]。秀丽线虫位于eri-6和eri-7位点之间的正向重复序列介导了这两个独立基因间的TS,从而形成ERI-6/7蛋白的完整编码序列[28]。
|
长方形表示外显子,直线表示内含子或外含子 Rectangles indicate exon, lines indicate intron or outron 图 4 互补配对结构及序列的反式剪接 Figure 4 Complementary structure and its trans-splicing |
低等真核生物的SL-型TS是在pre-mRNA的5′端加上一个帽状结构,不改变其原有的编码作用,因此不影响蛋白质组的复杂性和多样性,该种剪接主要对mRNA进行表达调控[29],不涉及到编码蛋白质的功能性通路。本文主要对哺乳动物TS及其嵌合体的功能进行介绍。
TS把两个或多个不同来源的pre-RNA分子剪接成一个嵌合分子,这种来源的嵌合RNA也称为tsRNA(trans-splicing RNA),嵌合点的位置存在着以下几种可能:(1)位于UTR区,导致一个亲本基因的5′或者3′ UTR发生改变;(2)位于编码区内部,但破坏了原有的读码框,导致移码融合(frame-shift fusion)。大部分TS属于该种形式[12],所产生的嵌合子可以作为长链非编码RNA发挥作用;也可能在嵌合点下游形成提前终止密码子,从而被生物体内的监测系统——无义介导的mRNA降解机制——降解掉;(3)位于编码区内部,并且不破坏两个基因的读码框,即发生了框内融合,嵌合子编码产生融合蛋白。因此,通过形成tsRNA,TS能够影响亲本基因表达、形成新蛋白质或非编码RNA,在细胞活力和生长、基因表达调控、信号转导等一系列生物过程中发挥着重要的调控作用。
3.1 反式剪接调控基因表达人酰基辅酶A:胆固醇酰基转移酶1(acyl-coenzyme A:cholesterol acyltransferase 1,ACAT1)基因通过TS形成的一种转录变异体(4.3-kb mRNA),几乎存在于所有组织细胞内[30]。通过起始密码子的选择性使用,4.3-kb mRNA翻译产生正常的ACAT1酶(50 ku)和N-端含有额外序列的一种亚型(56 ku),该亚型的活性是正常酶(50 ku)的30%[31]。因此,ACAT1通过产生嵌合RNA来竞争使用pre-mRNA,从而调控正常序列表达、影响酶活性。小鼠5号染色体上的非编码RNA(Dmr)和19号染色体上的Dmrt1基因形成的嵌合子,编码产生缺少C端的Dmrt1蛋白,Dmr主要充当嵌合子的3′ UTR,促进TS、下调正常Dmrt1蛋白表达量[32]。HongrES2是大鼠附睾组织中鉴定的嵌合、非编码RNA,其下游基因为附睾特异表达的CES7,HongrES2具有5′帽子结构和3′ poly(A)尾,类似于mRNA前体,加工成熟后的产物为microRNA样小RNA(microRNA-like small RNA),能够抑制CES7基因表达[33]。
3.2 反式剪接调控细胞生长和癌症发生Hirano和Noda[34]利用简并PCR在精子cDNA文库中分离得到了减数分裂同源重组基因Msh4的7条cDNA,其中3条(Msh4β、ε和δ)是基因间TS产物,主要表达于精子中,Msh4β和ε在脑、心、胸腺和卵巢等组织中也有少量表达;进一步研究发现,Msh4δ能在精子发生过程中诱导细胞程序性死亡。嵌合分子CYCLIN D1-TROP2编码产生截短的CYCLIN D1(细胞周期蛋白D1)和TROP2(滋养层细胞表面抗原-2),在原代培养的细胞中异源低表达CYCLIN D1-TROP2,促进细胞增殖、延长寿命,高表达则能导致细胞转化[35]。CHD2和CHMP1A基因分别位于人15和16号染色体上,二者编码的蛋白质都调控染色质/DNA结构,嵌合分子CHD2-CHMP1A在多种肿瘤组织和细胞系中表达,通过RNAi技术敲低其在HBL-100细胞系中的表达后,细胞生长受到明显抑制[36]。在肾癌和结肠癌细胞中检测到的tsADK-DHX8能够调控细胞生长[36]。
细胞过度生长和细胞周期改变都是癌症的标志[37],tsRNA与癌症发生、发展存在着密切关系,可以作为肿瘤诊断的生物标记[38]。约90%的前列腺癌细胞都表达TMEM79-SMG5嵌合分子,在健康的对照组织中不表达[39]。嵌合分子MN1-FLI和NIPBL-HOXB9被注入到小鼠骨髓后,能够诱发白血病[40]。CYCLIN D1-TROP2是一种强致癌因子,能促进侵袭性肿瘤生长[35]。Xie等[41]通过对多种组织和癌细胞检测发现,除了嵌合分子PAX3-FOXO1,还有些tsRNA在横纹肌肉瘤细胞系和临床样本中特异表达,并具有与PAX3-FOXO1相同的瞬时表达模式。此外,原发性大肠癌[42]、急性髓样白血病[43]等疾病中都鉴定出了相关嵌合分子。
3.3 反式剪接介导染色体重排TS和染色体重排存在着一定的联系。染色体易位[t(7;17)(p15q21)]形成的嵌合分子JAZF1-JJAZ1与子宫内膜间质肿瘤具有明显相关[44],研究发现,在染色体组成正常非肿瘤细胞内也存在着tsJAZF1-JJAZ1[45]。tsJAZF1-JJAZ1在子宫内膜基质细胞发育中受到严格的表达调控,嵌合子的转录量明显低于亲本基因,翻译产生的嵌合蛋白具有抗凋亡作用[45]。人、鼠上的荧光原位杂交分析发现[46],位于15号染色体上的原癌基因Myc和12号染色体上的免疫球蛋白重链基因Igh共同出现在同一个转录工厂(transcription factory)上,具备发生TS的条件。而在浆细胞瘤和伯基特淋巴瘤患者中,这两个位点非常频繁地发生易位。人2号和13号染色体易位形成的嵌合分子PAX3-FOXO1,是腺泡状横纹肌肉瘤(alveolar rhabdomyosarcoma, ARMS)的诊断标志物和治疗靶标[47],其编码的嵌合蛋白诱导肌肉生成、并抑制其分化产生成熟肌肉组织[48]。没有发生易位的间充质干细胞和胎儿肌肉组织均能形成tsPAX3-FOXO1[49]。在间充质干细胞中,该嵌合分子在其他生肌因子转录之前瞬时表达;在胎儿肌肉组织中,其相对表达量远低于横纹肌肉瘤细胞系。过表达PAX3-FOXO1导致MyoD和Myogenin两种肌肉标记物持续表达,并最终形成ARMS的肌肉发育和过表达症状。综上,研究者认为,tsRNA具有重要的生理功能,是机体正常生长发育所必须的;同时可作为骨架促进染色体间相互作用,是染色体发生重排的前提条件[50];而染色体重排造成的嵌合子表达失调是导致癌转化的原因[45]。
3.4 反式剪接维持胚胎干细胞多能性Wu等[51]在生物信息学分析基础上,通过两种逆转录酶(MMLV和AMV)催化的RT-PCR反应及RNase保护试验,在人胚胎干细胞(human embryonic stem cell, hESC)中鉴定了4个tsRNA——tsCSNK1G3、tsARHGAP5、tsFAT1和tsRMST,他们均在hESC体外分化过程中差异表达。其中,tsRMST在hESC和多种体细胞(皮肤成纤维细胞、毛乳头细胞和颗粒细胞)重编程形成的诱导性多能干细胞中高表达,但在肌肉、肝、肾等10种分化的组织中不表达。利用小发卡RNA (small hairpin RNA, shRNA)敲低tsRMST表达,hESC中多能性基因(NANOG、POU5F1、SOX2和TCF7L1)的表达受到显著抑制,而种系特异的转录因子(GATA6和PAX6)表达量增加,进一步分析证明了tsRMST通过招募NANOG和PRC2复合体抑制种系特异基因表达,维持hESC多能性。
3.5 反式剪接的其他功能G蛋白信号调节因子(regulaotrs of G-protein signaling, RGS)是G蛋白信号转导通路中的负性调节因子,能够与一些受体、效应分子相互作用,调节G蛋白信号通路。tsRGS12嵌合分子在组织中特异表达,其编码产物以细胞周期依赖的方式定位于核点,与减数分裂中期的染色体形成有关,过表达该嵌合分子导致细胞内形成不规则核与多核。此外,TS改变了正常RGS12基因的表达量,形成了新蛋白质,必然会对G蛋白的信号转导功能产生影响[52]。
对嵌合蛋白的结构分析表明,嵌合子含有完整蛋白结构域的几率非常显著地高于随机序列,暗示着TS及其产物在生命活动中具有一定作用[53]。有些嵌合分子的编码蛋白与亲本蛋白互相竞争,扰乱原有的蛋白互作网络[53-54]。随着TS产物作用的揭示,人们逐渐认识到其在生理、病理活动中的重要性,研究范畴也在逐渐扩大,从最初的癌症相关研究扩展到生命活动的多个领域,但Huang等[55]通过定量PCR分析发现,嵌合RNAs和衰老之间不存在相关性,这可能和样本数及检测的嵌合RNAs数量较少有关。
3.6 反式剪接在疾病治疗中的作用基于SMaRT的产生机制,人们开发了基因靶向疗法,基本原理是设计合成靶向内源、缺陷mRNA前体的RNA分子——pre-mRNA反式剪接分子(pre-mRNA trans-splicing molecule, PTM),其上含有与靶内含子互补的配对序列、催化剪接反应的人工内含子、以及替代缺陷序列的正确cDNA片段。PTM被注入到体内(细胞)后,首先利用互补配对序列识别、结合内源pre-mRNA,然后在剪接体介导下,内源pre-mRNA与PTM进行TS形成一个嵌合分子,在此过程中外源cDNA替代了内源pre-mRNA的缺陷序列,从而达到修复缺陷RNA的目的[56]。
脊髓性肌萎缩(spinal muscular atrophy, SMA)是一种神经退行性疾病,是导致婴儿期死亡的主要遗传因素,由运动神经元存活基因1(survival motor neuron 1, SMN1)缺失导致[57-58]。人基因组中含有SMN1和SMN2两个基因,二者具有完全相同的开放读码框,但SMN2的大部分转录本都是可变剪接产物,编码产生的截短蛋白稳定性差、没有功能[59]。SMN2基因存在于所有的SMA患者中,校正SMN2基因的可变剪接位点、抑制其可变剪接,是SMA分子治疗的可行思路[60]。Coady和Lorson[61]把人工合成的、靶向修复SMN2的PTM注射入SMA小鼠的侧脑室,在体内实现了SMN功能蛋白的挽救性表达,减轻了小鼠SMA症状,延长了生存期。
TS在遗传性疾病治疗方面的巨大潜力引起了人们的广泛研究兴趣,目前已经利用PTM对亨廷顿疾病[62]、营养不良大疱性表皮松懈症[63]、肥厚性心肌病[64]、镰状细胞贫血病[65]、B细胞急性淋巴细胞白血病[66]、直肠癌[67]、先天性肌肉营养不良症[68]等多种疾病的遗传因子修复方面进行了试验研究,并得到了预期效果。在病毒性疾病的治疗方面,TS也展现了良好的应用前景。Ingemarsdotter等[69]构建了靶向人类免疫缺陷病毒(human immunodeficiency virus, HIV)RNA SSs位点的TS载体,并成功地在携带HIV的细胞内获得了tsRNA,实现了选择性杀死感染细胞。以期TS介导的基因靶向疗法将会实现临床上的应用。
4 小结与展望TS及其嵌合RNAs在一系列生理、病理活动中发挥着重要作用,在正常细胞中的表达量较低且受到严格的时空调控,在特定条件下或癌细胞等细胞类型中的表达失调,导致染色体易位和肿瘤发生。但当前研究多集中在利用高通量测序结合生物信息学方法鉴定癌症相关tsRNA和试验验证层面,缺乏研究深度。研究的对象主要是人和小鼠,TS在农业动物领域方面的作用及应用前景有待揭示。进一步阐明哺乳动物TS在基本生命活动中的作用及产生机制,有助于深入揭示其生理/病理学意义,更好地为畜禽遗传改良和人类遗传性疾病的靶向治疗服务。
由于表达量较低,传统的RNA酶保护试验、Northern blot、Western blot等可信度高的分析方法往往灵敏度不够;而基于逆转录的检测方法(如RT-PCR)又容易形成假阳性,在一定程度上阻碍了TS在非肿瘤细胞中的研究,需要对相关试验技术进行改进和完善。近年来发展起来的直接RNA测序方法、Hi-C三维基因组研究技术、蛋白质数据分析方法为TS及其嵌合RNA研究提供了新的工具。鉴于其在生命活动中的重要性以及相关试验方法的不断改进、完善,TS必将会成为遗传学研究中的一个重要内容。
| [1] | LEI Q, LI C, ZUO Z X, et al. Evolutionary insights into RNA trans-splicing in vertebrates[J]. Genome Biol Evol, 2016, 8(3): 562–577. DOI: 10.1093/gbe/evw025 |
| [2] | KRCHÑÁKOVÁ Z, KRAJOVI J, VESTEG M. On the possibility of an early evolutionary origin for the spliced leader trans-splicing[J]. J Mol Evol, 2017, 85(1-2): 37–45. DOI: 10.1007/s00239-017-9803-y |
| [3] | PREUÉER C, JAÉ N, GÜNZL A, et al.Pre-mRNA splicing in Trypanosoma brucei: Factors, mechanisms, and regulation[C]//BINDEREIF A.RNA Metabolism in Trypanosomes.Berlin, Heidelberg: Springer, 2012: 49-77. |
| [4] | ALLEN M A, HILLIER L W, WATERSTON R H, et al. A global analysis of C.elegans trans-splicing[J]. Genome Res, 2011, 21(2): 255–264. DOI: 10.1101/gr.113811.110 |
| [5] | MCMANUS C J, DUFF M O, EIPPER-MAINS J, et al. Global analysis of trans-splicing in Drosophila[J]. Proc Natl Acad Sci U S A, 2010, 107(29): 12975–12979. DOI: 10.1073/pnas.1007586107 |
| [6] | SHAO W, ZHAO Q Y, WANG X Y, et al. Alternative splicing and trans-splicing events revealed by analysis of the Bombyx mori transcriptome[J]. RNA, 2012, 18(7): 1395–1407. DOI: 10.1261/rna.029751.111 |
| [7] | CAUDEVILLA C, SERRA D, MILIAR A, et al. Natural trans-splicing in carnitine octanoyltransferase pre-mRNAs in rat liver[J]. Proc Natl Acad Sci U S A, 1998, 95(21): 12185–12190. DOI: 10.1073/pnas.95.21.12185 |
| [8] | DI TOMMASO S, ANTONACCI R, CICCARESE S, et al. Extensive analysis of D-J-C arrangements allows the identification of different mechanisms enhancing the diversity in sheep T cell receptor β-chain repertoire[J]. BMC Genomics, 2010, 11: 3. DOI: 10.1186/1471-2164-11-3 |
| [9] | SULLIVAN P M, PETRUSZ P, SZPIRER C, et al. Alternative processing of androgen-binding protein RNA transcripts in fetal rat liver:identification of a transcript formed by trans splicing[J]. J Biol Chem, 1991, 266(1): 143–154. |
| [10] | MA E S K, WAN T S K, AU C H, et al. Next-generation sequencing and molecular cytogenetic characterization of ETV6-LYN fusion due to chromosomes 1, 8 and 12 rearrangement in acute myeloid leukemia[J]. Cancer Genet, 2017, 218-219: 15–19. DOI: 10.1016/j.cancergen.2017.09.001 |
| [11] | MITTAL V K, MCDONALD J F. De novo assembly and characterization of breast cancer transcriptomes identifies large numbers of novel fusion-gene transcripts of potential functional significance[J]. BMC Med Genomics, 2017, 10: 53. DOI: 10.1186/s12920-017-0289-7 |
| [12] | BABICEANU M, QIN F J, XIE Z Q, et al. Recurrent chimeric fusion RNAs in non-cancer tissues and cells[J]. Nucleic Acids Res, 2016, 44(6): 2859–2872. DOI: 10.1093/nar/gkw032 |
| [13] | FANG W W, WEI Y, KANG Y B, et al. Detection of a common chimeric transcript between human chromosomes 7 and 16[J]. Biol Direct, 2012, 7: 49. DOI: 10.1186/1745-6150-7-49 |
| [14] | GREWE F, VIEHOEVER P, WEISSHAAR B, et al. A trans-splicing group Ⅰ intron and tRNA-hyperediting in the mitochondrial genome of the lycophyte Isoetes engelmannii[J]. Nucleic Acids Res, 2009, 37(15): 5093–5104. DOI: 10.1093/nar/gkp532 |
| [15] | GLANZ S, KÜCK U. Trans-splicing of organelle introns-a detour to continuous RNAs[J]. Bioessays, 2009, 31(9): 921–934. DOI: 10.1002/bies.v31:9 |
| [16] | RANDAU L, MÜNCH R, HOHN M J, et al. Nanoarchaeum equitans creates functional tRNAs from separate genes for their 5'-and 3'-halves[J]. Nature, 2005, 433(7025): 537–541. DOI: 10.1038/nature03233 |
| [17] | MA L, YANG S L, ZHAO W M, et al. Identification and analysis of pig chimeric mRNAs using RNA sequencing data[J]. BMC Genomics, 2012, 13: 429. DOI: 10.1186/1471-2164-13-429 |
| [18] | HANNON G J, MARONEY P A, NILSEN T W. U small nuclear ribonucleoprotein requirements for nematode cis-and trans-splicing in vitro[J]. J Biol Chem, 1991, 266(34): 22792–22795. |
| [19] | PREUΒER C, ROSSBACH O, HUNG L H, et al. Genome-wide RNA-binding analysis of the trypanosome U1 snRNP proteins U1C and U1-70K reveals cis/trans-spliceosomal network[J]. Nucleic Acids Res, 2014, 42(10): 6603–6615. DOI: 10.1093/nar/gku286 |
| [20] | STERN M Z, GUPTA S K, SALMON-DIVON M, et al. Multiple roles for polypyrimidine tract binding (PTB) proteins in trypanosome RNA metabolism[J]. RNA, 2009, 15(4): 648–665. DOI: 10.1261/rna.1230209 |
| [21] | DE GAUDENZI J G, JÄGER A V, IZCOVICH R, et al. Insights into the regulation of mRNA processing of polycistronic transcripts mediated by DRBD4/PTB2, a trypanosome homolog of the polypyrimidine tract-binding protein[J]. J Eukaryot Microbiol, 2016, 63(4): 440–452. DOI: 10.1111/jeu.12288 |
| [22] | PHILIPPE L, PANDARAKALAM G C, FASIMOYE R, et al. An in vivo genetic screen for genes involved in spliced leader trans-splicing indicates a crucial role for continuous de novo spliced leader RNP assembly[J]. Nucleic Acids Res, 2017, 45(14): 8474–8483. DOI: 10.1093/nar/gkx500 |
| [23] | GUPTA S K, KOSTI I, PLAUT G, et al. The hnRNP F/H homologue of Trypanosoma brucei is differentially expressed in the two life cycle stages of the parasite and regulates splicing and mRNA stability[J]. Nucleic Acids Res, 2013, 41(13): 6577–6594. DOI: 10.1093/nar/gkt369 |
| [24] | MURPHY W J, WATKINS K P, AGABIAN N. Identification of a novel Y branch structure as an intermediate in trypanosome mRNA processing:Evidence for trans splicing[J]. Cell, 1986, 47(4): 517–525. DOI: 10.1016/0092-8674(86)90616-1 |
| [25] | MICHAELI S. Trans-splicing in trypanosomes:Machinery and its impact on the parasite transcriptome[J]. Future Microbiol, 2011, 6(4): 459–474. DOI: 10.2217/fmb.11.20 |
| [26] | DIXON R J, EPERON I C, SAMANI N J. Complementary intron sequence motifs associated with human exon repetition:A role for intragenic, inter-transcript interactions in gene expression[J]. Bioinformatics, 2007, 23(2): 150–155. |
| [27] | KAMIKAWA R, INAGAKI Y, TOKORO M, et al. Split introns in the genome of Giardia intestinalis are excised by spliceosome-mediated trans-splicing[J]. Curr Biol, 2011, 21(4): 311–315. DOI: 10.1016/j.cub.2011.01.025 |
| [28] | FISCHER S E J, BUTLER M D, PAN Q, et al. Trans-splicing in C.elegans generates the negative RNAi regulator ERI-6/7[J]. Nature, 2008, 455(7212): 491–496. DOI: 10.1038/nature07274 |
| [29] | YANG Y F, ZHANG X Q, MA X H, et al. Trans-splicing enhances translational efficiency in C.elegans[J]. Genome Res, 2017, 27(9): 1525–1535. DOI: 10.1101/gr.202150.115 |
| [30] | LI B L, LI X L, DUAN Z J, et al. Human acyl-CoA:Cholesterol acyltransferase-1(ACAT-1) gene organization and evidence that the 4.3-kilobase ACAT-1 mRNA is produced from two different chromosomes[J]. J Biol Chem, 1999, 274(16): 11060–11071. DOI: 10.1074/jbc.274.16.11060 |
| [31] | YANG L, LEE O, CHEN J, et al. Human acyl-coenzyme A:Cholesterol acyltransferase 1(acat1) sequences located in two different chromosomes (7 and 1) are required to produce a novel ACAT1 isoenzyme with additional sequence at the N terminus[J]. J Biol Chem, 2004, 279(44): 46253–46262. DOI: 10.1074/jbc.M408155200 |
| [32] | ZHANG L, LU H, XIN D Z, et al. A novel ncRNA gene from mouse chromosome 5 trans-splices with Dmrt1 on chromosome 19[J]. Biochem Biophys Res Commun, 2010, 400(4): 696–700. DOI: 10.1016/j.bbrc.2010.08.130 |
| [33] | NI M J, HU Z H, LIU Q, et al. Identification and characterization of a novel non-coding RNA involved in sperm maturation[J]. PLoS One, 2011, 6(10): e26053. DOI: 10.1371/journal.pone.0026053 |
| [34] | HIRANO M, NODA T. Genomic organization of the mouse Msh4 gene producing bicistronic, chimeric and antisense mRNA[J]. Gene, 2004, 342(1): 165–177. DOI: 10.1016/j.gene.2004.08.016 |
| [35] | GUERRA E, TREROTOLA M, DELL' ARCIPRETE R, et al. A bicistronic CYCLIN D1-TROP2 mRNA chimera demonstrates a novel oncogenic mechanism in human cancer[J]. Cancer Res, 2008, 68(19): 8113–8121. DOI: 10.1158/0008-5472.CAN-07-6135 |
| [36] | PLEBANI R, OLIVER G R, TREROTOLA M, et al. Long-range transcriptome sequencing reveals cancer cell growth regulatory chimeric mRNA[J]. Neoplasia, 2012, 14(11): 1087–1096. DOI: 10.1593/neo.121342 |
| [37] | HANAHAN D, WEINBERG R A. Hallmarks of cancer:The next generation[J]. Cell, 2011, 144(5): 646–674. DOI: 10.1016/j.cell.2011.02.013 |
| [38] | ZHOU J H, LIAO J, ZHENG X X, et al. Chimeric RNAs as potential biomarkers for tumor diagnosis[J]. BMB Rep, 2012, 45(3): 133–140. DOI: 10.5483/BMBRep.2012.45.3.133 |
| [39] | KANNAN K, WANG L G, WANG J H, et al. Recurrent chimeric RNAs enriched in human prostate cancer identified by deep sequencing[J]. Proc Natl Acad Sci U S A, 2011, 108(22): 9172–9177. DOI: 10.1073/pnas.1100489108 |
| [40] | DANG J, NANCE S, MA J, et al. AMKL chimeric transcription factors are potent inducers of leukemia[J]. Leukemia, 2017, 31(10): 2228–2234. DOI: 10.1038/leu.2017.51 |
| [41] | XIE Z Q, BABICEANU M, KUMAR S, et al. Fusion transcriptome profiling provides insights into alveolar rhabdomyosarcoma[J]. Proc Natl Acad Sci U S A, 2016, 113(46): 13126–13131. DOI: 10.1073/pnas.1612734113 |
| [42] | LEE J R, KWON C H, CHOI Y, et al. Transcriptome analysis of paired primary colorectal carcinoma and liver metastases reveals fusion transcripts and similar gene expression profiles in primary carcinoma and liver metastases[J]. BMC Cancer, 2016, 16: 539. DOI: 10.1186/s12885-016-2596-3 |
| [43] | ENDO A, TOMIZAWA D, AOKI Y, et al. EWSR1/ELF5 induces acute myeloid leukemia by inhibiting p53/p21 pathway[J]. Cancer Sci, 2016, 107(12): 1745–1754. DOI: 10.1111/cas.2016.107.issue-12 |
| [44] | KOONTZ J I, LEE SORENG A, NUCCI M, et al. Frequent fusion of the JAZF1 and JJAZ1 genes in endometrial stromal tumors[J]. Proc Natl Acad Sci U S A, 2001, 98(11): 6348–6353. DOI: 10.1073/pnas.101132598 |
| [45] | LI H, WANG J L, MOR G, et al. A neoplastic gene fusion mimics trans-splicing of RNAs in normal human cells[J]. Science, 2008, 321(5894): 1357–1361. DOI: 10.1126/science.1156725 |
| [46] | OSBORNE C S, CHAKALOVA L, MITCHELL J A, et al. Myc dynamically and preferentially relocates to a transcription factory occupied by Igh[J]. PLoS Biol, 2007, 5(8): e192. DOI: 10.1371/journal.pbio.0050192 |
| [47] | SORENSEN P H B, LYNCH J C, QUALMAN S J, et al. PAX3-FKHR and PAX7-FKHR gene fusions are prognostic indicators in alveolar rhabdomyosarcoma:A report from the children's oncology group[J]. J Clin Oncol, 2002, 20(11): 2672–2679. DOI: 10.1200/JCO.2002.03.137 |
| [48] | GRAF FINCKENSTEIN F, SHAHBAZIAN V, DAVICIONI E, et al. PAX-FKHR function as pangenes by simultaneously inducing and inhibiting myogenesis[J]. Oncogene, 2008, 27(14): 2004–2014. DOI: 10.1038/sj.onc.1210835 |
| [49] | YUAN H L, QIN F J, MOVASSAGH M, et al. A chimeric RNA characteristic of rhabdomyosarcoma in normal myogenesis process[J]. Cancer Discov, 2013, 3(12): 1394–1403. DOI: 10.1158/2159-8290.CD-13-0186 |
| [50] | ZAPHIROPOULOS P G. Trans-splicing in higher eukaryotes:Implications for cancer development?[J]. Front Genet, 2011, 2: 92. |
| [51] | WU C S, YU C Y, CHUANG C Y, et al. Integrative transcriptome sequencing identifies trans-splicing events with important roles in human embryonic stem cell pluripotency[J]. Genome Res, 2014, 24(1): 25–36. DOI: 10.1101/gr.159483.113 |
| [52] | CHATTERJEE T K, FISHER R A. Novel alternative splicing and nuclear localization of human RGS12 gene products[J]. J Biol Chem, 2000, 275(38): 29660–29671. DOI: 10.1074/jbc.M000330200 |
| [53] | FRENKEL-MORGENSTERN M, LACROIX V, EZKURDIA I, et al. Chimeras taking shape:Potential functions of proteins encoded by chimeric RNA transcripts[J]. Genome Res, 2012, 22(7): 1231–1242. DOI: 10.1101/gr.130062.111 |
| [54] | FRENKEL-MORGENSTERN M, VALENCIA A. Novel domain combinations in proteins encoded by chimeric transcripts[J]. Bioinformatics, 2012, 28(12): i67–i74. DOI: 10.1093/bioinformatics/bts216 |
| [55] | HUANG R, KUMAR S, LI H. Absence of correlation between chimeric RNA and aging[J]. Genes (Basel), 2017, 8(12): E386. DOI: 10.3390/genes8120386 |
| [56] | BERGER A, MAIRE S, GAILLARD M C, et al. mRNA trans-splicing in gene therapy for genetic diseases[J]. Wiley Interdiscip Rev RNA, 2016, 7(4): 487–498. DOI: 10.1002/wrna.1347 |
| [57] | CRAWFORD T O, PARDO C A. The neurobiology of childhood spinal muscular atrophy[J]. Neurobiol Dis, 1996, 3(2): 97–110. DOI: 10.1006/nbdi.1996.0010 |
| [58] | OSKOUI M, LEVY G, GARLAND C J, et al. The changing natural history of spinal muscular atrophy type Ⅰ[J]. Neurology, 2007, 69(20): 1931–1936. DOI: 10.1212/01.wnl.0000290830.40544.b9 |
| [59] | LEFEBVRE S, BVRGLEN L, REBOULLET S, et al. Identification and characterization of a spinal muscular atrophy-determining gene[J]. Cell, 1995, 80(1): 155–165. DOI: 10.1016/0092-8674(95)90460-3 |
| [60] | SUMNER C J. Therapeutics development for spinal muscular atrophy[J]. NeuroRx, 2006, 3(2): 235–245. DOI: 10.1016/j.nurx.2006.01.010 |
| [61] | COADY T H, LORSON C L. Trans-splicing-mediated improvement in a severe mouse model of spinal muscular atrophy[J]. J Neurosci, 2010, 30(1): 126–130. DOI: 10.1523/JNEUROSCI.4489-09.2010 |
| [62] | RINDT H, TOM C M, LORSON C L, et al. Optimization of trans-splicing for huntington's disease RNA therapy[J]. Front Neurosci, 2017, 11: 544. DOI: 10.3389/fnins.2017.00544 |
| [63] | PEKING P, KOLLER U, DUARTE B, et al. An RNA-targeted therapy for dystrophic epidermolysis bullosa[J]. Nucleic Acids Res, 2017, 45(17): 10259–10269. DOI: 10.1093/nar/gkx669 |
| [64] | PRONDZYNSKI M, KRÄMER E, LAUFER S D, et al. Evaluation of MYBPC3 trans-splicing and gene replacement as therapeutic options in human iPSC-derived cardiomyocytes[J]. Mol Ther Nucleic Acids, 2017, 7: 475–486. DOI: 10.1016/j.omtn.2017.05.008 |
| [65] | UCHIDA N, WASHINGTON K N, MOZER B, et al. RNA Trans-splicing targeting endogenous β-globin pre-messenger RNA in human erythroid cells[J]. Hum Gene Ther Methods, 2017, 28(2): 91–99. DOI: 10.1089/hgtb.2016.077 |
| [66] | UCKUN F M, QAZI S, MA H, et al. CD22△E12 as a molecular target for corrective repair using RNA trans-splicing:Anti-leukemic activity of a rationally designed RNA trans-splicing molecule[J]. Integr Biol (Camb), 2015, 7(2): 237–249. DOI: 10.1039/C4IB00221K |
| [67] | HE X X, LIAO J Z, LIU F, et al. Functional repair of p53 mutation in colorectal cancer cells using trans-splicing[J]. Oncotarget, 2015, 6(4): 2034–2045. |
| [68] | AZIBANI F, BRULL A, ARANDEL L, et al. Gene therapy via trans-splicing for LMNA-related congenital muscular dystrophy[J]. Mol Ther Nucleic Acids, 2018, 10: 376–386. DOI: 10.1016/j.omtn.2017.12.012 |
| [69] | INGEMARSDOTTER C K, PODDAR S, MERCIER S, et al. Expression of herpes simplex virus thymidine kinase/ganciclovir by RNA Trans-splicing induces selective killing of HIV-producing cells[J]. Mol Ther Nucleic Acids, 2017, 7: 140–154. DOI: 10.1016/j.omtn.2017.03.004 |



